2. 福建省家畜传染病防治与生物技术重点实验室, 龙岩 364000;
3. 福建农林大学动物科技学院, 福州 350002
2. Fujian Provincial Key Laboratory for the Prevention and Control of Animal Infectious Diseases and Biotechnology, Longyan 364000, China;
3. College of Animal Science, Fujian Agriculture and Forestry University, Fuzhou 350002, China
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)引起的一种高度接触性传染病,给全世界的养猪业造成了巨大的经济损失[1-3]。PRRSV依据遗传进化的差异可分为两个基因型,分别是以LV毒株为代表的PRRSV-1和以VR-2332毒株为代表的PRRSV-2,我国流行的毒株主要以PRRSV-2为主[4-6]。根据2010年Shi对PRRSV-2的分类,我国主要流行的毒株分为4个谱系(lineage),即lineage1(NADC30-like, Sublineage1.8)、lineage3(QYYZ-like)、lineage5(VR2332-like)和Sublineage 8.7(JXA1-like和CH-1a-like)[5-8]。1996年,郭宝清等[9]首次从流产胎儿中分离出PRRSV,随后迅速传播至全国各个省份。2006年,我国多个省份的猪场暴发了高发病率和高死亡率的HP-PRRSV,给我国的养猪业带来前所未有的经济损失[2-3]。2012年,我国出现了与美国毒株NADC30高度同源的NADC30-like PRRSV(Sublineage 1.8),该类毒株主要引起怀孕母猪流产和新生仔猪呼吸道疾病,其基因组高度变异且易于其他毒株发生重组,导致现有的商品化疫苗无法为其提供良好的免疫保护[10-12]。2015年,我国报道出现Lineage 3 PRRSV,该类毒株主要流行于我国的华南地区,近年来,呈现小范围流行的趋势[12-14]。2017年,国内出现了新流行的NADC34-like PRRSV(Sublineage 1.5),该类毒株的特征是在Nsp2基因编码序列中存在100个氨基酸(aa)的连续缺失,目前在多个省份报道[15-18]。
近年来,随着NADC30-like毒株的广泛流行,PRRSV变异明显加快,且毒株间频频发生重组,毒株呈现遗传多样性,极大地影响了猪群健康状况和生产性能[6, 8, 10-14]。福建省是中国的养猪大省,养猪业是当地的支柱产业,大部分学者对福建省PRRSV的流行病学调查主要集中在ORF5、Nsp2和ORF7基因,很少从全基因组角度去分析PRRSV的流行特点[14, 19-21]。因此,作者选择2017—2021年分别从福建省5个地市的规模化猪场采集的疑似感染PRRS猪的组织病料,进行PRRSV的分离及全基因组序列测定,从全基因组角度研究PRRSV的流行情况和分子特征,同时监控新毒株的出现,为PRRS的防控提供数据支持和理论基础。
1 材料与方法 1.1 样品来源及细胞2017—2021年,从福建省龙岩市、南平市、莆田、泉州、漳州等5个地市45个规模化猪场采集疑似感染PRRS的脾、肺、淋巴结等组织样品和血清样品。稳定高效表达猪CD163的MARC-145细胞(MARC-145CD163)由福建省家畜传染病防治与生物技术重点实验室构建并保存[8]。
1.2 主要试剂及细胞RNA提取试剂盒、普通琼脂糖凝胶DNA回收试剂盒购自天根生化科技(北京)有限公司;pEASY-Blunt Simple Cloning Vector购自北京全式金生物技术有限公司;反转录酶HiScriptⓇ III Reverse Transcriptase和高保真的DNA聚合酶2×PhantaⓇ Max Master Mix (Dye Plus) 购自南京诺唯赞生物科技有限公司公司;山羊抗鼠lgG荧光抗体(FITC标记)购自碧云天生物技术有限公司;PRRSV N蛋白单克隆抗体(MAb)由福建省家畜传染病防治与生物技术重点实验室制备并保存。
1.3 病毒分离与鉴定取适量的肺、脾、淋巴结等组织加入离心管中,以1∶5的比例加入PBS进行研磨,冻融2次后12 000 r·min-1离心10 min,将经0.22 μm滤器过滤的滤液和血清接种于长满单层的MARC-145CD163细胞的六孔板,PBS洗涤3次后加入DMEM维持溶液,37 ℃ 5% CO2条件下培养,每天观察细胞病变(CPE),待CPE达到80%左右时收毒。用荧光定量RT-PCR检测方法和间接免疫荧光(IFA)对分离的病毒进行鉴定[22]。
1.4 PRRSV全基因组扩增根据VR-2332、JXA1、NADC30和QYYZ毒株的序列设计6对重叠基因片段简并引物,用于扩增PRRSV全基因组序列(表 1)。同时根据参考文献[23-24]的用于扩增PRRSV全基因组序列的引物对本研究的基因序列进行验证。按照RNA提取试剂盒操作说明书提取病毒液RNA,以制备的cDNA为模板进行PRRSV全基因各片段PCR扩增。将回收、纯化的目的基因,克隆至pEASY-Blunt Simple载体,将鉴定为阳性的重组菌液由北京睿博兴科生物技术有限公司进行测序。
|
|
表 1 扩增PRRSV全基因组的引物序列 Table 1 Sequences of primers for amplification ofthe PRRSV genome |
利用DNAStar 7.0软件分析32个福建PRRSV分离株与参考毒株的相似性以及氨基酸缺失突变情况。使用MEGA 7.0软件中的邻接法对ORF5基因及全基因组进行遗传进化分析。利用RDP 4.10软件和Simplot 3.51软件对32个福建PRRSV分离株进行重组分析。
2 结果 2.1 病毒分离与鉴定将PRRSV阳性病料接种于MARC-145CD163细胞,在3~4 d后可以产生细胞圆缩、聚集等典型的细胞病变。荧光定量RT-PCR检测和IFA试验结果表明,共成功分离32株PRRSV毒株。
2.2 分离毒株全基因组分析将PRRSV基因组的各片段测序结果进行拼接,结果显示, 32株PRRSV基因组大小为14 938~ 15 439 bp(去除ployA)。基因组相似性分析结果显示,福建省32个PRRSV分离株之间核苷酸相似性为82.4%~99.4%,差异较大;与代表毒株VR-2332、JXA1、CH-1a、NADC30、QYYZ、JXA1 P80和LV的核苷酸相似性分别为84.4%~91.2%、82.9%~99%、83.7%~94.9%、84.2%~95.3%、81.2%~87.8%、82.7%~99.4%和59.7%~60.5%。此外,PRRSV2/CN/C2/2019、PRRSV2/CN/F3/2020与JXA1-R疫苗株的核苷酸相似性最高(为98.7%~99.4%),而与JXA1核苷酸相似性为98.3%~99.0%。
为进一步分析PRRSV基因组特性,将32株PRRSV与代表毒株VR-2332、CH-1a、JXA1、NADC30、QYYZ和LV进行比对分析。结果显示,与参考毒株相比较,32个毒株的ORF1b相对ORF1a较保守。ORF1a和ORF1b所编码的非结构蛋白中Nsp2的变异程度最大,与代表毒株之间基因序列相似性较低[nt/aa,(69.9%~99.2%)/(61.2%~98.9%)]。ORF2~ORF7编码PRRSV的结构蛋白,多重比对结果显示,基因组中GP3、GP5a和GP5变异程度较大(>15%),而M和N蛋白相对保守。
2.3 遗传进化分析用生物软件MEGA7.0对福建省32株PRRSV和15株代表毒株进行序列比对分析,分别构建PRRSV全基因组和ORF5基因的系统遗传进化树,结果显示,福建省呈现多种谱系毒株并存,两者的符合率为75%(24/32)(图 1)。8株PRRSV分型结果不一致,其中ORF5基因的遗传演化分析结果显示,6个分离株(PRRSV2/CN/L3/2021、PRRSV2/CN/L4/2020、PRRSV2/CN/L5/2018、PRRSV2/CN/G7/2018、PRRSV2/CN/G9/2018和PRRSV2/CN/F2/2019)分类在谱系3中,PRRSV2/CN/H5/2020和PRRSV2/CN/Q9/2021分别属于谱系1和谱系5;而全基因的遗传进化结果显示,3个分离株(PRRSV2/CN/H5/2020、PRRSV2/CN/Q9/2021和PRRSV2/CN/L5/2018)分类在谱系8.7中,3个分离株(PRRSV2/CN/G7/2018、PRRSV2/CN/G9/2018和PRRSV2/CN/F2/2019)分类在谱系1中,而PRRSV2/CN/L3/2021和PRRSV2/CN/L4/2020分化较大,处于谱系1和谱系3之间(图 1)。
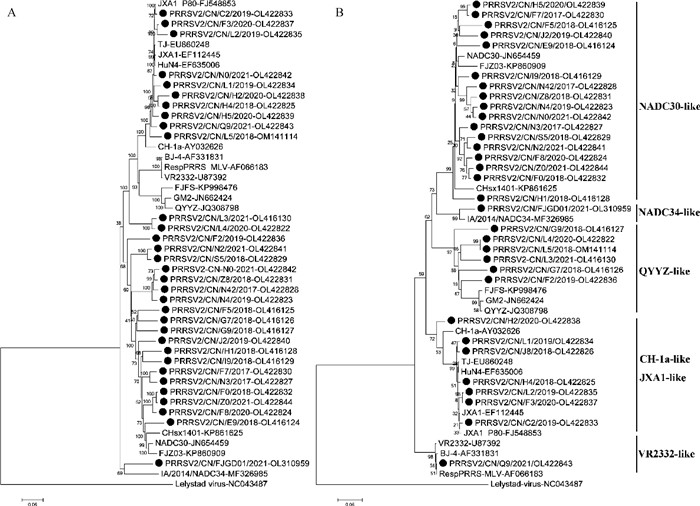
|
图 1 福建省32株PRRSV-2(●)全基因组(A)和ORF5基因(B)与参考毒株系统进化树分析 Fig. 1 Phylogenetic trees based on complete sequence (A) and ORF5 gene (B) of 32 PRRSV-2 strains and reference strains |
对32个PRRSV分离株的Nsp2基因推导的氨基酸序列进行比对分析,结果显示,福建地区PRRSV Nsp2基因呈现不同程度的缺失(图 2)。与参考毒株VR-2332相比,以PRRSV2/CN/E9/2018为代表的23个分离株在aa 325―435、aa 483和aa 504―522存在131 aa的不连续缺失(111+1+19 aa),与代表毒株NADC30缺失位置一致,其中,两个分离株(PRRSV2/CN/G7/2018和PRRSV2/CN/G9/2018)还出现了额外的24 aa缺失,两个分离株(PRRSV2/CN/L3/2021和PRRSV2/CN/L4/2020)在224―225位点插入了两个氨基酸KI(图 2A)。另外,以PRRSV2/CN/H2/2020为代表的8个分离株在aa 481和aa 534―562存在30 aa的不连续缺失(1+29 aa),与JXA1为代表的HP-PRRSV缺失位置一致(图 2B)。
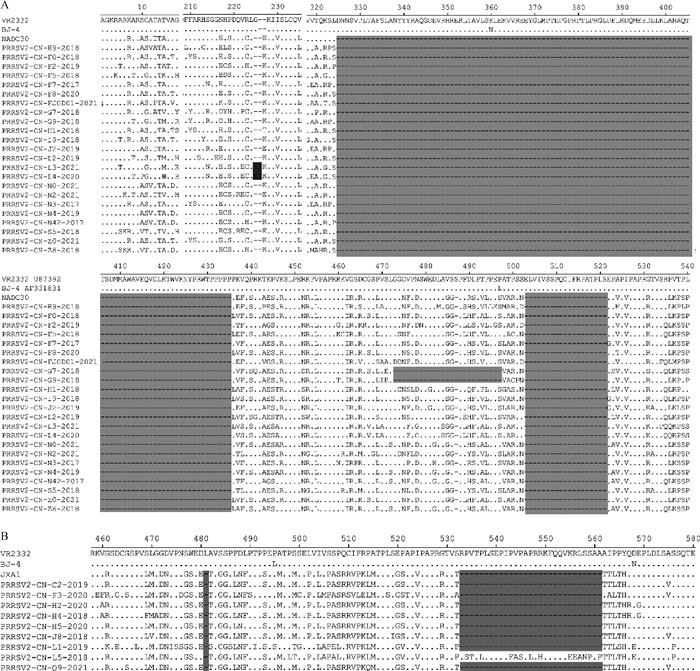
|
图A中阴影区域:224-225位点处是插入氨基酸,其他处为类NADC30毒株Nsp2氨基酸缺失模式;图B中阴影区域:HP-PRRSV Nsp2氨基酸缺失模式 In Fig.A, the shaded area at 224-225 sites indicate inserated amino acids, and the others show the amino acid deletion pattern of Nsp2 of NADC30 like strains; In Fig.B, the shaded areas show the HP-PRRSV Nsp2 amino acid deletion mode 图 2 Nsp2氨基酸序列分析 Fig. 2 Amino acid sequence analysis of Nsp2 between Fujian isolates and representative strains |
32个分离株ORF5基因核苷酸长度为603~606 bp,编码200~201 aa。与代表毒株VR-2332相比,PRRSV2/CN/I9/2018毒株在第59位点插入了一个氨基酸H。GP5蛋白中Q13和R151与PRRSV毒力相关,福建省24个毒株存在R151K突变(图 3)。福建省谱系3中的毒株在中和表位区(aa 37―45)高度变异,均存在H38Y和L39S的突变,大部分谱系8.7的毒株存在L39I的突变(图 3)。此外,与其他代表毒株相比,PRRSV2/CN/L3/202等6个毒株与谱系3代表毒株存在几处独有的氨基酸突变:F25S、A26I、N33G、L39S、S66C、A92S、L117F、A129F、L152I和R199H(图 3)。32个分离株有3~5个潜在的糖基化位点,其中,N44和N51比较保守(图 3)。
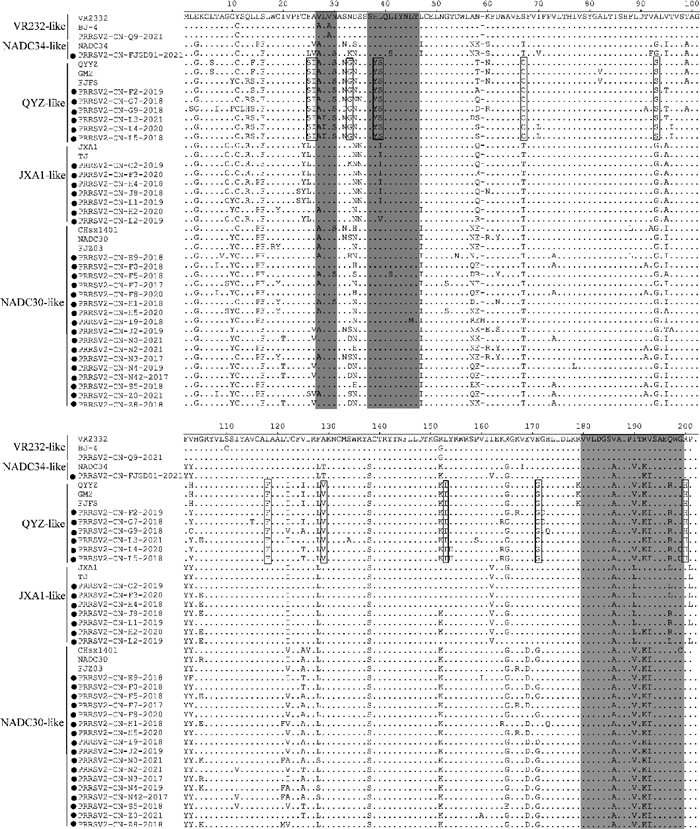
|
阴影区域:预测的抗原区域;黑色框:L3 PRRSV GP5蛋白重要氨基酸变异位点 Shaded area: Predicted antigen areas; Black box: Important amino acids of L3 PRRSV GP5 protein 图 3 32株PRRSV(●)与代表毒株ORF5蛋白氨基酸序列分析 Fig. 3 Amino acid sequence analysis of GP5 between Fujian isolates and representative strains |
使用重组分析软件RDP4.10和SimPlot 3.51对32个PRRSV-2全基因组进行重组分析。将分离毒株作为目的比对序列,各亚群代表毒株(CH-1a、JXA1、NADC30、VR-2332、QYYZ和NADC34)和疫苗株(CH-1R、JXA1 P80、TJM-F92、HuN4-F112和RespPRRS MLV)作为参考序列进行重组分析,并且对重组位点两端序列做遗传进化分析,验证重组事件的发生。结果显示,32株PRRSV-2中27株存在重组现象,共表现出9种不同的重组方式,分别为①Sublineage 1.8和Sublineage 8.7;②Sublineage 1.8、Sublineage 1.5和Sublineage 8.7;③Sublineage1.8、Lineage5和Sublineage8.7;④Sublineage1.8、Lineage3和Sublineage8.7;⑤Sublineage1.8、Lineage3、Lineage5和Sublineage8.7;⑥Lineage5和Sublineage8.7;⑦Sublineage1.8、Lineage3和Lineage5;⑧Sublineage1.8和Lineage3;⑨Sublineage8.7和Lineage3。福建省27个重组毒株中,12个毒株与疫苗株发生了重组;19个毒株以NADC30-like毒株为主要亲本,其他谱系的毒株为次要亲本重组而来。其中,PRRSV2/CN/FJGD01/202来源于NADC30-like(Sublineage 1.8)、NADC34-like(Sublineage1.5)和JXA1-like(Sublineage 8.7)之间的重组,PRRSV2/CN/G9/2018毒株来源于L1+L3+L5+L8四个谱系毒株的重组,表明随着NADC30-like PRRSV的不断变异,PRRSV重组模式呈现越来越复杂多样化。对27个重组毒株的重组断裂点进行统计分析,结果显示,重组断裂点主要分布在Nsp1、Nsp2、Nsp9和ORF3基因,值得注意的是PRRSV/CN/S5/2018和PRRSN2/CN/N2/2021两个毒株的重组断裂点多达16~17个。
3 讨论PRRS是严重影响我国养猪业的重要疫病。由于缺乏3′→5′核酸外切酶的校对功能,使得PRRSV的核苷酸替换频率是已报道的RNA病毒中最高的[25]。PRRSV自1996年首次在我国报道后,迅速传播至全国各个省份,给全国的养猪业造成巨大的经济损失。研究表明,我国PRRSV多种基因亚型并存,且频繁突变、不断发生重组,导致现有的商品化疫苗无法对PRRSV产生良好的保护,大大增加了PRRS的防控难度[10, 12-14, 26],因此需要对PRRSV进行流行病学调查,了解其流行情况和进化趋势,有助于制定针对性防控策略。
福建省32株PRRSV全基因组核苷酸相似性为82.4%~99.4%,差异性较大,尤其是PRRSV2/CN/G9/2018、PRRSV2/CN/L3/2021和PRRSV2/CN/L4/2020与NCBI数据库中PRRSV序列的核苷酸相似性较低(88.7%~91.7%),可能导致现有的PRRSV疫苗不能对其产生交叉保护,因此有必要对这3个毒株的生物学特性、致病性及疫苗免疫保护力进行深入研究。福建省32个PRRSV分离株分布在不同的进化谱系,主要流行毒株为Lineage 1和Lineage 8,不同地区或不同猪场检测到的Lineage 1毒株相似性差异比较大,可能是由于该类毒株的低保真度及缺乏有效的疫苗免疫保护,导致类NADC30毒株不断变异进化[26]。2018年,Lineage 1 PRRSV毒株复杂多样,处于同一谱系不同的进化分支,这可能是因为生猪及猪肉制品的大范围的调运,猪场没有或缺乏有效的检测手段而引入新毒株,部分猪场无序使用减毒活疫苗,导致毒株不断变异。2019―2021年,Lineage 1 PRRSV的检出率和分离率呈现下降的趋势,这可能与非洲猪瘟流行的背景下,猪场更加注重生物安全及实施闭群饲养的策略有关,使PRRSV类型及分布等生态特点发生改变[27]。PRRSV2/FJGD01/2021毒株为福建省新发现的一类毒株,属于Sublineage1.5(类NADC34毒株),这可能与省内外频繁的生猪贸易或引种有关,造成了PRRSV的传播和扩散,表明福建省PRRSV起源复杂,毒株呈现呈现多种亚型并存的局面,近年来类NADC34毒株检出比例持续增加,已有多个省份检测到该类毒株,可能会成为我国流行毒株,应当加强对这类毒株的检测[15]。研究表明,HP-PRRSV疫苗株在自然条件下毒力可以返强[28]。本研究中PRRSV2/CN/C2/2019和PRRSV2/CN/F3/2020两株毒株基因组核苷酸相似性与疫苗株JXA1-R最高(98.7%~99.4%),遗传进化分析显示两个毒株与JXA1-R亲缘关系较近,处于同一进化分支。回溯性调查表明,PRRSV2/CN/C2/2019和PRRSV2/CN/F3/2020分离于JXA1-R疫苗免疫的猪场,表明两个毒株可能来源于疫苗株JXA1-R。
ORF5所编码的GP5蛋白,是变异最大的结构蛋白,与病毒中和能力相关,本研究32株PRRSV的主要中和表位存在广泛的变异,潜在的N-糖基化位点也存在差异,可能会导致毒株逃避疫苗的中和作用,造成免疫失败。ORF5基因具有高度可变性,在一定程度上可以反映整个基因组的变异程度,因此ORF5基因常用来对PRRSV基因组分型[6-8, 10, 14, 18-21, 26, 29-31]。本研究32株PRRSV分别基于全基因组和ORF5基因进行遗传演化分析,结果表明符合率为75%(24/32),表明ORF5基因虽然可作为诊断标记为进化提供信息,但由于其长度和毒株重组限制了在详细的分子流行病学调查中的用途,存在一定误差[26, 32-33],因此,对PRRSV进行流行病学调查,最准确的应当从全基因组角度进行分析。
基因重组和突变是PRRSV进化的重要机制,近年来NADC30-like毒株与我国田间毒株甚至商品化弱毒疫苗株发生了广泛的重组,比如JL580、14LY01-FJ、SHpd1/2018和FJXS15的重组方式为L1+L8,SCcd17和SCN17重组方式为L1+L5+L8,SD17-38、SCcd16和SCya17重组方式为L1+L3+L8,而FJLIUY来源于4个谱系间的重组(L1+ L3+L5+L8),重组后毒株的生物学特性和致病性均发生一定程度的变化[34-36]。本研究中32株PRRSV-2中27株存在重组现象,重组方式主要为L1+L8和L1+L3+L8,并且出现了更为复杂的PRRSV重组毒株,比如PRRSV2/CN/FJGD01/202来自于3个谱系(Sublineage 1.8+ Sublineage1.5+Sublineage 8.7)之间的重组,而PRRSV2/CN/G9/2018来自于4个谱系(L1+L3+L5+L8)间的重组,这可能会进一步加剧猪场防控的PRRS的难度。福建省PRRSV呈现不同重组方式,其原因可能是猪场毒株类型复杂多样,部分猪场存在两个或两个以上的PRRSV,加之疫苗的免疫压力,从而导致毒株发生较为复杂的进化[14, 36]。此外,27株重组毒株的重组热点主要分布在与RNA复制有关的Nsp1、Nsp2、Nsp9和ORF3基因[37],预示PRRSV在这些位置发生重组可能更易于病毒的复制、生存和传播。
综上所述,福建省PRRSV主要流行毒株为以NADC30-like PRRSV为主要亲本的重组毒株,重组方式复杂多样,可能会导致疫苗无法起到很好的保护效果,从而加大了PRRS的防控难度,因此猪场在防控PRRS时,不能只依靠疫苗进行防控,应加强猪场的生物安全措施,定期对猪场的PRRSV全基因组进行分析。总之,本研究有助于进一步了解PRRSV的变异情况,为研制更有效的疫苗提供数据支持。
4 结论本研究共分离得到32株PRRSV,序列分析表明PRRSV重组频繁(27/32)且重组模式复杂多样,主要流行毒株以NADC30-like PRRSV为主要亲本的重组毒株。对PRRSV进行分型时应当以全基因组的结果为准。
| [1] |
NEUMANN E J, KLIEBENSTEIN J B, JOHNSON C D, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States[J]. J Am Vet Med Assoc, 2005, 227(3): 385-392. DOI:10.2460/javma.2005.227.385 |
| [2] |
TIAN K G, YU X L, ZHAO T Z, et al. Emergence of fatal PRRSV variants: unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark[J]. PLoS One, 2007, 2(6): e526. DOI:10.1371/journal.pone.0000526 |
| [3] |
TONG G Z, ZHOU Y J, HAO X F, et al. Highly pathogenic porcine reproductive and respiratory syndrome, China[J]. Emerg Infect Dis, 2007, 13(9): 1434-1436. DOI:10.3201/eid1309.070399 |
| [4] |
KUHN J H, LAUCK M, BAILEY A L, et al. Reorganization and expansion of the nidoviral family Arteriviridae[J]. Arch Virol, 2016, 161(3): 755-768. DOI:10.1007/s00705-015-2672-z |
| [5] |
GUO Z H, CHEN X X, LI R, et al. The prevalent status and genetic diversity of porcine reproductive and respiratory syndrome virus in China: a molecular epidemiological perspective[J]. Virol J, 2018, 15(1): 2. DOI:10.1186/s12985-017-0910-6 |
| [6] |
GAO J C, XIONG J Y, YE C, et al. Genotypic and geographical distribution of porcine reproductive and respiratory syndrome viruses in mainland China in 1996-2016[J]. Vet Microbiol, 2017, 208: 164-172. DOI:10.1016/j.vetmic.2017.08.003 |
| [7] |
SHI M, LAM T T Y, HON C C, et al. Phylogeny-based evolutionary, demographical, and geographical dissection of North American type 2 porcine reproductive and respiratory syndrome viruses[J]. J Virol, 2010, 84(17): 8700-8711. DOI:10.1128/JVI.02551-09 |
| [8] |
LIU J K, XU Y, LIN Z F, et al. Epidemiology investigation of PRRSV discharged by faecal and genetic variation of ORF5[J]. Transbound Emerg Dis, 2021, 68(4): 2334-2344. DOI:10.1111/tbed.13894 |
| [9] |
郭宝清, 陈章水, 刘文兴, 等. 从疑似PRRS流产胎儿分离PRRSV的研究[J]. 中国畜禽传染病, 1996(2): 1-5. GUO B Q, CHEN Z S, LIU W X, et al. Isolation and identification of porcine reproductory and respiratory syndrome (PRRS) virus from aborted fetuses suspected of PRRS[J]. Chinese Journal of Infectious Diseases of Livestock and Poultry, 1996(2): 1-5. (in Chinese) |
| [10] |
BAI X F, WANG Y Z, XU X, et al. Commercial vaccines provide limited protection to NADC30-like PRRSV Infection[J]. Vaccine, 2016, 34(46): 5540-5545. DOI:10.1016/j.vaccine.2016.09.048 |
| [11] |
ZHOU L, WANG Z C, DING Y P, et al. NADC30-like strain of porcine reproductive and respiratory syndrome virus, China[J]. Emerg Infect Dis, 2015, 21(12): 2256-2257. DOI:10.3201/eid2112.150360 |
| [12] |
张洪亮, 张文立, 许浒, 等. 2014年~2019年PRRSV主要流行毒株在我国的变化[J]. 中国预防兽医学报, 2020, 42(5): 512-516. ZHANG H L, ZHANG W L, XU H, et al. The changes of mainly endemic PRRSV in China during 2014-2019[J]. Chinese Journal of Preventive Veterinary Medicine, 2020, 42(5): 512-516. (in Chinese) |
| [13] |
郭振华, 陈鑫鑫, 李睿, 等. 中国猪繁殖与呼吸综合征病毒流行历史及现状[J]. 畜牧兽医学报, 2018, 49(1): 1-9. GUO Z H, CHEN X X, LI R, et al. The prevalent history and current status of porcine reproductive and respiratory syndrome in China[J]. Chinese Journal of Animal and Veterinary Sciences, 2018, 49(1): 1-9. (in Chinese) |
| [14] |
刘建奎, 范佳霖, 魏春华, 等. 2009─2017年福建省猪繁殖与呼吸综合征病毒分子流行病学调查和遗传变异分析[J]. 中国兽医科学, 2018, 48(5): 631-640. LIU J K, FAN J L, WEI C H, et al. Genetic diversity and evolutionary characteristics of PRRSV in Fujian province from 2009 to 2017[J]. Chinese Veterinary Science, 2018, 48(5): 631-640. (in Chinese) |
| [15] |
XU H, LI C, LI W S, et al. Novel characteristics of Chinese NADC34-like PRRSV during 2020-2021[J]. Transbound Emerg Dis, 2022, 69(5): e3215-e3224. DOI:10.1111/tbed.14485 |
| [16] |
LIU J K, WEI C H, LIN Z F, et al. Full genome sequence analysis of a 1-7-4-like PRRSV strain in Fujian Province, China[J]. PeerJ, 2019, 7: e7859. DOI:10.7717/peerj.7859 |
| [17] |
XIE C Z, HA Z, NAN F L, et al. Characterization of porcine reproductive and respiratory syndrome virus (ORF5 RFLP 1-7-4 viruses) in northern China[J]. Microb Pathog, 2019, 140: 103941. |
| [18] |
ZHANG H L, ZHANG W L, XIANG L R, et al. Emergence of novel porcine reproductive and respiratory syndrome viruses (ORF5 RFLP 1-7-4 viruses) in China[J]. Vet Microbiol, 2018, 222: 105-108. DOI:10.1016/j.vetmic.2018.06.017 |
| [19] |
魏春华, 夏伟, 李娜, 等. 福建省谱系3 PRRSV的分离及其ORF5基因序列分析[J]. 中国兽医杂志, 2019, 55(4): 20-24. WEI C H, XIA W, LI N, et al. Isolation and identification of lineage 3 PRRSV and sequence analysis of ORF5 gene[J]. Chinese Journal of Veterinary Medicine, 2019, 55(4): 20-24. (in Chinese) |
| [20] |
王隆柏, 张志灯, 车勇良, 等. 变异型PRRSV-FJ09A毒株ORF5和NSP2基因的遗传变异分析及致病性[J]. 福建农业学报, 2012, 27(10): 1032-1038. WANG L B, ZHANG Z D, CHE Y L, et al. Genetic variations of ORF5, NSP2 of variant PRRSV-FJ09A strain and it's pathogenicity[J]. Fujian Journal of Agricultural Sciences, 2012, 27(10): 1032-1038. DOI:10.3969/j.issn.1008-0384.2012.10.002 (in Chinese) |
| [21] |
王小敏, 何孔旺, 张文文, 等. 2008—2010年中国华东地区PRRSV ORF5基因遗传变异分析[J]. 中国农业科学, 2011, 44(18): 3886-3894. WANG X M, HE K W, ZHANG W W, et al. Genetic variation of the ORF5 gene of porcine reproductive and respiratory syndrome virus in East China from 2008 to 2010[J]. Scientia Agricultura Sinica, 2011, 44(18): 3886-3894. DOI:10.3864/j.issn.0578-1752.2011.18.020 (in Chinese) |
| [22] |
刘建奎. 福建省PRRSV分子流行病学调查及新毒株的致病性研究[J]. 广州: 华南农业大学, 2017. LIU J K. Molecular epidemiology inves tigation of PRRSV and pathogenicity of novel PRRSV in Fujian province[J]. Guangzhou: South China Agricultural University, 2017. |
| [23] |
李天宇, 常亚飞, 阮坤祥, 等. 一株类NADC30猪繁殖与呼吸综合征病毒的全基因组序列分析[J]. 中国动物传染病学报, 2020, 28(2): 39-47. LI T Y, CHANG Y F, RUAN K X, et al. Genomic analysis of a nadc30-like porcine reproductive and respiratory syndrome virus[J]. Chinese Journal of Veterinary Parasitology, 2020, 28(2): 39-47. (in Chinese) |
| [24] |
ZHOU L, KANG R M, JI G S, et al. Molecular characterization and recombination analysis of porcine reproductive and respiratory syndrome virus emerged in southwestern China during 2012-2016[J]. Virus Genes, 2018, 54(1): 98-110. DOI:10.1007/s11262-017-1519-y |
| [25] |
HANADA K, SUZUKI Y, NAKANE T, et al. The origin and evolution of porcine reproductive and respiratory syndrome viruses[J]. Mol Biol Evol, 2005, 22(4): 1024-1031. DOI:10.1093/molbev/msi089 |
| [26] |
YU F, YAN Y, SHI M, et al. Phylogenetics, genomic recombination, and NSP2 Polymorphic patterns of porcine reproductive and respiratory syndrome virus in China and the United States in 2014-2018[J]. J Virol, 2020, 94(6): e01813-19. |
| [27] |
杨汉春, 周磊, 周信荣. 2020年猪病流行情况与2021年流行趋势及防控对策[J]. 猪业科学, 2021, 38(2): 50-52. YANG H C, ZHOU L, ZHOU X R. Overview of swine diseases prevalence in 2020, epidemic tendency and control strategy in 2021[J]. Swine Industry Science, 2021, 38(2): 50-52. (in Chinese) |
| [28] |
JIANG Y F, XIA T Q, ZHOU Y J, et al. Characterization of three porcine reproductive and respiratory syndrome virus isolates from a single swine farm bearing strong homology to a vaccine strain[J]. Vet Microbiol, 2015, 179(3-4): 242-249. |
| [29] |
ZHOU L, YANG X R, TIAN Y, et al. Genetic diversity analysis of genotype 2 porcine reproductive and respiratory syndrome viruses emerging in recent years in China[J]. BioMed Res Int, 2014, 2014: 748068. |
| [30] |
SHI M, LAM T Y, HON C C, et al. Molecular epidemiology of PRRSV: a phylogenetic perspective[J]. Virus Res, 2010, 154(1-2): 7-17. |
| [31] |
CHA S H, CHANG C C, YOON K J. Instability of the restriction fragment length polymorphism pattern of open reading frame 5 of porcine reproductive and respiratory syndrome virus during sequential pig-to-pig passages[J]. J Clin Microbiol, 2004, 42(10): 4462-4467. |
| [32] |
FRIAS-DE-DIEGO A, JARA M, PECORARO B M, et al. Whole genome or single genes? A phylodynamic and bibliometric analysis of PRRSV[J]. Front Vet Sci, 2021, 8: 658512. |
| [33] |
DUDAS G, BEDFORD T. The ability of single genes vs full genomes to resolve time and space in outbreak analysis[J]. BMC Evol Biol, 2019, 1(19): 232. |
| [34] |
LIU J K, WEI C H, LIN Z F, et al. Recombination in lineage 1, 3, 5 and 8 of porcine reproductive and respiratory syndrome viruses in China[J]. Infect Genet Evol, 2019, 68: 119-126. |
| [35] |
谭祥梅, 吴瑕, 陈鹏飞, 等. 1株重组猪繁殖与呼吸综合征病毒的基因组特征[J]. 畜牧兽医学报, 2020, 51(12): 3181-3186. TAN X M, WU X, CEHN P F, et al. Genome characterization of a recombinant porcine reproductive and respiratory syndrome virus[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(12): 3181-3186. (in Chinese) |
| [36] |
周泷, 康润敏, 张毅, 等. 8株分离自四川、贵州地区猪繁殖与呼吸综合征病毒毒株的全基因组特征分析[J]. 畜牧兽医学报, 2020, 51(6): 1382-1390. ZHOU L, KANG R M, ZHANG Y, et al. Whole genome analysis of eight porcine reproductive and respiratory syndrome viruses isolated in Sichuan and Guizhou provinces[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(6): 1382-1390. (in Chinese) |
| [37] |
VAN AKEN D, ZEVENHOVEN-DOBBE J, GORBALENYA A E, et al. Proteolytic maturation of replicase polyprotein pp1a by the nsp4 main proteinase is essential for equine arteritis virus replication and includes internal cleavage of nsp7[J]. J Gen Virol, 2006, 87(12): 3473-3482. |
(编辑 白永平)



