2. 西北农林科技大学动物科技学院, 杨凌 712100;
3. 青海省格尔木市畜牧兽医站, 格尔木 816000;
4. 青海省格尔木市乌图美仁乡畜牧兽医站, 格尔木 816000
2. College of Animal Science and Technology, Northwest Agriculture and Forestry University, Yangling 712100, China;
3. Station of Animal Science and Veterinary Medicine of Golmud City of Qinghai Province, Golmud 816000, China;
4. Station of Animal Science and Veterinary Medicine of Wutumeiren Town in Golmud City of Qinghai Province, Golmud 816000, China
柴达木牛体型中等, 行动灵活, 体质结实, 结构匀称, 耐粗饲, 抗病力强,主产于青海省柴达木盆地边缘的都兰县、乌兰县和格尔木市等高海拔地区,是我国优良的地方黄牛品种之一[1]。先前基于生化遗传标记的研究表明,柴达木牛与蒙古牛具有较近的亲缘关系[2]。蒙古牛是在蒙古高原的大陆性严寒气候条件下,蒙古牧民历经多个世代的辛勤选育而形成的一个古老的地方品种[3],现广泛分布于蒙古国、俄罗斯及中国的内蒙古、东北、华北北部和西北地区,具有遗传性稳定、抗病耐粗饲、宜牧、抓膘能力强、适应性强等生物学特性,是利用放牧条件生存、繁殖、提供畜产品的一个地方特色黄牛品种[4-5]。
Y染色体是动物雄性个体所特有的性染色体,其单倍型完整, 突变率低, 不易受重组和回复突变等因素的影响,遵循父系遗传, 常用于探究动物父系遗传多样性、父系起源和驯化历史等研究[6]。牛Y染色体包括拟常染色体区(pseudoautosomal region,PAR)和雄性特异区(male-specific region,MSY),其中MSY区不与X染色体发生重组,是研究其父系起源的重要区域[7]。近年来, 众多关于家牛Y染色体SNPs标记(Y-SNPs) 和Y染色体特异性微卫星(Y-STRs)标记的研究,将家牛分为3种Y染色体单倍型组(即Y1、Y2和Y3)[8-10]。在全基因组水平上,Chen等[11]利用Y染色体基因组SNPs信息对世界家牛的父系单倍型组进行了更为精细的分类,将世界家牛的父系单倍型组细分为5个父系来源,即Y1、Y2a、Y2b、Y3a和Y3b,丰富了世界家牛的Y染色体遗传多样性并明确了其父系遗传背景。然而,对于柴达木牛和蒙古牛2个地方黄牛品种的父系遗传研究,目前大多只采用Y-SNPs或Y-STRs方法进行了探究[12-18]。尽管Chen等[11]初步分析了5头柴达木牛和7头蒙古牛的Y染色体基因组SNPs,但由于其所用样本数少,故难以准确地揭示2个黄牛品种的父系遗传多样性及遗传差异。鉴于此,本研究对柴达木牛和蒙古牛Y染色体基因组MSY区的X-退化区单拷贝基因进行全基因组SNP扫描分析,深入探究2个地方黄牛品种间的父系遗传多样性差异及其父系遗传背景,为后续开展2个地方黄牛品种种质资源的合理保护、开发和利用提供基础材料。
1 材料与方法 1.1 样品采集及数据来源22头柴达木牛公牛血样采自青海省海西蒙古族藏族自治州的乌兰县、都兰县、茫崖市、格尔木市和大柴旦行政区;同时,在我国内蒙古自治区采集8头、蒙古国采集15头共计23头蒙古牛公牛血样,于-80℃超低温冰箱冷冻保存。用苯酚-氯仿法提取基因组DNA并检测其浓度和纯度,将高质量的基因组DNA送往北京诺禾致源生物信息科技有限公司,构建长度为350 bp的文库,利用Illumina HiSeq 2000 PE150测序平台进行全基因组重测序。测序数据已上传至NCBI的SRA数据库(https://submit.ncbi.nlm.nih.gov/subs/sra/)中,项目号为PRJNA886843。此外,本研究选取Chen等[11]发表的代表家牛5种Y染色体单倍型组的基因组数据(SRR1365144、SRR5507257、SRR934415、SRR5507285和SRR2016752)作为参照。
1.2 Y染色体基因组数据分析将22头柴达木牛与23头蒙古牛基因组重测序的原始数据以及从NCBI下载的5个对照的家牛Y染色体基因组原始数据进行整合,用TRIMMOMATIC软件[19]进行质量检测控制,使用BWA-MEM软件[20]将质控后的clean reads比对到普通牛(Bos taurus)参考基因组(ARS-UCD1.2)上。使用GATK3.8软件[21]上的GenotypeGVCFs模块对22头柴达木牛、23头蒙古牛和5个家牛参照Y染色体基因组数据进行分型。对这50头黄牛Y染色体基因组MSY区的X-退化区数据,使用vcftools-0.1.16软件[22]提取其单拷贝基因SNP进行分析。Y染色体X-退化区SNP用snpEFF软件[23]进行注释,单倍型多样度(Hd±SD)和核苷酸多样度(Pi±SD)用DnaSP v5软件[24]进行计算。将所提取的SNP数据集用编写的脚本转换成fasta格式,并使用Muscle 3.8.31软件[25]对序列进行比对拉伸。使用RAxML-master v8.2.12软件[26]构建柴达木牛和蒙古牛的Y染色体基因组系统发育树,通过FigTree v1.4.3软件(http://tree.bio.ed.ac.uk/software/figtree/)实现可视化,最后使用R软件的pegas包[27]利用Muscle的fasta序列绘制Y-SNP单倍型网络图以揭示不同单倍型(组)间的系统发育关系。
2 结果 2.1 柴达木牛与蒙古牛Y染色体基因组SNP多态性全基因组重测序结果显示,22头柴达木牛基因组测序深度在8.14×~10.77×之间,平均测序深度为8.89×,而Y染色体基因组测序深度在3.52×~7.32×之间;23头蒙古牛基因组测序深度在9.30×~13.57×之间,平均测序深度为11.39×,Y染色体基因组测序深度范围为5.02×~ 8.05×之间。对22头柴达木牛、23头蒙古牛和5个家牛组成的数据集参照Chen等[11]研究中提取的745个Y染色体基因组X-退化区的单拷贝基因变异位点进行提取,共从这745个位点中提取到436个位点,snpEFF软件注释结果表明,共包含8个Y染色体单拷贝基因,分别是DDX3Y、EIF1AY、ZRSR2Y、EIF2S3Y、OFD1Y、USP9Y、UTY和ZFY。转换和颠换比值(Ts/Tv)为1.49,表明转换比例大于颠换比例,符合碱基替换的特征。柴达木牛和蒙古牛在这436个位点中去除存在信息缺失的位点后共有70个位点存在多态性。多样性参数计算结果表明:柴达木牛Y染色体基因组平均核苷酸差异数(K)为10.965,单倍型多样度(Hd±SD)为0.610±0.093,核苷酸多样度(Pi±SD)为0.074±0.015;而蒙古牛的Y染色体基因组平均核苷酸差异数为31.621,单倍型多样度为0.925±0.025,核苷酸多样度为0.137±0.013(表 1),提示蒙古牛相比柴达木牛具有更为丰富的Y染色体遗传多样性。
|
|
表 1 柴达木牛与蒙古牛Y染色体基因组单倍型多样度和核苷酸多样度 Table 1 Haplotype and nucleotide diversities of Y-chromosome genome in Qaidam cattle and Mongolian cattle |
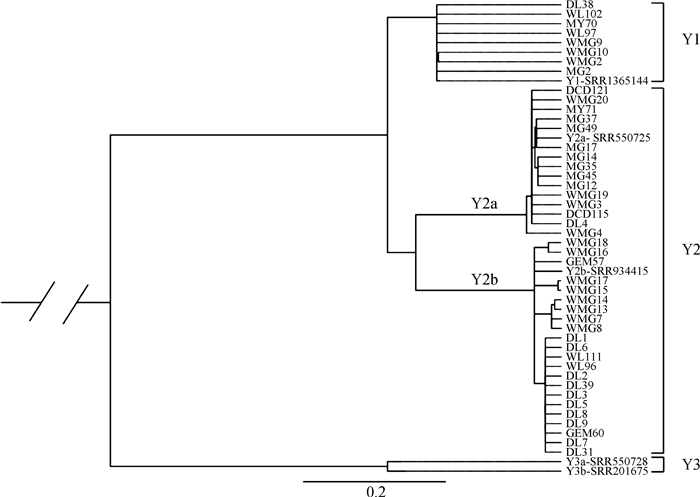
将50个个体436个位点的SNP数据集用编写的脚本转换成fasta格式,以5个已报道的家牛Y染色体基因组序列为参照[11, 28],构建柴达木牛与蒙古牛个体间Y染色体基因组最大似然树(图 1),结果表明,柴达木牛和蒙古牛都只有普通牛父系血统,包括Y1和Y2(包括Y2a和Y2b)两种单倍型组,拥有两个普通牛父系起源。在22头柴达木牛中,主要单倍型组为Y2b亚单倍型组,占63.6%(14/22),Y1单倍型组和Y2a亚单倍型组均占18.2%(4/22);而在23头蒙古牛中Y2a亚单倍型组个体最多,占47.8%(11/23),其次为Y2b亚单倍型组,比例为34.8%(8/23),Y1单倍型组个体最少,占17.4%(4/23)(表 1、表 2、图 1和图 2)。说明蒙古牛和柴达木牛均以Y2单倍型组为主,并包含少量的Y1单倍型组的个体,具有相似的父系遗传结构组成。略有区别的是柴达木牛以Y2b为优势亚单倍型组,而蒙古牛则是以Y2a为优势亚单倍型组,体现出两个黄牛品种在相似的父系遗传结构组成中也存在着一定程度的差异。
|
图 1 基于Y染色体基因组变异构建的柴达木牛和蒙古牛系统进化树 Fig. 1 Phylogenetic tree based on the Y-chromosome genomic variations of Qaidam cattle and Mongolian cattle |
|
|
表 2 柴达木牛与蒙古牛单倍型(组)信息 Table 2 Y-chromosome haplotypes/haplogroups information of Qaidam cattle and Mongolian cattle |
|
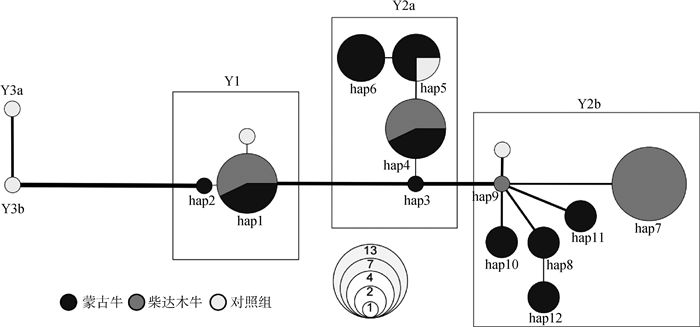
图 2 柴达木牛和蒙古牛Y染色体基因组单倍型(组)网络图 Fig. 2 Network diagram of Y-chromosome haplotypes/haplogroups of Qaidam cattle and Mongolian cattle |
除5个对照个体外,本研究在柴达木牛和蒙古牛Y染色体基因组单倍型分析中共确定了12种单倍型(即hap1~hap12)(表 2)。单倍型Network网络图中(图 2),上述12种单倍型中有2种单倍型(即hap1和hap2)属于Y1单倍型组,4种单倍型(即hap3~hap6)属于Y2a亚单倍型组,6种单倍型(即hap7~hap12)属于Y2b亚单倍型组。总体来看,柴达木牛拥有4种单倍型,蒙古牛有10种单倍型,有2种单倍型为两个品种所共有,其结果与构建的个体间最大似然树结果完全吻合。
3 讨论测序技术的不断更新使得动物Y染色体单倍型组的分类也更为详细,可以清晰地看出同一单倍型组又能分为不同的亚单倍型组。由于柴达木牛和蒙古牛Y染色体基因组尚未系统研究,因此,本研究利用全基因组重测序数据,对柴达木牛和蒙古牛Y染色体MSY的X-退化区Y-SNPs进行扫描和聚类分析,结果显示:柴达木牛和蒙古牛品种均只有普通牛父系起源,这与中国北方牛主要为普通牛类型的结论基本一致[29]。Li等[16-17]和Yue等[18]对蒙古牛Y染色体进行分子标记分析,结果表明蒙古牛父系起源由Y2单倍型组构成。Xia等[30]的研究发现,蒙古牛除了Y2单倍型组外还有少量Y1单倍型组。Chen等[11]对7头蒙古牛Y染色体基因组的分析,发现均为Y2a亚单倍型组。本研究结果与前人基本一致,略有不同的是,在本研究蒙古牛中除检测到Y2a亚单倍型组和少量Y1单倍型组外,还新检测到了Y2b亚单倍型组。Y1单倍型组的低水平存在可能是外来肉牛品种杂交所致。本研究新检测到蒙古牛含有Y2b亚单倍型组,其原因可能是Chen等[11]所使用的蒙古牛样本数较少的缘故。马志杰等[12-14]利用多种分子标记对柴达木牛Y染色体进行分析,表明柴达木牛含有Y1和Y2两个父系支系(单倍型组)。Chen等[11]的研究中5头柴达木牛均属于Y2a亚单倍型组。本研究结果显示,柴达木牛由Y1和Y2单倍型组构成,其中Y2单倍型组包括Y2a和Y2b两个亚单倍型组,且以Y2b为主,这与马志杰等[12-14]的研究结果一致,而与Chen等[11]的结果稍存在差异,其原因可能与后者使用的试验个体少、群体单一有关。
单倍型多样度和核苷酸多样度是评估动物群体遗传多样性的重要指数,其数值越高,群体遗传多样性就越丰富。本研究中,柴达木牛的核苷酸多样度(0.074±0.015)明显低于蒙古牛的核苷酸多样度(0.137±0.013),说明蒙古牛拥有更为丰富的父系遗传变异。在先前的研究中,马志杰等[14]研究发现,柴达木牛Y染色体单倍型多样度为0.337±0.061,低于中国黄牛品种的平均水平,提示柴达木牛单倍型多样度比较贫乏。Xia等[30]的研究结果显示,蒙古牛Y染色体单倍型多样度为0.550±0.038,蒙古牛为我国北方黄牛品种中单倍型多样度最高的群体。本研究中,柴达木牛Y染色体单倍型多样度为0.610±0.093,仍低于同为我国北方黄牛的蒙古牛(0.925±0.025),这可能与柴达木牛所处地理位置偏僻、有效群体数量下降、品种退化、牛群多为封闭性选育[31]等因素相关。而蒙古牛主要分布在蒙古高原及其毗邻地区,牧草丰美、地域开阔,为其基因交流带来极大的便利,使蒙古牛品种拥有丰富的遗传多样性。
整体来看,柴达木牛和蒙古牛虽然只有普通牛父系血统,但其具有较为复杂的父系起源。据柴达木牛品种形成和培育的历史资料记载[32],柴达木牛由蒙古牛风土驯化而成,近代受哈萨克牛、中亚瘤牛(Bos indicus)的影响,建国后又受荷斯坦牛、西门塔尔牛、秦川牛、三河牛等品种改良影响,导致柴达木牛形成较宽泛的父系遗传基础和起源。由本研究单倍型(组)网络关系结果(图 2)可以看出,柴达木牛与蒙古牛具有相同的Y1和Y2a的单倍型(即hap1和hap4),这也证实了柴达木牛具有一部分蒙古牛的父系起源;而柴达木牛和蒙古牛在Y2b亚单倍型组中的差异也说明了柴达木牛的Y2b亚单倍型组并不来源于蒙古牛,说明柴达木牛拥有自己独特的父系来源和特色。
4 结论本研究利用二代重测序技术系统分析了柴达木牛和蒙古牛两个品种的父系遗传结构及其多样性,发现柴达木牛和蒙古牛具有相似的父系遗传组成,与柴达木牛由蒙古牛风土驯化而成的历史资料记载基本一致。但与蒙古牛相比,柴达木牛拥有自己独特的父系来源信息。柴达木牛相比蒙古牛父系遗传多样度较为贫乏,考虑到其父系遗传多样性较低,故建议加大柴达木牛品种内群体间种公牛的基因交流,并制定长远的选种选配方案,提高其遗传多样性水平,这对柴达木牛遗传资源的合理保护和利用具有实际意义。蒙古牛作为我国北方代表性地方黄牛品种,其父系遗传多样性丰富,有利于蒙古牛的遗传资源保护和开发利用。
| [1] |
完么才郎. 青海柴达木黄牛种质特性的调查分析[J]. 畜牧兽医科技信息, 2011(8): 124-125. WANME C L. Investigation and analysis of the germplasm characteristics of Qinghai Qaidam Cattle[J]. Chinese Journal of Animal Husbandry and Veterinary Medicin, 2011(8): 124-125. DOI:10.3969/J.ISSN.1671-6027.2011.08.084 (in Chinese) |
| [2] |
张才骏, 张海峰, 晃生玉. 柴达木黄牛与我国其它品种黄牛亲缘关系的探讨[J]. 中国畜牧杂志, 2001, 37(6): 9-11. ZHANG C J, ZHANG H F, HUANG S Y. Study on the blood relationship among Chaidamu and other breeds of yellow cattle in China[J]. Chinese Journal of Animal Science, 2001, 37(6): 9-11. DOI:10.3969/j.issn.0258-7033.2001.06.003 (in Chinese) |
| [3] |
齐昱, 邢燕平, 潘静, 等. 基于转录组数据的蒙古牛皮肤组织抗寒相关信号通路及候选基因的筛选[J]. 畜牧兽医学报, 2017, 48(12): 2301-2313. QI Y, XING Y P, PAN J, et al. Screening of cold Tolerance-related signaling pathways and candidate genes in Mongolia cattle skin tissues based on transcriptome data[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(12): 2301-2313. DOI:10.11843/j.issn.0366-6964.2017.12.010 (in Chinese) |
| [4] |
马哈沙提·吐苏别克. 关于蒙古牛改良沿革及肉牛饲养技术路线[J]. 畜禽业, 2020, 31(3): 14. MA H S T. On the history of Mongolian cattle improvement and the technical route of beef cattle rearing[J]. Livestock and Poultry Industry, 2020, 31(3): 14. DOI:10.19567/j.cnki.1008-0414.2020.03.007 (in Chinese) |
| [5] |
王小梅, 查嘎巴拉. 自然放牧条件下锡林郭勒盟地区蒙古牛胴体性状的研究[J]. 当代畜禽养殖业, 2019(11): 12-14. WANG X M, CHA G B L. Study on carcass traits of Mongolian cattle in Xilinguolemeng region under natural grazing conditions[J]. Modern Animal husbandry, 2019(11): 12-14. DOI:10.3969/j.issn.1005-5959.2019.11.004 (in Chinese) |
| [6] |
CORTEZ D, MARIN R, TOLEDO-FLORES D, et al. Origins and functional evolution of Y chromosomes across mammals[J]. Nature, 2014, 508(7497): 488-493. DOI:10.1038/nature13151 |
| [7] |
董依萌, 王天骄, 刘华淼, 等. 线粒体DNA和Y染色体主要基因的研究进展[J]. 安徽农业科学, 2020, 48(8): 18-22. DONG Y M, WANG T J, LIU H M, et al. Research progress on the major genes of mitochondria and Y chromosome[J]. Journal of Anhui Agricultural Sciences, 2020, 48(8): 18-22. DOI:10.3969/j.issn.0517-6611.2020.08.004 (in Chinese) |
| [8] |
常振华, 黄洁萍, 徐苹, 等. 中国黄牛Y-STRs遗传多样性与起源研究[J]. 中国牛业科学, 2012, 38(3): 9-13. CHANG Z H, HUANG J P, XU P, et al. Genetic diversity and ori n based on Y-STRs in Chinese cattle[J]. China Cattle Science, 2012, 38(3): 9-13. DOI:10.3969/j.issn.1001-9111.2012.03.004 (in Chinese) |
| [9] |
常振华, 卫利选, 张润锋, 等. 中国黄牛Y-SNPs遗传多样性与起源研究[J]. 畜牧兽医学报, 2011, 42(11): 1537-1542. CHANG Z H, WEI L X, ZHANG R F, et al. Genetic diversity and origin based on Y-SNPs in Chinese cattle[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(11): 1537-1542. (in Chinese) |
| [10] |
GÖTHERSTRÖM A, ANDERUNG C, HELLBORG L, et al. Cattle domestication in the Near East was followed by hybridization with aurochs bulls in Europe[J]. Proc Biol Sci, 2005, 272(1579): 2345-2350. |
| [11] |
CHEN N B, CAI Y D, CHEN Q M, et al. Whole-genome resequencing reveals world-wide ancestry and adaptive introgression events of domesticated cattle in East Asia[J]. Nat Commun, 2018, 9(1): 2337. DOI:10.1038/s41467-018-04737-0 |
| [12] |
马志杰, 胡双龙, 李瑞哲, 等. 柴达木黄牛Y染色体单倍型组构成及父系起源——基于USP9Y基因多态性的分析[J]. 中国畜牧杂志, 2017, 53(6): 36-39. MA Z J, HU S L, LI R Z, et al. Y Chromosome haplogroups and paternal origin of Qaidam cattle: based on the Polymor phism analysis of USP9Y gene[J]. Chinese Journal of Animal Science, 2017, 53(6): 36-39. (in Chinese) |
| [13] |
马志杰, 夏小婷, 陈生梅, 等. 基于Y染色体ZFY-10标记多态性探究柴达木黄牛父系支系组成和起源[J]. 中国农业大学学报, 2018, 23(6): 70-76. MA Z J, XIA X T, CHEN S M, et al. Explore the patrilineal branches and origin of Qaidam cattle based on the polymorphism of Y chromosome ZFY-10 marker[J]. Journal of China Agricultural University, 2018, 23(6): 70-76. (in Chinese) |
| [14] |
马志杰, 李瑞哲, 夏小婷, 等. 柴达木黄牛Y-SNPs遗传多样性、群体遗传结构及父系起源[J]. 基因组学与应用生物学, 2018, 37(5): 1920-1925. MA Z J, LI R Z, XIA X T, et al. Y-SNPs genetic diversity, population genetic structure and paternal origin of Qaidam cattle[J]. Genomics and Applied Biology, 2018, 37(5): 1920-1925. (in Chinese) |
| [15] |
杨秋蕾, 马志杰, 李广祯, 等. 牛种间UTY基因内含子19(UTY19)序列遗传变异研究[J]. 青海畜牧兽医杂志, 2020, 50(6): 7-10, 14. YANG Q L, MA Z J, LI G Z, et al. Study on genetic variations of intron19 of UTY Gene (UTY19) among bovine interspecific[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 2020, 50(6): 7-10, 14. (in Chinese) |
| [16] |
LI R, XIE W M, CHANG Z H, et al. Y chromosome diversity and paternal origin of Chinese cattle[J]. Mol Biol Rep, 2013, 40(12): 6633-6636. DOI:10.1007/s11033-013-2777-y |
| [17] |
LI R, ZHANG X M, CAMPANA M G, et al. Paternal origins of Chinese cattle[J]. Anim Genet, 2013, 44(4): 446-449. DOI:10.1111/age.12022 |
| [18] |
YUE X P, LI R, LIU L, et al. When and how did Bos indicus introgress into Mongolian cattle?[J]. Gene, 2014, 537(2): 214-219. |
| [19] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. |
| [20] |
HOUTGAST E J, SIMA V M, BERTELS K, et al. Hardware acceleration of BWA-MEM genomic short read mapping for longer read lengths[J]. Comput Biol Chem, 2018, 75: 54-64. |
| [21] |
MCKENNA A, HANNA M, BANKS E, et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Res, 2010, 20(9): 1297-1303. |
| [22] |
DANECEK P, AUTON A, ABECASIS G, et al. The variant call format and VCFtools[J]. Bioinformatics, 2011, 27(15): 2156-2158. |
| [23] |
CINGOLANI P, PLATTS A, WANG L L, et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3[J]. Fly (Austin), 2012, 6(2): 80-92. |
| [24] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. |
| [25] |
EDGAR R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Res, 2004, 32(5): 1792-1797. |
| [26] |
STAMATAKIS A. RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics, 2014, 30(9): 1312-1313. |
| [27] |
PARADIS E. pegas: an R package for population genetics with an integrated-modular approach[J]. Bioinformatics, 2010, 26(3): 419-420. |
| [28] |
XIA X T, ZHANG S J, ZHANG H J, et al. Assessing genomic diversity and signatures of selection in Jiaxian Red cattle using whole-genome sequencing data[J]. BMC Genomics, 2021, 22(1): 43. |
| [29] |
陈宁博, 雷初朝. 从DNA角度认识中国黄牛的起源和利用历史[J]. 第四纪研究, 2022, 42(1): 92-100. CHEN N B, LEI C Z. The origins and utilization history of Chinese cattle as revealed by DNA analysis[J]. Quaternary Sciences, 2022, 42(1): 92-100. (in Chinese) |
| [30] |
XIA X, YAO Y, LI C, et al. Genetic diversity of Chinese cattle revealed by Y-SNP and Y-STR markers[J]. Anim Genet, 2019, 50(1): 64-69. |
| [31] |
尚有安, 王照忠, 杨坤. 德令哈市柴达木黄牛品种资源调查[J]. 畜牧与兽医, 2012, 44(8): 106-107. SHANG Y A, WANG Z Z, YANG K. A survey on the resources of the breed of Qaidam cattle in Delingha[J]. Animal Husbandry & Veterinary Medicine, 2012, 44(8): 106-107. (in Chinese) |
| [32] |
国家畜禽遗传资源委员会. 中国畜禽遗传资源志: 牛志[M]. 北京: 中国农业出版社, 2011. National Livestock and Poultry Genetic Resources Committee. Animal genetic resources in China: Bovines[M]. Beijing: China Agriculture Press, 2011. (in Chinese) |
(编辑 孟培)



