猪瘟(classical swine fever,CSF)是由猪瘟病毒(classical swine fever virus,CSFV)引起的一种传染性极强且临床以高热、出血和免疫抑制为表征的烈性传染病,给养猪业造成巨大的经济损失[1]。CSFV属于黄病毒科、瘟病毒属,其直径为40~60 nm,基因组大小约为12.5 kb[2]。CSFV编码产生4个结构蛋白(Core、Erns、E1和E2)和8个非结构蛋白(Npro、p7、NS2、NS3、NS4A、NS4B、NS5A和NS5B)[3]。CSFV NS5A是一种多功能的磷酸化蛋白,包括497个氨基酸,定位于细胞的内质网,具有参与病毒复制和调节宿主细胞信号通路的作用,包括细胞周期、细胞内新陈代谢、细胞内mRNA翻译、蛋白质稳定表达、血管生成和通透性以及胞内蛋白折叠等[4-5]。NS5A与鸟苷酸结合蛋白1互作抑制GTPase活性,进而影响猪瘟病毒的早期复制[6]。Rab1A作为CSFV组装所必须的蛋白与NS5A存在协同作用,促进病毒粒子的组装;缺失则导致CSFV的毒力下降[7]。NS5A在细胞内的分布经由Rab18的调节,其复合物参与CSFV RNA复制和病毒粒子组装[8]。NS5A一方面与热休克蛋白Hsp27相互作用进而负调控病毒复制[9];另一方面与热休克蛋白HSP70共定位于细胞质中,其N端氨基酸(29~240)与HSP70结合形成的复合物促进CSFV的复制[10]。此外,HSP90AB1/HSPCB与NS5A也存在相互作用[11]。GRP78与NS5A相互作用,在激活细胞未折叠蛋白反应中扮演重要角色,促进猪瘟病毒的复制[12]。NS5A能与ARFGAP互作促进CSFV复制,另一方面NS5A诱导的内质网应激可经由ARFGAP降低[13]。
研究表明,猪瘟病毒通过激活宿主细胞自噬促进其增殖[14-15]。自噬作为细胞程序性死亡的一种方式,表现为将宿主细胞中受损蛋白质及损伤、衰老的细胞器进行批量降解,而后循环利用细胞内营养物质,参与调节代谢平衡、维持细胞内环境稳态,促进生物体正常生长发育和分化[16-17]。自噬相关蛋白Beclin1、P62/SQSTM1及LC3等,在试验中经常被用作自噬的标记物[18]。这些基因通常介导自噬小体的形成,并与溶酶体结合[19]。自噬相关基因按其功能分类可分为诱导自噬,囊泡成核、延伸、形成和再循环的自噬过程,以及针对修饰蛋白或受损细胞器的选择性自噬降解[20]。Beclin1因其24.4%的氨基酸序列与酵母自噬蛋白Atg6相同,成为第一个被证实的正向调节自噬的哺乳动物基因之一[21]。Beclin1在PI3K激酶ClassⅢ复合物中扮演核心角色,该复合物由Vps34、Vps15、Beclin1三个主要亚基组成,作为自噬起始的复合物,其在自噬小体的形成中不可或缺[22]。目前已知Beclin1参与调节多个信号通路,包括PI3K-AKT-mTOR、AMPK、JNK及MAPK等通路[23]。研究表明,Beclin1在宿主感染丙型肝炎病毒(hepatitis C virus,HCV)引起的慢性肝炎病例中起到抑制病毒增殖作用[24]。在Fu等[25]的研究中,Beclin1等自噬相关基因表达增加可通过调节自噬促进病毒增殖。经研究HCV NS5A通过活化蛋白C激酶1受体受体与III类PI3K复合物相互作用[26]。
但Beclin1蛋白在CSFV诱发宿主细胞发生自噬的过程中是否发挥作用及其作用机制,还未见研究报道。本研究旨在探究宿主蛋白Beclin1在CSFV NS5A激活细胞自噬反应过程中所发挥的影响。研究结果表明,Beclin1蛋白促进CSFV增殖的机制是通过与CSFV NS5A的相互作用调控PI3K/Akt通路来实现,为猪瘟的有效防控提供理论依据。
1 材料与方法 1.1 材料1.1.1 生物材料 猪瘟病毒CSFV Shimen株,猪肾细胞系(ST)和非洲绿猴肾细胞(Vero)由本实验室保存。真核表达载体pDsRed1-N1购自Clontech公司,pcDNATM 3.1(+)购自Invitrogen公司。
1.1.2 主要试剂 DMEM培养基、TMB显色液、T4 DNA连接酶均购自Thermo Fisher Scientific公司;HRP标记羊抗鼠IgG和HRP标记羊抗兔IgG购自Sigma公司;胎牛血清(FBS)购自LONSERA公司;限制性内切酶BamHⅠ、EcoRⅠ和Xho Ⅰ购自NEB公司;超纯RNA提取试剂盒购自江苏康为世纪生物科技有限公司;AxyPrep DNA凝胶回收试剂盒及AxyPrep质粒DNA小量提取试剂盒均购自康宁生命科学(吴江)有限公司;Trans1-T1感受态细胞、反转录试剂盒、EasyTaqⓇ DNAPolymerase、琼脂糖等均购自北京全式金生物技术有限公司;AceQ qPCR Probe Master Mix购自南京诺唯赞生物科技股份有限公司;DL2000 Marker,DL10000 Marker均购自TaKaRa公司;青霉素/链霉素/两性霉素B溶液(无菌)购自生工生物工程(上海)有限公司。
1.1.3 主要仪器设备 PCR扩增仪及实时荧光定量PCR仪,杭州博日科技有限公司;电泳仪、泳凝胶成像系统,上海天能科技有限公司;CO2恒温培养箱,Thermo Fisher Scientific公司;倒置荧光光显微镜,Olympus Corporation公司;倒置光学显微镜,Leica仪器有限公司;超净工作台,苏州净化设备有限公司。
1.2 方法1.2.1 细胞培养 在无菌离心管中加入5 mL DMEM培养液备用,从液氮罐中取出冻存的ST细胞迅速放入37 ℃水浴中溶化至冰水混合状态,轻轻吹打数次转入准备好的离心管中,1 000 r·min-1室温离心2 min,将沉淀转入含有10%FBS的DMEM中,37 ℃、5% CO2培养24~48 h,显微镜下观察细胞生长状况,在细胞呈现单层生长时传代。弃去培养液,PBS缓慢清洗2~3次,加入胰蛋白酶消化2~3 min,当细胞呈现大部分脱落后加入血清终止消化,反复吹打至细胞成单个,加入培养液继续培养。
1.2.2 荧光定量PCR检测方法 将处于对数生长期的猪源细胞,在其汇集度在70%左右时更换DMEM基础培养基;接种1个moi量的CSFV,吸附1 h后,更换含有10%FBS的DMEM培养基作用6、12、24、36、48 h后收集细胞。参照江苏康为世纪生物科技有限公司生产的试剂盒的说明书的步骤提取总RNA,提取完成后将之反转录为cDNA。反转录体系:5×TranScriptⓇRT Buffer 2 μL,TranScriptⓇRT 0.5 μL,RI 0.25 μL,dNTPs 2 μL,6 nt 1 μL,Total mRNA 4.25 μL。反转录条件:25 ℃ 5 min,42 ℃ 30 min,85 ℃ 5 min。反转录产物-20 ℃保存备用。
参照GenBank数据库中公布的Sus scrofa的Beclin1、Vps34、Vps15、ATG14L、UVRAG的基因序列进行综合分析,设计设计PCR扩增的上、下游引物,送交南京金斯瑞生物科技有限公司合成。引物序列见表 1。
|
|
表 1 PCR引物序列 Table 1 The sequence of PCR primers |
将反转录得到的cDNA稀释为100 ng·μL-1,按照SYBR Green PCR MasterMix操作说明,利用上述合成的荧光定量引物分别对CSFV感染细胞中Beclin1、Vps34、Vps15、ATG14L、UVRAG mRNA水平进行相对荧光定量,RT-PCR反应体系:2×AceQⓇqPCR SYBR GREEN Master Mix 10 μL,cDNA 2 μL,Forward Primer 0.5 μL,Reverse Primer 0.5 μL,dd H2O补齐至20 μL。每个样品重复3次。反应程序:50 ℃ 2 min,95 ℃ 2 min;98 ℃ 15 s,58 ℃ 15 s,72 ℃ 1 min,40个循环。
1.2.3 重组表达载体的构建 取获得的ST细胞的cDNA,使用设计的特异性引物扩增带有不同酶切位点的Beclin1基因的片段。通过BamHⅠ、EcoRⅠ和XhoⅠ等酶作用进行双酶切后与相应载体连接分别克隆至对应载体用于后续试验。
1.2.4 Western blot 细胞质、细胞核蛋白的提取参照上海生工生物工程技术服务有限公司生产的试剂盒说明书的步骤进行:收集CSFV感染6、12、24、36、48 h的ST细胞,用预冷的PBS洗后,用细胞刮刀收集细胞,1 000 r·min-1离心2~3 min,弃上清留沉淀备用。将新鲜配制的200 μL RIPA(含2 μL 100×PMSF)裂解液裂解蛋白,冰上裂解10~15 min,将充分裂解的蛋白于4 ℃、12 000 r·min-1离心15 min,让细胞碎片沉入EP管底,吸取含有蛋白的上清至新的预冷的EP管里,抽取适量的上清利用BCA法测定蛋白浓度。剩余的上清蛋白液中加入一定量的5×SDS PAGE Loading buffer,金属浴100 ℃10 min使蛋白质变性。依据所测目的蛋白大小配制对应浓度的SDS-PAGE胶,电泳恒压120 V 90 min,完毕后冰上转印至PVDV膜上,电泳恒流120 mA 90 min,然后采用5%脱脂奶粉室温封闭2 h;TBST缓冲液洗膜2~3次,每次5 min;4 ℃过夜孵育一抗;TBST缓冲液漂洗3次,每次5 min;加入二抗室温孵育2 h,TBST缓冲液漂洗3次,每次15 min。将清洗干净的PVDV膜置于ECL发光成像仪中显影照相,分析结果并保存。
1.2.5 免疫共沉淀 将NS5A-Flag和Beclin1-Myc重组载体共转染ST细胞48 h后,收集细胞,用RIPA(含1% 100×PMSF)裂解液裂解蛋白,冰上裂解10~15 min。根据Thermo ScientificTM PierceTM c-Myc Tag co-IP说明书,将裂解所获蛋白液与抗c-Myc或抗Flag琼脂糖亲和凝胶进行免疫共沉淀试验。简要步骤如下:将200 μL细胞裂解液与10 μL琼脂糖浆混合,并使用150 μL TBS(25 mmol·L-1 Tris盐酸,0.15 mol·L-1氯化钠,pH7.2)稀释的50 μL阳性对照作为阳性对照,在4 ℃下孵育过夜。免疫沉淀用TBST洗涤3次,并在2×非还原样品缓冲液中重悬。煮沸5 min后,在样品中加入2 μL的2-ME,用特异性抗体进行免疫印迹分析。
1.2.6 GST-pulldown 按照GST-Pulldown说明书,IPTG诱导BL21原核表达pEGX-6p-1-Beclin1和pEGX-6p-1空载体;用预冷的200 μL TBS重悬菌体,离心后加入200 μL Pulldown裂解液,冰上裂解30 min,离心收集上清;将上清液与GST琼脂糖珠孵育2 h,离心收集GST-p17-琼脂糖复合沉淀物;收集已在真核细胞中表达的NS5A-Flag细胞蛋白,加入适量pulldown裂解液,冰上裂解30 min,离心收集上清(含NS5A-Flag蛋白);将收集的NS5A-Flag上清液和GST-Beclin1琼脂糖复合物中孵育过夜;离心弃上清,得到Beclin1-NS5A结合复合在此复合物中加入EB洗液(含谷胱甘肽),离心吸取40 μL上清,加入10 μL 5×蛋白上样缓冲液,通过Western blot检测目的蛋白。
1.2.7 激光共聚焦显微镜方法 将细胞爬片置于75%乙醇中消毒15 min后放入24孔板中,在24孔板内接种ST细胞,当细胞密度达到70%~90%时,将重组质粒Beclin1-Myc和NS5A-Flag共同转染至ST细胞。转染48 h后,弃去培养基后PBS清洗3次,每次5 min。预冷的无水甲醛固定15 min,弃掉固定液,PBS清洗3次,每次5 min;5%脱脂奶封闭1 h;吸弃封闭液,孵育一抗(兔抗Myc单抗和鼠抗Flag单抗);孵育二抗(FITC标记的羊抗兔IgG、Cy3标记的羊抗鼠IgG);避光加入DAPI(1∶2 000稀释)染色15~20 min;利用倒置荧光显微镜观察结果;指甲油封片后滴一滴10%甘油,用激光共聚焦显微镜DIPA、FITC、Cy3激发光激发荧光,观察Beclin1与NS5A位置分布。
1.2.8 数据统计分析 所有试验至少有3个独立重复,所有数据结果以“x±s”表示,采用GraphPad Prism软件进行数据的统计分析。*. P<0.05,表示显著性差异;**.P<0.01或***.P<0.001,表示差异极显著。
2 结果 2.1 真核表达载体构建Beclin1基因的PCR扩增:收取ST细胞RNA样后,加入Trizon后提取RNA并反转录成cDNA,用EasyTaqⓇ DNAPolymerase酶PCR扩增Beclin1全长,取部分产物进行电泳;由图 1A所示条带大小和预期相符。
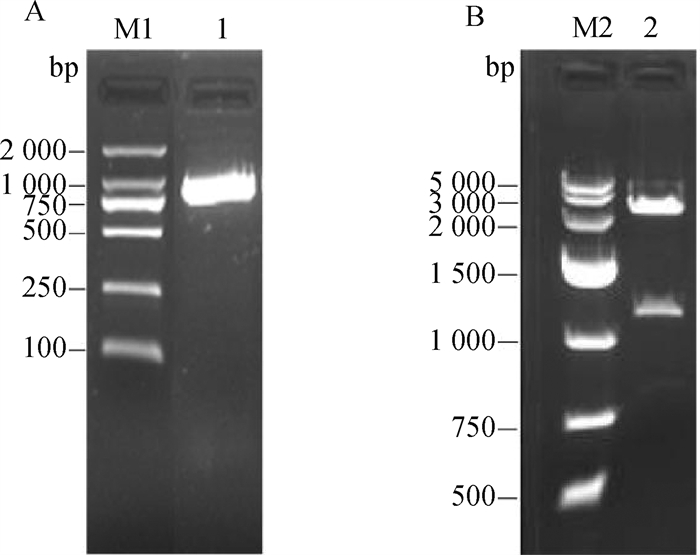
|
M1. DL2000 DNA相对分子质量标准;1.Beclin1基因PCR扩增;M2.DL5000 DNA相对分子质量标准;2.重组载体双酶切鉴定 M1. DL2000 DNA marker; 1. PCR amplification of Beclin1 gene; M2. DL5000 DNA marker; 2. Identification of recombinant vector by double enzyme digestion 图 1 Beclin1-RedN1重组载体构建 Fig. 1 Construction of recombinant vector Beclin1-RedN1 |
重组质粒双酶切鉴定:将Beclin1全长产物与pDsRedN1空载体双酶切后,22 ℃连接1 h并进行转化,将菌液PCR鉴定为阳性的菌液扩大培养,提取质粒后采用对应酶进行双酶切并进行电泳鉴定。结果见图 1B,条带大小与预期一致。
2.2 猪瘟病毒及NS5A蛋白上调Beclin1蛋白表达ST细胞在感染CSFV及转染表达NS5A蛋白后,于不同时间点分别收取RNA及蛋白样品,利用qRT-PCR及Western blot分别检测Beclin1转录及蛋白水平变化,结果如图 2显示,CSFV感染及NS5A蛋白均能显著上调Beclin1的表达水平。
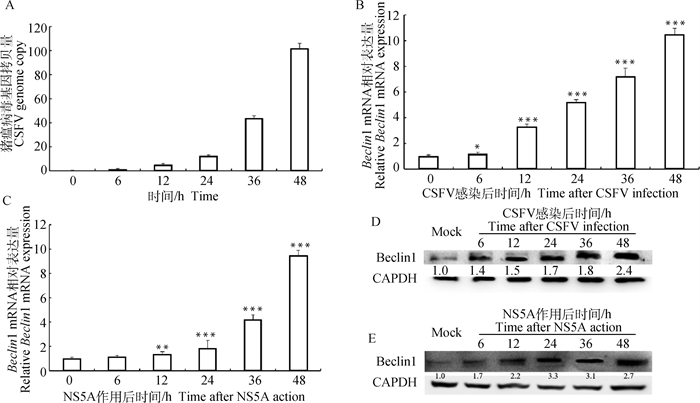
|
A. qRT-PCR检测CSFV增殖量;B. qRT-PCR检测CSFV对Beclin1表达影响;C. qRT-PCR检测NS5A对Beclin1表达影响;D. Western blot检测CSFV对Beclin1表达影响;D. Western blot检测NS5A对Beclin1表达影响。与对照组相比,*.P<0.05;**.P<0.01;***.P<0.001 A. qRT-PCR to detect the proliferation of CSFV; B. qRT-PCR detection of the effect of CSFV on Beclin1 expression; C. qRT-PCR detection of the effect of NS5A on Beclin1 expression; D. Western blot detection of the effect of CSFV on Beclin1 expression; D. Western blot detection of the effect of NS5A on Beclin1 expression. Compared with control, *.P < 0.05;**.P < 0.01;***.P < 0.001 图 2 猪瘟病毒与NS5A蛋白上调Beclin1表达 Fig. 2 CSFV and NS5A protein up-regulated Beclin1 expression |
将Beclin1-RedN1真核表达载体转入ST细胞,实现对Beclin1蛋白的过表达,同时,设置空载体转染组作为对照;之后感染CSFV,于不同时间点收取细胞样品,结果如图 3所示,CSFV复制水平显著提高。相反,利用si-RNA敲低Beclin1表达后,CSFV增殖受到显著抑制。
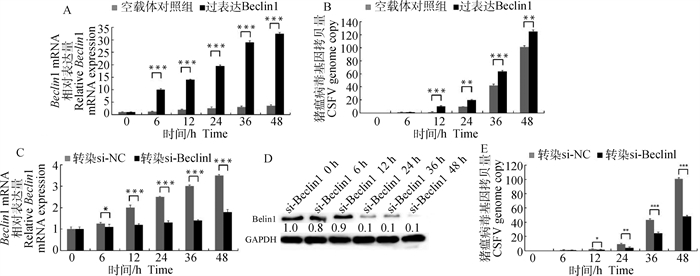
|
A.过表达Beclin1后qRT-PCR检测Beclin1表达水平;B.过表达Beclin1后qRT-PCR检测CSFV增殖水平;C.敲低Beclin1后qRT-PCR检测Beclin1表达水平;D.敲低Beclin1后Western blot检测Beclin1表达水平;E. qRT-PCR检测CSFV增殖水平;**.P<0.01;***.P<0.001 A. qRT-PCR detection of Beclin1 expression level after overexpression of Beclin1; B. qRT-PCR detection of CSFV proliferation level after overexpression of Beclin1; C. qRT-PCR detection of Beclin1 expression level after knock down of Beclin; D. Western blot detection of Beclin1 expression level after knock down of Beclin; E. qRT-PCR detection of CSFV proliferation level after knock down of Beclin. **.P < 0.01;***.P < 0.001 图 3 Beclin1促进CSFV复制 Fig. 3 Beclin1 promotes CSFV replication |
进行免疫共沉淀试验验证Beclin1蛋白是否与NS5A相互作用。将Beclin1-Myc和NS5A-Flag质粒共转染ST细胞24 h,细胞裂解液与抗Flag亲和凝胶免疫共沉淀。最后,用蛋白印迹法检测复合物中的蛋白。结果显示,Beclin1-Myc和NS5A-Flag蛋白相互作用(图 4)。
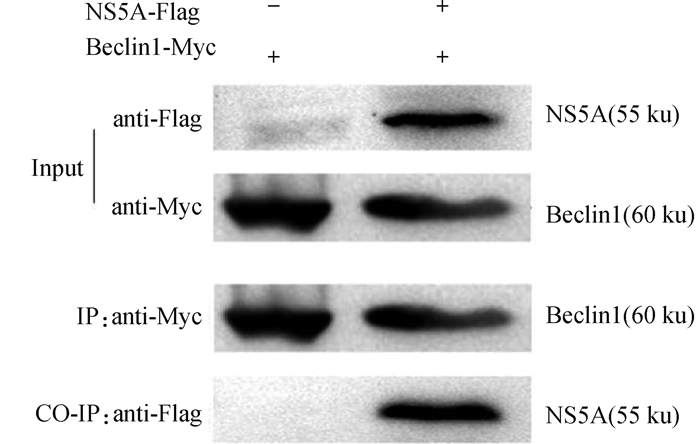
|
用NS5A-Flag和Beclin1-Myc质粒共转染ST细胞48 h后收取蛋白样。样品经抗Flag的抗体免疫沉淀,然后进行免疫印迹分析 The protein samples were collected after co-transfection of ST cells with NS5A-Flag and Beclin1-Myc plasmids for 48 h. Cell lysates were immunoprecipitated with anti-Flag antibodies, followed by Western blot analysis 图 4 CSFV NS5A与Beclin1相互作用 Fig. 4 CSFV NS5A interacts with Beclin1 |
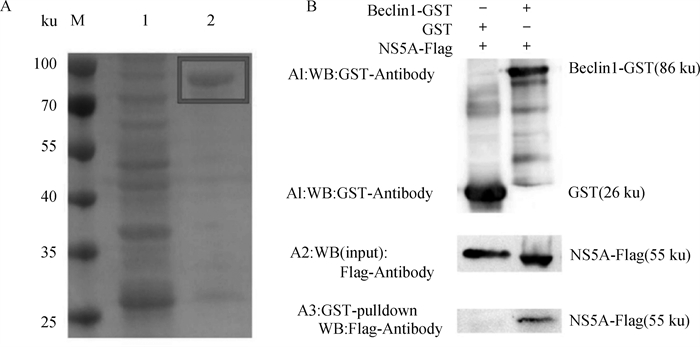
采用GST-pulldown试验,进一步证实Beclin1与NS5A之间的相互作用在体外条件下是否成立。重组全长Beclin1-GST融合蛋白和GST蛋白分别在大肠杆菌BL21(DE3)菌株中诱导表达,并将NS5A-Flag转染到ST细胞中。采用GST-pulldown法检测Beclin1与NS5A之间的关系。如图所示,在Beclin1-GST复合物中检测到NS5A-Flag(图 5)。该结果说明Beclin1在体外可以与NS5A相互作用。
|
A. GST空载体及Beclin1-GST重组质粒原核诱导表达;B. GST-pulldown试验验证蛋白互作 A. Prokaryotic expression of GST empty vector and Beclin1-GST recombinant plasmid; B. GST-pulldown experiment to verify protein interaction 图 5 GST-Pulldown研究Beclin1与CSFV NS5A相互作用 Fig. 5 GST-Pulldown study of Beclin1 interaction with CSFV NS5A |
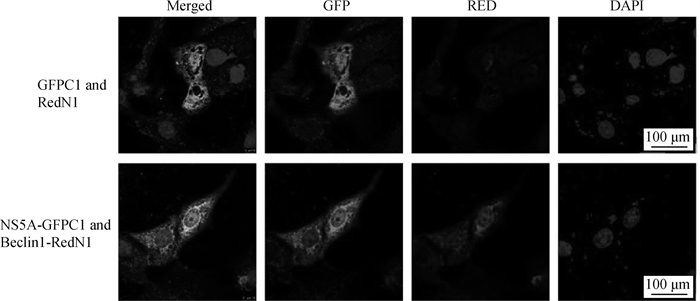
利用激光共聚焦显微镜观察Beclin1与NS5A在ST细胞中的亚细胞定位,在ST细胞中共转染Beclin1-RedN1和NS5A-GFPC1真核表达载体。36 h后制备细胞爬片,利用多聚甲醛固定后试验DAPI对细胞核进行染色,最后在激光共聚焦显微镜下进行观察。如图所示,在共转染Beclin1-RedN1和NS5A-GFPC1的细胞(黄色斑点)中,NS5A与细胞Beclin1有明显的共定位(图 6)。
|
用Beclin1-RedN1和NS5A-GFPC1共转染ST细胞,同时设置空白载体RedN1与GFPC1作为对照 ST cells were co-transfected with Beclin1-RedN1 and NS5A-GFPC1, while the blank vector RedN1 and GFPC1 were set as controls 图 6 NS5A与Beclin1蛋白细胞共定位观察 Fig. 6 Observation of co-localization of NS5A and Beclin1 protein in cells |
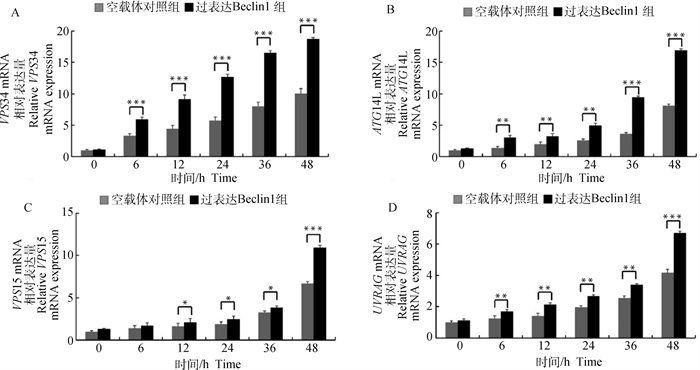
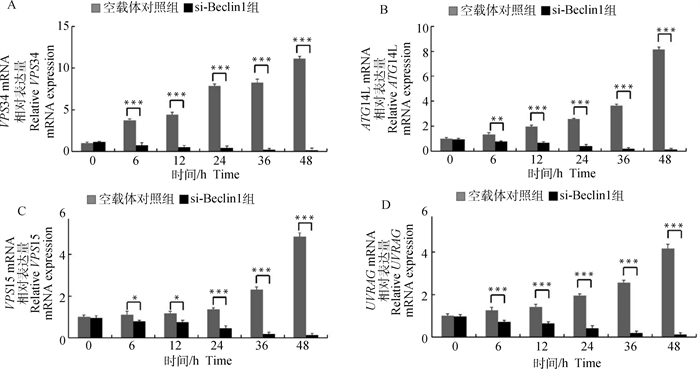
收取感染CSFV不同时间点ST细胞样品,利用qRT-PCR方法检测PI3K/Akt通路相关因子VPS34、VPS15、ATG14L与UVRAG等基因表达情况。结果如图 7所示,PI3K/Akt通路相关因子都呈现出不同程度的升高趋势,其中VPS34表达变化水平最高。当利用siRNA敲低Beclin1表达水平后,感染CSFV,收取不同时间点细胞样品,利用qRT-PCR检测PI3K/Akt通路相关因子VPS34、VPS15、ATG14L与UVRAG等基因表达情况。结果如图 8所示,VPS34、VPS15、ATG14L与UVRAG基因上调表达受到明显抑制。
|
A. qRT-PCR检测VPS34表达;B. qRT-PCR检测ATG14L表达;C. qRT-PCR检测VPS15表达;D. qRT-PCR检测UVRAG表达。*.P<0.05;**.P<0.01;***.P<0.001 A. The expression of VPS34 was detected by qRT-PCR; B. The expression of ATG14L was detected by qRT-PCR; C. The expression of VPS15 was detected by qRT-PCR; D. The expression of UVRAG was detected by qRT-PCR. *.P < 0.05;**.P < 0.01;***.P < 0.001 图 7 qRT-PCR方法检测过表达Beclin1基因后PI3K/Akt通路相关因子 Fig. 7 qRT-PCR method to detect PI3K/Akt pathway related factors after overexpression of Beclin1 gene |
|
A. qRT-PCR检测VPS34表达;B. qRT-PCR检测ATG14L表达;C. qRT-PCR检测VPS15表达;D. qRT-PCR检测UVRAG表达;**.P<0.01;***.P<0.001 A. The expression of VPS34 was detected by qRT-PCR; B. The expression of ATG14L was detected by qRT-PCR; C. The expression of VPS15 was detected by qRT-PCR; D. The expression of UVRAG was detected by qRT-PCR; **.P < 0.01; ***.P < 0.001 图 8 qRT-PCR方法检测敲低Beclin1基因后PI3K/Akt通路相关因子 Fig. 8 qRT-PCR method to detect PI3K/Akt pathway related factors after knockdown of Beclin1 gene |
CSFV感染可以诱导宿主细胞发生完全自噬,自噬体的激活有助于病毒的复制及其病毒颗粒的成熟[14, 27]。CSFV通过调控多种信号通路来完成感染与复制。PI3K/Akt信号通路在细胞的众多病理生理过程中扮演重要角色[28]。当前研究证实,与CSFV同属于黄病毒科的丙型肝炎病毒(HCV)可通过活化PI3K/Akt通路来影响其自身复制[29]。研究发现,CSFV感染可通过活化PI3K/Akt通路来促进CSFV复制[30]。CSFV NS5A蛋白定位于宿主细胞的内质网,对CSFV的生长和病毒RNA的合成具有重要意义[4]。LC3作为主要的自噬标记物常用于评估自噬溶酶体的形成,其作为PI3K/Akt通路上的自噬相关蛋白,与CSFV NS5A蛋白存在相互作用并可诱导完全自噬[15]。本文所研究的Beclin1蛋白,在PI3K激酶ClassⅢ(Class Ⅲ phosphatidylinositol 3-Kinase)复合物中扮演核心角色,该复合物由Vps34、Vps15、Beclin1三个主要亚基组成,作为自噬起始的复合物,在自噬小体的形成中不可或缺[22]。Twu等[31]研究证实,Beclin1敲低细胞系中,HCV和SARS-CoV-2的复制减少。PI3K复合物中的ATG14L的敲低/敲除可抑制HCV复制[32]。另一研究表明,Beclin1作为VPS34活性调控的关键因子,两者的降低均减少HCV复制[33]。有研究表明,HCV NS5A通过活化蛋白C激酶1(RACK1)受体与Ⅲ类PI3K复合物相互作用[26]。
本研究证实,ST细胞感染CSFV或转染表达NS5A蛋白后,Beclin1在转录及蛋白水平上表达均显著上升。通过免疫共沉淀及GST-pulldown等试验证实Beclin1与NS5A存在相互作用。当过表达Beclin1时,CSFV的复制水平显著增强;作为另一佐证,ST细胞干扰Beclin1表达后CSFV复制量显著减少。
此外,Funderburk等[34]研究证实Beclin1及其相关蛋白VPS34、VPS15、UVRAG结合, 诱导自噬小体的形成。UVRAG的表达与VPS34酶活性的增加相关,提示Beclin1-UVRAG-VPS34-VPS15复合物在自噬调节中发挥了作用。与此同时,细胞内Atg14L的存在主要依赖于Beclin1蛋白,Beclin1的缺失会大大降低Atg14L蛋白水平,这对自噬小体的形成至关重要[34]。本研究采用RT-PCR初步证实Beclin1的过表达及敲低影响VPS34、VPS15、ATG14L及UVRAG等级联因子的表达,结果呈正相关。VPS34基因的mRNA表达在感染后6 h呈现明显的持续上升趋势,差异极其显著(P<0.001)(图 7);相反,当ST细胞转染si-Beclin1后感染CSFV,VPS34基因的mRNA表达水平呈现下降趋势(图 8)。VPS15作为自噬早期复合体的关键调控因子的另一基因,当ST细胞过表达Beclin1后感染CSFV,其mRNA表达水平相较于转染空载体接毒组上调较为缓慢;VPS15基因的mRNA表达在感染后12 h呈现明显的持续上升趋势,差异显著(P<0.05);相反,当ST细胞转染si-Beclin1后感染CSFV,VPS15基因的mRNA表达水平呈现轻度下降趋势。ATG14L作为另一自噬早期复合体结合蛋白,当ST细胞过表达Beclin1后感染CSFV,其mRNA表达水平相较于转染空载体接毒组上调显著;ATG14L基因的mRNA表达在感染后6 h呈现较明显的持续上升趋势(图 7)。相反,当ST细胞转染si-Beclin1后感染CSFV,ATG14L基因的mRNA表达水平下降趋势较明显(图 8)。UVRAG与Beclin-1/PI3KC3复合体相关,其能促进PI3KC3酶促活性和自噬。当ST细胞过表达Beclin1后感染CSFV,其mRNA表达水平相较于转染空载体接毒组上调趋势并不显著;UVRAG基因的mRNA表达在感染后24 h呈现较明显的持续上升趋势,差异显著(P<0.05)(图 7)。相反,当ST细胞转染si-Beclin1后感染CSFV,UVRAG基因的mRNA表达水平下降趋势显著(图 8)。上述结果证实,Beclin1等自噬相关因子通过形成PI3K自噬复合体促进猪瘟病毒复制。
综上所述,本研究证实Beclin1蛋白可通过活化PI3K/Akt信号通路并与CSFV NS5A蛋白互作来促进CSFV复制,为猪瘟的有效防控提供理论依据。
4 结论本研究证实了ST细胞感染CSFV或转染表达NS5A蛋白后,均可上调细胞Beclin1及PI3K/Akt通路相关因子表达水平。此外,作者发现Beclin1蛋白对CSFV复制起到明显促进作用;且Beclin1蛋白能够与CSFV NS5A发生相互作用。提示作为自噬体的重要起始蛋白Beclin1蛋白,通过调控PI3K/Akt通路中的相关蛋白促进病毒的增殖。
| [1] |
尹春博. 简述猪瘟研究进展[J]. 中国畜牧兽医文摘, 2016, 32(10): 129, 126. YIN C B. A brief description of the research progress of swine fever[J]. Chinese Animal Husbandry and Veterinary Digest, 2016, 32(10): 129. (in Chinese) |
| [2] |
BECHER P, RAMIREZ R A, ORLICH M, et al. Genetic and antigenic characterization of novel pestivirus genotypes: implications for classification[J]. Virology, 2003, 311(1): 96-104. DOI:10.1016/S0042-6822(03)00192-2 |
| [3] |
LINDENBACH B D, THIEL H J, RICE C M. Flaviviridae: the viruses and their replication[M]//KNIPE D M, HOWLEY P M. Fields Virology. 5th ed. Philadelphia: Lippincott Williams & Wilkins, 2007: 1101-1152.
|
| [4] |
DONG W, LV H F, WANG Y F, et al. The effect of classical swine fever virus NS5A and NS5A mutants on oxidative stress and inflammatory response in swine testicular cells[J]. Res Vet Sci, 2017, 112: 89-96. DOI:10.1016/j.rvsc.2017.01.007 |
| [5] |
DONG X Y, TANG S Q. Classical swine fever virus NS5A protein changed inflammatory cytokine secretion in porcine alveolar macrophages by inhibiting the NF-κB signaling pathway[J]. Virol J, 2016, 13: 101. DOI:10.1186/s12985-016-0545-z |
| [6] |
LI L F, YU J H, LI Y F, et al. Guanylate-binding protein 1, an interferon-induced GTPase, exerts an antiviral activity against classical swine fever virus depending on its GTPase activity[J]. J Virol, 2016, 90(9): 4412-4426. DOI:10.1128/JVI.02718-15 |
| [7] |
LIN J H, WANG C B, LIANG W L, et al. Rab1A is required for assembly of classical swine fever virus particle[J]. Virology, 2018, 514: 18-29. DOI:10.1016/j.virol.2017.11.002 |
| [8] |
ZHANG L, ZHAO D, JIN M X, et al. Rab18 binds to classical swine fever virus NS5A and mediates viral replication and assembly in swine umbilical vein endothelial cells[J]. Virulence, 2020, 11(1): 489-501. DOI:10.1080/21505594.2020.1767356 |
| [9] |
LING S F, LUO M Y, JIANG S N, et al. Cellular Hsp27 interacts with classical swine fever virus NS5A protein and negatively regulates viral replication by the NF-κB signaling pathway[J]. Virology, 2018, 518: 202-209. DOI:10.1016/j.virol.2018.02.020 |
| [10] |
ZHANG C C, KANG K, NING P B, et al. Heat shock protein 70 is associated with CSFV NS5A protein and enhances viral RNA replication[J]. Virology, 2015, 482: 9-18. DOI:10.1016/j.virol.2015.02.014 |
| [11] |
XIE B M, ZHAO M Q, SONG D, et al. Induction of autophagy and suppression of type I IFN secretion by CSFV[J]. Autophagy, 2021, 17(4): 925-947. DOI:10.1080/15548627.2020.1739445 |
| [12] |
ZHANG C C, ZHAO F X, GUO M J, et al. CSFV protein NS5A activates the unfolded protein response to promote viral replication[J]. Virology, 2020, 541: 75-84. DOI:10.1016/j.virol.2019.12.006 |
| [13] |
ZHANG L, JIN M X, SONG M Z, et al. ARFGAP1 binds to classical swine fever virus NS5A protein and enhances CSFV replication in PK-15 cells[J]. Vet Microbiol, 2021, 255: 109034. DOI:10.1016/j.vetmic.2021.109034 |
| [14] |
PEI J J, ZHAO M Q, YE Z D, et al. Autophagy enhances the replication of classical swine fever virus in vitro[J]. Autophagy, 2014, 10(1): 93-110. DOI:10.4161/auto.26843 |
| [15] |
ZHANG C C, WANG X L, SUN J H, et al. Autophagy induced by the N-terminus of the classic swine fever virus nonstructural protein 5A protein promotes viral replication[J]. Front Microbiol, 2021, 12: 733385. DOI:10.3389/fmicb.2021.733385 |
| [16] |
SHANG Y Y, YAO M, ZHOU Z W, et al. Alisertib promotes apoptosis and autophagy in melanoma through p38 MAPK-mediated aurora a signaling[J]. Oncotarget, 2017, 8(63): 107076-107088. DOI:10.18632/oncotarget.22328 |
| [17] |
PARZYCH K R, KLIONSKY D J. An overview of autophagy: morphology, mechanism, and regulation[J]. Antioxid Redox Signal, 2014, 20(3): 460-473. DOI:10.1089/ars.2013.5371 |
| [18] |
HIRANO S, KANNO S. Relevance of autophagy markers to cytotoxicity of zinc compounds in macrophages[J]. Toxicol in Vitro, 2020, 65: 104816. DOI:10.1016/j.tiv.2020.104816 |
| [19] |
LIN L T, DAWSON P W H, RICHARDSON C D. Viral interactions with macroautophagy: a double-edged sword[J]. Virology, 2010, 402(1): 1-10. DOI:10.1016/j.virol.2010.03.026 |
| [20] |
LEVINE B, KLIONSKY D J. Development by self-digestion: molecular mechanisms and biological functions of autophagy[J]. Dev Cell, 2004, 6(4): 463-477. DOI:10.1016/S1534-5807(04)00099-1 |
| [21] |
LIANG X H, JACKSON S, SEAMAN M, et al. Induction of autophagy and inhibition of tumorigenesis by beclin 1[J]. Nature, 1999, 402(6762): 672-676. DOI:10.1038/45257 |
| [22] |
XIE Z P, KLIONSKY D J. Autophagosome formation: core machinery and adaptations[J]. Nat Cell Biol, 2007, 9(10): 1102-1109. DOI:10.1038/ncb1007-1102 |
| [23] |
宋嘉宁, MUHAMMAD T, 张文婷, 等. 自噬相关蛋白激酶的研究进展[J]. 河北师范大学学报(自然科学版), 2021, 45(6): 612-619. SONG J N, MUHAMMAD T, ZHANG W T, et al. Research progress of autophagy-associated protein kinases[J]. Journal of Hebei Normal University (Natural Science), 2021, 45(6): 612-619. (in Chinese) |
| [24] |
EHSAN N A, MOSBEH A M, ELKHADRY S W, et al. Altered protein and gene expression of beclin-1 correlates with poor prognosis of Hcv-associated hepatocellular carcinoma in Egyptian patients[J]. Asian Pac J Cancer Prev, 2021, 22(4): 1115-1122. DOI:10.31557/APJCP.2021.22.4.1115 |
| [25] |
FU N, DU H J, LI D D, et al. Clusterin contributes to hepatitis C virus-related hepatocellular carcinoma by regulating autophagy[J]. Life Sci, 2020, 256: 117911. DOI:10.1016/j.lfs.2020.117911 |
| [26] |
LEE J S, TABATA K, TWU W I, et al. RACK1 mediates rewiring of intracellular networks induced by hepatitis C virus infection[J]. PLoS Pathog, 2019, 15(9): e1008021. DOI:10.1371/journal.ppat.1008021 |
| [27] |
康恺, 林鸷, 高海慧, 等. 猪瘟病毒促进细胞自噬并利于病毒增殖[J]. 畜牧兽医学报, 2014, 45(9): 1481-1487. KANG K, LIN Z, GAO H H, et al. Classical swine fever virus promotes cell autophagy which facilitates virus proliferation[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(9): 1481-1487. (in Chinese) |
| [28] |
HOLM C K, RAHBEK S H, GAD H H, et al. Influenza A virus targets a cGAS-independent STING pathway that controls enveloped RNA viruses[J]. Nat Commun, 2016, 7: 10680. DOI:10.1038/ncomms10680 |
| [29] |
LI D, LEI C Q, XU Z S, et al. Foot-and-mouth disease virus non-structural protein 3A inhibits the interferon-β signaling pathway[J]. Sci Rep, 2016, 6: 21888. DOI:10.1038/srep21888 |
| [30] |
董小英, 彭国良, 许崇波, 等. miR-21通过PI3K/Akt通路抑制猪瘟病毒复制的机制的研究[J]. 中国预防兽医学报, 2016, 38(8): 595-599. DONG X Y, PENG G L, XU C B, et al. miR-21 inhibition of classical swine fever virus replication through the PI3K/Akt signaling pathway[J]. Chinese Journal of Preventive Veterinary Medicine, 2016, 38(8): 595-599. (in Chinese) |
| [31] |
TWU W I, LEE J Y, KIM H, et al. Contribution of autophagy machinery factors to HCV and SARS-CoV-2 replication organelle formation[J]. Cell Rep, 2021, 37(8): 110049. DOI:10.1016/j.celrep.2021.110049 |
| [32] |
DOWDLE W E, NYFELER B, NAGEL J, et al. Selective VPS34 inhibitor blocks autophagy and uncovers a role for NCOA4 in ferritin degradation and iron homeostasis in vivo[J]. Nat Cell Biol, 2014, 16(11): 1069-1079. DOI:10.1038/ncb3053 |
| [33] |
GASSEN N C, PAPIES J, BAJAJ T, et al. SARS-CoV-2-mediated dysregulation of metabolism and autophagy uncovers host-targeting antivirals[J]. Nat Commun, 2021, 12(1): 3818. DOI:10.1038/s41467-021-24007-w |
| [34] |
FUNDERBURK S F, WANG Q J, YUE Z Y. The Beclin 1-VPS34 complex-at the crossroads of autophagy and beyond[J]. Trends Cell Biol, 2010, 20(6): 355-362. DOI:10.1016/j.tcb.2010.03.002 |
(编辑 孟培))



