天然免疫是生物体免疫系统的第一道防线,在感知和防御病原微生物感染的过程中发挥着重要作用[1-2]。模式识别受体(pattern recognition receptors,PRRs)是天然免疫系统的重要组成部分,机体凭借其识别病原体、凋亡宿主细胞和受损衰老细胞表面特定分子结构[3]。根据蛋白质结构域的同源性,目前发现的PRRs可以分为以下五种:Toll样受体(toll-like receptors,TLRs)、维甲酸诱导基因Ⅰ型受体家族(retinoic acid-inducible gene I-like receptors,RLRs)、核酸结合寡聚结构域样受体(nucleotide-binding oligomerization domain-like receptors,NLRs)、C-型凝集素受体(C-type lectin receptors,CLRs)和黑色素瘤缺乏因子2样受体(absent in melanoma 2-like receptors,ALRs)[4-6]。其中细胞质内表达的NLRs在被激活后会组装成为一种叫做炎性小体的多聚蛋白复合体[7]。典型的炎性小体最少由一种NLR家族蛋白、凋亡相关斑点样蛋白(apoptosis-associated speck-like protein containing a CARD,ASC)和半胱氨酸蛋白酶-1(caspase-1)组成。其中NLR蛋白作为感受器蛋白,受到外源刺激后激活,通过同型蛋白结构域的相互作用与接头蛋白ASC结合进行信号传导。随后通过其C端的CASP活化募集结构域(caspase activating and recruitment domain, CARD)招募并活化caspase-1前体。活化的caspase-1将IL-1β前体和IL-18前体加工为其活性形式并且释放到细胞外,从而引起炎症[7]。此外,在经典炎性小体通路中活化的caspase-1可以将gasdermin D(GSDMD)切割成N端和C端,进一步引起细胞焦亡[8-10]。NLRP1是首个被发现的能形成炎性小体的PRRs,但相对于同家族的NLRP3,对于其调控机制以及生物学活性方面的研究一直较少[11]。本文将聚焦NLRP1的激活机制以及其感知病毒感染的研究进行综述。
1 NOD样受体的结构与功能NLRs作为细胞质内的PRRs,广泛存在于脊椎动物和非脊椎动物体内,是一类广泛、复杂的信号调节因子[12-13]。它能够直接识别病原相关分子模式(pathogen-associated molecular patterns,PAMPs)或者损伤相关分子模式(damage-associated molecular patterns,DAMPs),与之结合并通过调控一系列信号级联反应参与机体的天然免疫过程,对激活宿主的防御反应十分重要[14]。迄今为止,在人类中已发现23种NLRs蛋白,而在小鼠体内至少有34种NLR旁系同源物[15]。NLRs家族蛋白的结构高度保守,包括一个中心核苷酸结合寡聚结构域(central nucleotide-binding oligomerization domain,NACHT) 和一个决定底物特异性的C-末端富含亮氨酸的重复序列(leucine-rich repeat,LRR)[16]。NACHT结构域拥有着dNTP酶的活性,并介导着ATP依赖的自身寡聚化[15]。LRR结构域主要起到识别并结合PAMPs和DAMPs的作用,但大多数的NLR蛋白尚未通过试验证明这一点[17]。此外,从NLRC4的晶体结构中发现LRR结构域能够使NLR蛋白维持在一种自抑制的状态[18]。除了NACHT和LRR两个结构域外,NLRs还拥有着不同的N末端效应结构域,包括酸性反式激活结构域(acidic transactivation domain, AD)、杆状病毒抑制剂重复结构域(baculovirus inhibitor of apoptosis protein repeat domain, BIR)、CARD结构域和pyrin结构域(pyrin domain, PYD),它们主要与各种适配器分子和下游信号结合,介导信号转导。
基于不同的N末端效应结构域,NLRs被分为5个亚家族:NLRA、NLRB、NLRC、NLRP和NLRX[19]。NLRA亚家族的N端为AD结构域,目前仅包含一个成员,即Ⅱ类主要组织相容性复合物反式激活因子(class Ⅱ major histocompatibility complex transactivator, CⅡTA)。CⅡTA是NLRs中唯一一个转录调控因子,主要诱导Ⅰ类和Ⅱ类主要组织相容性复合体的表达,其激活受到一系列翻译后修饰的动态调控,如乙酰化、磷酸化和泛素化等[15, 20-21]。NLRB亚家族的N端为BIR结构域,目前在人类体内的成员也仅有一个,即神经元细胞凋亡抑制蛋白(neuronal apoptosis inhibitory protein,NAIP)。NAIP可以通过激活丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)通路来抑制CASP3、CASP7和CASP9的活性,从而起到阻止细胞凋亡的作用[22-25]。NLRC亚家族的N端为CARD结构域,包括5位成员,分别是NOD1、NOD2、NLRC3、NLRC4和NLRC5。其中NOD1和NOD2是NLRC亚家族的主要代表成员,分别具有一个(NOD1)或两个(NOD2)N端CARD结构域,能够识别细胞内的细菌肽聚糖基序[26]。它们能与早期核内体结合并寡聚化,从而导致下游的NF-κB和MAPK信号通路的激活[27]。NLRP是NLRs中最大的亚家族,其N端为PYD结构域,能够招募ASC作为炎性小体激活的支架蛋白[15]。该亚家族目前包含14种蛋白,其中部分NLRP成员能够感知来自细菌、病毒和真菌的PAMPs,形成炎性小体并进一步激活caspase-1引起炎症或细胞焦亡[27-28]。NLRX亚家族在NLRs中较为特殊,与其他NLR蛋白相比,其N端包含一个不同的结构域。NLRX1是NLRX家族的唯一成员,其N端为一个线粒体靶向序列,允许其靶向线粒体基质,并参与线粒体活性氧(reactive oxygen species,ROS)的形成[29]。此外,NLRX1参与对炎症反应和Ⅰ型干扰素信号通路的调节,在抗病毒免疫反应中发挥重要作用。如在丙肝病毒感染中负调节线粒体抗病毒信号蛋白(mitochondrial antiviral signaling protein,MAVS)并且通过抑制TRAF6蛋白与NF-κB激酶抑制剂(inhibitor of NF-κB kinase,IKK)的结合来减弱NF-κB信号通路[30]。
2 NLRP1的结构NLRP1是NLRP亚家族的一员,与其他NLRP蛋白相比,NLRP1拥有一些独特的结构域。以人类NLRP1(hNLRP1)为例,hNLRP1位于LRR之后的C端包含了一个被称为Function-to-Find domain(FIIND)的结构域和一个CARD结构域[31](图 1)。这些结构域使hNLRP1成为相对分子质量最大的NLR家族成员,包含1 473个氨基酸,相对分子质量为165.9 ku[32]。
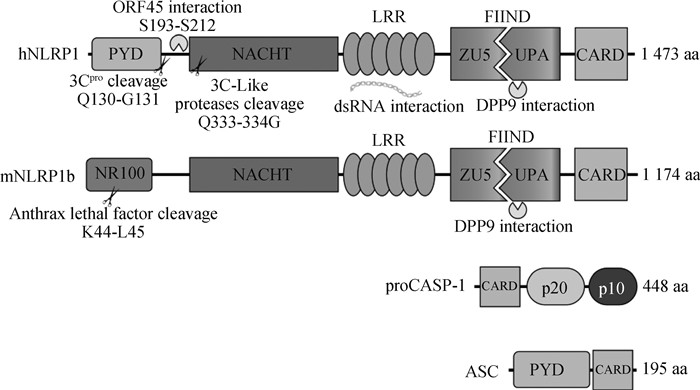
|
图 1 NLRP1蛋白的结构域(扫描文章首页OSID码可查看彩图) Fig. 1 The structural domains of NLRP1 protein (The color picture can be found by scanning the OSID code on the front page of the article) |
虽然hNLRP1同时拥有一个CARD和一个PYD,但已有报道称NLRP1的PYD是自我抑制的,并且研究表明hNLRP1的激活只与C端CARD有关,在ASC不存在的情况下hNLRP1不能诱导pro-caspase-1的活化[33-35]。拥有FIIND结构域是NLRP1的另一个特征。FIIND由ZU5和UPA两个亚结构域组成,并且在翻译后会进行“自裂解”,从而形成两个非共价相连的多肽链[35-36]。hNLRP1的FIIND结构域在F1212和S1213之间进行“自裂解”,并且位于1 186位的组氨酸是该自裂解过程所必须的保守氨基酸,这表明FIIND结构域具有蛋白酶的功能[32, 35, 37]。FIIND结构域在NLRP1与ASC的相互作用中发挥重要作用,并对NLRP1炎性小体的形成至关重要。与其他NLR家族成员类似,NLRP1的LRR结构域具有抑制功能,阻止炎性小体的自激活,直到发生模式识别为止,而NACHT结构域是促进寡聚化的核苷酸结合结构域[38]。
编码NLRP1的基因存在于大多数哺乳动物中,但NLRP1蛋白结构在不同物种中具有多样性。小鼠和人类的NLRP1基因和蛋白之间存在显著的结构差异。在人体内,NLRP1基因位于17号染色体且仅有一个,尽管目前已发现多个剪切变异体;而在小鼠体内NLRP1位于11号染色体上且具有高度多态性,其中以同源基因NLRP1a、NLRP1b和NLRP1c为研究对象的报道最为常见[39]。不同于hNLRP1,这些小鼠同源基因所编码的NLRP1蛋白缺乏N端的PYD结构域,取而代之的是一个NR100结构域(图 1)。有研究报道,炭疽杆菌分泌的关键毒力因子炭疽致死因子(anthrax lethal factor,LF)能在该结构域切割NLRP1b,并引起NLRP1b炎性小体的激活[40]。即使hNLRP1蛋白在不同物种间具有多样性,但由于目前仍缺乏一种能够完全复现hNLRP1蛋白的动物模型,因此对其他动物NLRP1蛋白的研究对奠定NLRP1的生物学基础仍至关重要。
3 NLRP1的激活机制 3.1 通过蛋白酶体途径降解并激活NLRP1炭疽杆菌产生的重要致病因子炭疽致死毒素由两部分组成:可作为炭疽疫苗的保护性抗原蛋白和锌依赖的金属蛋白酶致死因子LF。早在20世纪90年代,LF就被发现可以引起小鼠巨噬细胞的死亡,并且N端规则蛋白酶体途径的抑制剂可以阻断由LF诱导的巨噬细胞死亡[41-42]。2006年,Boyden等[43]成功定位NLRP1基因区域为LF诱导的巨噬细胞死亡的原因,并且在NLRP1b的5个高度多态性的等位基因中,只有NLRP1b1和NLRP1b5对LF刺激有反应。LF可以直接切割mNLRP1特有的NR100结构域的N端区域,在K44和L45残基之间发生裂解,该位点突变会导致LF无法激活NLRP1[44]。随后Chavarría-Smith等[34]将烟草蚀纹病毒(tobacco etch virus,TEV)蛋白酶位点引入对LF不敏感的mNLRP1A和hNLRP1中,发现TEV蛋白酶成功诱导了它们的裂解和激活。因此即便LF目前仅对mNLRP1b起到激活作用,但N端规则仍可以看做是引起NLRP1激活的普遍机制。然而N端规则蛋白酶体降解引起NLRP1激活的具体分子机制仍然有待发掘。2019年,Chui等[45]通过CRISPR-Cas9技术筛选LF诱导细胞焦亡所需的宿主基因,并发现了与mNLRP1b N端规则降解所关联的特定因子——E3连接酶UBR2。同年,Sandstrom所在的小组[46]提出了一个被称为“功能降解”的模型来解释这种蛋白酶体依赖的炎性小体激活机制,即LF在mNLRP1bN端K44和L45残基之间切割,暴露了一个不稳定的新N端,而该N端被泛素连接酶UBR2识别和泛素化,随后mNLRP1b的N端片段被蛋白酶体降解,但由于FIIND结构域存在“自裂解”阻止了mNLRP1b的C端片段与NLRP1 N端片段的同时降解,因此具有生物活性的C端UPA-CARD片段被释放后进一步引起了炎性小体的激活。
在病原体与宿主的长期斗争中,病原体可能已经进化出多种机制以破坏哺乳动物的NLR蛋白(包括但不限于LF),从而逃避天然免疫系统的检测[47]。而这种被称为“功能降解”的激活机制表明NLRP1 N末端的破坏会导致炎性小体的激活和免疫反应的诱导,因此NLRP1极有可能作为其他哺乳动物NLRP蛋白的“诱饵蛋白”来保护机体[46, 48]。而除了LF,由志贺氏痢疾杆菌编码的效应蛋白IpaH7.8也可以激活mNLRP1b,并且符合“功能降解”模型[46]。但与LF不同的是,IpaH7.8由于本身就是E3泛素连接酶,可以直接泛素化mNLRP1b,所以不依赖于UBR2和N端规则途径。综上所述,是否存在其他病原体效应因子通过“功能降解”模型激活NLRP1(尤其是hNLRP1)仍有待进一步探索和发掘。
3.2 通过抑制DPP9激活NLRP1DPP9是二肽基肽酶(DPPs)家族的成员[16, 49]。二肽基肽酶是一类重要的胞内蛋白酶,能够从多肽的N末端切割X-脯氨酸二肽。2017年,Okondo等[50]发现抑制DPP8/9可以诱导小鼠巨噬细胞的焦亡,而作为DPP9抑制剂的Val-boroPro(VbP,也被命名为Talabostat或PT-100)和1G244均能够激活NLRP1炎性小体[51-52]。在当时,通过抑制DPP9激活NLRP1炎性小体的分子机制是令人相当困惑的,因为DPP9没有通过发挥其二肽基肽酶的功能直接切割NLRP1来抑制NLRP1炎性小体的组装,且VbP等抑制剂也没有通过“功能降解”模型激活NLRP1[45, 51, 53]。Zhong等[52]通过免疫沉淀试验和质谱分析发现hNLRP1与DPP9存在相互作用,并且两者互作的区域位于FIIND结构域,而同样拥有该结构域的其他哺乳动物的NLRP1蛋白也被报道能与DPP9发生相互作用[54]。随后的研究则表明,DPP9的结合及其催化活性是抑制NLRP1炎性小体激活所必需的[52]。因此VbP等小分子抑制剂减缓了DPP9对NLRP1的抑制作用,并导致下游炎性小体的激活。
2021年两项背靠背的研究从结构角度研究了NLRP1与DPP9的相互作用,并进一步解释了抑制DPP9激活NLRP1炎性小体的分子机制。Huang等[55]和Hollingsworth等[56]分别报道了hNLRP1-DPP9和mNLRP1-DPP9的冷冻电镜结构。虽然来自不同的哺乳动物,但两者都形成了一种三元复合物:一个具有全长完整结构的NLRP1A,一个仅有UPA-CARD结构的NLRP1B和DPP9。NLRP1A的UPA亚结构域的第一个β链插入到ZU5折叠中,这使N端和C端片段结合并处于自抑制状态。而NLRP1B的UPA-CARD片段通过其N端结合到DPP9的底物结合位点中,因此被DPP9隔离并保持在抑制状态。并且两组研究都表明DPP9除了结合NLRP1A和NLRP1B外,还需要其催化活性来抑制炎性小体的激活。而小分子抑制剂VbP可以直接在DPP9底物结合位点竞争和取代NLRP1B,导致具有生物活性的C端UPA-CARD片段被释放,从而进一步引起了炎性小体的激活。
3.3 弓形虫和部分代谢抑制剂激活NLRP1弓形虫(Toxoplasma gondii)是一种专性的胞内寄生虫,可感染各种温血动物。弓形虫依赖的NLRP1激活在人和啮齿动物中均有报道,并且在大鼠中的研究最为深入[57-59]。Cirelli等[57]发现能抵抗弓形虫感染的大鼠具有对弓形虫敏感的巨噬细胞,这种巨噬细胞在弓形虫感染后被快速诱导焦亡。而后续的NLRP1敲低试验也证明弓形虫诱导的焦亡确实依赖于NLRP1。
弓形虫耐药大鼠具有对弓形虫敏感的巨噬细胞,这表明巨噬细胞焦亡对感染具有保护作用。弓形虫感染引起炎性小体的激活并不是NLRP1特异的,其也被报道同时激活NLRP3炎性小体[60]。有关弓形虫诱导NLRP3激活的分子机制尚未被广泛研究,有研究报道称这可能与弓形虫分泌的ROP7蛋白有关[61]。
在Duncan等[62]的报道中,NLRs家族蛋白的NACHT结构域可以结合ATP,这也表明细胞代谢水平很可能与炎性小体的激活存在一定联系。Liao等[63]发现糖酵解抑制剂2DG(2-deoxyglucose)和电子传递链抑制剂叠氮化钠的共同处理可以在HT1080细胞中特异性激活NLRP1b而不是NLRP3或NLRP6。但是与弓形虫感染引起的NLRP1激活一致,2DG加叠氮化钠的处理组合并没有引起NLRP1直接的N端裂解,这表明它们很可能不是通过“功能降解”模型途径来激活NLRP1的,具体的分子机制尚待进一步研究。
4 病毒感染引起的NLRP1激活机制作为细胞质内的模式识别受体,NLRP1和其他PRR家族成员一样起到识别入侵病原的作用。在检测到病原的入侵后,NLRP1会诱导炎性小体复合物的形成。这些炎性小体复合物可以进一步诱导细胞焦亡,阻滞病原体的复制周期,从而保护宿主。在与外界病原的长期对抗中,NLRP1已进化出了许多感知和检测病原的手段,其中N端具备的不同蛋白酶切割位点可能就是其中之一。如上文提到,LF因为可以特异性识别和切割mNLRP1b的K44-L45位点而成为首个被报道的可以引起NLRP1激活的病原相关因子。无独有偶,2020年,Robinson等[64]发现人鼻病毒(human rhinovirus,HRV)的3C蛋白酶可以特异性切割hNLRP1 N端的Q130-G131。通过试验不同的病毒以及生物信息学的预测,Tsu等[65]进一步确定了NLRP1的Q130-G131是多种小RNA病毒科成员的3C蛋白酶的共同切割位点,如柯萨奇病毒B3型(coxsachievirus B3,CVB3)、人鼻病毒、肠道病毒D68(enterovirus D-68,EV-D68)、脊髓灰质炎病毒1型(poliovirus 1)和罗沙病毒A2型(Rosavirus A2)。NLRP1的3C切割位点正在迅速进化,在不同灵长类动物甚至是人类群体内出现了不同程度的变异[66]。而在近期,冠状病毒拥有的3C样蛋白酶(3C-like protease)也被发现能通过切割NLRP1的N端而激活NLRP1,并且该现象在SARS-CoV-2中也得到证实[67]。与3C蛋白酶不同的是,3C样蛋白酶切割的是Q333位点而非Q130位点。上述几种病毒激活NLRP1的模式非常符合“功能降解”模型,NLRP1被切割而形成的新N端通过蛋白酶体途径被逐步降解,具有生物活性的C端UPA-CARD片段被释放逐步形成炎性小体复合物,进而引起细胞焦亡以保护宿主受到病毒进一步感染。值得注意的是,与NLRP1的C端结构域组成非常相似的人类CARD8炎性小体也被报道可通过病毒蛋白酶的切割而被激活[68-69]。
近期,Yang等[70]报道了一种新的独立于经典蛋白酶体切割途径的NLRP1激活机制。该研究表明,双链DNA病毒卡波济氏肉瘤病毒(Kaposi’s sarcoma herpesvirus,KSHV)的ORF45蛋白可以以非蛋白酶依赖的方式激活hNLRP1,而位于PYD结构域和NACHT结构域之间的Linker1部分是hNLRP1的自抑制和非蛋白酶依赖性激活所必需的。Linker1和UPA亚结构域之间的相互作用抑制了含有DPP9或非DPP9的自抑制复合物中hNLRP1的激活。而ORF45与Linker1的结合取代了UPA,使之从Linker1-UPA复合物中去除,并诱导hNLRP1的C端结构域的释放,最后用于炎性小体组装。ORF45依赖的NLRP1炎性小体的激活在灵长类动物中是保守的,但在mNLRP1b炎性小体中却没有观察到这种现象。
除了感知病毒蛋白酶的切割,NLRP1还被发现可以通过感知病毒双链RNA(dsRNA),进一步被激活从而组装成炎性小体[71]。西门立克森林病毒(Semliki forest virus,SFV)在入侵细胞后生成的dsRNA能够与hNLRP1的LRR结构域直接相互作用,并导致其NACHT结构域获得ATP酶活性,但在mNLRP1b中未观察到此现象。与弓形虫感染以及2DG和叠氮化钠等代谢抑制剂一致,dsRNA引起的NLRP1激活并没有蛋白N端切割降解的过程,但都引起了细胞内代谢水平的失衡,因此要明确dsRNA激活NLRP1的具体机制有待进一步的研究。
5 小结与展望虽然NLRP1是最早发现能组装成炎性小体的蛋白[11],但相对于其他NLRs家族蛋白,对其生物学特性还了解甚少。目前已经发现了多种NLRP1激活因子,包括LF、IpaH7.8、弓形虫感染、代谢抑制剂、VbP以及多种病毒的蛋白等。综合这些因子的激活机制,可以将它们大致分为三类:第一类可以称为“直接激活因子”,其本身直接或间接参与修饰和降解NLRP1的N端片段,比如LF、IpaH7.8、小RNA病毒科成员的3C蛋白、冠状病毒的3C蛋白酶等;第二类则是“间接激活因子”,它们通过干扰细胞内的代谢水平从而引起NLRP1激活,比如弓形虫感染、部分代谢抑制剂和SFV的dsRNA等;第三类激活因子不同于上述两类,可称为“DPP9抑制相关的激活因子”,如VbP、1G244等DPP9抑制剂。通过对NLRP1-DPP9三元复合物的解析,有学者提出了抑制DPP9导致NLRP1-DPP9三元复合物的不稳定进而激活NLRP1的激活模式[55-56]。这种模式的提出为后续对NLRP1激活的研究提供了新的方向。无论是“直接激活因子”或“间接激活因子”,都有可能在整个过程中破坏或占据了NLRP1和DPP9的结合面,这都会促进NLRP1的激活(图 2)。
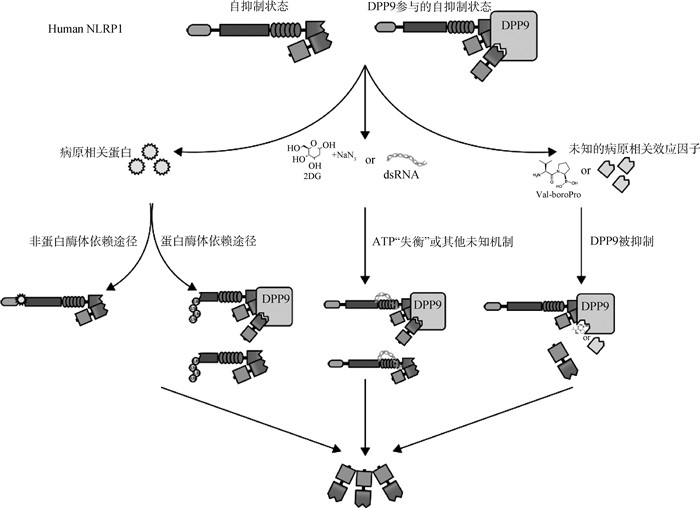
|
图 2 NLRP1炎性小体的激活模式(扫描文章首页OSID码可查看彩图) Fig. 2 The modes of NLRP1 activation (The color picture can be found by scanning the OSID code on the front page of the article) |
炎症反应失调在大多数人类疾病中都有所体现,而NLRP1在参与形成炎性小体后会促进炎症反应,自然与这些疾病的发生密不可分[72-74]。目前已发现NLRP1与人类皮肤病和神经性疾病均有所联系[75-76],虽然专门针对NLRP1炎性小体激活的治疗方法尚未得到广泛开发,但NLRP3抑制剂目前正在被设计用于同样与NLRP1过度激活有关的疾病,如动脉粥样硬化、阿尔茨海默病和类风湿性关节炎[77-78]。而随着对NLRP1调控机制的研究,加深了人们对这一重要的模式识别受体的了解,也有助于理解潜在的疾病治疗以及相关的药物开发。
回顾NLRP1调控机制方面的研究,不难发现NLRP1可以通过感知各种类型的胞内信号,从而激活炎症反应以保护宿主。与那些通过相同配体结合域和相同分子激活模式而适配于不同配体的受体相比,NLRP1感知不同信号的机制更为复杂。正是因为如此,NLRP1激活通路中还存在许多未解之谜,在这个领域中仍有大量的问题值得研究。比如,在弓形虫感染和部分代谢抑制剂作用激活NLRP1通路时并未引起NLRP1本身的降解,虽已有研究报道其与细胞内代谢水平的紊乱有关[62],但具体机制仍不得而知,这很可能是一个目前仍未知的炎性小体激活通路。此外,有报道称NLRP6可作为ISG并在抵抗肠道病毒入侵的过程中发挥重要作用,这提示作为同为NLRs家族一员的NLRP1也可能存在类似的抗病毒作用[79]。进一步探索这些可能性并最终定义NLRP1炎性小体在宿主防御中发挥的作用将是非常有意义的。
| [1] |
THAISS C A, ZMORA N, LEVY M, et al. The microbiome and innate immunity[J]. Nature, 2016, 535(7610): 65-74. DOI:10.1038/nature18847 |
| [2] |
DEMARIA O, CORNEN S, DAËRON M, et al. Harnessing innate immunity in cancer therapy[J]. Nature, 2019, 574(7776): 45-56. DOI:10.1038/s41586-019-1593-5 |
| [3] |
LI D Y, WU M H. Pattern recognition receptors in health and diseases[J]. Sig Transduct Target Ther, 2021, 6(1): 291. DOI:10.1038/s41392-021-00687-0 |
| [4] |
KANNEGANTI T D. Intracellular innate immune receptors: life inside the cell[J]. Immunol Rev, 2020, 297(1): 5-12. DOI:10.1111/imr.12912 |
| [5] |
DIAMOND M S, KANNEGANTI T D. Innate immunity: the first line of defense against SARS-CoV-2[J]. Nat Immunol, 2022, 23(2): 165-176. DOI:10.1038/s41590-021-01091-0 |
| [6] |
FITZGERALD K A, KAGAN J C. Toll-like receptors and the control of immunity[J]. Cell, 2020, 180(6): 1044-1066. DOI:10.1016/j.cell.2020.02.041 |
| [7] |
BROZ P, DIXIT V M. Inflammasomes: mechanism of assembly, regulation and signalling[J]. Nat Rev Immunol, 2016, 16(7): 407-420. DOI:10.1038/nri.2016.58 |
| [8] |
SHI J J, ZHAO Y, WANG K, et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death[J]. Nature, 2015, 526(7575): 660-665. DOI:10.1038/nature15514 |
| [9] |
KAYAGAKI N, STOWE I B, LEE B L, et al. Caspase-11 cleaves gasdermin D for non-canonical inflammasome signalling[J]. Nature, 2015, 526(7575): 666-671. DOI:10.1038/nature15541 |
| [10] |
BURDETTE B E, ESPARZA A N, ZHU H, et al. Gasdermin D in pyroptosis[J]. Acta Pharm Sin B, 2021, 11(9): 2768-2782. DOI:10.1016/j.apsb.2021.02.006 |
| [11] |
MARTINON F, BURNS K, TSCHOPP J. The inflammasome: a molecular platform triggering activation of inflammatory caspases and processing of proIL-β[J]. Mol Cell, 2002, 10(2): 417-426. DOI:10.1016/S1097-2765(02)00599-3 |
| [12] |
SHAW M H, REIMER T, KIM Y G, et al. NOD-like receptors (NLRs): bona fide intracellular microbial sensors[J]. Curr Opin Immunol, 2008, 20(4): 377-382. DOI:10.1016/j.coi.2008.06.001 |
| [13] |
TING J P Y, LOVERING R C, ALNEMRI E S, et al. The NLR gene family: a standard nomenclature[J]. Immunity, 2008, 28(3): 285-287. DOI:10.1016/j.immuni.2008.02.005 |
| [14] |
LUPFER C, KANNEGANTI T D. The expanding role of NLRs in antiviral immunity[J]. Immunol Rev, 2013, 255(1): 13-24. DOI:10.1111/imr.12089 |
| [15] |
DANIS J, MELLETT M. Nod-like receptors in host defence and disease at the epidermal barrier[J]. Int J Mol Sci, 2021, 22(9): 4677. DOI:10.3390/ijms22094677 |
| [16] |
QIAN X K, ZHANG J, LI X D, et al. Research progress on dipeptidyl peptidase family: structure, function and xenobiotic metabolism[J]. Curr Med Chem, 2022, 29(12): 2167-2188. DOI:10.2174/0929867328666210915103431 |
| [17] |
BENTHAM A, BURDETT H, ANDERSON P A, et al. Animal NLRs provide structural insights into plant NLR function[J]. Ann Bot, 2017, 119(5): 698-702. |
| [18] |
HU Z H, YAN C Y, LIU P Y, et al. Crystal structure of NLRC4 reveals its autoinhibition mechanism[J]. Science, 2013, 341(6142): 172-175. DOI:10.1126/science.1236381 |
| [19] |
MOTTA V, SOARES F, SUN T, et al. NOD-like receptors: versatile cytosolic sentinels[J]. Physiol Rev, 2015, 95(1): 149-178. DOI:10.1152/physrev.00009.2014 |
| [20] |
LIU P, LU Z W, LIU L L, et al. NOD-like receptor signaling in inflammation-associated cancers: from functions to targeted therapies[J]. Phytomedicine, 2019, 64: 152925. DOI:10.1016/j.phymed.2019.152925 |
| [21] |
MASTERNAK K, MUHLETHALER-MOTTET A, VILLARD J, et al. CⅡTA is a transcriptional coactivator that is recruited to MHC class Ⅱ promoters by multiple synergistic interactions with an enhanceosome complex[J]. Genes Dev, 2000, 14(9): 1156-1166. DOI:10.1101/gad.14.9.1156 |
| [22] |
MAIER J K X, LAHOUA Z, GENDRON N H, et al. The neuronal apoptosis inhibitory protein is a direct inhibitor of caspases 3 and 7[J]. J Neurosci, 2002, 22(6): 2035-2043. DOI:10.1523/JNEUROSCI.22-06-02035.2002 |
| [23] |
DAVOODI J, GHAHREMANI M H, ES-HAGHI A, et al. Neuronal apoptosis inhibitory protein, NAIP, is an inhibitor of procaspase-9[J]. Int J Biochem Cell Biol, 2010, 42(6): 958-964. DOI:10.1016/j.biocel.2010.02.008 |
| [24] |
SANNA M G, DA SILVA CORREIA J, DUCREY O, et al. IAP suppression of apoptosis involves distinct mechanisms: the TAK1/JNK1 signaling cascade and caspase inhibition[J]. Mol Cell Biol, 2002, 22(6): 1754-1766. DOI:10.1128/MCB.22.6.1754-1766.2002 |
| [25] |
HAUSMANN A, BÖCK D, GEISER P, et al. Intestinal epithelial NAIP/NLRC4 restricts systemic dissemination of the adapted pathogen Salmonella typhimurium due to site-specific bacterial PAMP expression[J]. Mucosal Immunol, 2020, 13(3): 530-544. DOI:10.1038/s41385-019-0247-0 |
| [26] |
CORREA R G, MILUTINOVIC S, REED J C. Roles of NOD1 (NLRC1) and NOD2 (NLRC2) in innate immunity and inflammatory diseases[J]. Biosci Rep, 2012, 32(6): 597-608. DOI:10.1042/BSR20120055 |
| [27] |
VELLOSO F J, TROMBETTA-LIMA M, ANSCHAU V, et al. NOD-like receptors: major players (and targets) in the interface between innate immunity and cancer[J]. Biosci Rep, 2019, 39(4): BSR20181709. DOI:10.1042/BSR20181709 |
| [28] |
SCHRODER K, TSCHOPP J. The inflammasomes[J]. Cell, 2010, 140(6): 821-832. DOI:10.1016/j.cell.2010.01.040 |
| [29] |
TATTOLI I, CARNEIRO L A, JéHANNO M, et al. NLRX1 is a mitochondrial NOD-like receptor that amplifies NF-κB and JNK pathways by inducing reactive oxygen species production[J]. EMBO Rep, 2008, 9(3): 293-300. DOI:10.1038/sj.embor.7401161 |
| [30] |
STOKMAN G, KORS L, BAKKER P J, et al. NLRX1 dampens oxidative stress and apoptosis in tissue injury via control of mitochondrial activity[J]. J Exp Med, 2017, 214(8): 2405-2420. DOI:10.1084/jem.20161031 |
| [31] |
TSCHOPP J, MARTINON F, BURNS K. NALPs: a novel protein family involved in inflammation[J]. Nat Rev Mol Cell Biol, 2003, 4(2): 95-104. DOI:10.1038/nrm1019 |
| [32] |
YU C H, MOECKING J, GEYER M, et al. Mechanisms of NLRP1-mediated autoinflammatory disease in humans and mice[J]. J Mol Biol, 2018, 430(2): 142-152. DOI:10.1016/j.jmb.2017.07.012 |
| [33] |
ZHONG F L, MAMAÏ O, SBORGI L, et al. Germline NLRP1 mutations cause skin inflammatory and cancer susceptibility syndromes via inflammasome activation[J]. Cell, 2016, 167(1): 187-202.e17. DOI:10.1016/j.cell.2016.09.001 |
| [34] |
CHAVARRÍA-SMITH J, MITCHELL P S, HO A M, et al. Functional and evolutionary analyses identify proteolysis as a general mechanism for NLRP1 inflammasome activation[J]. PLoS Pathog, 2016, 12(12): e1006052. DOI:10.1371/journal.ppat.1006052 |
| [35] |
FINGER J N, LICH J D, DARE L C, et al. Autolytic proteolysis within the function to find domain (FⅡND) is required for NLRP1 inflammasome activity[J]. J Biol Chem, 2012, 287(30): 25030-25037. DOI:10.1074/jbc.M112.378323 |
| [36] |
XUE Y S, ENOSI TUIPULOTU D, TAN W H, et al. Emerging activators and regulators of inflammasomes and pyroptosis[J]. Trends Immunol, 2019, 40(11): 1035-1052. DOI:10.1016/j.it.2019.09.005 |
| [37] |
D'OSUALDO A, WEICHENBERGER C X, WAGNER R N, et al. CARD8 and NLRP1 undergo autoproteolytic processing through a ZU5-like domain[J]. PLoS One, 2011, 6(11): e27396. DOI:10.1371/journal.pone.0027396 |
| [38] |
FAUSTIN B, LARTIGUE L, BRUEY J M, et al. Reconstituted NALP1 inflammasome reveals two-step mechanism of caspase-1 activation[J]. Mol Cell, 2007, 25(5): 713-724. DOI:10.1016/j.molcel.2007.01.032 |
| [39] |
LILUE J, DORAN A G, FIDDES I T, et al. Sixteen diverse laboratory mouse reference genomes define strain-specific haplotypes and novel functional loci[J]. Nat Genet, 2018, 50(11): 1574-1583. DOI:10.1038/s41588-018-0223-8 |
| [40] |
CHAVARRÍA-SMITH J, VANCE R E. Direct proteolytic cleavage of NLRP1B is necessary and sufficient for inflammasome activation by anthrax lethal factor[J]. PLoS Pathog, 2013, 9(6): e1003452. DOI:10.1371/journal.ppat.1003452 |
| [41] |
ROBERTS J E, WATTERS J W, BALLARD J D, et al. Ltx1, a mouse locus that influences the susceptibility of macrophages to cytolysis caused by intoxication with Bacillus anthracis lethal factor, maps to chromosome 11[J]. Mol Microbiol, 1998, 29(2): 581-591. DOI:10.1046/j.1365-2958.1998.00953.x |
| [42] |
WICKLIFFE K E, LEPPLA S H, MOAYERI M. Killing of macrophages by anthrax lethal toxin: involvement of the N-end rule pathway[J]. Cell Microbiol, 2008, 10(6): 1352-1362. DOI:10.1111/j.1462-5822.2008.01131.x |
| [43] |
BOYDEN E D, DIETRICH W F. Nalp1b controls mouse macrophage susceptibility to anthrax lethal toxin[J]. Nat Genet, 2006, 38(2): 240-244. DOI:10.1038/ng1724 |
| [44] |
LEVINSOHN J L, NEWMAN Z L, HELLMICH K A, et al. Anthrax lethal factor cleavage of Nlrp1 is required for activation of the inflammasome[J]. PLoS Pathog, 2012, 8(3): e1002638. DOI:10.1371/journal.ppat.1002638 |
| [45] |
CHUI A J, OKONDO M C, RAO S D, et al. N-terminal degradation activates the NLRP1B inflammasome[J]. Science, 2019, 364(6435): 82-85. DOI:10.1126/science.aau1208 |
| [46] |
SANDSTROM A, MITCHELL P S, GOERS L, et al. Functional degradation: a mechanism of NLRP1 inflammasome activation by diverse pathogen enzymes[J]. Science, 2019, 364(6435): eaau1330. DOI:10.1126/science.aau1330 |
| [47] |
TAABAZUING C Y, GRISWOLD A R, BACHOVCHIN D A. The NLRP1 and CARD8 inflammasomes[J]. Immunol Rev, 2020, 297(1): 13-25. DOI:10.1111/imr.12884 |
| [48] |
MITCHELL P S, SANDSTROM A, VANCE R E. The NLRP1 inflammasome: new mechanistic insights and unresolved mysteries[J]. Curr Opin Immunol, 2019, 60: 37-45. DOI:10.1016/j.coi.2019.04.015 |
| [49] |
WILSON C H, ZHANG H E, GORRELL M D, et al. Dipeptidyl peptidase 9 substrates and their discovery: current progress and the application of mass spectrometry-based approaches[J]. Biol Chem, 2016, 397(9): 837-856. DOI:10.1515/hsz-2016-0174 |
| [50] |
OKONDO M C, JOHNSON D C, SRIDHARAN R, et al. DPP8 and DPP9 inhibition induces pro-caspase-1-dependent monocyte and macrophage pyroptosis[J]. Nat Chem Biol, 2017, 13(1): 46-53. DOI:10.1038/nchembio.2229 |
| [51] |
OKONDO M C, RAO S D, TAABAZUING C Y, et al. Inhibition of Dpp8/9 activates the Nlrp1b inflammasome[J]. Cell Chem Biol, 2018, 25(3): 262-267.e5. DOI:10.1016/j.chembiol.2017.12.013 |
| [52] |
ZHONG F L, ROBINSON K, TEO D E T, et al. Human DPP9 represses NLRP1 inflammasome and protects against autoinflammatory diseases via both peptidase activity and FⅡND domain binding[J]. J Biol Chem, 2018, 293(49): 18864-18878. DOI:10.1074/jbc.RA118.004350 |
| [53] |
GRISWOLD A R, CIFANI P, RAO S D, et al. A chemical strategy for protease substrate profiling[J]. Cell Chem Biol, 2019, 26(6): 901-907.e6. DOI:10.1016/j.chembiol.2019.03.007 |
| [54] |
JOHNSON D C, TAABAZUING C Y, OKONDO M C, et al. DPP8/DPP9 inhibitor-induced pyroptosis for treatment of acute myeloid leukemia[J]. Nat Med, 2018, 24(8): 1151-1156. DOI:10.1038/s41591-018-0082-y |
| [55] |
HUANG M H, ZHANG X X, TOH G A, et al. Structural and biochemical mechanisms of NLRP1 inhibition by DPP9[J]. Nature, 2021, 592(7856): 773-777. DOI:10.1038/s41586-021-03320-w |
| [56] |
HOLLINGSWORTH L R, SHARIF H, GRISWOLD A R, et al. DPP9 sequesters the C terminus of NLRP1 to repress inflammasome activation[J]. Nature, 2021, 592(7856): 778-783. DOI:10.1038/s41586-021-03350-4 |
| [57] |
CIRELLI K M, GORFU G, HASSAN M A, et al. Inflammasome sensor NLRP1 controls rat macrophage susceptibility to Toxoplasma gondii[J]. PLoS Pathog, 2014, 10(3): e1003927. DOI:10.1371/journal.ppat.1003927 |
| [58] |
EWALD S E, CHAVARRIA-SMITH J, BOOTHROYD J C. NLRP1 is an inflammasome sensor for Toxoplasma gondii[J]. Infect Immun, 2014, 82(1): 460-468. DOI:10.1128/IAI.01170-13 |
| [59] |
WITOLA W H, MUI E, HARGRAVE A, et al. NALP1 influences susceptibility to human congenital toxoplasmosis, proinflammatory cytokine response, and fate of Toxoplasma gondii-infected monocytic cells[J]. Infect Immun, 2011, 79(2): 756-766. DOI:10.1128/IAI.00898-10 |
| [60] |
GORFU G, CIRELLI K M, MELO M B, et al. Dual role for inflammasome sensors NLRP1 and NLRP3 in murine resistance to Toxoplasma gondii[J]. mBio, 2014, 5(1): e01117-13. |
| [61] |
ZHU L J, QI W J, YANG G, et al. Toxoplasma gondii rhoptry protein 7 (ROP7) interacts with NLRP3 and promotes inflammasome hyperactivation in THP-1-derived macrophages[J]. Cells, 2022, 11(10): 1630. DOI:10.3390/cells11101630 |
| [62] |
DUNCAN J A, BERGSTRALH D T, WANG Y H, et al. Cryopyrin/NALP3 binds ATP/dATP, is an ATPase, and requires ATP binding to mediate inflammatory signaling[J]. Proc Natl Acad Sci U S A, 2007, 104(19): 8041-8046. DOI:10.1073/pnas.0611496104 |
| [63] |
LIAO K C, MOGRIDGE J. Activation of the Nlrp1b inflammasome by reduction of cytosolic ATP[J]. Infect Immun, 2013, 81(2): 570-579. DOI:10.1128/IAI.01003-12 |
| [64] |
ROBINSON K S, TEO D E T, TAN K S, et al. Enteroviral 3C protease activates the human NLRP1 inflammasome in airway epithelia[J]. Science, 2020, 370(6521): eaay2002. DOI:10.1126/science.aay2002 |
| [65] |
TSU B V, BEIERSCHMITT C, RYAN A P, et al. Diverse viral proteases activate the NLRP1 inflammasome[J]. Elife, 2021, 10: e60609. DOI:10.7554/eLife.60609 |
| [66] |
NOZAKI K, LI L P, MIAO E A. Innate sensors trigger regulated cell death to combat intracellular infection[J]. Ann Rev Immunol, 2022, 40: 469-498. DOI:10.1146/annurev-immunol-101320-011235 |
| [67] |
PLANÈS R, PINILLA M, SANTONI K, et al. Human NLRP1 is a sensor of pathogenic coronavirus 3CL proteases in lung epithelial cells[J]. Mol Cell, 82(13): 2385-2400.
|
| [68] |
WANG Q K, GAO H B, CLARK K M, et al. CARD8 is an inflammasome sensor for HIV-1 protease activity[J]. Science, 2021, 371(6535): eabe1707. DOI:10.1126/science.abe1707 |
| [69] |
HOLLINGSWORTH L R, DAVID L, LI Y, et al. Mechanism of filament formation in UPA-promoted CARD8 and NLRP1 inflammasomes[J]. Nat Commun, 2021, 12(1): 189. DOI:10.1038/s41467-020-20320-y |
| [70] |
YANG X, ZHOU J F, LIU C R, et al. KSHV-encoded ORF45 activates human NLRP1 inflammasome[J]. Nat Immunol, 2022, 23(6): 916-926. DOI:10.1038/s41590-022-01199-x |
| [71] |
BAUERNFRIED S, SCHERR M J, PICHLMAIR A, et al. Human NLRP1 is a sensor for double-stranded RNA[J]. Science, 2021, 371(6528): eabd0811. DOI:10.1126/science.abd0811 |
| [72] |
TUPIK J D, NAGAI-SINGER M A, ALLEN I C. To protect or adversely affect?The dichotomous role of the NLRP1 inflammasome in human disease[J]. Mol Aspects Med, 2020, 76: 100858. DOI:10.1016/j.mam.2020.100858 |
| [73] |
ALEHASHEMI S, GOLDBACH-MANSKY R. Human autoinflammatory diseases mediated by NLRP3-, Pyrin-, NLRP1-, and NLRC4-inflammasome dysregulation updates on diagnosis, treatment, and the respective roles of IL-1 and IL-18[J]. Front Immunol, 2020, 11: 1840. DOI:10.3389/fimmu.2020.01840 |
| [74] |
SAND J, FENINI G, GROSSI S, et al. The NLRP1 inflammasome pathway is silenced in cutaneous squamous cell carcinoma[J]. J Invest Dermatol, 2019, 139(8): 1788-1797.e6. DOI:10.1016/j.jid.2019.01.025 |
| [75] |
FENINI G, KARAKAYA T, HENNIG P, et al. The NLRP1 inflammasome in human skin and beyond[J]. Int J Mol Sci, 2020, 21(13): 4788. DOI:10.3390/ijms21134788 |
| [76] |
YAP J K Y, PICKARD B S, CHAN E W L, et al. The role of neuronal NLRP1 inflammasome in Alzheimer's disease: bringing neurons into the neuroinflammation game[J]. Mol Neurobiol, 2019, 56(11): 7741-7753. DOI:10.1007/s12035-019-1638-7 |
| [77] |
MULLARD A. NLRP3 inhibitors stoke anti-inflammatory ambitions[J]. Nat Rev Drug Discov, 2019, 18(6): 405-407. DOI:10.1038/d41573-019-00086-9 |
| [78] |
DE BRITO TOSCANO E C, ROCHA N P, LOPES B N A, et al. Neuroinflammation in Alzheimer's disease: focus on NLRP1 and NLRP3 inflammasomes[J]. Curr Protein Pept Sci, 2021, 22(8): 584-598. DOI:10.2174/1389203722666210916141436 |
| [79] |
WANG P H, ZHU S, YANG L, et al. Nlrp6 regulates intestinal antiviral innate immunity[J]. Science, 2015, 350(6262): 826-830. DOI:10.1126/science.aab3145 |
(编辑 白永平)



