2. 江苏省家禽科学研究所, 江苏省家禽遗传育种重点实验室, 扬州 225125;
3. 江苏省家禽科学研究所科技创新有限公司, 扬州 211412
2. Key Laboratory for Poultry Genetics and Breeding of Jiangsu Province, Jiangsu Institute of Poultry Science, Yangzhou 225125, China;
3. Jiangsu Poultry Science Institute S & T Innovation Co. Ltd., Yangzhou 211412, China
黄羽肉鸡产业是我国畜牧业的支柱产业之一,出栏数约占肉鸡总数的一半,为满足城乡居民肉食品供应和养殖户增收做出了重要的贡献。根据生长速度和出栏日龄,黄羽肉鸡一般分为快速型(49~70 d)、中速型(71~90 d)和慢速型(91~180 d)三类[1]。快速型黄羽肉鸡生产时间短,饲料报酬高,具有最低的生产成本,并且肉质优于白羽肉鸡,因此越来越受到消费者的喜爱。随着肉鸡消费逐步转向冰鲜上市,快速型黄羽肉鸡能够更好地适应屠宰需求,市场份额逐渐增加,其选育提高也越来越受到育种人员的关注。
生长速度和肉品质是影响快速型肉鸡经济效益最重要的两类性状。虽然肉鸡体重在过去几十年中得到了显著提高,但由于瓶颈效应的存在,进一步的提升存在明显困难[2-3]。在实际选育工作中,肉色、剪切力等肉质性状由于遗传力低、测量难度大等原因,常规选择遗传进展明显滞后[4-5]。因此,深入鉴定与生长和肉品质性状相关的分子标记,并基于此开展分子选育,对于加快肉鸡品种选育进展有着重要的意义。
近年来,全基因组关联分析(genome-wide associated study,GWAS)已成为研究人类疾病和畜禽经济性状遗传调控机制的重要手段[6-7]。在肉鸡上,已有许多学者针对重要经济性状开展了相关研究,包括屠宰性能[8]、体重[9-10]、抗病性状等[11-12],并报道了大量的候选SNPs和基因。然而,大部分研究集中于国外品种和部分地方品种,在快速型黄羽肉鸡品种上的研究较为有限。
本研究选择本团队与江苏立华牧业股份有限公司合作培育的立华麻黄鸡终端父系种鸡作为研究对象,该品系具有生长速度快、优质、早熟的特点,60日龄(配套系上市日龄)体重可达到2.5 kg。利用重测序获得393只公鸡的基因组遗传变异信息,通过GWAS方法获得与体重和肉品质性状显著关联的候选基因和位点,为快速型黄羽肉鸡选育提高提供理论基础。
1 材料与方法 1.1 试验动物本研究选择393只立华麻黄终端父系公鸡作为试验材料,饲养于江苏立华牧业股份有限公司金坛种鸡场。试验鸡群均同批饲养于同一鸡舍,采用公司标准的种鸡饲粮进行饲喂,试验期自由饮水和采食。采用常规光照和免疫程序。对所有试验鸡进行翅静脉采血保存备用。
1.2 体重和肉品质测定鸡群饲养至60日龄(上市日龄),禁食12 h后对所有个体进行称重。随后进行屠宰,统一采集左侧胸肌样品进行剪切力、系水力、pH和肉色测定。肉品质测定方法参考《NY/T 1333—2007畜禽肉质的测定》进行。剪切力测定:使用C-LM4型电脑测控式肌肉嫩度仪进行测定,胸肌样品修剪成长条状(厚0.5 cm,宽1.0 cm),测定3次取平均值。系水力测定:使用YYW-2型应变控制式无侧限压力仪对样品进行加压测定,取新鲜胸肌肉样1 g左右(w1)放置于上下压台间,并于样品上下垫8层滤纸,施压35 kg,保持5 min后,去掉压力和滤纸,测定肉样重量(w2),计算系水力(w1-w2)/w1。pH测定:使用胴体肌肉pH测定仪PH-STAR,屠宰后45 min测定胸肌pH,选取3个不同位置取平均值。肉色测定:使用NR10QC-3nh色差仪进行肉色测定,在相同环境条件下,选取3个不同位置进行3次测定,分别记录亮度值(L*)、红度值(a*)、黄度值(b*)。
1.3 全基因组重测序使用TIANGEN基因组试剂盒提取和纯化高质量的基因组DNA,通过凝胶迁移(1%琼脂糖凝胶)测定DNA降解和污染程度,DNA纯度采用Nano Photometer分光光度计测定,DNA浓度通过NanoDrop荧光仪进行定量。将合格样品送至北京博致格雅生物科技有限公司建库,采用MGISEQ-2000平台进行全基因组重测序,测序深度为10×。获得原始测序数据后,使用FASTP(v.0.20.0)[13]进行过滤,去除低质量reads,并对得到的Clean Reads进行质量控制,后用BWA(v0.7.17)[14]软件将质控后得到的reads比对至参考基因组GRcg7b。用Picard软件将重复比对的reads移除。通过GATK4[15]软件分析获得高质量的SNP位点。
1.4 全基因组关联分析使用Plink v1.9[16]软件对所得SNP数据进行质量控制,合格的个体和位点用于GWAS分析。筛选标准为剔除基因频率缺失高于2%的SNP(geno < 0.02),个体基因频率缺失超过4%的个体(mind < 0.04),次等位基因频率小于0.05(MAF < 0.05),哈代温伯格频率小于0.000 1(hwe < 1×10-4),杂合率平均值中偏离3倍标准差的个体(mean±3SD)。利用GEMMA(v0.98.5)[17]软件进行关联分析,模型如下:
| $ y=\boldsymbol{W} \alpha+\boldsymbol{X} \beta+\mu+e \text {; } $ |
其中,y是表型向量;W是协方差矩阵;α是以第一、二、三主成分作为的协变量;X是固定效应矩阵;β是有效SNP位点的数量;μ代表随机效应向量;e代表残差。
使用Plink(v1.90p)软件中的参数(-indep-pairwise 50 10 0.2)来推断独立检验的有效SNP数量,最终推断出有效独立检验的SNPs为432 529个。多重检验后,采用Bonferroni校正法来设定显著阈值,全基因组水平显著阈值和潜在显著阈值分别为1.15×10-7(0.05/432 529)和2.31×10-6(1/432 529)。使用Cmplot程序包对GWAS结果进行可视化。通过Bedtools (v2.30.0)[18]软件和参考基因组GRcg7b对显著位点的上、下游100 kb进行基因注释。
2 结果 2.1 表型描述统计本研究使用的393只立华父系麻黄鸡7种表型的基本统计描述如表 1所示。体重、剪切力、系水力、pH、肉色L值、a值和b值的变异系数分别为8.18%、22.17%、10.45%、5.57%、4.91%、69.57%、22.58%。其中a值变异达到高度水平,剪切力和b值为中等变异性状,表明这些性状具有较大的遗传选择潜力。本群体中体重的变异系数较低,可能由于该品系经过系统的体重选择导致。
|
|
表 1 立华父系麻黄鸡性状表型值统计结果 Table 1 Statistical results of phenotypic values of Lihua paternal ephedra chickens |
本研究获得的原始下机数据共包含16 411 878个SNPs和393个个体,首先剔除SNP缺失率大于2%的位点1 767 209个;2个个体因缺失率大于2%而被剔除;以MAF < 0.05和HWE < 1×10-4为标准,剔除位点106 298个;根据杂合度剔除个体3个。经过质控后,剩余388个个体和10 266 594个SNPs可用于后续的关联分析。结合异常表型值的剔除,最终用于各性状GWAS分析个体数为:体重382个,剪切力361个,系水力354个,pH 360个,L值365个,a值362个,b值364个。
2.3 体重性状全基因组关联分析利用GEMMA软件构建混合线性模型对382个个体的体重性状进行关联分析。如图 1和表 2所示,本研究鉴定到2个与体重显著关联的SNPs位点,分别位于4号和28号染色体上,通过对SNP上、下游基因进行搜索注释,发现了11个基因位于与体重显著关联的SNPs位点上、下游100 kb的区域,包括FSTL3、DOCK11、IL13RA1、STK26等基因。而与体重潜在显著关联的位点有52个,主要分布于4、5、19、28号染色体上,位于NPFF、PFKM、MBNL3等基因附近。
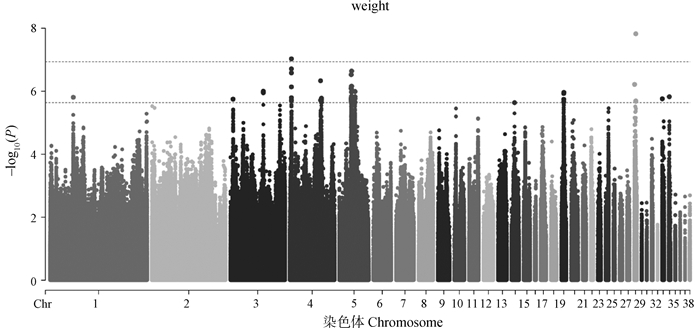
|
图 1 体重性状的全基因组关联分析 Fig. 1 Genome-wide association study of body weight trait |
|
|
表 2 关联SNPs注释结果 Table 2 Associated SNPs annotation results |
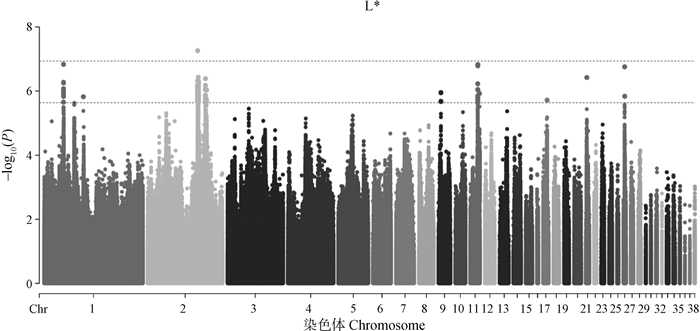
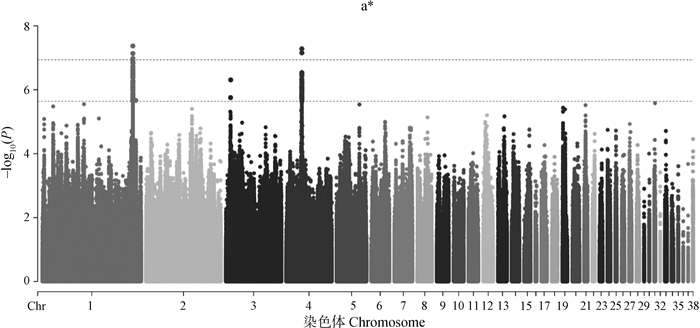
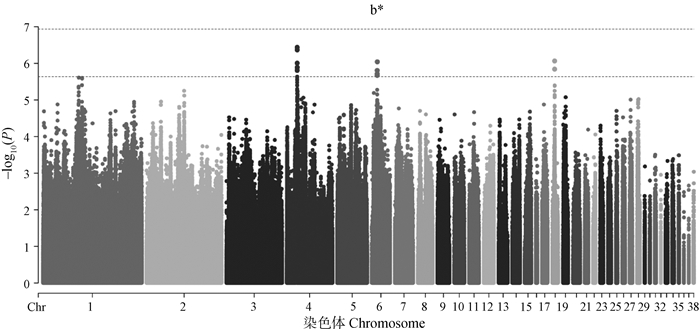
肉品质关联分析结果如图 2-7和表 2所示。结果发现位于4号染色体的1个位点与剪切力性状显著相关;还发现15个位点与剪切力性状存在潜在显著关联,主要分布于4号和19号染色体上,位于SLC13A5等基因附近。与系水力显著相关的位点有1个,位于17号染色体PTGS1基因附近;与系水力存在潜在关联的位点有25个,主要分布在1号、14号和17号染色体,位于MDFIC等基因附近。共发现与pH性状潜在关联的位点有36个,主要分布在2号和6号染色体上,位于FGD3、AQP1等基因附近。位于2号染色体的1个SNP与肉色L值存在显著关联,与a值存在显著关联的位点有5个,主要分布在1号和4号染色体上。与L*、a*、b*值存在潜在关联的位点分别有124个、83个和13个。
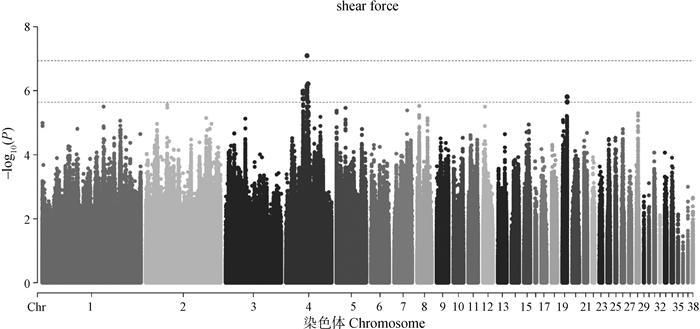
|
图 2 剪切力性状的全基因组关联分析 Fig. 2 Genome-wide association study of shear force trait |
|
图 3 系水力性状的全基因组关联分析 Fig. 3 Genome-wide association study of water binding capacity trait |
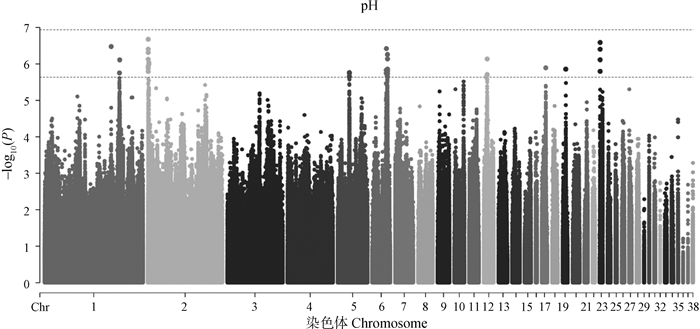
|
图 4 pH的全基因组关联分析 Fig. 4 Genome-wide association study of pH |
|
图 5 L*值的全基因组关联分析 Fig. 5 Genome-wide association study of L* value |
|
图 6 a*值的全基因组关联分析 Fig. 6 Genome-wide association study of a* value |
|
图 7 b*值的全基因组关联分析 Fig. 7 Genome-wide association study of b* value |
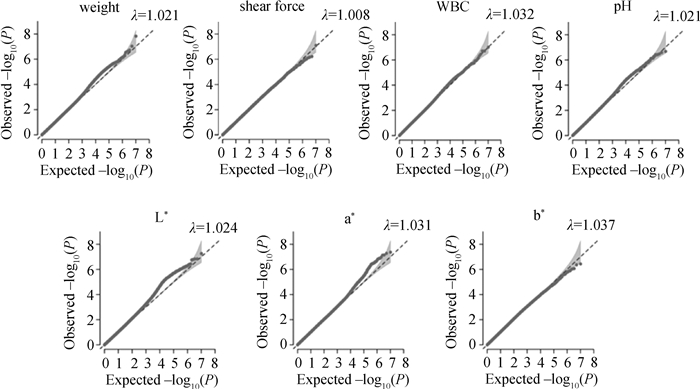
本试验QQ-plot分析结果见图 8,各性状膨胀系数介于1.008~1.037之间,表明本研究试验群体中不存在明显的种群分层现象。
|
图 8 QQ-plot分析 Fig. 8 QQ-plot analysis |
由于传统的饮食习惯,我国肉鸡生产呈现出独特的产业结构。随着活禽交易的逐步减少,消费者开始接受冰鲜鸡肉产品,使得育种企业和研究人员对兼顾成本和品质的快速型黄羽肉鸡品种的关注日益增加。利用GWAS方法挖掘与重要性状相关的分子标志并用于分子育种,是加快畜禽品种选育进展的有效措施。目前许多研究已经应用GWAS对京海黄鸡[19]、汶上芦花鸡[20]、茶花鸡、狼山鸡、竹丝鸡[21]等品种的体重性状开展了研究。鸡肉品质方面,许多学者已经开展了肌肉糖原与血糖水平[22-23]、肌内脂肪[24]、pH[25]、胸肌脂肪酸组成[26]、胸肌游离氨基酸和核苷酸组成[27]等性状的关联研究,为相关性状的选育提高积累了重要的基础数据。
本研究利用全基因组重测序,对393只立华父系麻黄鸡的60日龄体重和肉品质性状进行了关联分析。结果共发现了10个与目标性状显著关联的SNPs位点,338个潜在关联位点。这些与体重相关的SNPs主要位于4号、5号、19号和28号染色体上,其中以4号染色体上的位点最为丰富。先前的研究也发现,与体重有关的位点集中在1号和4号染色体上,张涛等[28]基于简化基因组测序对京海黄鸡上市体重进行研究时,筛选出了9个位于4号染色体75.641~79.592 Mb区域的关联位点,这说明本研究构建的模型相对准确。
在本研究中,发现一个与体重显著关联的位点位于28号染色体,其临近基因为FSTL3。FSTL3是一种分泌型糖蛋白,属于卵泡抑素模块蛋白家族,与肥胖水平和脂肪细胞炎症发生相关。血清中的FSTL3水平可反映脂肪中FSTL3表达水平及肥胖相关指标,被认为是内脏肥胖的生物标志[29]。此外,FSTL3的过表达减少了肥胖发展过程中的脂肪积累,并可能通过抑制肌肉生长抑制素等因子来控制体重[30]。在潜在关联位点中,rs43415532位于4号染色体上,其临近基因为MBNL3。MBNL3属于肌盲样蛋白家族成员,与肌肉发育和RNA的剪接调控密切相关。研究发现,MBNL3能够通过调控Myod的转录参与肌细胞的分化过程,抑制肌管的形成,并拮抗肌原素和肌球蛋白重链的表达。MBNL3过表达也会导致肌肉发育和收缩相关的基因表达下调[31]。一项研究报道了MBNL3敲除的小鼠出现肌肉再生功能的明显受损[32]。另外,位于34号染色体附近的一个体重相关SNP注释到基因NPFF和PFKM。NPFF是一种由两个氨基酸(Phe-Leu)组成的神经肽,在中枢神经系统中发挥多种作用。研究表明,NPFF可能通过调控相关激素的分泌来控制动物的摄食行为[33]。PFKM是磷酸化果糖激酶,在糖酵解途径中起重要作用,并在肌肉组织中广泛表达。Tan等[34]报道该基因与鸡和猪的肌肉发育有关。综上所述,本研究推测FSTL3、MBNL3、NPFF、PFKM可能通过控制脂肪沉积、肌肉发育和摄食行为参与快速型黄羽肉鸡的生长发育,因此可作为候选基因进行进一步研究。
目前,用于评价鸡肉品质的常见指标包括剪切力、系水力、pH和肉色。肉色通常被认为是评价肉质的首要指标,直接影响消费者的购买选择,一般用L(亮度)、a(红度)和b(黄度)来衡量。本研究共鉴定到220个与肉色相关联的位点,位点数量多于前人报道的结果[35-36],可能是由于本研究选择重测序作为分型工具,可用于关联的位点更多。本研究发现一个与胸肌黄度值相关的位点位于PRKG1基因附近。Zheng等[37]研究发现,PRKG1可以通过调控MYOD、MYOG和MEF2C的表达,从而参与肌肉品质性状的调控。剪切力是另一项重要的肉质指标,可直接反映肉的鲜嫩程度。本研究发现一个与剪切力显著关联的位点位于SLC13A5基因附近,该基因是溶质载体家族成员,在脂肪酸合成中起重要作用[38]。此外,Luo等[39]报道SLC13A5与肌内脂肪含量(IMF)显著相关,而IMF是影响肉类多汁性、嫩度和风味的关键因素之一[40]。系水力即肌肉的保水能力[41],它决定了肉的多汁性、营养组成、肉色外观、酸碱度和其他感官特性[42]。此外,系水力与pH存在联系。本研究发现了一个与系水力相关位点附近的基因为MDFIC。MDFIC属于一个包含富含半胱氨酸的c端结构域的小家族蛋白,研究发现该基因可能通过PI3K/AKT等通路调控肌细胞增殖分化,从而参与肌肉功能的调控[43-44]。本研究还发现AQP1基因旁的一个SNP与鸡肉的pH潜在相关。AQP编码一类水通道蛋白,参与水的跨膜运输。在一项关于猪肉品质的GWAS研究中也报道了AQP1与滴水损失显著关联[45]。另一个与pH潜在关联的位点位于FGD3基因附近。Marchesi等[46]在白条纹鸡肉上的研究中发现,FGD3基因在患病和正常个体的胸肌组织中表达存在显著差异,提示该基因与这种鸡肉问题的发生有关。本试验中,pH性状关联信号较强,与之相关的位点集中于1、2、5、6、12和23号染色体。葡萄糖的代谢途径之一即为合成肝糖原与肌糖原,肌糖原分解为乳酸影响pH,已有研究发现鸡肌肉糖原和血糖水平关联位点位于1、2、3、6和12号染色体[22-23],与本试验结果相似,提示相关位点在肉质调控中发挥关键作用。根据以上结果,认为PRKG1、SLC13A5、MDFIC、AQP1、FGD3等基因可能是快速型黄羽肉鸡肉品质性状的候选基因。然而,具体的作用机制仍需进一步进行试验验证。
4 结论本研究基于立华麻黄鸡全基因组重测序数据进行了GWAS分析,在与体重性状关联的结果中,筛选到2个显著关联,52个潜在关联的位点;肉品质性状结果中,筛选到8个显著关联,296个潜在关联的位点;并注释到99个与体重、225个与肉品质相关的功能基因。其中FSTL3、MBNL3、NPFF、PFKM、PRKG1、SLC13A5、MDFIC、FGD3、AQP1等基因可作为肉鸡体重和肉质性状的关键候选基因。本研究为快速型黄羽肉鸡重要经济性状提供了重要的遗传变异位点和候选基因,可以为相关品种开展分子育种提供理论基础。
| [1] |
蒋守群, 苟钟勇, 李龙, 等. 国家农业行业标准《黄羽肉鸡营养需要量》技术解析[J]. 饲料工业, 2022, 43(16): 59-64. JIANG S Q, GOU Z Y, LI L, et al. Technique introduction of national agricultural industry standard "Nutrient Requirements of Yellow Chicken"[J]. Feed Industry, 2022, 43(16): 59-64. (in Chinese) |
| [2] |
HUO W R, WENG K Q, LI Y, et al. Comparison of muscle fiber characteristics and glycolytic potential between slow-and fast-growing broilers[J]. Poult Sci, 2022, 101(3): 101649. DOI:10.1016/j.psj.2021.101649 |
| [3] |
WENG K Q, LI Y, HUO W R, et al. Comparative phosphoproteomic provides insights into meat quality differences between slow-and fast-growing broilers[J]. Food Chem, 2022, 373: 131408. DOI:10.1016/j.foodchem.2021.131408 |
| [4] |
褚震荣, 周辅民. 不同饲养方式对武定鸡肉品质的影响[J]. 特种经济动植物, 2022, 25(12): 1-3. CHU Z R, ZHOU F M. Effects of different feeding methods on the quality of Wuding chicken[J]. Special Economic Animals and Plants, 2022, 25(12): 1-3. (in Chinese) |
| [5] |
姜宏正, 杨德智, 马中华, 等. 儋州鸡胸肌肉色性状全基因组关联分析[J]. 中国畜牧杂志, 2022, 58(6): 117-122. JIANG H Z, YANG D Z, MA Z H, et al. Genome-wide association study of meat color traits in Danzhou chickens[J]. Chinese Journal of Animal Science, 2022, 58(6): 117-122. (in Chinese) |
| [6] |
张笑科, 廖伟莉, 陈信佑, 等. 杜洛克猪生长性状全基因组关联分析及候选基因鉴定[J]. 畜牧兽医学报, 2023, 54(5): 1868-1876. ZHANG X K, LIAO W L, CHEN X Y, et al. Genome-wide association study for identifying candidate genes of growth traits in Duroc pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2023, 54(5): 1868-1876. (in Chinese) |
| [7] |
乔贤, 张磊, 李晓凯, 等. 全基因组关联分析在畜禽重要经济性状中的研究进展[J]. 家畜生态学报, 2017, 38(10): 1-9. QIAO X, ZHANG L, LI X K, et al. Progress of genome-wide association analysis on important economic traits of livestock and poultry[J]. Journal of Domestic Animal Ecology, 2017, 38(10): 1-9. (in Chinese) |
| [8] |
HUANG S W, HE Y T, YE S P, et al. Genome-wide association study on chicken carcass traits using sequence data imputed from SNP array[J]. J Appl Genet, 2018, 59(3): 335-344. DOI:10.1007/s13353-018-0448-3 |
| [9] |
DOU D C, SHEN L Y, ZHOU J M, et al. Genome-wide association studies for growth traits in broilers[J]. BMC Genom Data, 2022, 23(1): 1. DOI:10.1186/s12863-021-01017-7 |
| [10] |
WANG K J, HU H F, TIAN Y D, et al. The chicken pan-genome reveals gene content variation and a promoter region deletion in IGF2BP1 affecting body size[J]. Mol Biol Evol, 2021, 38(11): 5066-5081. DOI:10.1093/molbev/msab231 |
| [11] |
HABIMANA R, NGENO K, OKENO T O, et al. Genome-wide association study of growth performance and immune response to Newcastle disease virus of indigenous chicken in Rwanda[J]. Front Genet, 2021, 12: 723980. DOI:10.3389/fgene.2021.723980 |
| [12] |
LI D F, LIAN L, QU L J, et al. A genome-wide SNP scan reveals two loci associated with the chicken resistance to Marek's disease[J]. Anim Genet, 2013, 44(2): 217-222. DOI:10.1111/j.1365-2052.2012.02395.x |
| [13] |
BUSH S J. Read trimming has minimal effect on bacterial SNP-calling accuracy[J]. Microb Genom, 2020, 6(12): e000434. |
| [14] |
LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760. DOI:10.1093/bioinformatics/btp324 |
| [15] |
NIARÉ K, GREENHOUSE B, BAILEY J A. An optimized GATK4 pipeline for Plasmodium falciparum whole genome sequencing variant calling and analysis[J]. Malar J, 2023, 22(1): 207. DOI:10.1186/s12936-023-04632-0 |
| [16] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [17] |
ZHOU X, STEPHENS M. Genome-wide efficient mixed-model analysis for association studies[J]. Nat Genet, 2012, 44(7): 821-824. DOI:10.1038/ng.2310 |
| [18] |
QUINLAN A R. BEDTools: The Swiss-army tool for genome feature analysis[J]. Curr Protoc Bioinformatics, 2014, 47: 11.12.1-11.12.34. |
| [19] |
张涛, 樊庆灿, 张向前, 等. 京海黄鸡体组成性状的全基因组关联分析[J]. 畜牧兽医学报, 2015, 46(9): 1502-1514. ZHANG T, FAN Q C, ZHANG X Q, et al. A genome-wide association study on body composition of Jinghai yellow chicken[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(9): 1502-1514. (in Chinese) |
| [20] |
KUMAR V, KATO N, URABE Y, et al. Genome-wide association study identifies a susceptibility locus for HCV-induced hepatocellular carcinoma[J]. Nat Genet, 2011, 43(5): 455-458. DOI:10.1038/ng.809 |
| [21] |
YUAN Y Y, PENG D Z, GU X R, et al. Polygenic basis and variable genetic architectures contribute to the complex nature of body weight-A genome-wide study in four Chinese indigenous chicken breeds[J]. Front Genet, 2018, 9: 229. DOI:10.3389/fgene.2018.00229 |
| [22] |
LIU X J, LIU L, WANG J, et al. Genome-wide association study of muscle glycogen in Jingxing yellow chicken[J]. Genes (Basel), 2020, 11(5): 497. DOI:10.3390/genes11050497 |
| [23] |
刘晓静, 刘璐, 王杰, 等. 鸡血糖性状的全基因组关联分析[J]. 畜牧兽医学报, 2020, 51(6): 1187-1195. LIU X J, LIU L, WANG J, et al. Genome-wide association study of chicken blood glucose traits using whole genome resequencing[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(6): 1187-1195. (in Chinese) |
| [24] |
WANG Y L, LIU L, LIU X J, et al. SLC16A7 Promotes triglyceride deposition by de novo Lipogenesis in chicken muscle tissue[J]. Biology (Basel), 2022, 11(11): 1547. |
| [25] |
BIHAN-DUVAL E L, HENNEQUET-ANTIER C, BERRI C, et al. Identification of genomic regions and candidate genes for chicken meat ultimate pH by combined detection of selection signatures and QTL[J]. BMC Genomics, 2018, 19(1): 294. DOI:10.1186/s12864-018-4690-1 |
| [26] |
FAN S X, YUAN P T, LI S H, et al. Genetic architecture and key regulatory genes of fatty acid composition in Gushi chicken breast muscle determined by GWAS and WGCNA[J]. BMC Genomics, 2023, 24(1): 434. DOI:10.1186/s12864-023-09503-1 |
| [27] |
KIM M, CHO E, MUNYANEZA J P, et al. Genome-wide association study for the free amino acid and nucleotide components of breast meat in an F2 crossbred chicken population[J]. J Anim Sci Technol, 2023, 65(1): 57-68. DOI:10.5187/jast.2022.e96 |
| [28] |
张涛, 王文浩, 张向前, 等. 基于简化基因组测序的京海黄鸡上市体重全基因组关联分析[J]. 农业生物技术学报, 2015, 23(8): 1067-1075. ZHANG T, WANG W H, ZHANG X Q, et al. Genome-wide association study on market weight based on specificlocus amplified fragment sequencing (SLAF-seq) in Jinghai yellow chicken (Gallus gallus)[J]. Journal of Agricultural Biotechnology, 2015, 23(8): 1067-1075. (in Chinese) |
| [29] |
LI X Y, ZHANG H W, MA X J, et al. FSTL3 is highly expressed in adipose tissue of individuals with overweight or obesity and is associated with inflammation[J]. Obesity (Silver Spring), 2023, 31(1): 171-183. DOI:10.1002/oby.23598 |
| [30] |
BRANDT C, HANSEN R H, HANSEN J B, et al. Over-expression of Follistatin-like 3 attenuates fat accumulation and improves insulin sensitivity in mice[J]. Metabolism, 2015, 64(2): 283-295. DOI:10.1016/j.metabol.2014.10.007 |
| [31] |
LEE K S, SMITH K, AMIEUX P S, et al. MBNL3/CHCR prevents myogenic differentiation by inhibiting MyoD-dependent gene transcription[J]. Differentiation, 2008, 76(3): 299-309. DOI:10.1111/j.1432-0436.2007.00209.x |
| [32] |
POULOS M G, BATRA R, LI M Y, et al. Progressive impairment of muscle regeneration in muscleblind-like 3 isoform knockout mice[J]. Hum Mol Genet, 2013, 22(17): 3547-3558. DOI:10.1093/hmg/ddt209 |
| [33] |
ZHANG L, KOLLER J, GOPALASINGAM G, et al. NPFF signalling is critical for thermosensory and dietary regulation of thermogenesis[J]. Neuropeptides, 2022, 96: 102292. DOI:10.1016/j.npep.2022.102292 |
| [34] |
TAN X F, HE Y, HE Y Q, et al. Comparative proteomic analysis of glycolytic and oxidative muscle in pigs[J]. Genes (Basel), 2023, 14(2): 361. DOI:10.3390/genes14020361 |
| [35] |
SUN Y F, ZHAO G P, LIU R R, et al. The identification of 14 new genes for meat quality traits in chicken using a genome-wide association study[J]. BMC Genomics, 2013, 14(1): 458. DOI:10.1186/1471-2164-14-458 |
| [36] |
杨欣婷, 郑麦青, 谭晓冬, 等. 快大型黄羽肉鸡肉品质性状的遗传参数估计和关键基因挖掘[J]. 畜牧兽医学报, 2021, 52(9): 2416-2428. YANG X T, ZHENG M Q, TAN X D, et al. Genetic parameters estimation and key genes identification for meat quality traits of fast-growing yellow-feather meat-type chickens[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(9): 2416-2428. (in Chinese) |
| [37] |
ZHENG Y, LIU T, LI Q, et al. Integrated analysis of long non-coding RNAs (lncRNAs) and mRNA expression profiles identifies lncRNA PRKG1-AS1 playing important roles in skeletal muscle aging[J]. Aging (Albany NY), 2021, 13(11): 15044-15060. |
| [38] |
LI L H, LI H S, GARZEL B, et al. SLC13A5 is a novel transcriptional target of the pregnane X receptor and sensitizes drug-induced steatosis in human liver[J]. Mol Pharmacol, 2015, 87(4): 674-682. DOI:10.1124/mol.114.097287 |
| [39] |
LUO W Z, CHENG D X, CHEN S K, et al. Genome-wide association analysis of meat quality traits in a porcine Large White×Minzhu intercross population[J]. Int J Biol Sci, 2012, 8(4): 580-595. DOI:10.7150/ijbs.3614 |
| [40] |
MADEIRA M S, COSTA P, ALFAIA C M, et al. The increased intramuscular fat promoted by dietary lysine restriction in lean but not in fatty pig genotypes improves pork sensory attributes[J]. J Anim Sci, 2013, 91(7): 3177-3187. DOI:10.2527/jas.2012-5424 |
| [41] |
张玉仙, 王文利, 曹允, 等. 不同浓度的同源益生素对肉鸡抗氧化能力的影响[J]. 中国兽医杂志, 2017, 53(8): 22-25. ZHANG Y X, WANG W L, CAO Y, et al. Effects of differentlevels of homologous probiotics on Antioxidant capacity of Broilers[J]. Chinese Journal of Veterinary Medicine, 2017, 53(8): 22-25. (in Chinese) |
| [42] |
袁艳枝, 邓文, 金瑶瑶, 等. 猪肉品质评定指标及影响因素的研究进展[J]. 黑龙江畜牧兽医, 2020(1): 31-35, 40. YUAN Y Z, DENG W, JIN Y Y, et al. Research progress on evaluation indices and influencing factors of pork quality[J]. Heilongjiang Animal Science and Veterinary Medicine, 2020(1): 31-35, 40. (in Chinese) |
| [43] |
HU X, XING Y S, REN L, et al. bta-miR-23a regulates the myogenic differentiation of fetal bovine skeletal muscle-derived progenitor cells by targeting MDFIC gene[J]. Genes (Basel), 2020, 11(10): 1232. DOI:10.3390/genes11101232 |
| [44] |
HUANG W L, GUO L J, ZHAO M X, et al. The inhibition on MDFIC and PI3K/AKT pathway caused by miR-146b-3p triggers suppression of myoblast proliferation and differentiation and promotion of apoptosis[J]. Cells, 2019, 8(7): 656. DOI:10.3390/cells8070656 |
| [45] |
JI J X, ZHOU L S, HUANG Y Z, et al. A whole-genome sequence based association study on pork eating quality traits and cooking loss in a specially designed heterogeneous F6 pig population[J]. Meat Sci, 2018, 146: 160-167. DOI:10.1016/j.meatsci.2018.08.013 |
| [46] |
MARCHESI J A P, IBELLI A M G, PEIXOTO J O, et al. Whole transcriptome analysis of the pectoralis major muscle reveals molecular mechanisms involved with white striping in broiler chickens[J]. Poult Sci, 2019, 98(2): 590-601. DOI:10.3382/ps/pey429 |
(编辑 郭云雁)



