人类基因组约有7% 的顺式调控元件作为基因表达调节的开关[1]。基因的激活或抑制是顺式调控元件上多个转录因子和辅因子协同作用的结果。现已证实转录因子通过激活或抑制调控基因表达,在细胞增殖、分化、衰老和凋亡等功能调控中起到关键作用。YY1是进化保守的C2H2型锌指蛋白,属于GLI-Krüppel蛋白家族,最初由Shi等[2]、Seto等[3]、Park和Atchison[4]多个研究组同时克隆并描述。YY-1在中国古代哲学中代表阴阳或正负,反映了该蛋白质在基因调节中发挥的双重功能,这取决于其与DNA的结合位点或细胞类型。YY1在动物体内广泛表达,HiChIP结果表明[5],在哺乳动物细胞中,YY1通常占据增强子-启动子相互作用的位点,偶尔也占据绝缘子相互作用的位点。且研究发现大约10% 的哺乳动物基因受该转录因子调控[6-8],因此YY1对基因的调控具有重要意义。本文将综述YY1的结构与功能、作用机制及其在动物繁殖调控过程中的研究进展,以期为深入挖掘该基因的功能及研究动物的繁殖调控机理提供理论参考。
1 YY1蛋白结构及特点YY1是调控真核细胞生命活动的关键转录因子。YY1在某些无脊椎动物和所有脊椎动物广泛表达且高度保守[9]。在人类基因组中,YY1位于人的14号染色体,包含5个外显子,编码414个氨基酸,预测分子量为44 ku,然而,蛋白质在SDS凝胶上迁移为65~68 ku,这可能与其空间结构有关。YY1基因表达的双重调控功能由其特殊的结构(N端激活域与C端抑制域)来决定的。YY1的N端1~100氨基酸序列[10],包含一段由11个连续的酸性氨基酸残基组成的区域、一段由12个不间断的组氨酸残基组成的区域(主要参与核定位)、一段富含甘氨酸和丙氨基酸的区域,一段富含脯氨酸和谷氨酰胺的区域[11]。YY1中心区域被证明参与转录抑制:176~200氨基酸序列组成的HID结构域;201~225氨基酸序列组成的REPO结构域,能够招募PcG蛋白到DNA并且引起组蛋白H3赖氨酸27甲基化[12-13]。YY1的C端氨基酸序列为298~414,由锌指结构域与某些特殊结构域构成的4个C2H2型锌指蛋白结构区域,C2H2型锌指蛋白既在转录过程中调控基因的表达,也参与转录后的修饰过程,转录后的修饰过程能提高锌指蛋白的抑制作用[14]。热力学和动力学研究表明,虽然YY1的N端片段本身不与DNA结合,但它的存在影响YY1与NDA结合参数,进而影响YY1对靶基因的调控。YY1靶向的基因范围广泛,大约10% 的哺乳动物基因受该转录因子调控,大多数YY1结合位点靠近转录起始位点,有时也出现在调控基因的远端。它通常绑定到具有以下位点的DNA序列[15-16]:GGCGCCATnTT、CCGCCATnTT、CGCCATnTT、GGCGCCATnTT、GCCATnTT、CnCCATnTT、GnCGACATnTT、CCGCCATnTT、TACGCCATtTTG、CGCCATCTT、CGCCATTTT和GCCATnTT(n表示任意碱基)。最新研究表明YY1可以与单链RNA结合,但具有低序列特异性[17]。
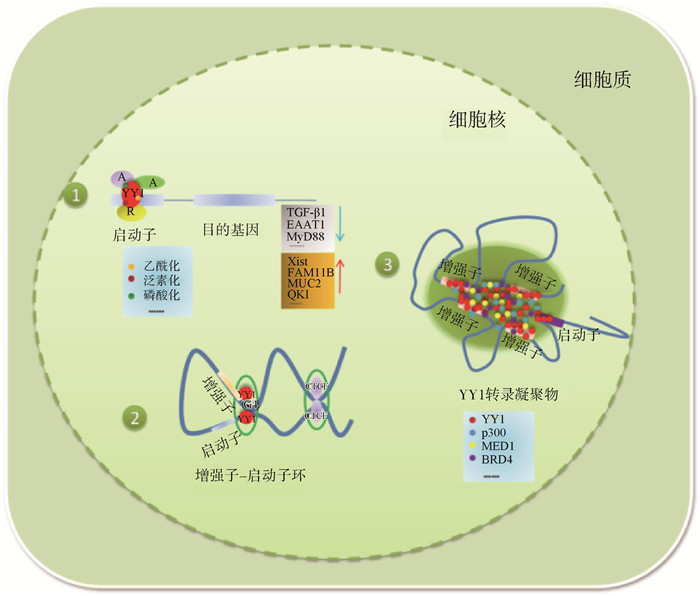
2 转录因子YY1的作用机制通过分析总结YY1的结构和功能得知,YY1调控基因表达的方式主要有:1)YY1作为传统转录因子调控基因表达;2)YY1通过调控染色质结构影响基因表达;3)YY1通过相分离机制调控基因的表达。
2.1 YY1作为传统转录因子调控基因表达2.1.1 YY1直接与基因启动子区结合调控基因表达 首先,YY1作为一种传统的DNA结合转录因子调控基因的表达,YY1能够直接与启动子结合抑制或者激活下游基因表达。例如,YY1与TGF-β1启动子结合能有效抑制TGF-β1的表达[18]。YY1可以直接与Xist(X-inactive-specific transcript,Xist)基因的5′调控区结合,激活Xist的转录,导致染色体失活,但YY1仅与未甲基化的Xist等位基因结合[19]。乳腺癌细胞中,YY1可与FAM111B基因启动子结合,增强其转录活性,加速了乳腺癌的进展[20]。胰腺β细胞中,YY1结合到Ins1和Ins2外显子2的增强子区域,激活胰岛素转录,进而调控葡萄糖稳态[21]。抗猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)感染过程中,YY1与MUC2启动子的结合[22]促进MUC2的表达,降低仔猪对PEDV的易感性,但该位点的调控作用受甲基化影响。YY1可以直接结合在PTX3基因的启动子区,还可以与PRMT1关联一起募集到PTX3启动子上[23]。Nhlh2参与卵泡发育等繁殖活动,而YY1可以直接结合到该基因的启动子上调控Nhlh2的表达[24]。截止目前,对YY1功能的研究多集中在其对DNA的直接调控上,但通常情况下YY1不是单独起作用的,YY1通常与其他的辅助因子共同发挥作用。
2.1.2 YY1与其他辅助因子结合后共同调控基因表达 除了直接与启动子结合外,YY1能够与其他辅助因子结合共同调控基因表达。在小鼠神经元中,YY1及其相互作用蛋白Brd4激活了谷氨酸信号的上游调节因子Senp1,在神经元可塑性中起着关键作用[25]。另外,YY1能够破坏p53和辅激活因子p300之间的相互作用,阻断p300依赖的乙酰化且降低p53的稳定[26]。不仅如此,YY1可与p53直接物理相互作用,而槲皮素能够直接与p53竞争结合YY1,降低YY1与p53蛋白对接能量进而发挥抗癌作用[27]。功能试验表明,circ-RCCD调控心脏搏动细胞簇的形成,主要通过将YY1招募到MyD88的启动子上来抑制MyD88的水平,促进心肌细胞分化[28]。QKI是一种可在上皮-间质转化过程中调节环状RNA形成的剪接因子,与肝细胞癌发生密切相关,YY1通过与p65和p300形成复合物增强QKI的转录和翻译,是治疗肝癌的潜在靶点[29]。在HepG2细胞中,RBM25是YY1高效靶向全基因组所必需的,YY1和RBM25之间存在广泛的共结合,RBM25首先与靶基因启动子相互作用,然后将YY1招募到这些启动子上,共同调控基因的转录[30]。
2.1.3 YY1的转录后修饰对基因表达调控的影响 YY1的生物学功能受其翻译后修饰的调控,如乙酰化、泛素化和磷酸化等。首先,YY1的转录活性可以通过乙酰化或去乙酰化来调控。蛋白质赖氨酸乙酰化是调节蛋白质结构和功能的翻译后修饰,GCN5在YY1的两个残基(K392和K393)上乙酰化,破坏了YY1锌指和DNA之间的相互作用维持结构蛋白的表达,从而正向调节肌肉完整性[31]。另外,YY1蛋白可以在两个区域被p300或PCAF乙酰化。在体外,YY1的C端结构域的乙酰化降低了DNA结合能力,而中心结构域的乙酰化是YY1作为完全阻滞剂所必需的[32]。在小鼠前神经外胚层发育过程中,Otx2激活需要YY1的乙酰化[33]。E3连接酶Smurf2可以泛素化YY1,抑制其下游基因VEGFA和Snail1表达[34]。另外,JWA是一种有效的环境反应基因,具有促凋亡作用。三阴性乳腺癌中JAC1引起YY1泛素化激活JWA,解除YY1对JWA的抑制[35]。磷酸化使蛋白结构发生变化引起蛋白质活性的改变,CK2α催化YY1第118位丝氨酸磷酸化干扰了Caspase 7对其的切割,促进癌细胞存活。YY1是Src家族激酶介导酪氨酸磷酸化的一个靶点,研究表明YY1第383位酪氨酸磷酸化影响它结合DNA和RNA的能力[36],但对于负责YY1去磷酸化的特定酪氨酸磷酸酶,以及体内酪氨酸磷酸化YY1的定位和动态变化需要进一步研究。
2.2 YY1通过调控染色质结构影响基因表达大量的证据表明,染色质结构在基因调控中起到重要作用,但对于基因启动子和增强子之间互相作用的调控蛋白了解很少。基因及其调控元件的大DNA环的形成需要CTCF-CTCF相互作用,但大多数情况下这种结构蛋白并不参与启动子和增强子之间的相互作用。通过研究发现,YY1在所有被检测的细胞中普遍表达,且占据增强子和启动子位置,YY1的二聚化促进DNA环的形成[37],推测YY1介导的增强子-启动子相互作用可能是哺乳动物基因调控的一般特征。YY1在不同类型的细胞中通常占据活跃的增强子和启动子,扰动YY1结合会破坏增强子-启动子环[37]。同样,敲低神经祖细胞中的YY1后,许多3D结构被破坏,这表明YY1可能是一种连接基因启动子和增强子的结构蛋白[38]。作为增强子和启动子的连接蛋白,YY1与其招募的共激活因子和增强子元件共同作用,增强子-启动子相互作用稳定,激活下游靶基因的转录。例如:YY1与许多转录辅激活因子相互作用,如EP300、BRD4、MED1等,并在稳定增强子-启动子环中发挥关键作用。分析YY1序列时,发现其TAD结构域(topologically associating domain,TAD)具有较高的结构紊乱倾向,它的大多数结合位点都靠近转录起始位点,只有少数位点位于调控基因的远端[39-41]。然而,在YY1-单倍缺陷淋巴母细胞系中,差异表达最多的基因是由YY1远端结合位点控制的[39]。为了进一步揭示YY1影响增强子-启动子互作的机制,研究发现DNA的鸟嘌呤四联体的结构(G4)是YY1促进增强子与启动子之间环形成的具体结合位置[42],同时也表明YY1除了结合双链DNA的共识序列外,还能够直接与G4直接结合对基因表达调控起作用。
2.3 YY1通过相分离机制调控基因的表达液-液相分离是指溶液相的组分由于溶解度的差异而分离,自2009年Brangwynne等[43]通过P颗粒液体的性质证明了液-液相分离的存在,已有研究表明相分离现象在细胞中普遍存在,是研究活细胞基因调控的新方法。越来越多的研究发现,相分离与基因的转录调控密切相关[44-46]。转录控制可能是由相分离进行驱动的[47],共激活蛋白MED1和BRD4在超增强子处发生相分离形成液滴样物质[45],胚胎干细胞多能性转录因子OCT4、雌激素受体(estrogen receptor,ER)和酵母转录因子GCN4与Mediator形成相分离的液滴样凝聚物,并且需要相同的氨基酸或配体来激活相分离。Boija等[44]认为,内在无序区域介导的相分离与辅激活剂是转录因子活化结构域激活基因的一种机制。大量研究一致表明,YY1参与形成增强子或超级增强子,调节生物过程多种基因的表达,然而,YY1如何招募、协调共激活因子和染色质增强子元件,组装增强子簇或超级增强子的调控机制还不清楚。目前,试验数据表明了YY1在体外和细胞内进行液-液相分离的能力[48]。YY1已被报道通过促进增强子活性来调节基因表达,而相分离是增强子机制的一个特征[49],YY1蛋白在细胞内形成液-液凝聚物,YY1的组氨酸簇对于其产生相分离和维持细胞招募Pol Ⅱ转录复合体非常重要,将H3K9me3隔离在凝聚物外,增强FOXM1转录[50]。生化试验表明,转录因子Twist1和YY1以相分离的形式形成转录复合体。Twist1-YY1-p300形成核相分离凝聚体,当Twist1和YY1蛋白共存时,辅激活剂p300刺激转录因子Twist1和YY1的相分离液滴物质在细胞内形成Twist1/YY1/p300复合物,可在miR-9超级增强子上形成相分离的凝聚体[51],发挥转录激活功能。这种类型的结合调节miR-9的表达,从而增加细胞迁移和扩散,促进HCC中的EMT。目前关于YY1相分离的研究不多,相信随着科研的不断进步,将深入探讨YY1如何通过相分离调控转录,为研究发育和疾病过程中基因表达调控机制提供新思路。
总结前人研究,可以将YY1作用机制概括如图 1所示:1)YY1作为传统的转录因子直接结合在基因启动子区,或者通过招募其他辅因子结合到靶基因启动子区共同调控基因的表达,或者通过调控YY1转录后修饰改变YY1的表达进而对下游基因进行调控;2)YY1作为启动子-增强子重要的结构物质,维持启动子-增强子环,G4是其与DNA结合的具体位点;3)YY1通过转录复合物与超级增强子形成液滴凝聚物,以相分离的形式调控下游基因的表达。
|
A. 激活因子;R. 抑制因子 A. Activation factor; R. Inhibitory factor 图 1 YY1调控基因表达的作用机制[37, 42, 50] Fig. 1 The mechanism of YY1 regulating gene expression[37, 42, 50] |
在哺乳动物中,随着研究的不断深入,无论是卵巢发育与精子生成,还是胚胎发育,亦或生殖激素及受体表达的调控,YY1在其中都扮演着至关重要的角色。
3.1 YY1对卵巢发育的影响信号分子和转录调节因子是卵泡发生、成熟和排卵期不可缺乏的,YY1作为一种重要的转录因子,在调节卵泡发育过程中起着重要的作用。2011年,Griffith等[52]通过构建卵母细胞YY1(CKO-YY1)特异性敲除小鼠,发现YY1是卵母细胞成熟和颗粒细胞扩张及卵母细胞-颗粒细胞通信所必需的。此外,有团队研究发现YY1基因在恒河猴的卵母细胞及胚胎发育过程中表达丰富,揭示其可能调控相关重要基因的转录[53]。鸡的全基因组关联分析显示YY1处于与排卵数性状的显著相关的候选区域[54],但其与鸡产蛋数之间的关系需要进一步探究。近期研究发现,基质细胞源性因子-l(stromal cell-derived factor-l,SDF1)处理增加了YY1和TET1的mRNA和蛋白质水平,对卵母细胞质量至关重要,可能增强成熟卵母细胞支持后续胚胎发育的能力[55]。然而,敲低多囊卵巢综合征小鼠卵巢颗粒细胞YY1促进细胞增殖,抑制细胞凋亡[56]。盛中伟等[57]发现,12~13周龄文昌母鸡的卵巢迅速发育是其性成熟启动时期,而qRT-PCR检测结果表明YY1此时在下丘脑组织中的表达水降低,提示YY1可能参与了下丘脑性腺轴对鸡性成熟启动的调节。高婷等[58]研究发现,六味地黄丸能够治疗小鼠卵巢储备功能减退,而YY1是六味地黄丸发挥作用的潜在靶点。以上研究表明,YY1在卵巢发育过程中的功能比较复杂,可能与细胞的状态和类型相关,这些研究发现有利于扩展研究者对卵巢发育分子机制的理解。
3.2 YY1对精子发生的影响YY1不仅参与雌性动物卵巢发育调控,还在雄性性腺的发育和精子发生中起着关键作用。研究发现,小鼠雄性性腺的发育过程中,YY1也扮演着重要的角色。YY1在精子发生的不同阶段表达量不同,这取决于生精小管的发育阶段。Kim等[59]在2016年的研究中发现,YY1强烈定位于精原干细胞,YY1与CP2c共定位促进精子发生,同时发现YY1对精原干细胞的干性至关重要,但在成熟的精子中不表达。利用RNAi技术敲除小鼠睾丸中的YY1基因,引起DNA修复缺陷造成精母细胞单倍染色体形成,非整倍体增加,粗线期细胞死亡,说明YY1在小鼠精子减数分裂过程中起重要作用[60]。同样,有研究发现在鸡胚睾丸中,YY1在精原细胞和支持细胞中表达,而在成年鸡睾丸中,YY1在生精细胞和支持细胞中表达[61],而在精子中不表达,这与小鼠中的研究结果一致。由此可见,YY1可能通过影响鸡睾丸中支持细胞内的细胞因子来调控鸡性腺的发育和功能,进而影响精精子的发生。
3.3 YY1对胚胎发育的调控胚胎发育过程中,YY1在除1细胞胚胎外的所有组织中均表达,但主要定位在2细胞胚胎的细胞质及4细胞、8细胞、桑葚胚和囊胚期胚胎的细胞核[62],推测YY1对胚胎的调控可能从4细胞开始。YY1缺失并不影响OCT4和SOX2 mRNA的表达[62],但会影响两者的共定位, 推测乙酰化的YY1可能特异性调节OCT4/SOX2复合物形成,但具体机制未知。在小鼠胚胎干细胞中,YY1和OCT4具有协同作用,同时YY1与BAF复合物相互作用促进小鼠胚胎干细胞增殖[63]。Donohoe等[64]通过构建YY1敲除小鼠模型试验研究发现,缺乏YY1的胚胎植入子宫后未能发育到原肠胚期,导致胚胎在植入时死亡,说明了YY1在小鼠胚胎发育中的关键作用。PHLDA2(印迹基因)会影响山羊胎盘发育,而YY1和CpG甲基化均会抑制PHLDA2的表达,YY1更倾向与CpG-甲基化序列相结合来抑制PHLDA2的表达,进而对胎盘发育进行调节[65]。Rhee等[66]研究发现,虽然YY1不是胚胎内胚层衍生器官形成所必需的,但YY1在内胚层中是邻近中胚层血管生成所必需的,且YY1对小鼠胚胎血管内皮生长因子(vascular endothelial growth factor,VEGFA)的产生和卵黄囊的发育都具有重要的作用。YY1直接结合lncRNA Xist启动子,促进Xist在围产期小鼠卵巢中的表达[67],但siRNA介导的YY1表达下调可以抑制Xist的表达,上调X连锁基因的表达,提高克隆猪胚胎的发育效率[68]。除此之外,YY1还与胚胎的DNA甲基化有关,水牛植入前胚胎注射YY1过表达载体不会影响囊胚发育率,但会提高DNA甲基化水平[69]。PGC7主要在卵母细胞中表达,PGC7和YY1相互作用能够抑制YY1对PRC2的募集,促进AKT与EZH2相互作用,减弱YY1与EZH2的相互作用,从而降低H3K27me3水平。在受精卵中,PGC7缺乏和AKT抑制剂均会促进EZH2进入原核,增加原核中H3K27me3的水平,抑制H3K27me3调控的合子激活基因的表达,最终影响两细胞胚胎的早期发育[70]。因此,YY1在胚胎发育过程中起重要的调控作用,但是YY1是如何调控胚胎发育生物学过程的,仍需要深入探究。
3.4 YY1调控生殖激素及其受体的表达YY1除了调控卵巢发育、精子发生和胚胎发育以外,还参与生殖激素的调控。芳香化酶(CYP19)是雌二醇合成的关键酶,大鼠CYP19启动子中含有YY1应答元件[71],但YY1的下调显著促进了多囊氏综合征小鼠卵巢颗粒细胞中雌激素的产生[56]。促卵泡激素受体(follicle-stimulating hormone receptor,FSHR)在卵巢颗粒细胞中特异性表达,并连接配体FSH和细胞内信号分子,如PKA和AKT,以调节雌性哺乳动物的卵泡发育和不孕症。FSHR已被确定为决定猪排卵率和多态性的主要候选基因,转录因子YY1通过与湖羊FSHR启动子的核心启动子结合而影响其转录活性,但不参与核心启动子内突变位点对转录活性的调控[72]。Ai等[73]证实,YY1能够直接结合在胆固醇调节元件结合蛋白(sterol regulatory element binding protein,SREBP) 启动子上。研究发现YY1-AR相互作用对YY1介导的AR转录活性至关重要,YY1是与雄激素反应元件结合复合物的必要组成部分[74-75]。ChIP、qPCR和YY1敲低/过表达试验表明,YY1在睾丸间质细胞中作为转录激活因子直接调节类固醇激素生成的关键基因StAR和3β-HSD的表达,最终协同调控睾酮的产生[76],但YY1有时可以作为类固醇生成基因的转录抑制因子,具体原因尚未揭示。
以上研究表明,YY1在动物繁殖调控中起到关键作用,但相关研究尚处于起步阶段,需开展相关研究进行深入探索。
4 结语与展望截止目前YY1蛋白结构基本明确,且在多个物种中高度保守,它通常绑定到保守的DNA结合序列上,但有新的研究发现YY1还可以结合到DNA的G4结构上影响启动子-增强子的结构。同样,不同物种中YY1的结合位点是否相同尚无定论。除了与DNA结合以外,有研究揭示YY1可以与单链RNA结合,但其结合序列的特异性不高,YY1与单链RNA结合位点的特征及功能目前还不清楚,因此YY1的作用机制要复杂的多。
对于YY1作用机制的研究多集中在传统的转录因子作用机理上,YY1既可以促进基因的表达也可以抑制基因的表达,因此,对于YY1作用机制的研究重点在于要确定YY1对基因表达的调控是正向的还是反向的。另外,一般YY1的靶基因启动子区含有多个结合位点,那么YY1和这些位点结合对于基因的表达调控是否一致也需要进一步揭示。YY1转录后修饰的方式多种多样,多数研究显示转录后修饰影响了YY1作为经典转录因子对下游靶基因的调控。生物体的调控网络比较复杂,DNA环的形成可以更好的解释增强子-启动子远距离互作的机制。YY1可以以CTCF介导DNA相互作用的方式促进基因启动子-增强子之间的相互作用[37],未来研究可关注YY1对染色质结构的影响。除此之外,最新的研究揭示YY1在液-液相分离中表现出重要的调控作用。目前,液-液相分离的研究已经成为生命科学领域的新焦点,但是对于相分离形成的机制仍知之甚少,因此YY1通过相分离影响基因表达的机理目前依然不清楚。
虽然YY1在癌症调控中的作用已经被广泛的证实,但YY1对动物繁殖调控影响的研究并不多,YY1是卵母细胞成熟、卵丘扩张的关键因子,对胚胎发育至关重要,可以调控睾酮、雌激素等的产生,因此YY1在动物繁殖调控中的作用比较复杂,有必要展开更多的试验进一步揭示YY1对动物繁殖调控的作用。
综上所述,YY1除了作为传统的转录因子发挥作用外,能够作为一种结构蛋白参与染色质环的形成,除此之外还通过相分离机制来调控基因表达。此综述使广大科研工作者对YY1功能机制的理解更全面。YY1参与动物卵巢发育、精子生成、胚胎发育、生殖激素调控及其受体表达等繁殖相关的重要生物学过程。但目前关于YY1在动物繁殖调控中作用机制的研究还不透彻,许多重要的问题还没进行有效的解答。因此,YY1在动物繁殖领域的功能和作用机理需要更加深入和全面的研究,以便为畜牧领域提供理论基础。
| [1] |
KREBS A R. Studying transcription factor function in the genome at molecular resolution[J]. Trends Genet, 2021, 37(9): 798-806. DOI:10.1016/j.tig.2021.03.008 |
| [2] |
SHI Y, SETO E, CHANG L S, et al. Transcriptional repression by YY1, a human GLI-Krüippel-related protein, and relief of repression by adenovirus E1A protein[J]. Cell, 1991, 67(2): 377-388. DOI:10.1016/0092-8674(91)90189-6 |
| [3] |
SETO E, SHI Y, SHENK T. YY1 is an initiator sequence-binding protein that directs and activates transcription in vitro[J]. Nature, 1991, 354(6350): 241-245. DOI:10.1038/354241a0 |
| [4] |
PARK K, ATCHISON M L. Isolation of a candidate repressor/activator, NF-E1 (YY-1, delta), that binds to the immunoglobulin kappa 3' enhancer and the immunoglobulin heavy-chain mu E1 site[J]. Proc Natl Acad Sci U S A, 1991, 88(21): 9804-9808. DOI:10.1073/pnas.88.21.9804 |
| [5] |
GOPALAKRISHNAN J, TESSNEER K L, FU Y, et al. Variants on the UBE2L3/YDJC autoimmune disease risk haplotype increase UBE2L3 expression by modulating CCCTC-binding factor and YY1 binding[J]. Arthrit Rheumatol, 2022, 74(1): 163-173. DOI:10.1002/art.41925 |
| [6] |
KHACHIGIAN L M. The Yin and Yang of YY1 in tumor growth and suppression[J]. Int J Cancer, 2018, 143(3): 460-465. DOI:10.1002/ijc.31255 |
| [7] |
GRÖNROOS E, TERENTIEV A A, PUNGA T, et al. YY1 inhibits the activation of the p53 tumor suppressor in response to genotoxic stress[J]. Proc Natl Acad Sci U S A, 2004, 101(33): 12165-12170. DOI:10.1073/pnas.0402283101 |
| [8] |
SUI G C, AFFAR E B, SHI Y J, et al. Yin Yang 1 is a negative regulator of p53[J]. Cell, 2004, 117(7): 859-872. DOI:10.1016/j.cell.2004.06.004 |
| [9] |
MELIALA I T S, HOSEA R, KASIM V, et al. The biological implications of Yin Yang 1 in the hallmarks of cancer[J]. Theranostics, 2020, 10(9): 4183-4200. DOI:10.7150/thno.43481 |
| [10] |
GORDON S, AKOPYAN G, GARBAN H, et al. Transcription factor YY1:structure, function, and therapeutic implications in cancer biology[J]. Oncogene, 2006, 25(8): 1125-1142. DOI:10.1038/sj.onc.1209080 |
| [11] |
ATCHISON M L, BASU A, ZAPRAZNA K, et al. Mechanisms of Yin Yang 1 in oncogenesis: the importance of indirect effects[J]. Crit Rev Oncog, 2011, 16(3-4): 143-161. DOI:10.1615/CritRevOncog.v16.i3-4.20 |
| [12] |
VERHEUL T C J, VAN HIJFTE L, PERENTHALER E, et al. The Why of YY1:mechanisms of transcriptional regulation by Yin Yang 1[J]. Front Cell Dev Biol, 2020, 8: 592164. DOI:10.3389/fcell.2020.592164 |
| [13] |
WILKINSON F H, PARK K, ATCHISON M L. Polycomb recruitment to DNA in vivo by the YY1 REPO domain[J]. Proc Natl Acad Sci U S A, 2006, 103(51): 19296-19301. DOI:10.1073/pnas.0603564103 |
| [14] |
樊星, 宋兴舜, 邢倩. 锌指蛋白转录因子YY1的功能研究进展[J]. 农业与技术, 2022, 42(8): 1-4. FAN X, SONG X S, XING Q. Progress in functional research on zinc finger protein transcription factor YY1[J]. Agriculture and Technology, 2022, 42(8): 1-4. DOI:10.19754/j.nyyjs.20220430001 (in Chinese) |
| [15] |
KIM J D, KIM J. YY1's longer DNA-binding motifs[J]. Genomics, 2009, 93(2): 152-158. DOI:10.1016/j.ygeno.2008.09.013 |
| [16] |
FIGIEL M, GÓRECKI A. Physical interaction of Human Yin Yang 1 protein with DNA[J]. Crit Rev Oncog, 2017, 22(1-2): 75-97. DOI:10.1615/CritRevOncog.2017020976 |
| [17] |
WAI D C C, SHIHAB M, LOW J K K, et al. The zinc fingers of YY1 bind single-stranded RNA with low sequence specificity[J]. Nucleic Acids Res, 2016, 44(19): 9153-9165. |
| [18] |
GAO P, LI L L, YANG L, et al. Yin Yang 1 protein ameliorates diabetic nephropathy pathology through transcriptional repression of TGFβ1[J]. Sci Transl Med, 2019, 11(510): eaaw2050. DOI:10.1126/scitranslmed.aaw2050 |
| [19] |
MAKHLOUF M, OUIMETTE J F, OLDFIELD A, et al. A prominent and conserved role for YY1 in Xist transcriptional activation[J]. Nat Commun, 2014, 5: 4878. DOI:10.1038/ncomms5878 |
| [20] |
LI W, HU S H, HAN Z Q, et al. YY1-induced transcriptional activation of FAM111B contributes to the malignancy of breast cancer[J]. Clin Breast Cancer, 2022, 22(4): e417-e425. DOI:10.1016/j.clbc.2021.10.008 |
| [21] |
LIU D, YANG K Y, CHAN V W, et al. YY1 regulates glucose homeostasis through controlling insulin transcription in pancreatic β-cells[J]. Diabetes, 2022, 71(5): 961-977. DOI:10.2337/db21-0695 |
| [22] |
XIAO Y Y, ZHOU Y J, SUN S Y, et al. Effect of promoter methylation on the expression of porcine MUC2 gene and resistance to PEDV infection[J]. Front Vet Sci, 2021, 8: 646408. DOI:10.3389/fvets.2021.646408 |
| [23] |
LATHORIA K, GOWDA P, UMDOR S B, et al. PRMT1 driven PTX3 regulates ferritinophagy in glioma[J]. Autophagy, 2023, 19(7): 1997-2014. DOI:10.1080/15548627.2023.2165757 |
| [24] |
温丽娟, 刘发伟, 林长光, 等. 猪Nhlh2基因启动子活性及其表达调控的初步研究[J]. 畜牧兽医学报, 2018, 49(4): 667-674. WEN L J, LIU F W, LIN C G, et al. Analysis of the promoter activity and expression regulation of porcine Nhlh2 gene[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(4): 667-674. (in Chinese) |
| [25] |
WU T, DONOHOE M E. Yy1 regulates Senp1 contributing to AMPA receptor GluR1 expression following neuronal depolarization[J]. J Biomed Sci, 2019, 26(1): 79. DOI:10.1186/s12929-019-0582-1 |
| [26] |
GRÖNROOS E, TERENTIEV A A, PUNGA T, et al. YY1 inhibits the activation of the p53 tumor suppressor in response to genotoxic stress[J]. Proc Natl Acad Sci U S A, 2004, 101(33): 12165-12170. DOI:10.1073/pnas.0402283101 |
| [27] |
GUAN H, ZHANG W Y, LIU H, et al. Quercetin induces apoptosis in HepG2 cells via directly interacting with YY1 to disrupt YY1-p53 interaction[J]. Metabolites, 2023, 13(2): 229. DOI:10.3390/metabo13020229 |
| [28] |
LIU Y W, GAO J F, XU M, et al. Circular RNA circ-RCCD promotes cardiomyocyte differentiation in mouse embryo development via recruiting YY1 to the promoter of MyD88[J]. J Cell Mol Med, 2022, 26(13): 3616-3627. DOI:10.1111/jcmm.17336 |
| [29] |
HAN J X, MENG J, CHEN S, et al. YY1 complex promotes quaking expression via super-enhancer binding during EMT of hepatocellular carcinoma[J]. Cancer Res, 2019, 79(7): 1451-1464. DOI:10.1158/0008-5472.CAN-18-2238 |
| [30] |
XIAO R, CHEN J Y, LIANG Z Y, et al. Pervasive chromatin-RNA binding protein interactions enable RNA-based regulation of transcription[J]. Cell, 2019, 178(1): 107-121.e18. DOI:10.1016/j.cell.2019.06.001 |
| [31] |
ADDICKS G C, ZHANG H B, RYU D, et al. GCN5 maintains muscle integrity by acetylating YY1 to promote dystrophin expression[J]. J Cell Biol, 2022, 221(2): e202104022. DOI:10.1083/jcb.202104022 |
| [32] |
YAO Y L, YANG W M, SETO E. Regulation of transcription factor YY1 by acetylation and deacetylation[J]. Mol Cell Biol, 2001, 21(17): 5979-5991. DOI:10.1128/MCB.21.17.5979-5991.2001 |
| [33] |
TAKASAKI N, KUROKAWA D, NAKAYAMA R, et al. Acetylated YY1 regulates Otx2 expression in anterior neuroectoderm at two cis-sites 90 kb apart[J]. EMBO J, 2007, 26(6): 1649-1659. DOI:10.1038/sj.emboj.7601619 |
| [34] |
FU S H, LAI M C, ZHENG Y Y, et al. MiR-195 inhibits the ubiquitination and degradation of YY1 by Smurf2, and induces EMT and cell permeability of retinal pigment epithelial cells[J]. Cell Death Dis, 2021, 12(7): 708. DOI:10.1038/s41419-021-03956-6 |
| [35] |
ZHAI Z R, REN Y L, SHU C J, et al. JAC1 targets YY1 mediated JWA/p38 MAPK signaling to inhibit proliferation and induce apoptosis in TNBC[J]. Cell Death Discov, 2022, 8(1): 169. DOI:10.1038/s41420-022-00992-9 |
| [36] |
RIMAN S, RIZKALLAH R, KASSARDJIAN A, et al. Phosphorylation of the transcription factor YY1 by CK2α prevents cleavage by caspase 7 during apoptosis[J]. Mol Cell Biol, 2012, 32(4): 797-807. DOI:10.1128/MCB.06466-11 |
| [37] |
WEINTRAUB A S, LI C H, ZAMUDIO A V, et al. YY1 is a structural regulator of enhancer-promoter loops[J]. Cell, 2017, 171(7): 1573-1588.e28. DOI:10.1016/j.cell.2017.11.008 |
| [38] |
BEAGAN J A, DUONG M T, TITUS K R, et al. YY1 and CTCF orchestrate a 3D chromatin looping switch during early neural lineage commitment[J]. Genome Res, 2017, 27(7): 1139-1152. DOI:10.1101/gr.215160.116 |
| [39] |
GABRIELE M, VAN SILFHOUT A T, GERMAIN P L, et al. YY1 haploinsufficiency causes an intellectual disability syndrome featuring transcriptional and chromatin dysfunction[J]. Am J Human Genet, 2017, 100(6): 907-925. DOI:10.1016/j.ajhg.2017.05.006 |
| [40] |
VARUM S, BAGGIOLINI A, ZURKIRCHEN L, et al. Yin Yang 1 orchestrates a metabolic program required for both neural crest development and melanoma formation[J]. Cell Stem Cell, 2019, 24(4): 637-653.e9. DOI:10.1016/j.stem.2019.03.011 |
| [41] |
ZURKIRCHEN L, VARUM S, GIGER S, et al. Yin Yang 1 sustains biosynthetic demands during brain development in a stage-specific manner[J]. Nat Commun, 2019, 10(1): 2192. DOI:10.1038/s41467-019-09823-5 |
| [42] |
LI L, WILLIAMS P, REN W D, et al. YY1 interacts with guanine quadruplexes to regulate DNA looping and gene expression[J]. Nat Chem Biol, 2021, 17(2): 161-168. DOI:10.1038/s41589-020-00695-1 |
| [43] |
BRANGWYNNE C P, ECKMANN C R, COURSON D S, et al. Germline P granules are liquid droplets that localize by controlled dissolution/condensation[J]. Science, 2009, 324(5935): 1729-1732. DOI:10.1126/science.1172046 |
| [44] |
BOIJA A, KLEIN I A, SABARI B R, et al. Transcription factors activate genes through the phase-separation capacity of their activation domains[J]. Cell, 2018, 175(7): 1842-1855.e16. DOI:10.1016/j.cell.2018.10.042 |
| [45] |
SABARI B R, DALL'AGNESE A, BOIJA A, et al. Coactivator condensation at super-enhancers links phase separation and gene control[J]. Science, 2018, 361(6400): eaar3958. DOI:10.1126/science.aar3958 |
| [46] |
CHONG S S, DUGAST-DARZACQ C, LIU Z, et al. Imaging dynamic and selective low-complexity domain interactions that control gene transcription[J]. Science, 2018, 361(6400): eaar2555. DOI:10.1126/science.aar2555 |
| [47] |
HNISZ D, SHRINIVAS K, YOUNG R A, et al. A phase separation model for transcriptional control[J]. Cell, 2017, 169(1): 13-23. DOI:10.1016/j.cell.2017.02.007 |
| [48] |
BOIJA A, KLEIN I A, SABARI B R, et al. Transcription factors activate genes through the phase-separation capacity of their activation domains[J]. Cell, 2018, 175(7): 1842-1855.e16. DOI:10.1016/j.cell.2018.10.042 |
| [49] |
HAHN S. Phase separation, protein disorder, and enhancer function[J]. Cell, 2018, 175(7): 1723-1725. DOI:10.1016/j.cell.2018.11.034 |
| [50] |
WANG W M, QIAO S Y, LI G Y, et al. A histidine cluster determines YY1-compartmentalized coactivators and chromatin elements in phase-separated enhancer clusters[J]. Nucleic Acids Res, 2022, 50(9): 4917-4937. DOI:10.1093/nar/gkac233 |
| [51] |
MENG J, HAN J X, WANG X R, et al. Twist1-YY1-p300 complex promotes the malignant progression of HCC through activation of miR-9 by forming phase-separated condensates at super-enhancers and relieved by metformin[J]. Pharmacol Res, 2023, 188: 106661. DOI:10.1016/j.phrs.2023.106661 |
| [52] |
GRIFFITH G J, TRASK M C, HILLER J, et al. Yin-Yang1 is required in the mammalian oocyte for follicle expansion[J]. Biol Reprod, 2011, 84(4): 654-663. DOI:10.1095/biolreprod.110.087213 |
| [53] |
ZHENG P, PATEL B, MCMENAMIN M, et al. Expression of genes encoding chromatin regulatory factors in developing rhesus monkey oocytes and preimplantation stage embryos: possible roles in genome activation[J]. Biol Reprod, 2004, 70(5): 1419-1427. DOI:10.1095/biolreprod.103.023796 |
| [54] |
ZHAO X R, NIE C S, ZHANG J X, et al. Identification of candidate genomic regions for chicken egg number traits based on genome-wide association study[J]. BMC Genomics, 2021, 22(1): 610. DOI:10.1186/s12864-021-07755-3 |
| [55] |
ZHAO H X, DONG Y Z, ZHANG Y X, et al. Supplementation of SDF1 during pig oocyte in vitro maturation improves subsequent embryo development[J]. Molecules, 2022, 27(20): 6830. DOI:10.3390/molecules27206830 |
| [56] |
HU M, GAO T, DU Y. MiR-98-3p regulates ovarian granulosa cell proliferation and apoptosis in polycystic ovary syndrome by targeting YY1[J]. Med Mol Morphol, 2022, 55(1): 47-59. DOI:10.1007/s00795-021-00307-4 |
| [57] |
盛中伟, 韩威, 李国辉, 等. 文昌鸡性成熟启动过程中下丘脑EED、YY1基因表达规律分析[J]. 中国家禽, 2016, 38(13): 48-50. SHENG Z W, HAN W, LI G H, et al. Analysis of EED and YY1 gene expression in hypothalamus of Wenchang chicken during Sexual maturity initiation[J]. China Poultry, 2016, 38(13): 48-50. (in Chinese) |
| [58] |
高婷, 仲佳雯, 秦岭, 等. 基于蛋白组学技术探究六味地黄丸治疗卵巢储备减退小鼠的作用机制[J]. 中国中药杂志, 2023, 48(12): 3224-3234. GAO T, ZHONG J W, QIN L, et al. Mechanism of Liuwei Dihuang Pills in treatment of mice with diminished ovarian reserve based on proteomics[J]. China Journal of Chinese Materia Medica, 2023, 48(12): 3224-3234. DOI:10.19540/j.cnki.cjcmm.20230202.704 (in Chinese) |
| [59] |
KIM J S, CHAE J H, CHEON Y P, et al. Reciprocal localization of transcription factors YY1 and CP2c in spermatogonial stem cells and their putative roles during spermatogenesis[J]. Acta Histochem, 2016, 118(7): 685-692. DOI:10.1016/j.acthis.2016.08.005 |
| [60] |
WU S, HU Y C, LIU H F, et al. Loss of YY1 impacts the heterochromatic state and meiotic double-strand breaks during mouse spermatogenesis[J]. Mol Cell Biol, 2009, 29(23): 6245-6256. DOI:10.1128/MCB.00679-09 |
| [61] |
CHEN L G, QIAO L Y, GUO Y, et al. Localization and regulatory function of Yin Yang 1 (YY1) in chicken testis[J]. Mol Genet Genomics, 2022, 297(1): 113-123. DOI:10.1007/s00438-021-01840-8 |
| [62] |
WALLINGFORD M C, HILLER J, ZHANG K, et al. YY1 is required for posttranscriptional stability of SOX2 and OCT4 proteins[J]. Cell Reprogram, 2017, 19(4): 263-269. DOI:10.1089/cell.2017.0002 |
| [63] |
WANG J, WU X G, WEI C, et al. YY1 Positively regulates transcription by targeting promoters and super-enhancers through the BAF complex in embryonic stem cells[J]. Stem Cell Reports, 2018, 10(4): 1324-1339. DOI:10.1016/j.stemcr.2018.02.004 |
| [64] |
DONOHOE M E, ZHANG X L, MCGINNIS L, et al. Targeted disruption of mouse Yin Yang 1 transcription factor results in peri-implantation lethality[J]. Mol Cell Biol, 1999, 19(10): 7237-7244. DOI:10.1128/MCB.19.10.7237 |
| [65] |
WANG R H, SU L F, YU S T, et al. Inhibition of PHLDA2 transcription by DNA methylation and YY1 in goat placenta[J]. Gene, 2020, 739: 144512. DOI:10.1016/j.gene.2020.144512 |
| [66] |
RHEE S, GUERRERO-ZAYAS M I, WALLINGFORD M C, et al. Visceral endoderm expression of Yin-Yang1 (YY1) is required for VEGFA maintenance and yolk sac development[J]. PLoS One, 2013, 8(3): e58828. DOI:10.1371/journal.pone.0058828 |
| [67] |
ZHOU M, LIU X Q, QIUKAI E, et al. Long non-coding RNA Xist regulates oocyte loss via suppressing miR-23b-3p/miR-29a-3p maturation and upregulating STX17 in perinatal mouse ovaries[J]. Cell Death Dis, 2021, 12(6): 540. DOI:10.1038/s41419-021-03831-4 |
| [68] |
DONG Y Z, WU X, PENG X T, et al. Knockdown of YY1 inhibits XIST expression and enhances cloned pig embryo development[J]. Int J Mol Sci, 2022, 23(23): 14572. DOI:10.3390/ijms232314572 |
| [69] |
任雪婷, 龚云, 黄时海, 等. 过表达YY1对水牛早期胚胎发育及DNA甲基化的影响[J]. 中国兽医科学, 2021, 51(11): 1447-1452. REN X T, GONG Y, HANG S H, et al. Effects of overexpression YY1 on buffalo early embryonic development and DNA methylation[J]. Chinese Veterinary Science, 2021, 51(11): 1447-1452. DOI:10.16656/j.issn.1673-4696.2021.0201 (in Chinese) |
| [70] |
HAO W J, HUANG C Y, LIU D F, et al. PGC7 regulates H3K27me3 modification by inhibiting the interaction of YY1 with PRC2[J]. Am J Physiol Cell Physiol, 2023, 325(1): C286-C299. DOI:10.1152/ajpcell.00041.2023 |
| [71] |
STOCCO C, KWINTKIEWICZ J, CAI Z L. Identification of regulatory elements in the Cyp19 proximal promoter in rat luteal cells[J]. J Mol Endocrinol, 2007, 39(4): 211-221. DOI:10.1677/JME-07-0026 |
| [72] |
DU X, GUO J, CAO Q Y, et al. A haplotype variant of Hu sheep follicle-stimulating hormone receptor promoter region decreases transcriptional activity[J]. Anim Genet, 2019, 50(4): 407-411. DOI:10.1111/age.12794 |
| [73] |
AI W D, LIU Y, WANG T C. Yin yang 1 (YY1) represses histidine decarboxylase gene expression with SREBP-1a in part through an upstream Sp1 site[J]. Am J Physiol Gastrointest Liver Physiol, 2006, 290(6): G1096-G1104. DOI:10.1152/ajpgi.00199.2005 |
| [74] |
WU S Q, ZHANG L Y, DENG J Q, et al. A novel micropeptide encoded by Y-linked LINC00278 links cigarette smoking and AR signaling in male esophageal squamous cell carcinoma[J]. Cancer Res, 2020, 80(13): 2790-2803. DOI:10.1158/0008-5472.CAN-19-3440 |
| [75] |
REN G L, ZHANG G C, DONG Z X, et al. Recruitment of HDAC4 by transcription factor YY1 represses HOXB13 to affect cell growth in AR-negative prostate cancers[J]. Int J Biochem Cell Biol, 2009, 41(5): 1094-1101. DOI:10.1016/j.biocel.2008.10.015 |
| [76] |
LIU C J, QU J Y, WU M Z, et al. Cypermethrin triggers YY1-mediated testosterone biosynthesis suppression[J]. Ecotoxicol Environ Saf, 2021, 225: 112792. DOI:10.1016/j.ecoenv.2021.112792 |
(编辑 郭云雁)



