2. 福建省农业科学院畜牧兽医研究所, 福建省禽病防治重点实验室, 福州 350013
2. Animal Disease Control Technology Center, Institute of Animal Husbandry and Veterinary Medicine, Fujian Academy of Agricultural Sciences, Fuzhou 350013, China
金黄色葡萄球菌(Staphylococcus aureus, SA)是重要的条件致病菌,通过获得SCCmec元件成为耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus, MRSA),已对人类和动物健康造成了严重影响,并引起了世界范围内的广泛关注[1]。家畜是MRSA的重要贮库[2],其中,猪是家畜相关MRSA(livestock-associated MRSA, LA-MRSA)的主要宿主,欧美和中国的猪源MRSA优势克隆分别为ST398型[3]和ST9型[4],两者均通过获得大量耐药基因而表现出了不同特征的广泛耐药。噬菌体的转导是介导SA耐药性传播的重要机制之一[5]。大部分SA菌株携带有稳定整合在染色体中的前噬菌体序列[6],但各克隆谱系中前噬菌体的流行情况不尽相同[7-8],不同克隆的适应性进化和流行也可能与之相关。
CC5型SA中Sa7int前噬菌体携带率较高[7-8],ST398型LA-MRSA中不携带Sa3int和Sa4int前噬菌体,但Sa2int较为流行[9]。Sa3int前噬菌体是ST398型LA-MRSA得以在人类定殖传播的关键因素[10],其定殖于动物后丢失Sa3int前噬菌体并获得甲氧西林和四环素耐药性[11],前噬菌体影响了ST398型MRSA在人或动物定殖的适应性[12]。研究表明,SA中许多耐药基因和移动元件可通过噬菌体转导进行水平传播[13]。噬菌体φ80α和噬菌体φ29可介导Ⅰ型和Ⅳ型SCCmec元件在MRSA USA300种群中传播[14]。噬菌体φ54可包装ST398型MRSA中的SCCmec元件[15]。此外,Sa2int、Sa3int、Sa5int、Sa7int型噬菌体介导的转导事件已有报道,其中,Sa2int型噬菌体φ11和φ80α已被广泛应用于SA研究[16]。前期研究表明,ST9型MRSA株间的基因组差异主要在于前噬菌体序列,且不包含Sa3int型噬菌体所携带的人类定殖相关的毒力因子[17]。然而,我国猪源ST9型MRSA中前噬菌体的流行情况,以及耐药基因可否通过噬菌体转导进行水平传播的研究仍未见报道。
因此,本研究基于已保存的我国猪源ST9型MRSA菌株及其全基因组数据,对前噬菌体的分布特征及序列结构进行比较分析;对前噬菌体进行诱导和转导试验,测定转导子体外适应性代价。本研究旨在阐明猪源ST9型MRSA中前噬菌体的流行情况,探究其介导耐药基因水平转移的可能性,分析前噬菌体对我国猪源MRSA耐药性发展和流行克隆形成的影响。
1 材料与方法 1.1 菌株和序列菌株来源于2010—2016年在我国11省分离保存并已全基因组测序的119株猪源ST9型MRSA[17-18],所有全基因组序列均已上传NCBI网络数据库(www.ncbi.nlm.nih.gov), 并发布于基因组登录编号PRJNA487590、PRJNA433075。此外,本研究增加了NCBI数据库中12株已发布的ST9型MRSA全基因组数据用以生物信息比较分析(表 1);在亲缘关系分析中,加入了NCBI中4株不同分型的MRSA噬菌体序列(表 1)。
|
|
表 1 16个菌株及噬菌体基因组数据登录编号 Table 1 The accession No. of 16 strain and phage genomes |
TSA、LA、LB培养基购自北京陆桥生物科技有限公司,蛋白酶K、溶菌酶、溶葡萄球菌素、DNase I、RNase A、SM buffer均购自生工生物工程(上海)有限公司,柠檬酸三钠、二水合氯化钙、氯仿均购自国药集团化学试剂有限公司,聚乙二醇、丝裂霉素C购自索莱宝(北京)生物科技有限公司,病毒基因组DNA快速提取试剂盒(R4173)购自广州美基生物科技有限公司,细菌基因组DNA/RNA快速提取试剂盒(DP302)购自天根生化科技(北京)有限公司。
1.3 前噬菌序列分析1.3.1 序列预测和分型 分别使用PHASTER[19]和Prophage Hunter[20]对以上MRSA菌株基因组序列进行前噬菌体区域预测。分别以PHASTER score大于120与Prophage Hunter score大于0.9为参考,对照序列中aatL和aatR位点、溶原性模块、复制模块、转录控制模块、头尾模块的位置来确定完整前噬菌体区域。根据SA噬菌体分型方法[7],使用Blastn[21]对完整前噬菌体序列进行比对分型。
1.3.2 基因筛查和亲缘关系分析 使用Blastn对完整前噬菌体序列与数据库Resfinder[22]和VFDB[23]分别进行耐药基因和毒力基因筛查。使用Easyfig v2.2.2[24]比较不同分型前噬菌体结构。使用MAFFT[25]将提取的前噬菌体序列对齐并建立矩阵,使用Fasttree[26]进行亲缘关系分析,使用iTOL[27]进行ggtree[28],对进化树进行可视化。
1.4 前噬菌体诱导1.4.1 诱导和浓缩 参照转导试验方法[29],在诱导株的新鲜培养物(OD600 nm = 0.3~0.5) 中加入丝裂霉素C至终浓度为1 μg·mL-1,同时设立无丝裂霉素的空白对照组,置于37 ℃以转速120 r·min-1下避光培养6~8 h。待菌液明显澄清后,加入氯仿[V(氯仿)∶V(菌液)=1∶10],震荡混匀15 min。在4 ℃条件下以10 000 r·min-1离心混合液10 min,小心吸出上清液。上清液通过0.22 μm孔径滤器进行灭菌,得到噬菌体悬浮液,置于4 ℃冰箱中备用。
在噬菌体悬浮液中加入适量DNase I(100 U·mL-1) 和RNase A(1 mg·mL-1),并在37 ℃孵育30 min,加入PEG8000[V(噬菌体)∶V(PEG8000)=4∶1]混匀,并于4 ℃静置过夜后,于16 000 r·min-1离心10 min, 获得噬菌体颗粒沉淀,加1 mL SM buffer重悬混匀。重悬液中加入1/10体积氯仿抽提,并于3 000 r·min-1离心15 min,取上清液,获得浓缩后的噬菌体悬浮液。
1.4.2 宿主测定和分离纯化 参照转导试验方法[29],使用双层平板法对噬菌体的宿主范围进行测定。于恒温56 ℃下,将1 mL指示菌株的新鲜培养物(OD600 nm = 0.3~0.5)与4 mL的0.7%LB琼脂混匀,均匀倾倒在已制备好的单层TSA(或LB)琼脂平板上,制备成双层平板。将5~8 μL噬菌体液滴加于双层平板上,随后置于37 ℃条件下, 静置培养24 h。
若双层平板上存在透明噬菌斑,则受试菌株对该噬菌体敏感。从上层琼脂切下噬菌斑,重悬于1 mL SM Buffer中,于4 ℃静置过夜。用10倍体积的对数生长期宿主菌株接种噬菌体液,于37 ℃恒温摇床中培养过夜,经少量氯仿抽提和低速离心,取上清液,获得噬菌体悬浮液。重复以上步骤直至噬菌斑大小、形状无明显差异为止。选择噬菌斑数量50~100个的平板进行计数,按该平板对应噬菌体稀释比例计算噬菌体液的滴度(PFU·mL-1)。
1.4.3 形态观察 将镀碳支持膜铜网放在封口膜上,吸取1滴纯化后的噬菌体液滴于铜网上,室温静置2~5 min。用带尖的滤纸从铜网边缘吸去多余噬菌体液,干燥1 min。滴加1滴2%磷钨酸溶液(pH7.2),染色1 min。用滤纸吸去多余染液,室温静置30 min后, 置于透射电镜下观察。
1.5 转导试验1.5.1 噬菌体、供体菌和受体菌选择 如表 2所示,选择宿主范围较广的4株噬菌体作为转导试验噬菌体;根据噬菌体的共有宿主,选择MRSA菌株LN-DL44-2和BJ-PNB40作为供体菌;因工程菌株RN4220具有对大部分抗生素的敏感性、不包含质粒、对噬菌体的易感性,所以选择其作为受体菌株。
|
|
表 2 转导试验中的噬菌体、供体菌和受体菌 Table 2 Phages, donors and recipient in transduction experiment |
1.5.2 转导颗粒获得和富集 参照转导试验方法[29],将0.5 mL供体菌过夜培养物稀释至5 mL含0.01 mol·L-1 CaCl2的LB培养基中,孵育至OD600nm为0.2~0.4,加入50 μL噬菌体液(加入体积可根据噬菌体滴度适当调整,使感染复数MOI值为0.1~1),继续在37 ℃下孵育至混合液清晰透明。加入200 μL氯仿混匀抽提并16 000 r·min-1离心10 min,取上清液,获得噬菌体悬浮液。按照“1.4.2”所述测定滴度,并重复以上步骤获得109PFU·mL-1左右的噬菌体液,用0.22 μm滤器过滤并保存。
1.5.3 转导 参照转导试验方法[30],根据供体菌株耐药表型和基因型,制作含0.01 mol·L-1 CaCl2的LB单一抗性平板,抗菌药物种类及用量见表 3。将受体菌株的过夜培养物稀释于10 mL含0.01 mol·L-1 CaCl2的LB肉汤中[V(菌液)∶V(肉汤)=1∶10],置于37 ℃,以转速180 r·min-1条件下培养4 h,使菌液OD600为0.8~1,以获得高浓度且生长状态良好的受体菌。受体菌于离心机中以1 500 g离心5 min,弃上清液。加入2 mL含0.01 mol·L-1 CaCl2的LB肉汤重悬沉淀。
|
|
表 3 抗性平板药物浓度 Table 3 Concentration of antibacterial agents in resistance medium |
重悬的受体菌液分为试验组和空白对照组,分别加入500 μL转导噬菌体液和肉汤,置于37 ℃以转速200 r·min-1条件下,培养30 min,进行转导。随后,加入适量的预冷柠檬酸钠溶液终止转导,混合液中柠檬酸钠终浓度为0.02 mol·L-1,溶液pH5.5。将混合物在4 ℃ 1 000 g离心15 min。加入300 μL含0.02 mol·L-1柠檬酸钠的预冷LB肉汤重悬沉淀,将重悬菌液均匀涂布于LA抗性琼脂平板(含0.02 mol·L-1柠檬酸钠,重复3个平行,每皿100 μL菌液),置于37 ℃静置培养24~36 h。对转导子数目计数,从3个独立的重复转导试验计算出转导子数(CFU)与噬菌斑形成单位数(PFU),计算转导频率。
挑选单个转导子菌落接种于相同抗性的LA琼脂平板(含0.02 mol·L-1柠檬酸钠),置于37 ℃静置培养24~36 h以确认并纯化转导子。
1.5.4 转导噬菌体及转导子的基因检测 根据DNA提取试剂盒说明,提取转导噬菌体颗粒及转导子的基因组DNA。其中,优化后的噬菌体DNA提取步骤如下:
将20 μL蛋白酶K与200 μL BB5溶液混匀15 s,随后加入200 μL噬菌体液混匀。混合物于56 ℃静置15 min后,加入250 μL无水乙醇并涡旋混匀,室温静置15 min。将以上内容物加入离心柱,在转速12 000 r·min-1条件下离心1 min。向离心柱中加入500 μL VHB溶液,在转速12 000 r·min-1条件下离心1 min,并重复该步骤1次。将离心柱和收集管在转速12 000 r·min-1条件下离心3 min,以去除残留乙醇。在室温下,将离心柱静置15 min后,向中央滤膜上滴加50 μL dd2H2O,并静置1 min。在转速12 000 r·min-1条件下离心1 min,洗脱获得噬菌体DNA。
对细菌噬菌体、转导噬菌体、供体菌株、受体菌株、转导子分别进行已探明耐药基因的检测(表 4)。反应条件:95 ℃ 5 min;95 ℃ 30 s,52~57 ℃ 30 s,72 ℃ 1~1.5 min,30个循环;72 ℃ 5 min。
|
|
表 4 耐药基因引物 Table 4 The primers of resistant genes |
1.6.1 药敏试验 以ATCC29213为质控菌株,根据美国临床实验室标准委员会(CLSI)推荐的微量肉汤稀释法测定供、受体菌及转导子对应抗生素敏感性。
1.6.2 生长曲线 取供、受体菌株及转导子过夜培养物1 mL稀释至100 mL的LB肉汤,置于恒温37 ℃培养至菌液OD600 nm为0.5。稀释菌液1 000倍后,于全自动酶标仪中37 ℃培养24 h,每30 min测定1次OD600 nm值,并绘制生长曲线。生长曲线上每株转导子的OD600 nm值、平均值和标准方差值均经单因素方差分析(one-way ANOVA analysis)和Tukey-Kramer分析进行分组检验,以比较各菌株体外生长速率的显著性差异。
1.6.3 体外竞争试验 对供体菌株及转导子共培养,取共培养物分别于LA平板和含4 μg·mL-1头孢西丁LA平板筛选培养,并进行菌落计数。参考Nielsen等[37]的计算公式计算适应性差异。
2 结果 2.1 前噬菌体的流行特征2.1.1 前噬菌体的流行情况 序列分析结果表明,猪源ST9型MRSA的前噬菌体携带率为78.6%(103/131株),其中,63株MRSA携带结构完整的前噬菌体序列,仅4株含两条及以上完整前噬菌体序列。
根据SA噬菌体分型方法,按照不同功能模块对完整结构的前噬菌体序列进行分型。结果如图 1所示,整合酶位点分型(int)中,Sa2int和Sa4int占47.7%和30.8%;抗阻遏物位点分型(ant)中,ant5占58.5%;复制位点分型(rep)中,dnaD2b占67.7%;尿嘧啶核酸酶位点分型(dut)中,dut2占86.2%;血清学位点分型(SG)中,SGA占97%;酰胺酶位点分型(ami)中,ami1和ami2占46.2%和50.8%。因此,猪源ST9型MRSA完整前噬菌体流行分型为Sa2int/Sa4int-ant5-dnaD2b-dut2-SGA-ami1/ami2,也可简化为整合酶分型Sa2int型和Sa4int型。
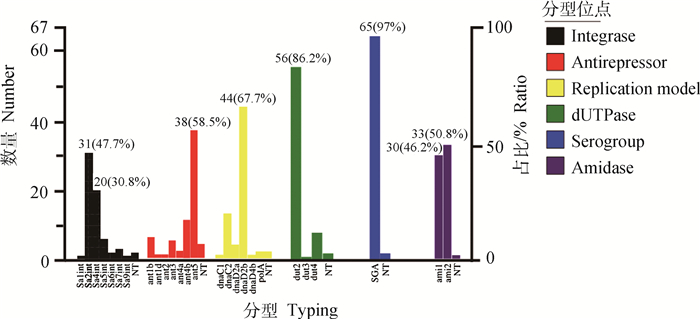
|
不同颜色代表噬菌体分型中的各相关模块 Different colors represent relevant modules in phage typing 图 1 猪源ST9型MRSA分离株前噬菌体分型 Fig. 1 The prophage typing of porcine MRSA ST9 isolates |
通过基因比对,发现所有前噬菌体序列均不含SA耐药基因,仅2.9%(3/103株)的前噬菌体序列含毒力基因。菌株GD-G39、GD-G44和SH-WF33所携带前噬菌体分别包含肠毒素基因簇seg-sei-sem-sen-seo-seu、血友病因子结合蛋白基因vwb、肠毒素基因seo。
2.1.2 前噬菌体的亲缘关系和结构特征 本研究对所有完整前噬菌体序列进行了亲缘关系分析。如图 2所示,前噬菌体序列被分为两个亲缘关系较远的进化枝,Clade-A和Clade-B间的平均核苷酸差异为21.5%。两个分支的组间、组内的前噬菌体分型、基因组长度和基因数量无明显相关。但来源于ST22型SA的前噬菌体φtp310-2、来源于ST80型SA的前噬菌体φ7410PVL与来源于ST398型SA的φstauST398-2均聚集于Clade-A,且同源性较高。

|
左侧标签分别代表噬菌体的整合酶位点分型、抗阻遏物位点分型、复制位点分型、尿嘧啶核酸酶位点分型、酰胺酶位点分型、血清学位点分型;右侧柱状图分别代表噬菌体的大小(蓝)和基因数量(绿);红色虚线和绿色虚线分别表示Clade-A和Clade-B株;灰色阴影区域代表除Sa2int和Sa4int前噬菌体外其他分型噬菌体 The left labels represent the integrase type, antirepressor type, replication module type, dUTPase type, amidase type and serological type of bacteriophage respectively; The right histogram represents the phage size (blue) and gene number (green) respectively; The red dotted line and the green dotted line indicate Clade-A and Clade-B strains respectively; The gray shaded area represents other phenotypic phages except for Sa2int and Sa4int prophages 图 2 猪源ST9型MRSA分离株完整前噬菌体亲缘关系 Fig. 2 The phylogeny of intact prophages in porcine MRSA ST9 isolates |
为探究ST9型MRSA前噬菌体的结构特征,对不同int分型前噬菌体进行了结构比较。如图 3所示,与噬菌体φStauST398-2(Sa2int)相比,φGD-G44(Sa1int)、φQD-CD9(Sa4int)和φGD-G51 (Sa5int)分别具有52%、65%和60%的相似度,φNX-T44(Sa2int)、φSD-YI040(Sa6int)、φLN-DL44-2(Sa7int)及φBA01611-1(Sa9int)具有较高同源性。所有前噬菌体均享有一段保守的核心功能区域:包装模块、形态模块和裂解模块,据此推测噬菌体介导转导的过程中存在非保守区域的基因交换。
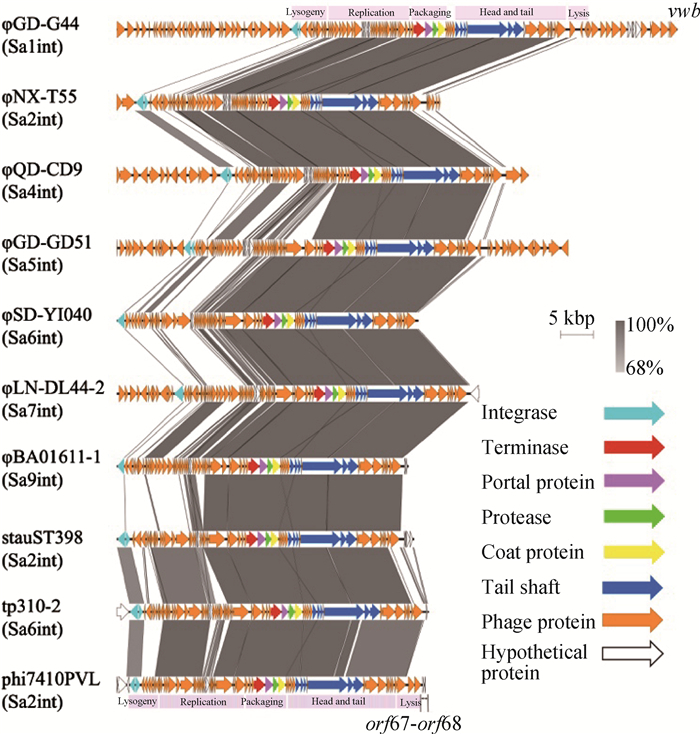
|
各色箭头代表编码不同噬菌体结构蛋白的基因;阴影部分代表不同噬菌体之间的序列相似度 The colored arrows represent the genes encoding different phage structural proteins; The shaded parts represent the sequence similarity among different bacteriophages 图 3 各整合酶分型前噬菌体基因结构比较 Fig. 3 Genomic structure comparison among prophages with each integrase typing |
对挑选的60株含完整前噬菌体的菌株进行诱导试验,结果表明,58株可被成功诱导为噬菌体,其中Sa2int型21株,Sa4int型噬菌体22株,其余分型的15株。所有噬菌体均在双层平板上呈现出直径约1 mm大小相似、形状规则的噬菌斑。以所有保存菌株作为测试菌株,进行噬菌体宿主范围测定。结果表明,所有噬菌体株宿主范围差异较大。挑选其中4株宿主范围较广的噬菌体株φQD-J1、φSD-PW88、φGD-G14、φGD-G51进行形态观察及转导试验。
电镜结果(图 4)显示,4株噬菌体均具六边形外廓头部,长度为75~100 nm;尾部较长,为200~300 nm。头尾轴向两端及尾部被磷钨酸深着色,富含双链DNA。根据SA噬菌体分类标准及国际病毒分类方法[38],这4株噬菌体可被归类于有尾噬菌体目(Caudovirales)长尾噬菌体科(Siphoviridae)。

|
图A~D分别代表φQD-J1、φSD-PW88、φGD-G14、φGD-G51电镜下形态 Figures A to D represent φQD-J1, φSD-PW88, φGD-G14, φGD-G51 morphology under electron microscope 图 4 透射电镜下的各噬菌体形态 Fig. 4 Morphology of each phage isolate under transmission electron micrograph |
转导试验结果表明,在MOI为0.5~1.0时,4株噬菌体均可获得供体菌LN-DL44-2中基因aadD、tet(L)和ccrC2,其中耐药基因aadD和tet(L)可被成功转导至受体菌株RN4220(图 5)。aadD和tet(L)分别介导RN4220对卡那霉素和四环素的耐药性。然而,以菌株BJ-PNB40为供体获得的转导噬菌体则不携带任何耐药基因及功能基因。4组噬菌体的转导频率较低,但Sa4int型噬菌体φQD-J1和φSD-PW88的转导频率较其他噬菌体略高(表 5)。
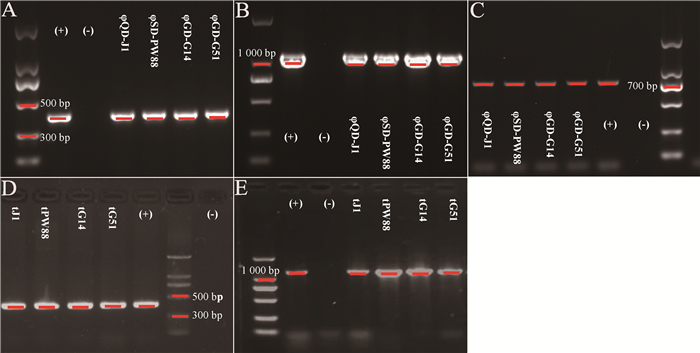
|
图A~C分别代表各转导噬菌体颗粒中携带aadD、tet(L)和ccrC2的基因检测结果,图D~E分别代表各转导子中携带aadD和tet(L)的基因检测结果 Figures A to C respectively represent the detection results of genes carrying aadD, tet (L) and ccrC2 in each transducing phage particle, and Figures D to E respectively represent the detection results of genes carrying aadD and tet(L) in each transductant 图 5 转导噬菌体颗粒和转导子中耐药基因检测结果 Fig. 5 The results of resistance genes detection in transducing phage particles and transductants |
|
|
表 5 转导试验结果 Table 5 The results of transduction experiment |
2.4.1 转导子的耐药性 药敏试验可知,相比于RN4220,4株噬菌体对应转导子tJ1、tPW88、tG14、tG51对卡那霉素MIC提高128倍~256倍,对四环素MIC提高32倍~64倍。
2.4.2 转导子的体外生长曲线 生长曲线如图 6所示。经统计学检验,4株转导子的体外生长速率分别与RN4220并无显著差异(P>0.05), 但所有转导子以及RN4220的生长速率均明显高于供体菌LN-DL44-2。
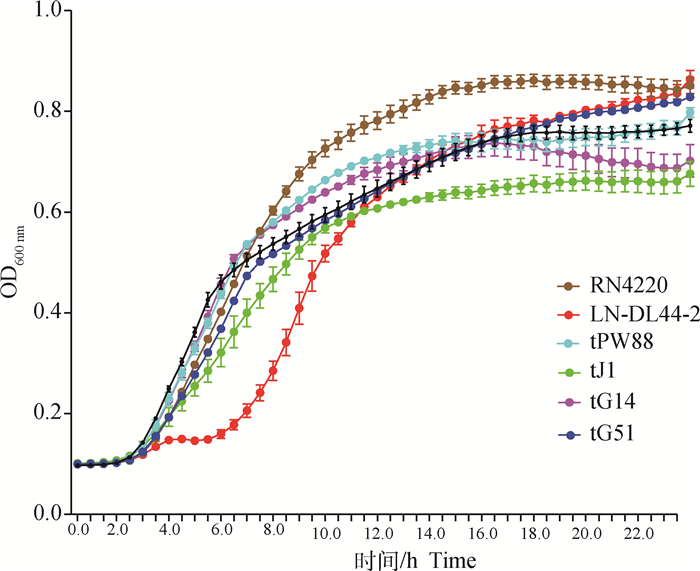
|
图 6 供体、受体菌株以及转导子的体外生长曲线 Fig. 6 In vitro growth curves of donor, recipient strains and transductants |
2.4.3 转导子的适应性代价 体外竞争性试验结果如表 6所示,RN4220与各转导子相比均显示出了较小的竞争性优势,各转导子的适应性代价(C)均在2%以内。
|
|
表 6 转导子的适应性代价 Table 6 The fitness cost of transductants |
本研究基于细菌全基因组序列,对猪源ST9型MRSA中前噬菌体进行了预测,对完整前噬菌体序列进行分型。在猪源ST9型MRSA的基因组中,前噬菌体的携带率达78.6%,符合SA菌株中前噬菌体的流行情况[7]。63株ST9型MRSA携带完整前噬菌体,由于超感染外排机制[39],其中,大部分菌株仅含1条完整前噬菌体序列。噬菌体的分型方法中,最保守的是整合酶基因(int)类型,与形态模块对应的血清学类型(SG)和酰胺酶类型(ami)。因此,我国猪源ST9型MRSA前噬菌体分型也可描述为Sa2int/Sa4int-SGA-ami1/ami2。本研究中,Sa2int和Sa4int型前噬菌体在ST9型MRSA基因组中为优势谱型,且所有菌株均不携带Sa3int前噬菌体。该流行情况与LA-MRSA ST398相似[9],同样缺失Sa3int前噬菌体。Sa2int前噬菌体通常携带编码杀白细胞介素的基因luk[7-8],但猪源ST9型MRSA中Sa2int前噬菌体却完全缺失luk。因此,Sa3int前噬菌体的丢失、前噬菌体中毒力基因的缺失可能对猪源ST9型MRSA在我国流行产生了一定程度的影响。我国猪源ST9型MRSA前噬菌体呈现出分型的多样性和较大的基因差异。噬菌体进化理论[40]指出,前噬菌体通过功能基因(模块)间交换而进化。SA前噬菌体具有3个特征导致其个体间差异:首先,SA噬菌体基因组中存在的镶嵌现象,使基因片段在模块间和模块内更加动态;第二,在各模块功能的基因之间插入了外源ORF;第三,噬菌体个体的特定基因、模块缺失,例如缺少ant基因。
噬菌体宿主范围和形态学鉴定是进行转导试验的必要前置步骤。本研究中各噬菌体宿主范围无明显规律。迄今,关于不同型别SA噬菌体宿主范围的相关研究未见报道。宿主范围的差异可能是因为SA噬菌体对宿主的裂解和转导被限制在亲缘关系相近的菌株间发生[41]。菌株对温和噬菌体不敏感的常见原因是超感染外排机制[39],SA在已有噬菌体整合至基因组中的情况下,对外源噬菌体保持一定的抵抗作用[41],超感染外排机制,可能是本研究中转导频率较低的原因。因此,噬菌体的宿主范围可能决定了其转导的成功与否。本研究中,鉴定了4株SA噬菌体。迄今为止,所有已知的SA噬菌体均属于有尾噬菌体目(Caudovirales)长尾噬菌体科(Siphoviridae)。本研究中的4株噬菌体均为血清学SGA型。研究已证明了SGA型噬菌体具有转导能力[42],并且也有SGA噬菌体成功转导的报道[43]。此外,细胞壁WTA表面受体是噬菌体转导的途径之一[44]。但由于宿主范围限制及RN4220更易接受外源DNA的特性,本研究中仅使用WTA蛋白缺陷的RN4220作为受体,这可能同样导致本研究中转导频率较低。
转导试验表明,氨基糖苷类耐药基因aadD和四环素类耐药基因tet(L)被噬菌体包装并转导至受体菌内。MIC结果与供体菌有所差异,可能是由于转导子中耐药基因侧翼序列的差异性而导致表达量的变化。噬菌体转导可能是aadD和tet(L)在ST9菌株中广泛分布的原因之一。SA中噬菌体可转导位于染色体序列上的基因,也同样可转导小型质粒[45]。本研究中噬菌体是包装含有耐药基因质粒或者是染色体上耐药基因而发生了转导,该过程不得而知,仍需后续深入研究。在转导噬菌体颗粒中检测到SCCmec-XII元件中的ccrC2基因,这表明噬菌体可能参与了SCCmec-XII的菌株间水平转移,可为ST9型MRSA中SCCmec-XII元件的广泛分布提供了一种合理的可能性。而噬菌体未包装SCCmec-XII的其它组分的原因尚不清楚。其原因可能是因为:1)SCCmec-XII的att序列不同于噬菌体中的att序列而无法同源重组;2)噬菌体中att序列被SCCmec-XII中的部分基团,例如ccrC2,所占据而阻止其它部分的包装;3)整合的过程属于同源重组并且需要特定活性重组酶,该种重组酶可能在供体菌株中不存在;4)SCCmec-XII序列相对较大,完整包装进噬菌体的难度较高。SA前噬菌体的基因组大小为30~50 kb[46], 而SCCmec-XII长度约为60 kb。研究表明,完整SCCmec元件的转导事件中,其SCCmec长度均 < 40 kb[14]。因此,噬菌体可能分别包装SCCmec元件中的部分基因而转导。
由生长曲线和竞争性试验结果显示,相比于受体菌,转导子几乎不产生适应性代价。与供体菌相比,在无抗条件下转导子仍保持着竞争性优势。细菌获得耐药性可引起某些生物学方面的损害,表现为生长速率降低[47]。而细菌往往对基因组改变导致的适应性降低会产生对应补偿机制。噬菌体整合对于ST9菌株成为优势克隆的对应机制仍未有深入研究。
4 结论我国猪源ST9型MRSA中前噬菌体流行率较高,均不携带耐药基因,少数携带毒力基因,系统发育关系上分为两个分支,主要分型为Sa2int和Sa4int型。ST9型MRSA的前噬菌体可被诱导为长尾噬菌体,可包装ccrC2基因至转导噬菌体颗粒,可包装且转导耐药基因aadD和tet(L)至受体菌,各型噬菌体转导频率无明显差异。转导子获得卡那霉素和四环素耐药性且几乎不产生适应性代价。
| [1] |
LAKHUNDI S, ZHANG K Y. Methicillin-resistant Staphylococcus aureus: molecular characterization, evolution, and epidemiology[J]. Clin Microbiol Rev, 2018, 31(4): e00020-18. |
| [2] |
VERKADE E, KLUYTMANS J. Livestock-associated Staphylococcus aureus CC398:Animal reservoirs and human infections[J]. Infect Genet Evol, 2014, 21: 523-530. DOI:10.1016/j.meegid.2013.02.013 |
| [3] |
VOSS A, LOEFFEN F, BAKKER J, et al. Methicillin-resistant Staphylococcus aureus in pig farming[J]. Emerg Infect Dis, 2005, 11(12): 1965-1966. DOI:10.3201/eid1112.050428 |
| [4] |
CUI S H, LI J Y, HU C Q, et al. Isolation and characterization of methicillin-resistant Staphylococcus aureus from swine and workers in China[J]. J Antimicrob Chemother, 2009, 64(4): 680-683. DOI:10.1093/jac/dkp275 |
| [5] |
LINDSAY J A. Staphylococcus aureus genomics and the impact of horizontal gene transfer[J]. Int J Med Microbiol, 2014, 304(2): 103-109. DOI:10.1016/j.ijmm.2013.11.010 |
| [6] |
LINDSAY J A, HOLDEN M T G. Staphylococcus aureus: Superbug, super genome?[J]. Trends Microbiol, 2004, 12(8): 378-385. DOI:10.1016/j.tim.2004.06.004 |
| [7] |
KAHÁNKOVÁ J, PANTǓČEK R, GOERKE C, et al. Multilocus PCR typing strategy for differentiation of Staphylococcus aureus siphoviruses reflecting their modular genome structure[J]. Environ Microbiol, 2010, 12(9): 2527-2538. DOI:10.1111/j.1462-2920.2010.02226.x |
| [8] |
GOERKE C, PANTUCEK R, HOLTFRETER S, et al. Diversity of prophages in dominant Staphylococcus aureus clonal lineages[J]. J Bacteriol, 2009, 191(11): 3462-3468. DOI:10.1128/JB.01804-08 |
| [9] |
PRICE L B, STEGGER M, HASMAN H, et al. Staphylococcus aureus CC398: Host adaptation and emergence of methicillin resistance in livestock[J]. mBio, 2012, 3(1): e00305-11. |
| [10] |
DIENE S M, CORVAGLIA A R, FRANÇOIS P, et al. Prophages and adaptation of Staphylococcus aureus ST398 to the human clinic[J]. BMC Genomics, 2017, 18(1): 133. DOI:10.1186/s12864-017-3516-x |
| [11] |
UHLEMANN A C, PORCELLA S F, TRIVEDI S, et al. Identification of a highly transmissible animal-independent Staphylococcus aureus ST398 clone with distinct genomic and cell adhesion properties[J]. mbio, 2012, 3(2): e00027-12. |
| [12] |
KRAUSHAAR B, HAMMERL J A, KIENÖL M, et al. Acquisition of virulence factors in livestock-associated MRSA: Lysogenic conversion of CC398 strains by virulence gene-containing phages[J]. Sci Rep, 2017, 7(1): 2004. DOI:10.1038/s41598-017-02175-4 |
| [13] |
UBELAKER M H, ROSENBLUM E D. Transduction of plasmid determinants in Staphylococcus aureus and Escherichia coli[J]. J Bacteriol, 1978, 133(2): 699-707. DOI:10.1128/jb.133.2.699-707.1978 |
| [14] |
SCHARN C R, TENOVER F C, GOERING R V. Transduction of staphylococcal cassette chromosome mec elements between strains of Staphylococcus aureus[J]. Antimicrob Agents Chemother, 2013, 57(11): 5233-5238. DOI:10.1128/AAC.01058-13 |
| [15] |
LEE S M, ENDER M, ADHIKARI R, et al. Fitness cost of staphylococcal cassette chromosome mec in methicillin-resistant Staphylococcus aureus by way of continuous culture[J]. Antimicrob Agents Chemother, 2007, 51(4): 1497-1499. DOI:10.1128/AAC.01239-06 |
| [16] |
MASLANOVA I, STRIBNA S, DOSKAR J, et al. Efficient plasmid transduction to Staphylococcus aureus strains insensitive to the lytic action of transducing phage[J]. FEMS Microbiol Lett, 2016, 363(19): fnw211. DOI:10.1093/femsle/fnw211 |
| [17] |
JIANG N S, WYRES K L, LI J, et al. Evolution and genomic insight into methicillin-resistant Staphylococcus aureus ST9 in China[J]. J Antimicrob Chemother, 2021, 76(7): 1703-1711. DOI:10.1093/jac/dkab106 |
| [18] |
SUN C T, CHEN B L, HULTH A, et al. Genomic analysis of Staphylococcus aureus along a pork production chain and in the community, Shandong Province, China[J]. Int J Antimicrob Agents, 2019, 54(1): 8-15. DOI:10.1016/j.ijantimicag.2019.03.022 |
| [19] |
ARNDT D, GRANT J R, MARCU A, et al. PHASTER: a better, faster version of the PHAST phage search tool[J]. Nucleic Acids Res, 2016, 44(W1): W16-W21. DOI:10.1093/nar/gkw387 |
| [20] |
SONG W C, SUN H X, ZHANG C, et al. Prophage Hunter: an integrative hunting tool for active prophages[J]. Nucleic Acids Res, 2019, 47(W1): W74-W80. DOI:10.1093/nar/gkz380 |
| [21] |
CAMACHO C, COULOURIS G, AVAGYAN V, et al. BLAST+: architecture and applications[J]. BMC Bioinformatics, 2009, 10: 421. DOI:10.1186/1471-2105-10-421 |
| [22] |
BORTOLAIA V, KAAS R S, RUPPE E, et al. ResFinder 4.0 for predictions of phenotypes from genotypes[J]. J Antimicrob Chemother, 2020, 75(12): 3491-3500. DOI:10.1093/jac/dkaa345 |
| [23] |
LIU B, ZHENG D D, ZHOU S Y, et al. VFDB 2022: a general classification scheme for bacterial virulence factors[J]. Nucleic Acids Res, 2022, 50(D1): D912-D917. DOI:10.1093/nar/gkab1107 |
| [24] |
SULLIVAN M J, PETTY N K, BEATSON S A. Easyfig: va genome comparison visualizer[J]. Bioinformatics, 2011, 27(7): 1009-1010. DOI:10.1093/bioinformatics/btr039 |
| [25] |
KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability[J]. Mol Biol Evol, 2013, 30(4): 772-780. DOI:10.1093/molbev/mst010 |
| [26] |
PRICE M N, DEHAL P S, ARKIN A P. FastTree: computing large minimum evolution trees with profiles instead of a distance matrix[J]. Mol Biol Evol, 2009, 26(7): 1641-1650. DOI:10.1093/molbev/msp077 |
| [27] |
LETUNIC I, BORK P. Interactive Tree Of Life (iTOL) v4: recent updates and new developments[J]. Nucleic Acids Res, 2019, 47(W1): W256-W259. DOI:10.1093/nar/gkz239 |
| [28] |
YU G C, LAM T T Y, ZHU H C, et al. Two methods for mapping and visualizing associated data on phylogeny using ggtree[J]. Mol Biol Evol, 2018, 35(12): 3041-3043. DOI:10.1093/molbev/msy194 |
| [29] |
KRAUSZ K L, BOSE J L. Bacteriophage transduction in Staphylococcus aureus: Broth-based method[J]. Methods Mol Biol, 2016, 1373: 63-68. |
| [30] |
OLSON M E. Bacteriophage transduction in Staphylococcus aureus[J]. Methods Mol Biol, 2016, 1373: 69-74. |
| [31] |
汪宇. 猪源MRSA和多药耐药基因cfr的流行情况调查和传播机制研究[D]. 北京: 中国农业大学, 2012. WANG Y. Study on the prevalence and mechanism of dissemination on MRSA and multidrug resistant gene cfr from swine[D]. China Agricultural University, 2012. (in Chinese) |
| [32] |
WU Z W, LI F, LIU D L, et al. Novel type XⅡ staphylococcal cassette chromosome mec harboring a new cassette chromosome recombinase, ccrC2[J]. Antimicrob Agents Chemother, 2015, 59(12): 7597-7601. DOI:10.1128/AAC.01692-15 |
| [33] |
李君. 猪源耐甲氧西林金黄色葡萄球菌流行病学特征及遗传进化分析[D]. 北京: 中国农业大学, 2017. LI J. Epidemiological characteristics and phylogenetic analysis of pig associated Methicillin-resistant Staphylococcus aureus[D]. Beijing: China Agricultural University, 2017. (in Chinese) |
| [34] |
CHEN C J, HSUEH P R, SU L H, et al. Change in the molecular epidemiology of methicillin-resistant Staphylococcus aureus bloodstream infections in Taiwan[J]. Diagn Microbiol Infect Dis, 2009, 65(2): 199-201. DOI:10.1016/j.diagmicrobio.2009.05.020 |
| [35] |
PAN E S, DIEP B A, CHARLEBOIS E D, et al. Population dynamics of nasal strains of methicillin-resistant Staphylococcus aureus and their relation to community-associated disease activity[J]. J Infect Dis, 2005, 192(5): 811-818. DOI:10.1086/432072 |
| [36] |
李德喜. 恶唑烷酮类耐药基因cfr和optrA在猪源MRSA和CoNS中流行及传播机制的研究[D]. 北京: 中国农业大学, 2016. LI D X. The epidemiological study on the oxazolidinone resistance genes cfr and optrA and theirs transmission mechanism among MRSA and CoNS isolates from swine[D]. Beijing: China Agricultural University, 2012. (in Chinese) |
| [37] |
NIELSEN K L, PEDERSEN T M, UDEKWU K I, et al. Fitness cost: a bacteriological explanation for the demise of the first international methicillin-resistant Staphylococcus aureus epidemic[J]. J Antimicrob Chemother, 2012, 67(6): 1325-1332. DOI:10.1093/jac/dks051 |
| [38] |
KWAN T, LIU J, DUBOW M, et al. The complete genomes and proteomes of 27 Staphylococcus aureus bacteriophages[J]. Proc Natl Acad Sci U S A, 2005, 102(14): 5174-5179. DOI:10.1073/pnas.0501140102 |
| [39] |
LABRIE S J, SAMSON J E, MOINEAU S. Bacteriophage resistance mechanisms[J]. Nat Rev Microbiol, 2010, 8(5): 317-327. DOI:10.1038/nrmicro2315 |
| [40] |
CANCHAYA C, PROUX C, FOURNOUS G, et al. Prophage genomics[J]. Microbiol Mol Biol Rev, 2003, 67(2): 238-276. DOI:10.1128/MMBR.67.2.238-276.2003 |
| [41] |
MCCARTHY A J, WITNEY A A, LINDSAY J A. Staphylococcus aureus temperate bacteriophage: Carriage and horizontal gene transfer is lineage associated[J]. Front Cell Infect Microbiol, 2012, 2: 6. |
| [42] |
DEGHORAIN M, VAN MELDEREN L. The Staphylococci phages family: an overview[J]. Viruses, 2012, 4(12): 3316-3335. DOI:10.3390/v4123316 |
| [43] |
WINSTEL V, LIANG C G, SANCHEZ-CARBALLO P, et al. Wall teichoic acid structure governs horizontal gene transfer between major bacterial pathogens[J]. Nat Commun, 2013, 4: 2345. DOI:10.1038/ncomms3345 |
| [44] |
XIA G Q, CORRIGAN R M, WINSTEL V, et al. Wall teichoic Acid-dependent adsorption of staphylococcal siphovirus and myovirus[J]. J Bacteriol, 2011, 193(15): 4006-4009. DOI:10.1128/JB.01412-10 |
| [45] |
TANG Y Y, NIELSEN L N, HVITVED A, et al. Commercial biocides induce transfer of prophage Φ13 from human strains of Staphylococcus aureus to livestock CC398[J]. Front Microbiol, 2017, 8: 2418. DOI:10.3389/fmicb.2017.02418 |
| [46] |
XIA G Q, WOLZ C. Phages of Staphylococcus aureus and their impact on host evolution[J]. Infect Genet Evol, 2014, 21: 593-601. DOI:10.1016/j.meegid.2013.04.022 |
| [47] |
MELNYK A H, WONG A, KASSEN R. The fitness costs of antibiotic resistance mutations[J]. Evol Appl, 2015, 8(3): 273-283. DOI:10.1111/eva.12196 |
(编辑 白永平)



