2. 中国农业大学动物科学技术学院, 北京 100193
2. College of Animal Science and Technology, China Agricultural University, Beijing 100193, China
猪RNA病毒的模板有正链病毒RNA模板、负链病毒RNA模板和全长正负链反基因组RNA模板,即双链RNA(double-stranded RNA,dsRNA)模板[1-2]。dsRNA作为单链RNA5 OAS)/ RNaseL系统[3-6]所识别,并诱导Ⅰ型干扰素(IFN-α / β)的产生。Poly I: C(Polyinosinic:polycytidylic acid)为多聚肌苷酸Poly(I)和多聚胞苷酸Poly(C)的共聚物,通常用于模拟病毒的复制中间体双链RNA(dsRNA)。通过合成不同长度的Poly I: C,研究dsRNA作为病毒或者免疫刺激剂转染宿主细胞的分子免疫机制。Liu等[7]认为短片段的polymer(~300 bp)不会激活MDA-5,但它是RIG-I潜在的配体。相反的,长片段的Poly I: C更偏好激活MDA-5[8],长度≥63 kb的PolyI: C通常用于研究RNA sensing。但关于dsRNA转染猪细胞的全基因组甲基化机制仍没有被研究。
全基因组甲基化是近些年发展较快的新兴学科,就像分子生物学的发展由单个基因进入全基因组的研究一样,DNA甲基化也由对少数位点的修饰分析进入了全基因组水平的“全景式”研究阶段[9]。DNA甲基化与人类发育和肿瘤等疾病密切相关,特别是CpG岛高甲基化导致抑癌基因转录失活。近些年,畜禽重要经济性状的DNA甲基化已经成为表观基因组学的重要研究内容之一[10-12]。差异甲基化区域(differential methylation region,DMR)是指在不同组织类型或疾病状态下的细胞水平上发生的差异甲基化区域[13-14]。2019年,Wang等[10]解析了PolyI: C结合Aza-CdR共同转染猪肾细胞的甲基化图谱,定量分析DMR和甲基化标记,揭示病毒模拟物Poly I: C对甲基化抑制剂Aza-CdR具有潜在的抑制作用,这些结果将提高人类对哺乳动物疾病的了解和诊断具有重要的实用价值。全基因组DNA甲基化图谱通过分析试验组与对照组的DMR,以寻找重要的表观遗传标记(epigenetic marker),这将有助于了解正常和疾病状态下不同基因相互作用的网络调控关系[13],不仅可为病毒感染、肿瘤和糖尿病等复杂疾病的深入研究提供新的理论依据,还可为环境因素、营养和衰老等研究提供新的方法。
DNA甲基化转移酶抑制剂5-氮脱氧胞苷[5-aza-2′-deoxycytidine(脱氧胞核嘧啶),Aza-CdR],具有很强的甲基化抑制作用,在细胞培养中可以介导细胞分化和基因表达[15]。含Aza-CdR的DNA与甲基化酶形成的稳定共价复合体使甲基基团不能转移到胞嘧啶,导致DNA甲基化反应受阻,从而使基因组甲基化水平降低。有研究表明,Aza-CdR会抑制DNA的复制、转录,还可能在哺乳动物细胞内诱发基因表达变化、细胞程序性死亡等,也可以抑制病毒RNA成熟所需要的甲基化反应。Aza-CdR常用于两个领域:1)在DNA甲基化抑制基因表达的生物功能试验中做验证对照,进而深入挖掘DNA甲基化标记;2)用于治疗某些特异的肿瘤。2019年,Chen等[15]利用5-Aza-CdR抑制DNMT1表达,增加肠癌细胞凋亡的速率。2015年,Roulois等[16]证明低剂量5-Aza-CdR可通过诱导病毒模拟物靶向应对结直肠癌启动细胞(CICs),这与部分源自内源性逆转录病毒元件的dsRNA诱导、MDA5/MAVS RNA识别通路的激活以及IRF7的下游激活有关。2009年,Koga等[17]利用MeDIP-chip方法鉴定5个新的人类黑色素瘤甲基化标记,并结合Aza-CdR进行验证。基于此,本研究利用Poly I: C和Aza-CdR转染猪肾上皮细胞系PK15,模拟dsRNA病毒的感染及抗病毒作用。通过与非转染的PK15细胞进行对比,分析猪肾细胞全基因组差异甲基化基因并筛选抗病毒相关通路,为肾感染的病毒性疾病研究提供分子依据,进而揭示Aza-CdR和dsRNA对猪肾细胞的甲基化效应。
1 材料与方法 1.1 试验用细胞及MeDIP-chip芯片设计以猪肾上皮细胞系PK15(购自中国武汉典型培养物保藏中心,细胞编号:3115CNCB00260)为试验材料,10%热灭活胎牛血清、100单位·mL-1青链霉素37 ℃、5% CO2条件下培养(具体培养方法参考文献[4])。当细胞生长至70%~80%时,饥饿培养2 h,以达到细胞平衡状态。使用Poly I: C(P)和Aza-CdR(A)转染PK15细胞,收集处理的细胞,进行DNA甲基化芯片检测和分析。共3组细胞:1)Mock细胞(C组);2)10 μg·mL-1 PolyI: C转染10 h的PK15细胞(P组);3)5 μmol·L-1 Aza-CdR转染6 h的PK15细胞(A组)。细胞的时间和剂量依赖试验结果见参考文献[18]。每组3个重复,共计9个样本,用于MeDIP-chip芯片检测。9张芯片来源于Nimbgen公司设计的Pig 385 K CpG Island plus Ensembl Promoter Array Methylation Chip,最终完成本试验猪DNA甲基化差异区域的注释。
1.2 PK15细胞DNA的抽提使用Wizard基因组DNA纯化试剂盒(Promega)提取细胞基因组DNA。利用NanoDropTM ND-2000c分光光度计测定DNA浓度后,再使用EZ DNA甲基化金标准试剂盒(Zymo Research)对每个样本1 μg基因组DNA进行重亚硫酸盐转化。用20 μL缓冲液洗脱转化的DNA用于免疫共沉淀。
1.3 DNA免疫共沉淀和芯片杂交基因组DNA经超声波破碎后,样本用于免疫共沉淀试验。每个样本的免疫共沉淀处理分为3份,第1份是Input DNA,第2份是MeDIP DNA,第3份是Mock IP DNA。分别对9张MeDIP芯片进行qPCR质检。1)Input试验:加入0.4 μg样本基因组DNA,不加任何抗体;2)MeDIP试验:加入2 μg样本基因组DNA和5 μg样本甲基化DNA抗体(抗体来源于猪Anti-5-methylcytidine antibody,Diagenode);3)Mock试验:加入5 μg的小鼠IgG和2 μg的样本DNA(小鼠IgG不能和样本DNA发生作用)。MeDIP DNA和Input DNA分别用Cy 5和Cy 3标记,检测合格后, 与猪385 K全基因组CpG岛(CGI)和启动子芯片进行杂交。
1.4 原始数据校正免疫共沉淀的样本经芯片杂交后得到原始数据(raw data),原始数据包括每个探针的荧光信号强度,这些数据以log2(MeDIP / Input)(即log2 ratio)及P value来表示。log2 ratio即MeDIP DNA的荧光信号和Input DNA的荧光信号比值的对数值。为了避免技术因素对试验结果的影响,以便更真实地反映样品间DNA甲基化的差异,试验对芯片得到的原始log2 ratio进行校正。利用中位数中心和分位数校正(median-centering and quantile normalization)方法对试验组中每个样品的log2 ratio值进行校正,经过校正后所有芯片数据的中值都在0左右,并且样本间在上下区间的分布也趋于相似,表明数据可用于后续分析。
1.5 芯片数据分析由NimbleScan v2.6(Roche NimbleGen,05933315001)计算log2ratio值。log2 ratio值代表每个探针在MeDIP DNA和Input DNA中的富集强度,P值代表每个探针的红绿信号差异是由非生物因素造成的概率,P值由修正的KS检验算法计算。P值越低,表示探针越有可能代表一个甲基化事件,进而找到富集的甲基化峰(enrichment peaks,EPs)。Peaks代表可能的DNA甲基化区域,由专业商用软件SignalMapv1.9(Roche NimbleGen,05225051001)计算。
1.6 差异甲基化区域计算多样本之间甲基化区域的比较由差异甲基化峰(differential enrichment peaks,DEPs)表示,通过两两组别之间的差异值log2 ratio计算M值,M值(也称作Peak DM value值)是指组间的差异甲基化峰值(DEPs),可代表两组之间的差异甲基化区域(DMR)。计算方法:M=Average(log2 MeDIP E / Input E)- Average(log2 MeDIP C / Input C), 其中,E代表试验组,C代表对照组。根据NimbleScan Algorithm判定DEPs时有两个条件:1)两组中至少有一组log2(MeDIP / Input) > 0.3和M > 0;2)在一个Peak中,两组中至少有一半的探针变异系数(coefficient variability,CV)≤0.8。本研究分析了PolyI: C vs. Control(P vs. C)和Aza-CdR vs. Control(A vs. C)两个比较组间的差异甲基化区域分布特征。
1.7 DNA bisulfite PCR和Bisifite clone sequencing应用Oligo 6.0软件设计目的基因和持家基因引物,使用Zymo Premix启动PCR热循环。PCR热循环条件:95 ℃ 10 min;94 ℃ 30 s,55 ℃ 45 s,72 ℃ 45 s,40~45循环;72 ℃ 10 min。PCR产物用于后续测序试验。Bisifite clone sequencing前试验主要包括PCR产物切胶回收,感受态细胞制备(CaCl2法),载体连接反应,质粒DNA转化。PCR产物克隆至pGM-T载体,使用ABI 377自动测序仪完成测序反应。挑取8~10个克隆用于Bisulfite克隆测序分析。
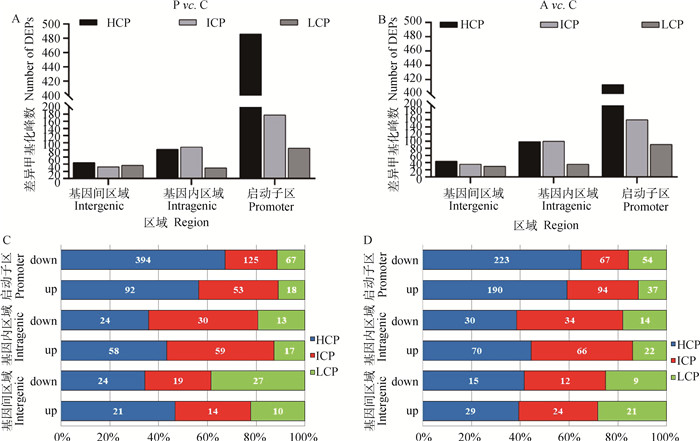
2 结果 2.1 Poly I: C和Aza-CdR转染猪PK15细胞的全基因组差异甲基化峰分布试验分析了两个比较组P vs. C和A vs. C基因间区域(intergenic)、基因内区域(introgenic)和启动子区(promoter)3个区域的差异甲基化峰(DEPs)分布。正常的猪肾上皮细胞在全基因组区域(intergenic、introgenic、promoter)均有DEPs分布,而Poly I: C(图 1A)和Aza-CdR(图 1B)分别转染PK15细胞的DEPs则主要在高密度CpG含量启动子(high-CpG-containing promoters,HCPs)增加显著,中等密度CpG含量启动子(inter mediate-CpG-containing promoters,ICPs)和低密度CpG含量启动子(low-CpG-containing promoters,LCPs)并没有显著增加。结果说明,两种化学物质处理细胞后,DNA甲基化的变化主要发生在启动子位置,特别是与启动子甲基化的密度相关联。
|
A. Poly I: C转染猪PK15细胞的差异甲基化峰(DEPs)数; B. Aza-CdR转染猪PK15细胞的DEPs数;C. Poly I: C转染猪PK15细胞的DEPs分布; D. Aza-CdR转染猪PK15细胞的DEPs分布 A. The number of differential methylation peaks (DEPs) of Poly I: C transfected porcine PK15 cells; B. The number of DEPs of porcine PK15 cells transfected with Aza-CdR; C. DEPs distribution of Poly I: C transfected porcine PK15 cells; D. DEPs in porcine PK15 cells transfected with Aza-CdR 图 1 Poly I: C和Aza-CdR转染猪PK15细胞的全基因组差异甲基化峰分布 Fig. 1 Genome differential methylation peaks in PK15 cells transfected with Poly I: C and Aza-CdR |
为更进一步确认富集的差异甲基化峰(DEPs)在全基因组的分布,试验将DEPs分为上调的甲基化峰和下调的甲基化峰。若差异探针分布在Poly I: C处理组(或Aza-CdR组)中而不分布在Control对照组中,则为上调的甲基化峰;若差异探针分布在Control对照组中而不分布在Poly I: C处理组(或Aza-CdR组)中,则为下调的甲基化峰。在两个比较组P vs. C和A vs. C中,高密度CpG岛启动子(HCPs)下调的DEPs较多,分别为394个下调DEPs(81%,92个上调DEPs,图 1C)和223个下调DEPs(54%,190个上调,图 1D),初步推断Poly I: C和Aza-CdR对猪PK5细胞均具有去甲基化作用,而这一现象在ICPs和LCPs中并没有被发现。
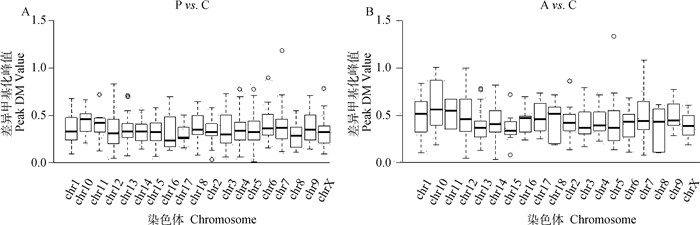
2.2 Poly I: C和Aza-CdR转染猪PK15细胞的染色体差异甲基化峰分布为进一步研究染色体上不同密度的CpG岛及甲基化峰在染色体上的分布情况,试验分析了甲基化峰值Peak DM value与染色体的关系。利用R软件绘制箱型图,结果如图 2所示,横轴代表不同的染色体,纵轴代表不同染色体上富集的差异甲基化峰值。结果说明,不同染色体上甲基化水平不同。当Poly I: C转染PK15细胞后,染色体水平上甲基化峰值均<0.5(图 2A);当Aza-CdR转染细胞后,染色体水平上甲基化峰值在0.5左右波动(图 2B)。结果证明,Poly I: C可能具有去甲基化作用,而Aza-CdR除了具有去甲基化作用,对某些区域也会上调甲基化,如1、10、11和18号染色体区域Peak DM value值(即M值)分别为0.52、0.58、0.57和0.56(图 2B)。
|
图 2 Poly I: C(A)和Aza-CdR(B)转染猪PK15细胞的染色体差异甲基化峰分布 Fig. 2 Chromosome differential methylation peaks in PK15 cells transfected with Poly I: C (A) and Aza-CdR(B) |
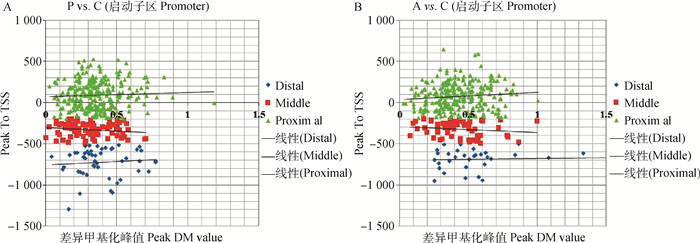
将启动子-1 500—+500 bp区划分为3个区域:-1 500—-500 bp(远端启动子,distal promoter),-500—-200 bp(中端启动子,middle promoter) 和-200—+500 bp(近端启动子和第一外显子,proximal promoter and first exson)。结果揭示,在P vs. C和A vs. C两个比较组中,-200—+500 bp区域中的甲基化阳性富集峰最多(绿色),其次为-500—-200 bp区域(红色),最后为-1 500—-500 bp(蓝色)(图 3)。结果表明,Poly I: C和Aza-CdR具有比较相似的启动子甲基化分布特征。
|
图 3 Poly I: C(A)和Aza-CdR(B)转染猪PK15细胞的启动子差异甲基化峰分布 Fig. 3 Promoter differential methylation peaks in PK15 cells transfected with Poly I: C (A) and Aza-CdR (B) |
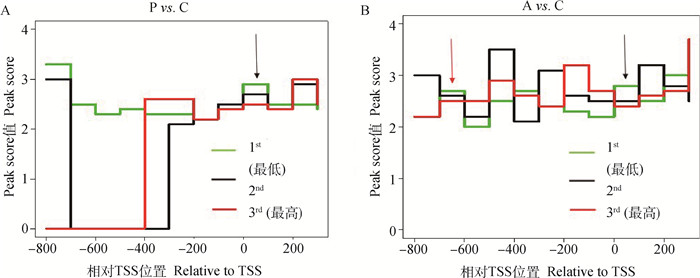
在差异甲基化区域DMR中,Peaks内探针的平均-lg(P值)反映了阳性富集的可能性大小(即Peak Score,Cut off = 2,P值<0.01)。若Peak Score ≥ 2,则这个Peak极显著代了一个甲基化事件。发现甲基化的富集程度在Poly I: C和Aza-CdR对Control组中的-200—+200 bp区域富集程度较高[图 4,- lg (P值) ≥ 2, P<0.01]。试验用3种不同颜色的线代表-800—+200 bp区域差异表达基因3种不同的表达水平,绿色线代表表达量最低(1 st),红色线代表表达量最高(3rd)。通过与表达谱差异基因的结合分析[10, 19],如图 4所示,在-200—+200 bp的近端启动子及第一外显子区域,甲基化富集水平(代表甲基化程度)与差异基因的表达水平存在负相关关系(图 4箭头所示)。结果表明,PK15细胞的DNA甲基化影响基因表达与甲基化修饰的位置相关。在0—+ 200 bp(P vs. C和A vs. C,图 4A和4B黑色箭头所示)区域和-800—-600 bp(A vs. C,图 4B红色箭头所示)区域内基因表达受DNA甲基化调控,即甲基化富集程度越高表达量越低;而在-600—0 bp区域,DNA甲基化不调控基因表达。
|
A. Poly I: C转染猪PK15细胞; B. Aza-CdR转染猪PK15细胞 A. Poly I: C transfected porcine PK15 cells; B. PK15 cells transfected with Aza-CdR 图 4 启动子差异甲基化峰的分布及其与表达的关系 Fig. 4 Promoter methylation peaks amd its relationship with gene expression |
图 5结果显示了差异甲基化峰在染色体上的富集情况[- lg (P值)≥2, P<0.01]。Poly I: C处理细胞的启动子中等密度的ICPs主要富集在9号和17号染色体上,而HCP和LCP富集程度较一致(图 5A);Aza-CdR处理细胞的启动子CpG岛密度与染色体分布无明显的相关性(图 5B)。
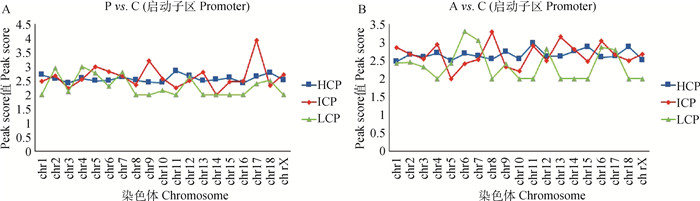
|
图 5 Poly I: C(A)和Aza-CdR(B)转染猪PK15细胞的染色体上启动子差异甲基化峰分布 Fig. 5 Chromosome promoter differential methylation peaks in PK15 transfected withPoly I: C (A) and Aza-CdR (B) |
和表达谱分析一样,本研究对差异甲基化基因进行功能分类,利用Gene Ontology(GO)注释结合fisher’s exact test(P值<0.05)检验方法进行分析。如图 6所示,这些在P vs. C比较组中上调和下调的差异甲基化基因都有各自独特的富集GO terms,基于P<0.05的条件下,结果揭示Poly I: C主要参与细胞生长调控、细胞成分组成及发育过程等,其中,富集程度最高的基因参与了巨噬细胞分化的调控(图 6A);Aza-CdR主要参与调控Rho信号转导、对病毒响应的防御、调控器官生长等(图 6B)。
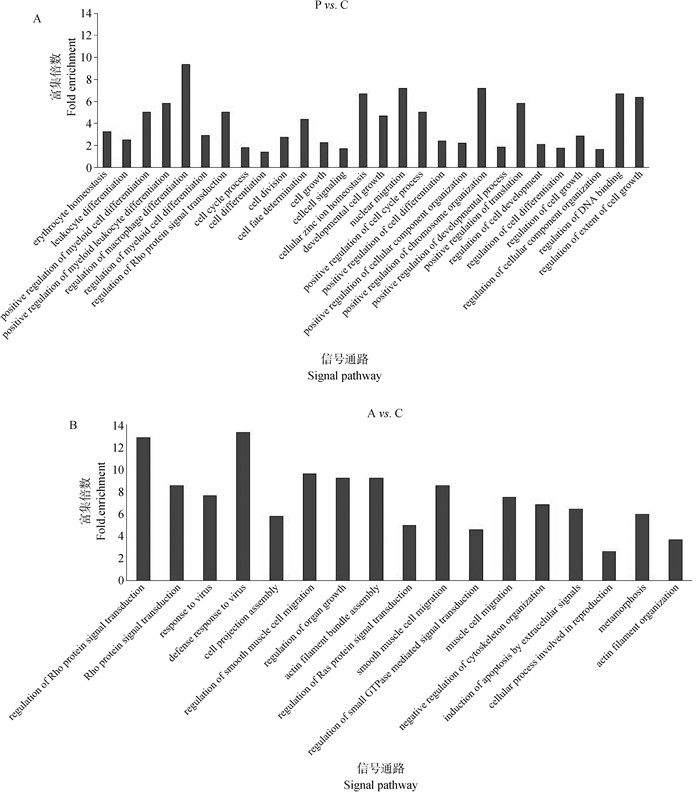
|
图 6 Poly I: C(A)和Aza-CdR(B)转染猪PK15细胞的差异甲基化基因GO分析 Fig. 6 GO analysis of differential methylation genes in PK15 cells transfected with Poly I: C (A) and Aza-CdR (B) |
分析P vs. C(表 1)和A vs. C(表 2)比较组中的差异甲基化基因发现,Poly I: C转染的细胞中Peroxisome Proliferator-Activated Receptor Gamma (PPARG)、Follistatin(FST)等基因启动子甲基化下调,主要参与细胞分化、发育的调控。Aza-CdR除了具有调节DNA甲基化的修饰作用外,它还具有调控其他表观遗传修饰、诱导细胞凋亡及参与机体免疫反应的作用,例如DFP3在调控心肌细胞发育方面发挥作用、BNIP3L参与病毒的繁殖等过程,这些基因的复杂调控可通过调节DNA的甲基化来抑制或增强基因表达。为了明确差异甲基化区域DMR,试验选择BCL2/腺病毒E1B相互作用蛋白3样基因(BNIP3L,BCL2/adenovirus E1B interacting protein 3-like)进行比对,利用NimbleGen公司的Signalmap软件分析基因启动子区富集的差异甲基化峰,结果显示,在14号染色体的10459946—10460615 bp区段共有669 bp Peak Length CG位点发生去甲基化(图 7)。
|
|
表 1 Poly I: C处理猪PK5细胞后启动子差异甲基化基因 Table 1 Promoter differential methylation genes in PK15 cells transfected with Poly I: C |
|
|
表 2 Aza-CdR处理猪PK5细胞后启动子差异甲基化基因 Table 2 Promoter differential methylation genes in PK15 cells transfected with Aza-CdR |
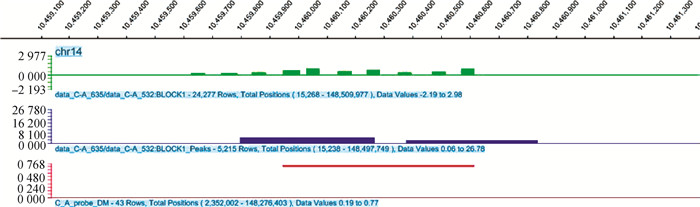
|
图 7 猪BNIP3L基因的启动子差异甲基化区域DMR Fig. 7 Promoter differential methylation region of porcine BNIP3L gene |
为了获得更精确的BNIP3L启动子甲基化差异信息,使用高敏感的bisulfite测序方法测定BNIP3L基因启动子区甲基化差异变化,检测上述区域20个CpG位点(Chr14: 10459946—10460615 bp)。与未转染的细胞(DNA甲基化程度为93.8%)相比,Poly I: C处理的DNA甲基化水平显著降低至35.0 %,Aza-CdR转染的细胞DNA甲基化程度降低至26.3%(图 8A)。
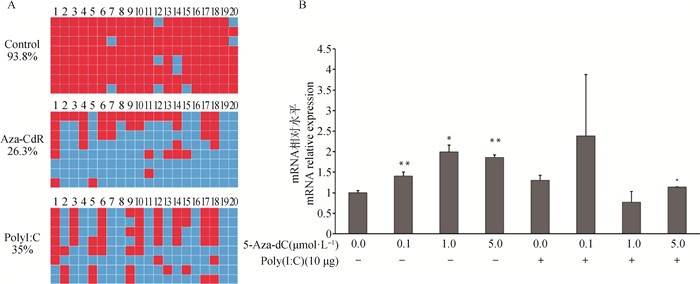
|
A. BNIP3L基因启动子甲基化克隆测序分析;B. BNIP3L基因mRNA表达量分析 A. Clone-sequencing of BNIP3L gene promoter; B. mRNA expression level of BNIP3L gene 图 8 猪BNIP3L基因的启动子差异甲基化区域DMR的验证 Fig. 8 Validation of promoter differential methylation region of porcine BNIP3L gene |
为了进一步验证PolyI: C通过去甲基化机制启动BNIP3L基因的转录表达,试验引入甲基化抑制剂Aza-CdR分析去甲基化与转录表达的关系。试验使用0.1、1.0、5.0 μmol·L-1 Aza-CdR处理细胞,同时在10.0 μg·mL-1 PolyI: C转染4 h后再使用Aza-CdR不同浓度处理细胞6 h,分析BNIP3L基因的转录表达。试验结果显示,0.1、1.0、5.0 μmol·L-1 Aza-CdR和10.0 μg·mL-1 Poly I: C单独处理细胞时均显著表达BNIP3L基因(P<0.05)(图 8B)。然而,5.0 μmol·L-1 Aza-CdR单独处理细胞时,与未处理的细胞相比,BNIP3L差异表达2倍以上,并达到及其差异显著水平(P<0.01)。
3 讨论本研究首次在全基因组范围研究猪肾细胞的DNA甲基化图谱的试验,获得了3组样本(Control细胞,Poly I: C转染细胞,Aza-CdR转染细胞)所有特有的全基因组甲基化分布特点。猪肾细胞全基因组DNA甲基化主要分布于5′调控区域。试验在组间比较后,特别是在P vs. C和A vs. C比较中发现DNA甲基化在基因组上的分布特征与CpG岛密度与距离TSS的位置有关。Poly I: C对PK15作用使得TSS附近200 bp(-200—+500 bp处,即近启动子区和第一外显子区)低甲基化启动子增多(图 3),可能这种低甲基化模式更利于转录因子的结合进而起始转录[20]。Tang等[21]报道dsRNA转染人胚肾细胞(HEK293T)和人肺上皮细胞(A549)后激活IL6启动子,增加IL6转录表达。该研究同时使用DNA甲基化转移酶抑制剂5-aza-2′-deoxycytidine处理细胞作为对照,IL6表达上调。这些结果表明dsRNA通过抑制DNMT活性从而诱导启动子去甲基化激活IL6表达,发挥抗流感病毒的作用。Li等[22]研究发现在流感病毒感染的细胞中,病毒中间产物dsRNA使得IL32启动子去甲基化增加转录因子CREB对IL32的结合上调其表达,证明dsRNA具有抑制DNMT1活性作用。通过抑制IL32的6个亚型均在病毒感染的细胞中阻碍病毒复制,特别是IL32-γ具有较强的抗病毒活性[22]。Wu等[23]使用深度测序技术对Poly I: C刺激和PRRSV感染的猪肺泡巨噬细胞(PAMs)进行miRNA转录组分析,以比较先天免疫激活和失活状态下的不同miRNA谱。测序后,在PAMs中观察到267个已知成熟miRNAs和64个新miRNAs,与模拟对照细胞相比,共197个miRNAs在Poly I: C刺激的PAMs中表达显著不同。Tran-Thi等[24]研究了猪卫星细胞(PSCs)在骨骼肌的构建、发育和自我更新中起着重要的作用。Poly I: C在刺激PSCs后0、12、24和48 h可模拟病毒感染的增殖和分化。
早在1980年,Jones和Taylor[25]就已发现5-氮杂胞苷(5-azacytidine)可以抑制活体细胞DNA甲基化,使DNA甲基化对自然状态的基因组DNA影响的研究成为可能。前期研究通过引入Aza-CdR研究病毒类似物dsRNA转染猪肾细胞后对全基因组表达的影响,结果揭示,Aza-CdR可逆转基因表达,这与基因的甲基化状态密切相关[10, 15-16]。Venolia等[26]早在1982年用5-azacytidine处理过细胞DNA转染其他细胞时,失活X染色体相关的次黄嘌呤酸核糖基转移酶基因得到表达,而对照的没有用5-azacytidine处理过的细胞DNA不能使该基因表达。另外,Aza-CdR对细胞的多样效应(主要包括DNA去甲基化及诱导细胞凋亡效应)具有细胞依赖以及时间剂量处理性特点,对细胞具有一定的毒性作用[27]。因此,在使用时要慎重考虑其特征。
前期研究利用Agilent猪4 × 44 K全基因组表达谱芯片分析了Poly I: C和Aza-CdR单独处理组(P vs. C和A vs. C)的差异表达基因的变化,在两个比较组中均筛选出与免疫反应、细胞发育分化及细胞凋亡相关的信号通路[19],这些通路中基因的表达差异可能与DNA甲基化的调控密切相关。与此同时,研究利用猪385 K MeDIP-chip芯片技术筛选得到DGAT、FST、BNIP3L、DPF3等与细胞发育分化、病毒复制、炎症反应相关的重要甲基化调控基因。这些结果揭示,Poly I: C和Aza-CdR对PK15细胞的去甲基化作用可调控免疫相关的信号通路。结合甲基化芯片与表达谱芯片研究结果发现,Aza-CdR具有负向调控病毒繁殖的功能,并且可减弱PolyI: C对细胞的反应[18, 28]。最近的研究证实,Lnc-Dpf3的新型长链非编码RNA可通过抑制树突状细胞体内迁移进而抑制炎症性疾病的发生发展[29]。BNIP3L的过表达可挽救线粒体自噬缺陷并对脑缺血损伤具有保护作用[30]。本研究也筛选到这两个关键基因,抗病毒相关基因BNIP3L和炎症反应相关基因DPF3,以期作为候选甲基化差异基因用于今后的验证试验,这些系统的分析将有利于理解病毒感染猪宿主细胞的免疫反应分子机理,为猪和其传染性病毒的互作提供遗传基础。
4 结论目前,使用Aza-CdR转染猪肾细胞的全基因组甲基化图谱仍未研究,本试验首次比较研究了Aza-CdR和dsRNA对猪肾细胞转染后的甲基化效应。试验初步揭示,Poly I: C与Aza-CdR的作用相似具有去甲基化作用,这与最近的一些报道相一致。Aza-CdR和dsRNA并不是对猪所有基因具有去甲基化作用,主要针对特有基因的特有启动子,譬如在与病毒复制有关基因BNIP3L和炎症反应相关基因DPF3的启动子区域中,这些特有启动子的CpG岛对Aza-CdR和dsRNA具有特别的敏感性。
| [1] |
隋维恺, 高艳, 杨茜茜, 等. PolyI: C刺激豚鹿(Axis porcinus)外周血淋巴细胞转录组分析[J]. 四川农业大学学报, 2021, 39(1): 114-120, 128. SUI W K, GAO Y, YANG X X, et al. Transcriptome analysis of hog deer (Axis porcinus) peripheral blood lymphocyte after PolyI: C challenge[J]. Journal of Sichuan Agricultural University, 2021, 39(1): 114-120, 128. (in Chinese) |
| [2] |
刘青珍, 李凌云, 齐义鹏, 等. RNA病毒基因组和转录复制多样性的分子基础[J]. 生物多样性, 2001, 9(3): 294-300. LIU Q Z, LI L Y, QI Y P, et al. Molecular mechanism for transcription and replication of RNA virus[J]. Biodiversity Science, 2001, 9(3): 294-300. DOI:10.3321/j.issn:1005-0094.2001.03.014 (in Chinese) |
| [3] |
HARRIS P, SRIDHAR S, PENG R, et al. Double-stranded RNA induces molecular and inflammatory signatures that are directly relevant to COPD[J]. Mucosal Immunol, 2013, 6(3): 474-484. DOI:10.1038/mi.2012.86 |
| [4] |
HÜSSER L, ALVES M P, RUGGLI N, et al. Identification of the role of RIG-I, MDA-5 and TLR3 in sensing RNA viruses in porcine epithelial cells using lentivirus-driven RNA interference[J]. Virus Res, 2011, 159(1): 9-16. DOI:10.1016/j.virusres.2011.04.005 |
| [5] |
ANDREJEVA J, CHILDS K S, YOUNG D F, et al. The V proteins of paramyxoviruses bind the IFN-inducible RNA helicase, mda-5, and inhibit its activation of the IFN-β promoter[J]. Proc Natl Acad Sci U S A, 2004, 101(49): 17264-17269. DOI:10.1073/pnas.0407639101 |
| [6] |
GIL J, ESTEBAN M. Induction of apoptosis by the dsRNA-dependent protein kinase (PKR): mechanism of action[J]. Apoptosis, 2000, 5(2): 107-114. DOI:10.1023/A:1009664109241 |
| [7] |
LIU J J, GUO Y M, HIROKAWA M, et al. A synthetic double-stranded RNA, poly I: C, induces a rapid apoptosis of human CD34+ cells[J]. Exp Hematol, 2012, 40(4): 330-341. DOI:10.1016/j.exphem.2011.12.002 |
| [8] |
KATO H, TAKEUCHI O, MIKAMO-SATOH E, et al. Length-dependent recognition of double-stranded ribonucleic acids by retinoic acid-inducible gene-I and melanoma differentiation-associated gene 5[J]. J Exp Med, 2008, 205(7): 1601-1610. DOI:10.1084/jem.20080091 |
| [9] |
MASALMEH R H A, TAGLINI F, RUBIO-RAMON C, et al. De novo DNA methyltransferase activity in colorectal cancer is directed towards H3K36 me3 marked CpG islands[J]. Nat Commun, 2021, 12(1): 694. DOI:10.1038/s41467-020-20716-w |
| [10] |
WANG X S, AO H, SONG M Y, et al. Identification of DNA methylation regulated novel host genes relevant to inhibition of virus replication in porcine PK15 cell using double stranded RNA mimics and DNA methyltransferase inhibitor[J]. Genomics, 2019, 111(6): 1464-1473. DOI:10.1016/j.ygeno.2018.09.020 |
| [11] |
刘国强, 罗建兴, 李春冬, 等. 绵羊肌肉组织中HOXB5和HOXC6基因的甲基化分析[J]. 中国畜牧杂志, 2022, 58(1): 133-137, 145. LIU G Q, LUO J X, LI C D, et al. Methylation analysis of HOXB5 and HOXC6 genes in sheep muscle tissue[J]. Chinese Journal of Animal Science, 2022, 58(1): 133-137, 145. (in Chinese) |
| [12] |
李庆贺. 鸡全基因组甲基化图谱及原鸡和家鸡启动子DNA甲基化保守性分析[D]. 北京: 中国农业大学, 2011. LI Q H. Genome-wide mapping of DNA methylation in chicken and analysis of promoter DNA methylation conservation between the wild and domestic chicken breeds[D]. Beijing: China Agricultural University, 2011. (in Chinese) |
| [13] |
NOVIK K L, NIMMRICH I, GENC B, et al. Epigenomics: genome-wide study of methylation phenomena[J]. Curr Issues Mol Biol, 2002, 4(4): 111-128. |
| [14] |
YANG Y L, FAN X H, YAN J Y, et al. A comprehensive epigenome atlas reveals DNA methylation regulating skeletal muscle development[J]. Nucleic Acids Res, 2021, 49(3): 1313-1329. DOI:10.1093/nar/gkaa1203 |
| [15] |
CHEN J Y, WU L X, XU H, et al. 5-Aza-CdR regulates RASSF1A by inhibiting DNMT1 to affect colon cancer cell proliferation, migration and apoptosis[J]. Cancer Manag Res, 2019, 11: 9517-9528. DOI:10.2147/CMAR.S229726 |
| [16] |
ROULOIS D, YAU H L, SINGHANIA R, et al. DNA-demethylating agents target colorectal cancer cells by inducing viral mimicry by endogenous transcripts[J]. Cell, 2015, 162(5): 961-973. DOI:10.1016/j.cell.2015.07.056 |
| [17] |
KOGA Y, PELIZZOLA M, CHENG E, et al. Genome-wide screen of promoter methylation identifies novel markers in melanoma[J]. Genome Res, 2009, 19(8): 1462-1470. DOI:10.1101/gr.091447.109 |
| [18] |
王晓铄, 俞英, 敖红, 等. 体外建立dsRNA结合甲基化转移酶抑制剂转染猪肾细胞的模型[J]. 中国畜牧杂志, 2017, 53(8): 94-99. WANG X S, YU Y, AO H, et al. The model of Aza-CdR treated porcine kidney cells after transfected with PolyI: C[J]. Chinese Journal of Animal Science, 2017, 53(8): 94-99. (in Chinese) |
| [19] |
WANG X S, AO H, ZHAI L W, et al. Genome-wide effects of DNA methyltransferase inhibitor on gene expression in double-stranded RNA transfected porcine PK15 cells[J]. Genomics, 2014, 103(5-6): 371-379. DOI:10.1016/j.ygeno.2013.10.005 |
| [20] |
SUN J F, JIANG Y, SHI Z X, et al. Proteomic alteration of PK-15 cells after infection by classical swine fever virus[J]. J Proteome Res, 2008, 7(12): 5263-5269. DOI:10.1021/pr800546m |
| [21] |
TANG B, ZHAO R, SUN Y, et al. Interleukin-6 expression was regulated by epigenetic mechanisms in response to influenza virus infection or dsRNA treatment[J]. Mol Immunol, 2011, 48(8): 1001-1008. DOI:10.1016/j.molimm.2011.01.003 |
| [22] |
LI W, SUN W, LIU L, et al. IL-32:a host proinflammatory factor against influenza viral replication is upregulated by aberrant epigenetic modifications during influenza a virus infection[J]. J Immunol, 2010, 185(9): 5056-5065. DOI:10.4049/jimmunol.0902667 |
| [23] |
WU J J, JI Z Y, QIAO M, et al. MicroRNA transcriptome analysis of poly I: C-stimulated and PRRSV-infected porcine alveolar macrophages[J]. J Appl Genetics, 2019, 60(3-4): 375-383. DOI:10.1007/s13353-019-00500-3 |
| [24] |
TRAN-THI T N, WANG S, ADETULA A A, et al. Gene expression profiling of porcine skeletal muscle satellite cells after poly(I: C) stimulation[J]. Gene, 2019, 695: 113-121. DOI:10.1016/j.gene.2018.12.059 |
| [25] |
JONES P A, TAYLOR S M. Cellular differentiation, cytidine analogs and DNA methylation[J]. Cell, 1980, 20(1): 85-93. DOI:10.1016/0092-8674(80)90237-8 |
| [26] |
VENOLIA L, GARTLER S M, WASSMAN E R, et al. Transformation with DNA from 5-azacytidine-reactivated X chromosomes[J]. Proc Natl Acad Sci U S A, 1982, 79(7): 2352-2354. DOI:10.1073/pnas.79.7.2352 |
| [27] |
ZHANG H H, HUANG B, CAO Y H, et al. Role of 5-Aza-CdR in mitomycin-C chemosensitivity of T24 bladder cancer cells[J]. Oncol Lett, 2017, 14(5): 5652-5656. |
| [28] |
王晓铄, 俞英, 刘会杰, 等. 基于表达谱芯片挖掘抗猪瘟病毒相关基因及其在不同猪瘟抗体水平下的表达差异[J]. 畜牧兽医学报, 2014, 45(1): 14-22. WANG X S, YU Y, LIU H J, et al. Mining anti classic swine fever virus-related genes based on microarray and analyzing their transcriptional expression under different CSF-antibody levels[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(1): 14-22. (in Chinese) |
| [29] |
LIU J, ZHANG X, CHEN K, et al. Lnc-Dpf3 impairs CCR7-mediated dendritic cell migration by inhibiting glycolysis[J]. Eur J Immunol, 2019, 49: 485. |
| [30] |
WU X L, ZHENG Y R, LIU M R, et al. BNIP3L/NIX degradation leads to mitophagy deficiency in ischemic brains[J]. Autophagy, 2021, 17(8): 1934-1946. DOI:10.1080/15548627.2020.1802089 |
(编辑 白永平)



