2. 天津市农业科学院畜牧兽医研究所, 天津 300000
2. Animal Husbandry Institute of Tianjin Academy of Agricultural Sciences, Tianjin 300000, China
杂种优势(heterosis)是一种广泛存在于动植物中的生物学现象,指具有遗传差异的两个亲本杂交,其F1代在生产性能、抗病性、抗逆性等方面一定程度上优于双亲的现象[1-2]。目前,杂种优势测定大多通过对不同亲本间杂种后代生产性能的评价,筛选出最佳的杂交亲本,但该方法耗费大量的人力、物力、财力和时间成本,且缺乏预见性。因此,寻找一种简单有效的方法,在育种早期预测出目标亲本间潜在的杂种优势,对提高育种效率,加快育种进程具有重要意义。
20世纪80年代,随着分子生物学技术的发展,DNA遗传标记兴起,这使得利用DNA遗传标记来预测和估计畜禽基因组杂种优势及其组分成为可能。吴常信[3]指出,通过分析品种或品系间DNA遗传多态,计算两个杂交品种或品系间的遗传距离来预测杂种优势,要比根据其它材料更稳定,更准确。随后随机扩增多态DNA(random amplified polymorphic DNA, RAPD)[4-5]、扩增片段长度多态性(amplified fragment length polymorphism, AFLP)[6]和微卫星DNA(simple sequence repeat, SSR)[7-10]等标记在畜禽杂种优势预测上得到广泛应用。近年来,随着快速、高通量SNP分型技术的发展,尤其SNP芯片问世,基于SNP标记研究畜禽遗传多态性[11]和关联分析[12-14]等来探究畜禽杂种优势的报道越来越多。Amuzu-Aweh等[15]等利用53 000个基因组SNPs对白来航鸡产蛋量、蛋重的杂种优势进行了预测,发现利用等位基因频率平方差(SDAF)预测产蛋量和蛋重的杂种优势准确性达到50%,节省了约50%的现场测试成本。徐忠等[16]等利用胴体与肉品质、繁殖、健康和生长4种性状的基因组特异性SNPs标记估算了杜洛克、大白和长白与我国地方猪种金华猪间的遗传距离,预测了金华猪不同杂交模式下的杂种优势。范婷婷等[17]则利用覆盖全基因组和与性状相关的SNPs标记预测了西门塔尔牛与我国8个地方黄牛品种间的杂种优势,结果发现西门塔尔牛与南丹牛杂交优势最高。这些研究虽然筛选出较优的杂交组合,但未对较优组合进行实际杂交效果验证。
近年来,由于肉牛业和奶业市场的双向需求,在实际生产中,西门塔尔牛、和牛常被用来杂交改良中低产荷斯坦牛以提高后代公牛的产肉性能,但它们与荷斯坦牛的杂种优势研究少有报道。因此,本研究以和牛、西门塔尔牛为父本,荷斯坦牛为母本构建不同的杂交组合,利用Illumina BovineHD770K和GGP Bovine 100 K基因芯片进行基因分型,分别用性状特异性SNPs和全基因组SNPs两种标记估算亲本间的遗传距离,预测各杂交组合在生长发育和胴体性状上的杂种优势,并构建较优组合的实际杂交群体,通过分析亲本间的一般配合力和特殊配合力来评估实际杂交效果,为指导牛的杂种优势利用和选种选配提供参考依据。
1 材料与方法 1.1 试验动物1.1.1 杂种优势预测群体 杂种优势预测群体包括和牛(Wagyu, W)、西门塔尔牛(Simmental, S)和荷斯坦牛(Holstein, H)。和牛来自辽宁省大连市甘井子区大连雪龙牧场,共464头;西门塔尔牛来自内蒙古锡林郭勒盟乌拉盖管理区牧场,共1 222头;荷斯坦牛来自天津市金三农农业科技开发有限公司和天津市东旭奶牛养殖专业合作社两个牧场,共43头。
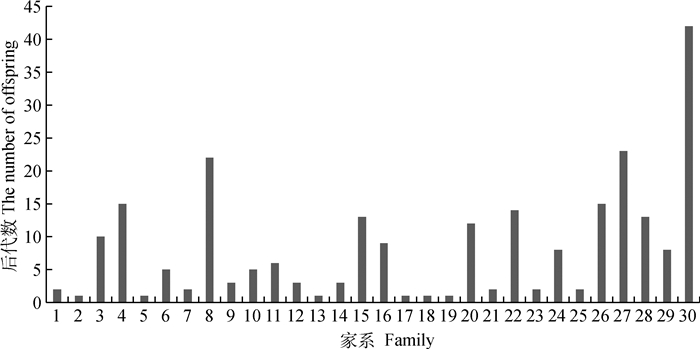
1.1.2 实际杂交群体 2017—2019年,利用30头德系西门塔尔牛冻精(German Simmental,GS)与天津市东旭奶牛养殖专业合作社、天津市宏乐奶牛养殖有限公司、天津市金三农农业科技开发有限公司荷斯坦母牛(Holstein, H)进行人工授精,获得261头F1代,并收集其初生重数据(图 1)。
|
图 1 30个德系西门塔尔牛不同家系的后代数 Fig. 1 The number of offspring of different families in German Simmental |
除30头德系西门塔尔牛使用冻精提取DNA外,所有试验牛均使用静脉采血,每头牛采集血液5 mL,与EDTA抗凝剂充分混匀后,放入低温采样箱运至实验室,于-20 ℃冰箱冷冻保存。采用血液/细胞/组织基因组DNA提取试剂盒(天根生化科技有限公司)提取DNA,并利用紫外分光光度计检测其浓度与纯度,检测合格的DNA分别送由北京康普森农业科技有限公司和纽勤生物科技(上海)有限公司进行基因分型。464头和牛与1 222头西门塔尔牛使用Illumina BovineHD770K芯片分型;30头德系西门塔尔牛、43头荷斯坦牛和261头F1代使用GGP Bovine100K芯片进行分型。
1.3 基因型数据质量控制利用PLINK v1.90[18-19]软件对基因型数据进行质量控制,先质控SNPs,然后再质控个体。质控标准如下:1)SNP位点位于常染色体;2)SNP位点的检出率(call rate)大于95%;3)SNP的最小等位基因频率(minor allele frequency, MAF)大于5%;4)位点哈代-温伯格平衡(hardy-weinberg equilibrium, HWE)检验概率大于10-6(P>10-6);5)个体SNP基因型检出率(call rate)大于90%。命令如下:plink --file filename -- geno 0.05 --hwe 0.000001-- mad 0.05 --mind 0.1 -- make-bed --out filename --cow -noweb。
对于缺失基因型的位点,利用Beagle[20-21]软件进行填充。命令:java -Xmx1000 m -jar beagle.jar unphased=filename.bgl missing=? out=outfilename。
质量控制后,和牛506 996个SNPs,西门塔尔牛675 029个SNPs,荷斯坦牛83 952个SNPs,德系西门塔尔牛81 411个SNPs,F1代83 723个SNPs用于后续分析。
1.4 杂种优势预测分析1.4.1 确定目的性状特异性SNPs位点 本研究以初生重(birth weight, BW)、周岁重(yearling weight, YW)、胴体重(carcass weight, CW)作为目的性状,将牛QTLs数据库(https://www.animalgenome.org/cgi-bin/QTLdb/BT/index)中关于生长性状的QTLs筛选出来,并利用Perl软件将目的性状QTLs与牛参考基因组进行比对,分别获得与初生重、周岁重、胴体重相关的3类基因集合。最后,通过R软件将检测到的基因芯片SNPs映射到这些基因上,从而筛选出3个性状特异性SNPs。
1.4.2 分析群体遗传结构 为避免连锁SNPs对群体结构分析的影响,利用PLINK软件剔除连锁不平衡系数(r2)大0.8的SNPs。然后分别对基因组SNPs及性状特异性SNPs进行主成分分析(principal component analysis,PCA)(参数:indep-pairwise 50 5 0.2),分析群体间的遗传结构;输出结果用R软件画图展现。
1.4.3 构建群体间性状IBS 依据质控后基因组SNP位点和目的性状特异性SNPs位点,按照性状类别,计算杂交组合2亲本群体间的同态相同概率(identity by state, IBS),即可能的子代纯合子概率。以群体间遗传距离D=1-IBS作为评定杂种优势高低的指标。亲本i与j之间的IBS为:
| $ I B S_{i j}=\frac{1}{m} \sum\nolimits_{n=1}^{m} \sum\nolimits_{k=1}^{s} p_{i n k} p_{j n k} $ |
式中,m为SNP的总个数;s表示第n个SNP位点的等位基因个数,一般为2;Pink表示第k个等位基因在第i个群体内第n个位点上的等位基因频率,Pjnk表示第k个等位基因在第j个群体内第n个位点上的等位基因频率[16]。
1.5 实际杂交群体配合力分析利用混合线性模型,计算德系西门塔尔(父本)与荷斯坦母牛(母本)杂交一代初生重性状的一般配合力(general combining ability, GCA)和特殊配合力(special combining ability, SCA)。
| $ {\rm{模型}}: \;\;y_{i j k}=\mu+g_{i}+g_{j}+s_{i j}+e_{i j k} $ |
其中,yijk为两亲本杂交后代的表型值,μ为杂交后代群体均值,gi为i亲本的一般配合力,gj为j亲本的一般配合力,sij为i亲本与j亲本间杂交的特殊配合力,eijk为随机误差,假设它独立地服从N(0, σe2)分布。利用R软件完成配合力的计算。
2 结果 2.1 品种间杂种优势预测分析2.1.1 目的性状特异性多态位点确定 将和牛、西门塔尔牛和荷斯坦牛的基因组SNPs位点与牛QTLs数据库进行比对,筛选出与牛生长发育及胴体性状相关的QTLs,并与牛基因组序列进行比对,筛选到与牛BW、YW和CW性状相关的特异性SNPs位点集合。和牛、西门塔尔牛和荷斯坦牛与目的性状相关的SNPs位点数见表 1。
|
|
表 1 3个群体中与目的性状相关的SNPS位点数 Table 1 Number of trait-specific SNPs in 3 populations |
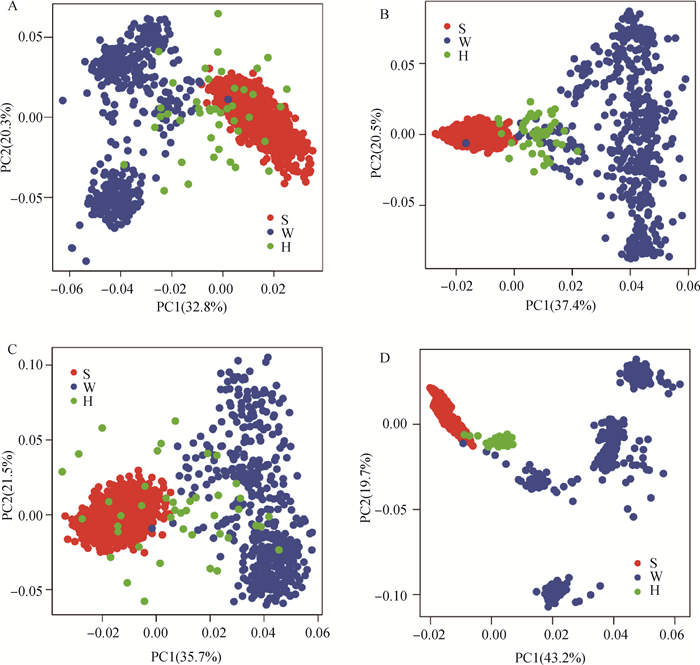
2.1.2 群体遗传结构分析 利用性状特异性和全基因组SNPs标记对和牛、西门塔尔牛及荷斯坦牛3个群体进行主成分分析,结果如图 2所示。由图 2可以看出,基于两种标记密度的主成分分析结果大致相同,和牛、西门塔尔牛、荷斯坦牛各自较为集中地聚在一起。同时,根据PC1发现荷斯坦牛与西门塔尔牛、和牛均存在一定程度的交互现象,但整体来看,和牛与荷斯坦牛的距离较近。这表明相对西门塔尔牛,和牛与荷斯坦牛的亲缘关系更近,遗传背景差异更小,这也与和牛的育种历史相符合[22-23]。
|
A.初生重相关SNPs的PCA;B. 周岁重相关SNPs的PCA;C. 胴体重相关SNPs的PCA;D.全基因组SNPs的PCA A. PCA of SNPs for birth weight; B. PCA of SNPs for yearling weight; C. PCA of SNPs for carcass weight; D. PCA of genome-wide SNPs 图 2 基于性状特异性和全基因组SNPs对和牛、西门塔尔牛及荷斯坦牛群体的主成分分析 Fig. 2 Principal component analysis of Wagyu, Simmental and Holstein based on trait-associated and genome-wide SNPs |
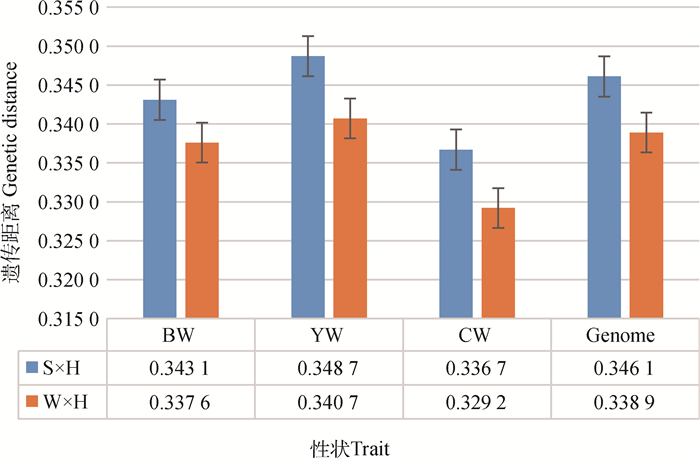
2.1.3 品种间杂交组合遗传距离比较 利用BW、YW和CW性状相关的特异性SNPs与全基因组SNPs分别计算西门塔尔牛♂×荷斯坦牛♀(S×H)与和牛♂×荷斯坦牛♀(W×H)亲本间遗传距离,结果见图 3。由图 3可以看出,在BW、YW和CW 3个性状上,S×H遗传距离分别为0.343 1、0.348 7和0.336 7,W×H亲本间遗传距离分别为0.337 6、0.340 7和0.329 2。在全基因组水平上,S×H和W×H亲本间杂交组合遗传距离为0.346 1和0.338 9,与相关SNPs估算的遗传距离差异不显著。由图 3可以发现,基于BW、YW和CW性状特异性SNPs与全基因组水平SNPs分析两个杂交组合遗传距离排序均相同,即为S×H > W×H。因此,西门塔尔牛与荷斯坦牛杂交获得的杂种优势可能性更大,这一结果与PCA分析结果相同。
|
图 3 西门塔尔牛、和牛与荷斯坦牛杂交组合的遗传距离比较 Fig. 3 Genetic distance comparison of Simmental×Holstein and Wagyu×Holstein |
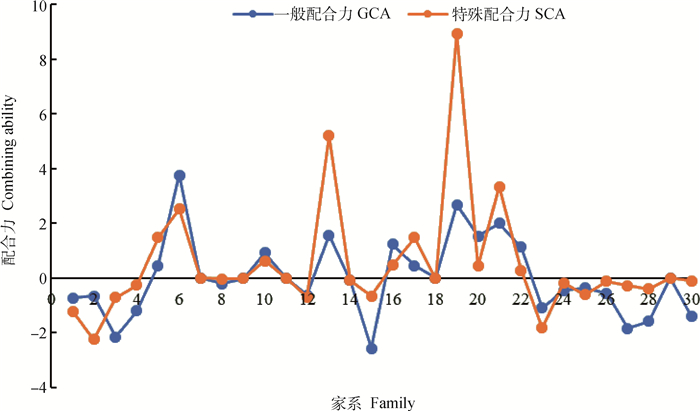
为了进一步验证较优组合S×H的实际杂种优势效果,2017—2019年期间,利用30头德系西门塔尔牛父本与荷斯坦母牛交配构建了实际杂交群体,获得261头F1代。本研究利用261头F1代初生重性状计算了德系西门塔尔牛与荷斯坦牛GCA和SCA,结果见图 4和表 2。由图 4可以看出,在初生重性状上,有10个父本的GCA效应值为正,父系6号(29617)的效应值最高,达到了3.760 9,其次为父系19号(12FFM13)的GCA达到了2.673 2,具体见表 2。对于不同父系杂交组合的SCA来看,SCA效应值的正负情况与GCA大致相符,父系13号(426989)、19号(12FFM13)、21号(FW13213107)杂交组合的SCA效应值较高,尤其父系19号(12FFM13)的SCA达到8.931 2。因此,在指导实际的选种选配中,为了获得较大的初生重,可优先选择德系西门塔尔牛父系6号(29617)、13号(426989)、19号(12FFM13)、21号(FW13213107)与荷斯坦牛配种。
|
图 4 不同父系初生重性状一般配合力和特殊配合力 Fig. 4 General and specific combining abilities of different paternal families for birth weight |
|
|
表 2 30个父本初生重性状的一般配合力和特殊配合力 Table 2 General and specific combining abilities of birth weight for 30 paternal families |
杂种优势因杂交后代具有生产性能、生活力和抗逆性等均高于双亲的特点,被广泛地运用在畜禽杂交育种中,例如我国利用西门塔尔牛培育了中国西门塔尔牛和蜀宣花牛等乳肉兼用新品种;利用夏洛莱牛培育了夏南牛和辽育白牛专门化肉牛品种。杂种优势利用成为提高我国地方黄牛生产性能的重要途径之一,杂种优势利用率越高,种畜的价值就越高,其后代的养殖利润就越大,而杂种优势利用率的评估则依赖于对种畜杂种优势大小的预测[24]。传统经典遗传学认为,亲本间的遗传差异是杂种优势产生的基础,亲本间遗传距离越远、品系纯度越高,后代表现出来的杂种优势就会越明显[25]。曹阳等[26]等利用微卫星标记分析了德国黄牛、利木赞牛、红安格斯牛、夏洛莱牛与当地西杂牛的遗传距离,发现4个引进品种与西杂牛的遗传距离为:德国黄牛>利木赞牛>红安格斯牛>夏洛莱牛,预测西杂牛与德国黄牛的杂种优势最高,利木赞次之,夏洛莱牛最低。范婷婷等[17]等利用牛770K SNPs高密度芯片分析了西门塔尔牛与我国8个地方牛品种间的遗传距离,预测西门塔尔牛与南丹牛在各性状上可获较高的杂种优势。Martinez-Boggio等[27]研究表明,安格斯♂×海福特♀的F1代母牛受胎率(artificial insemination probability,AIP)和产犊成功(calving success,CS)较高,而内洛尔牛♂×海福特♀的3~7岁杂交母牛的平均妊娠天数(days to calving,DC)和CS较高。其他畜禽,如猪[16, 28-29]、鸭[30]、鸡[31]、羊[32-33]等同样依据遗传距离得到了各组合杂种优势优劣结果。本研究利用全基因组水平SNPs和性状特异性的SNPs对3个群体遗传结构及不同亲本间的遗传距离进行了分析,预测了其杂种优势。结果表明, 无论是利用全基因组水平SNPs还是目的性状特异性SNPs位点分析3个群体PCA,均发现西门塔尔牛与荷斯坦牛的遗传距离较远,且预测杂种优势高。虽然利用性状特异性SNPs标记估算的初生重、胴体重性状遗传距离低于全基因组SNPs,但差异不显著。与全基因组水平相比,性状特异性SNPs标记减少了标记数量,但预测结果与全基因组SNPs预测结果相同,说明使用性状特异性SNPs标记可提高估计准确性[27]。另外,本研究中利用Illumina Bovine HD 770K和GGP Bovine 100K两款芯片分别对和牛、西门塔尔牛和荷斯坦牛群体进行基因分型,筛选得到和牛506 996个有效多态性SNPs位点,西门塔尔牛675 029个,荷斯坦牛83 952个,德系西门塔尔牛81 411个。从群体PCA和杂种优势预测结果看出,GGP Bovine 100K芯片标记数量能准确评估群体遗传结构和杂种优势预测,但检测成本却大幅降低75%左右。因此,在畜禽杂交优势预测中,可采用中低密度芯片来鉴定被预测亲本的基因型,从而减少杂交预测的试验成本。
3.2 基于配合力的杂交效果分析配合力(combing ability)是指一个亲本(纯系、自交系或被测材料)与另一系列具有遗传差异的亲本杂交所产生杂种一代性状表现中所起作用相对大小的度量,可分为一般配合力(GCA)和特殊配合力(SCA)。GCA是指亲本在所有杂交组合中表现出的平均配合效应,是亲本基因加性效应作用的结果,可稳定遗传。而SCA反映了基因的非加性效应,即基因显性、超显性和上位效应等相互作用的结果,不能稳定遗传,只能通过亲本杂交在F1代表现出来,是杂种优势的重要遗传基础。一般GCA高的亲本,其SCA也会比较高,后代杂种优势的表现也会更突出[34]。本研究为进一步验证杂种优势预测结果的准确性,针对初生重、周岁重和胴体重性状较优的S×H,进行了实际群体的杂交试验,根据后代初生重性状的GCA和SCA来验证杂种优势预测的准确性。结果发现,S×H杂交后代初生重性状表现出了较好的杂种优势,GCA较高的父本,其SCA同样也比较高,都表现为正向的效应值。赵宗胜等[34]报道,GCA效应值较高的安卡红肉鸡,其杂交组合的SCA也均为正值,为高效组合。在对兔子[35]、虹鳟[36]、凡纳滨对虾[37]、牡蛎[38]、黄瓜[39]、甘蓝[40]的研究等中同样也得到了类似的结果。由此证明,基于基因组和性状特异性SNPs预测杂种优势结果的准确性。同时,在实际群体的选配计划中,可选择GCA和SCA高的德系西门塔尔牛父系与荷斯坦牛杂交,以获得较高的初生重,例如GCA和SCA均为正值的父系6号(29617)、13号(426989)、19号(12FFM13)、21号(FW13213107)。
4 结论本研究基于覆盖全基因组和性状的特异性SNPs标记估算了西门塔尔牛、和牛与荷斯坦牛亲本间的遗传距离,预测出S×H为杂种优势较高的杂交组合;实际杂交群体的配合力测定发现,S×H的杂交后代在初生重性状上表现出较高的杂种优势。因此,西门塔尔牛与荷斯坦牛杂交可在初生重、周岁重和胴体重性状获得较高的杂种优势。
| [1] |
CHEN Z J. Molecular mechanisms of polyploidy and hybrid vigor[J]. Trends Plant Sci, 2010, 15(2): 57-71. DOI:10.1016/j.tplants.2009.12.003 |
| [2] |
张媛媛, 杨艳华, 陈克平. 杂种优势的研究进展[J]. 生命科学研究, 2016, 20(5): 447-454. ZHANG Y Y, YANG Y H, CHEN K P. Advances in heterosis research[J]. Life Science Research, 2016, 20(5): 447-454. (in Chinese) |
| [3] |
吴常信. 分子数量遗传学与动物育种[J]. 遗传, 1997(S1): 1-3. WU C X. Molecular quantitative genetics and animal breeding[J]. Hereditas(Beijing), 1997(S1): 1-3. (in Chinese) |
| [4] |
玉荣. 六个肉牛群体RAPD变异及其与杂种优势关系的研究[D]. 呼和浩特: 内蒙古农业大学, 2004. YU R. Studies of RAPD variability of six beef cattle population and its relationship to heterosis[D]. Huhhot: Inner Mongolia Agricultural University, 2004. (in Chinese) |
| [5] |
张凤伟, 蒋曹德, 邓昌彦, 等. RAPD标记遗传距离与猪杂种优势相关性的研究[J]. 中国农业科学, 2007, 40(1): 173-177. ZHANG F W, JIANG C D, DENG C Y, et al. Study on correlation between genetic distance and heterosis percentage of pig with RAPD marker[J]. Scientia Agricultura Sinica, 2007, 40(1): 173-177. DOI:10.3321/j.issn:0578-1752.2007.01.024 (in Chinese) |
| [6] |
苟本富. 利用AFLP标记遗传距离预测猪经济性状杂种优势的研究[J]. 中国畜牧杂志, 2009, 45(17): 13-15. GOU B F. Study on prediction of heterosis of pig economic traits by AFLP marker genetic distance[J]. Chinese Journal of Animal Science, 2009, 45(17): 13-15. (in Chinese) |
| [7] |
DI R, CHU M X, LI Y L, et al. Predictive potential of microsatellite markers on heterosis of fecundity in crossbred sheep[J]. Mol Biol Rep, 2012, 39(3): 2761-2766. DOI:10.1007/s11033-011-1032-7 |
| [8] |
ZHANG J H, XIONG Y Z, DENG C Y, et al. Correlation of individual heterozygosity of microsatellite marker loci with heterosis of growth traits in pig populations[J]. Agric Sci China, 2006, 5(8): 635-642. DOI:10.1016/S1671-2927(06)60104-1 |
| [9] |
孙少华, 桑润滋, 师守堃, 等. 肉牛杂交优势预测、评估及其应用研究[J]. 遗传学报, 2000, 27(7): 580-589. SUN S H, SANG R Z, SHI S K, et al. Prediction and evaluation of heterosis of beef cattle and their application[J]. Acta Genetica Sinica, 2000, 27(7): 580-589. (in Chinese) |
| [10] |
张建新, 岳文斌, 张雨. 利用微卫星DNA标记法进行5个肉用绵羊品种的多态性分析和杂种优势率预测[J]. 激光生物学报, 2010, 19(2): 218-223. ZHANG J X, YUE W B, ZHANG Y. Study on polymorphism and heterosis of five meat sheep breeds using microsatellite marker[J]. Acta Laser Biology Sinica, 2010, 19(2): 218-223. DOI:10.3969/j.issn.1007-7146.2010.02.015 (in Chinese) |
| [11] |
AKANNO E C, ABO-ISMAIL M K, CHEN L H, et al. Modelingheterotic effects in beef cattle using genome-wide SNP-marker genotypes1[J]. J Anim Sci, 2018, 96(3): 830-845. DOI:10.1093/jas/skx002 |
| [12] |
RIBEIRO A M F, SANGLARD L P, SNELLING W M, et al. Geneticparameters, heterosis, and breed effects for body condition score and mature cow weight in beef cattle[J]. J Anim Sci, 2022, 100(2): skac017. DOI:10.1093/jas/skac017 |
| [13] |
ZIMMERMANN M J, KUEHN L A, SPANGLER M L, et al. Breed and heterotic effects for mature weight in beef cattle[J]. J Anim Sci, 2021, 99(8): skab209. DOI:10.1093/jas/skab209 |
| [14] |
REZENDE F M, RODRIGUEZE, LEAL-GUTIÉRREZ J D, et al. Genomic approaches reveal pleiotropic effects in crossbred beef cattle[J]. Front Genet, 2021, 12: 627055. DOI:10.3389/fgene.2021.627055 |
| [15] |
AMUZU-AWEH E N, BIJMA P, KINGHORN B P, et al. Prediction of heterosis using genome-wide SNP-marker data: application to egg production traits in White Leghorn crosses[J]. Heredity (Edinb), 2013, 111(6): 530-538. DOI:10.1038/hdy.2013.77 |
| [16] |
徐忠, 张哲, 孙浩, 等. 金华猪与杜洛克、长白猪、大白猪杂交的杂种优势预测[J]. 中国畜牧杂志, 2019, 55(1): 64-67. XU Z, ZHANG Z, SUN H, et al. Heterosis prediction of Jinhua pig crossing with Duroc, Landrace pig and Large White pig[J]. Chinese Journal of Animal Science, 2019, 55(1): 64-67. (in Chinese) |
| [17] |
范婷婷, 陈燕, 张路培, 等. 西门塔尔牛与我国地方黄牛的杂种优势预测分析[J]. 畜牧兽医学报, 2021, 52(3): 653-661. FAN T T, CHEN Y, ZHANG L P, et al. Prediction of heterosis between Chinese Simmental beef cattle and Chinese local cattle[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(3): 653-661. (in Chinese) |
| [18] |
CHANG C C, CHOW C C, TELLIER L C A M, et al. Second-generation PLINK: rising to the challenge of larger and richer datasets[J]. Giga Science, 2015, 4(1): 7. DOI:10.1186/s13742-015-0047-8 |
| [19] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: A tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [20] |
BROWNING B L, BROWNING S R. A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals[J]. Am J Hum Genet, 2009, 84(2): 210-223. DOI:10.1016/j.ajhg.2009.01.005 |
| [21] |
BROWNING S R, BROWNING B L. Rapid and accurate haplotype phasing and missing-data inference for whole-genome association studies by use of localized haplotype clustering[J]. Am J Hum Genet, 2007, 81(5): 1084-1097. DOI:10.1086/521987 |
| [22] |
张英汉. 黑毛和牛地方类群培育成国际品种的经验分析[J]. 中国牛业科学, 2014, 40(4): 1-7. ZHANG Y H. Historical analysis of Black Wagyu from a native to a internationally recognized beef breed[J]. China Cattle Science, 2014, 40(4): 1-7. DOI:10.3969/j.issn.1001-9111.2014.04.001 (in Chinese) |
| [23] |
潘军. 日本和牛品种形成、将征与评价[J]. 黑龙江动物繁殖, 1994, 2(1): 4-6. PAN J. Origination and characteristics of Japanese cattle breeds[J]. Heilongjiang Journal of Animal Reproduction, 1994, 2(1): 4-6. (in Chinese) |
| [24] |
朱磅强, 何俊, 曾青华, 等. 动物杂种优势利用及其预测方法[J]. 中国猪业, 2021, 16(4): 22-26. ZHU B Q, HE J, ZENG Q H, et al. Utilization of animal heterosis and its prediction methods[J]. China Swine Industry, 2021, 16(4): 22-26. (in Chinese) |
| [25] |
张乐, 苗永旺. 畜禽杂种优势测定和评估方法分析[J]. 吉林农业, 2015(15): 78-79. ZHANG L, MIAO Y W. Evaluation and test of heterosis in livestock and poultry[J]. Agriculture of Jilin, 2015(15): 78-79. (in Chinese) |
| [26] |
曹阳, 张国梁, 李金龙, 等. 不同肉牛品种间杂种优势预测[J]. 安徽农业科学, 2010, 38(33): 18842. CAO Y, ZHANG G L, LI J L, et al. Heterosis prediction of Simmental cross cattle with other different beef breeds[J]. Journal of Anhui Agricultural Sciences, 2010, 38(33): 18842. DOI:10.3969/j.issn.0517-6611.2010.33.086 (in Chinese) |
| [27] |
MARTINEZ-BOGGIO G, LEMA M, AGUILAR I, et al. Reproductive performance of Angus, Hereford, Salers and Nellore crossbred females: additive and non-additive effects[J]. J Anim Breed Genet, 2021, 138(6): 688-697. DOI:10.1111/jbg.12553 |
| [28] |
柳丽华, 闵令江, 沈伟, 等. 野猪及其杂交群体的遗传多样性和遗传分化的研究[J]. 畜牧兽医学报, 2009, 40(2): 173-179. LIU L H, MIN L J, SHEN W, et al. Analysis of genetic structure in wild boar and their crossbred populations[J]. Acta Veterinaria et Zootechnica Sinica, 2009, 40(2): 173-179. (in Chinese) |
| [29] |
盛中华, 陈国宏, 潘玉春, 等. 基于基因组遗传标记的上海白猪(上系)杂交优势预测分析[J]. 上海农业学报, 2018, 34(6): 60-64. SHENG Z H, CHEN G H, PAN Y C, et al. Prediction and analysis of heterosis in Shanghai white pigs (Shang) based on genome-wide genetic markers[J]. Acta Agriculturae Shanghai, 2018, 34(6): 60-64. (in Chinese) |
| [30] |
胡继伟, 彭地莎, 胡瑞, 等. 4个微卫星标记的多态性与鸭肉用性能杂种优势的相关性研究[J]. 中国畜牧杂志, 2012, 48(19): 21-24. HU J W, PENG D S, HU R, et al. Correlation between polymorphism of four microsatellite markers and heterosis of duck meat performance[J]. Chinese Journal of Animal Science, 2012, 48(19): 21-24. (in Chinese) |
| [31] |
张庆桥, 代敏敏, 靳伟, 等. 微卫星标记预测太行鸡与其他高产商品蛋鸡亲本间的杂种优势[J]. 黑龙江畜牧兽医, 2019(11): 46-52. ZHANG Q Q, DAI M M, JIN W, et al. Prediction of heterosis between parents of Taihang Mountains chicken and other high-yield commercial layers by microsatellite markers[J]. Heilongjiang Animal Science and Veterinary Medicine, 2019(11): 46-52. (in Chinese) |
| [32] |
刘建斌, 李发弟, 杜立新, 等. 利用微卫星DNA多态性预测引进肉用绵羊品种杂种优势[J]. 畜牧兽医学报, 2010, 41(2): 147-154. LIU J B, LI F D, DU L X, et al. Forecast of heterosis of imported meat sheep by genetic polymorphism of microsatellite DNA[J]. Acta Veterinaria et Zootechnica Sinica, 2010, 41(2): 147-154. (in Chinese) |
| [33] |
罗惠娣, 毛杨毅, 张亚萍, 等. 无角陶赛特羊和地方绵羊品种微卫星多态性分析及品种间杂种优势预测[J]. 中国草食动物科学, 2013, 33(3): 11-15. LUO H D, MAO Y Y, ZHANG Y P, et al. Analysis on microsatellite polymorphisms of poll Dorset and local Sheep and the prediction of their crossbred superiority[J]. China Herbivore Science, 2013, 33(3): 11-15. (in Chinese) |
| [34] |
赵宗胜, 李大全, 李伟, 等. 利用配合力分析预测肉杂鸡的杂种优势[J]. 黑龙江畜牧兽医, 2012(5): 61-63. ZHAO Z S, LI D Q, LI W, et al. Prediction of heterosis of hybrid broilers by combining ability analysis[J]. Heilongjiang Animal Science and Veterinary Medicine, 2012(5): 61-63. (in Chinese) |
| [35] |
ADENAIKE A S, OSISANYA T O, OGUNSOLA O D, et al. Combining ability and inheritance of growth traits in rabbits[J]. J Biol Agric Heal, 2013, 3(13): 102-108. |
| [36] |
王炳谦, 谷伟, 高会江, 等. 利用配合力和微卫星标记预测虹鳟品系间的杂交优势[J]. 中国水产科学, 2009, 16(2): 206-213. WANG B Q, GU W, GAO H J, et al. Prediction of hybridization advantage among five strains of rainbow trout by combination ability and SSR markers[J]. Journal of Fishery Sciences of China, 2009, 16(2): 206-213. (in Chinese) |
| [37] |
王伦, 王崇懿, 刘建勇, 等. 凡纳滨对虾多代选育群体生长和耐综合胁迫性状的配合力及杂种优势分析[J]. 海洋学报, 2022, 44(4): 65-73. WANG L, WANG C Y, LIU J Y, et al. Analysis of combining ability and heterosis of growth and comprehensive stress tolerance traits of Litopenaeus vannamei multi-generation breeding population[J]. Haiyang Xuebao, 2022, 44(4): 65-73. (in Chinese) |
| [38] |
梁园鑫, 徐成勋, 刘士凯, 等. 长牡蛎"海大1号"与壳橙品系杂交子代的杂种优势及配合力分析[J/OL]. 水产学报: 1-9[2022-05-14]. https://kns.cnki.net/kcms/detail/31.1283.s.20220401.0813.002.html. LIANG Y X, XU C X, LIU S K, et al. Heterosis and combining ability of reciprocal hybrids between"Haida No. 1" strain and orange shell line of the Pacific oyster (Crassostreagigas)[J/OL]. Journal of Fisheries of China: 1-9[2022-05-14]. https://kns.cnki.net/kcms/detail/31.1283.s.20220401.0813.002.html. (in Chinese) |
| [39] |
王慧, 臧柯昕, 陈劲枫, 等. 欧洲温室型黄瓜品质性状杂种优势及配合力分析[J/OL]. 南京农业大学学报: 1-12[2022-05-14]. https://kns.cnki.net/kcms/detail/32.1148.s.20220401.1336.006.html. WANG H, ZANG K X, CHEN J F, et al. Analysis of heterosis and combining ability on quality traits in European greenhouse cucumber[J/OL]. Journal of Nanjing Agricultural University: 1-12[2022-05-14]. https://kns.cnki.net/kcms/detail/32.1148.s.20220401.1336.006.html. (in Chinese) |
| [40] |
冯羽飞, 郭艳, 黄炜, 等. 8个春甘蓝自交系主要农艺性状的配合力与杂种优势分析[J/OL]. 分子植物育种: 1-14[2022-05-14]. https://kns.cnki.net/kcms/detail/46.1068.S.20220329.1551.013.html. FENG Y F, GUO Y, HUANG W, et al. Analysis on combining ability and heterosis of main agronomic traits of eight spring cabbage inbred lines[J/OL]. Molecular Plant Breeding: 1-14[2022-05-14]. https://kns.cnki.net/kcms/detail/46.1068.S.20220329.1551.013.html. (in Chinese) |
(编辑 郭云雁)



