牛奶中乳蛋白的含量约为3%~5%,是牛奶的主要成分之一。因其几乎含有人体所需的全部必需氨基酸并易于吸收,满足机体生长和维持日常生命活动的需要,被认为是评价牛奶价值的重要指标之一[1-2]。如何提高牛奶中乳蛋白水平并改善牛奶中乳蛋白致敏性一直是学术界的焦点,也是生产者、消费者和乳品加工企业关注的热点。因此,通过分子遗传育种等现代手段改善牛奶中乳蛋白的品质是奶牛育种的重要目标之一。
乳蛋白主要是由酪蛋白和乳清蛋白组成,是由乳腺上皮细胞通过细胞膜上氨基酸转运载体吸收转运血液中游离氨基酸而合成[3]。乳蛋白的合成主要在粗面内质网的核糖体上开始,然后由信号肽引导进入内质网中,并在内质网和高尔基体内进行磷酸化和糖基化等化学修饰,再由分泌泡转运至上皮细胞顶膜,通过胞吐方式释放到细胞外的腺泡腔[3]。乳蛋白的合成受到多个信号通路及多种激素相互影响,如:氨基酸转运、蛋白酪氨酸激酶2-信号转导子和转录激活因子5(janus tyrosine kinase 2-signal transducer and activator of transcription 5, Jak2-Stat5)和雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)等信号通路,胰岛素、催乳素和生长激素等[4-6]。由此可见,各通路中的信号传递和激素进入细胞后的信号传递过程至关重要。
VPS28(vacuolar protein sorting 28)是一种液泡蛋白分选基因,是内体蛋白分选转运装置Ⅰ(endosomal sorting complex required for transport- Ⅰ,ESCRT- Ⅰ)的亚单位,与ESCRTs的稳定性有关[7]。泛素化是细胞中以单泛素蛋白和多泛素蛋白链为信号分子,将泛素蛋白结合到底物蛋白特定位点的一种翻译后修饰过程[8]。ESCRTs不仅能识别泛素化膜蛋白,将其转运至溶酶体进行降解,还参与蛋白酶体对胞质中多泛素化蛋白的降解[9-11]。在课题组前期研究中,发现VPS28基因与奶牛乳蛋白率和乳蛋白量均存在极显著关联(P<0.01),并发现该基因在奶牛的乳腺组织中特异性高表达[12-15],但VPS28基因对乳蛋白的调控尚无相关研究。因此,本研究以VPS28基因为奶牛乳蛋白性状的重要候选基因,采用RNAi干扰技术和iTRAQ比较蛋白质组学,分析并试图阐明VPS28基因调控乳蛋白合成的分子机制,为奶牛产奶性状的分子育种研究提供理论基础。
1 材料与方法 1.1 材料奶牛原代乳腺上皮细胞(BMECs)为原实验室冻存;DMEM/F12培养基、DMEM培养基、Opti-MEM培养基、胎牛血清、双抗(penicillin-streptomycin solution)购自美国Gibco公司;TRIzol购自美国Invitrogen公司;转染试剂X-treme GENE siRNA Transfection Reagent、SYBR Green Mix购自美国Roche公司;溶酶体抑制剂氯喹CQ(chloroquine,C668)购自美国Sigma-Aldrich公司;蛋白酶体抑制剂环氧霉素Epox(epoxomicin,BU-4061T)购自美国MCE公司;泛素抗体(ubiquition,sc53509)购自美国Santa Cruz公司;β-actin抗体(66009-Ig)购自美国Proteintech公司;HRS标记二抗(7076S)购自美国Cell Signaling Technology公司;蛋白酶体活性检测试剂盒(Proteasome-GloTM Chymo-trypsin-Like, Trypsin-Like and Caspase-Like Cell-Based Assays,G1180)购自美国Promega公司。
1.2 细胞培养将BMECs培养于DMEM/F12全培养基中(含有10%胎牛血清,100 U·mL-1青霉素-链霉素),并将其置于温度37 ℃,CO2浓度5%的细胞培养箱中。当细胞汇合度达到100%时,用0.25%胰酶进行消化并将BMECs以2.5×105个·孔-1传代接种至6孔板中,以备后续试验使用。
1.3 BMECs中VPS28基因的敲降培养BMECs 24 h,当细胞汇合度达80%时使用siRNA串联片段对VPS28基因进行敲降,首先,用150 μL Opti-MEM I Reduced Serum Medium分别稀释2 μg siRNA和20 μL X-treme GENE siRNA Transfection Reagent;然后将二者轻柔混匀并静置20 min;最后,缓慢滴加入细胞培养液中。6 h后更换为完全培养基,培养72 h后收集细胞[14]。
1.4 VPS28基因及乳蛋白合成相关基因的表达量检测结合KEGG网站(KEGG-Table of Contents, http://www.genome.jp/kegg/kegg2.html),本研究选择乳蛋白合成、泛素化-溶酶体和泛素化-蛋白酶体通路中11个基因作为候选基因,采用qRT-PCR方法检测这11个相关基因mRNA表达量的变化(具体基因和引物信息见表 1)。以GAPDH为内参基因,采用比较Ct值法即2-△△Ct(△△Ct =目的基因△Ct值-内参基因△Ct值)表示目的基因的相对表达量。采用美国ABI公司SYBR Green Ⅰ进行定量表达检测。反应体系及反应程序参照SYBR Green Ⅰ说明书,在ABI 7500荧光定量PCR仪上进行扩增。
|
|
表 1 目的基因与引物信息 Table 1 Target genes and primers information |
使用siRNA串联片段对BMECs中VPS28基因进行敲降后,收集细胞提取总蛋白,利用Western blot技术检测敲降前后BMECs中泛素蛋白的表达水平。配制10%聚丙烯酰胺凝胶,按40 μg·孔-1上样,70 V电泳0.5 h至分离胶处,调整电压为120 V继续电泳1 h。采用湿转法转膜,200 mA 1 h。采用5%脱脂奶粉封闭1 h后,分别加入泛素抗体(1∶200) 和β-actin抗体(1∶1 000)4 ℃孵育过夜,1× TBST洗涤3次,5 min·次-1。二抗(1∶2 000)摇晃孵育1 h,1×TBST洗涤3次,5 min·次-1,用ECL发光液显影,用Image J软件进行灰度值分析。
1.6 蛋白酶体活性的检测利用蛋白酶体活性检测试剂盒检测VPS28敲降组和未敲降对照组中胰蛋白酶样、糜蛋白酶样和半胱天冬酶样蛋白酶活性。配制荧光素酶检测液,室温孵育30 min。将准备好的细胞悬液加入白壁96孔板中,并加入等体积的检测液,室温孵育10 min后检测相对荧光信号值。
1.7 BMECs中溶酶体和蛋白酶体的抑制取10 mg溶酶体抑制剂氯喹CQ,加入0.387 7 mL超纯水,充分混匀配置为50 mmol·L-1 CQ处理液,置于-20 ℃保存;取100 μg蛋白酶体抑制剂Epox,溶于180.27 μL DMSO中,充分混匀配置为1 mmol·L-1的Epox处理液,置于-20 ℃保存。培养BMECs 24 h,当6孔板中细胞汇合度达80%时,在不同孔中分别加入2 μL CQ或20 μL Epox孵育细胞,24 h收集细胞。
1.8 比较蛋白质组学的分析使用siRNA串联片段对BMECs VPS28基因进行敲降后,收集细胞提取总蛋白,利用iTRAQ比较蛋白质组学分析敲降前后BMECs的差异表达蛋白。利用在线软件DAVID(https://david.ncifcrf.gov/)对分析得到的差异蛋白(≤0.8或≥ 1.25倍)进行富集分析,并利用在线软件STRING(https://www.string-db.org/)对差异蛋白进行互作分析。
1.9 数据分析所有试验数据均采用3次独立重复试验结果,以“均数±标准差”表示,两组数据差异使用SPSS 21.0软件进行独立样本T检验统计分析,P<0.05时认为具有统计学意义。
2 结果 2.1 敲降VPS28基因后乳蛋白相关基因、泛素蛋白的表达水平及蛋白酶体活性采用串联片段的siRNA对BMECs中VPS28基因进行敲降,在成功敲降72%后(图 1A、1B),检测了11个候选基因的表达趋势,结果如图 1C所示,酪蛋白基因CSN1S1、CSN2、CSN3、核糖体蛋白RPS8、泛素蛋白基因UBC及蛋白酶体相关基因PSMC3、PSMC5表达水平均显著上调(P<0.05),PSMD12表达水平显著下调(P<0.05),RPL13、UBE2L差异不显著;泛素蛋白表达水平及蛋白酶体活性如图 1D、1E所示,在敲降VPS28基因后,泛素蛋白显著上调(P<0.05),蛋白酶体活性下调。结果提示,VPS28基因可能通过泛素-蛋白酶体及泛素-溶酶体信号通路改变细胞中核糖体合成乳蛋白的水平。
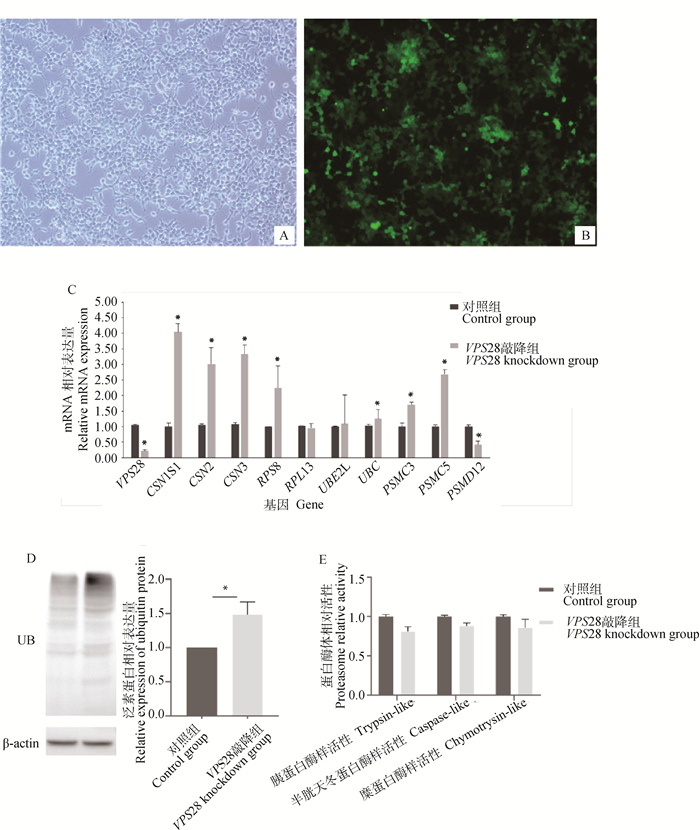
|
A. 空白细胞(10×);B. BMECs中转染siRNAs敲降VPS28基因(10×);C. 敲降VPS28基因后BMECs中乳蛋白相关基因的相对表达水平;D. 敲降VPS28基因后BMECs中泛素蛋白的相对表达水平;E. 敲降VPS28基因后BMECs中蛋白酶体相对活性 A. Blank cells (10×); B. Knockdown of VPS28 gene by transfection of siRNAs in BMECs (10×); C. Relative expression levels of milk protein-related genes in BMECs after knockdown of the VPS28 gene; D. Relative expression levels of ubiquitin proteins in BMECs after knockdown of the VPS28 gene; E. Relative proteasome activity in BMECs after knockdown of the VPS28 gene 图 1 敲降BMECs中VPS28基因及检测乳蛋白相关基因、泛素蛋白的表达水平及蛋白酶体活性 Fig. 1 The knockdown of the VPS28 gene with siRNAs in BMECs and detection of the mRNA expression levels of milk protein related genes, ubiquitin protein, and proteasome activity |
为验证VPS28基因可以通过泛素-溶酶体及泛素-蛋白酶体信号通路改变细胞中乳蛋白的合成,本研究通过蛋白酶体抑制剂Epox及溶酶体抑制剂CQ抑制细胞中蛋白酶体及溶酶体活性,并检测乳蛋白及核糖体相关基因的表达水平。结果如图 2所示,抑制细胞中蛋白酶体活性后,细胞中酪蛋白基因CSN1S1、CSN2、CSN3的相对表达量显著上调(P<0.05),核糖体蛋白RPL13的相对表达量显著下调(P<0.05);抑制细胞中溶酶体活性后酪蛋白相关基因表达不显著,核糖体相关基因RPS8显著上调(P<0.05)。结果表明,泛素-溶酶体及泛素-蛋白酶体信号通路确实可以改变核糖体活性及乳蛋白的合成。
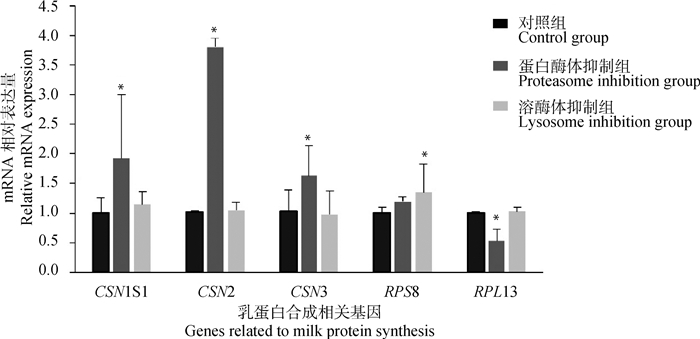
|
图 2 抑制BMECs中蛋白酶体及溶酶体活性后乳蛋白相关基因的相对表达水平 Fig. 2 Relative mRNA expression levels of milk protein-related genes after inhibition of proteasome and lysosome activities in BMECs |
为进一步确定VPS28基因通过泛素化途径调控BMECs中乳蛋白合成的调控水平及通路,本研究利用iTRAQ技术对敲降VPS28基因后的BMECs进行了蛋白谱分析。通过与未敲降的对照组相比,共发现129个差异表达蛋白,其中75个显著下调(下调倍数≤0.8倍),54个显著上调(上调倍数≥1.25倍)。对差异蛋白进行GO分析,如图 3A、B所示,在生物学过程中,下调蛋白主要富集在细胞质翻译(GO: 0002181)、高密度脂蛋白粒子组装(GO: 0043691)、胆固醇反向运输(GO: 0043691)及翻译(GO: 0006412),上调蛋白主要富集在基因表达正向调控(GO: 0010628)及过时氧化还原过程(GO: 0055114);在细胞组分中,下调蛋白主要富集在乳糜微粒(GO: 0042627)、胞质大核糖体亚基(GO: 0022625)、粘着斑(GO: 0005925)、细胞膜(GO: 0016020)及细胞核(GO: 0005730),上调蛋白主要富集在细胞外空间(GO: 0005615)、浆膜的重要组成部分(GO: 0005887)、膜结构组分(GO: 0016021)、胞外外泌体(GO: 0070062)及内质网膜组分(GO: 0005789);在分子功能中,下调蛋白主要富集在核糖体结构组成(GO: 0003735)、rRNA结合(GO: 0019843)、磷脂酰胆碱固醇-甾醇O-酰基转移酶活化剂(GO: 0060228)、胆固醇转运体活性(GO: 0017127)、胆固醇酯化的正向调节(GO: 0010873)及胆固醇结合(GO: 0015485),上调蛋白主要富集在核苷酸结合(GO: 0000166)、ATP结合(GO: 0005524)、poly-A-RNA结合(GO: 0044822)及金属离子结合(GO: 0005506)。对差异蛋白进行KEGG通路分析,如图 3C所示,下调蛋白主要富集在核糖体(bta03010)、溶酶体通路(bta04142)、剪切体(bta03040)等信号通路,上调蛋白主要富集在内质网的蛋白质加工(bta04141)、加压素调控的水重吸收(bta04962)、RNA转运(bta04962)和阿尔兹海默症(bta05010)等通路中。对差异蛋白进行STRING互作分析,如图 3D所示,差异蛋白主要集中在核糖体相关蛋白的互作上。结果表明,在BMECs中敲降VPS28基因可以显著降低核糖体中蛋白质的翻译及溶酶体的功能,同时可以提高细胞中基因的表达转录水平、内质网的蛋白质加工合成及泛素化介导的蛋白质降解过程。
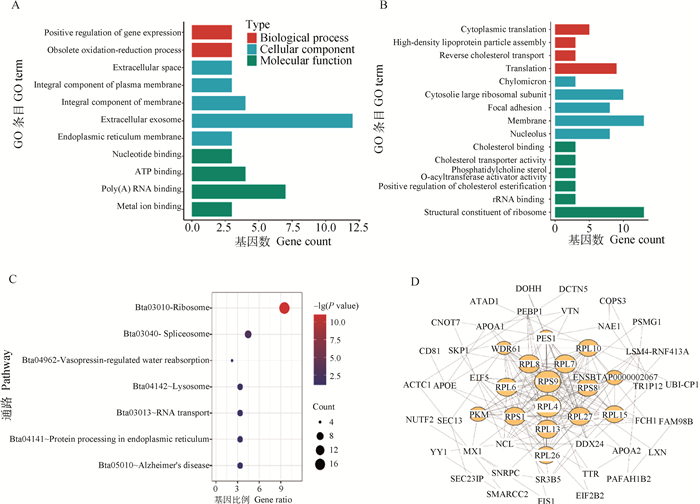
|
A. 上调差异表达蛋白的GO功能注释;B. 下调差异表达蛋白的GO功能注释;C. 差异表达蛋白的KEGG通路分析;D. 差异表达蛋白互作网络分析 A. GO functional annotation of up-regulated differentially expressed proteins; B. GO functional annotation of down-regulated differentially expressed proteins; C. Analysis of the KEGG pathway of differentially expressed proteins; D. Analysis of interaction networks of differentially expressed proteins 图 3 VPS28干扰前后BMECs中差异表达蛋白的富集分析 Fig. 3 Enrichment analysis of differentially expressed proteins in BMECs before and after VPS28 interference |
本研究以VPS28基因为影响奶牛乳蛋白性状的重要功能候选基因,在细胞水平对该基因进行功能研究,试图解释VPS28基因调控乳蛋白合成的重要作用,为该基因的功能研究及奶牛乳蛋白性状相关分子遗传标记的筛选提供重要理论依据。
VPS28是真核细胞中内体蛋白分选转运装置ESCRTs的亚单位,属于ESCRT-Ⅰ的顶端帽子结构[16-17]。VPS28可以通过GLUE结构域直接识别膜蛋白上的泛素蛋白并与之相结合,然后召集ESCRT-0、ESCRT-Ⅱ和ESCRT-Ⅲ组合形成超级复合体ESCRTs,再通过多囊体将泛素化的膜蛋白运输至溶酶体中进行降解,由此构成泛素-溶酶体通路,完成细胞中膜蛋白的泛素化降解过程[9-10]。同时,研究发现,泛素-溶酶体通路可以通过泛素化影响泛素-蛋白酶体通路的活性,对细胞中泛素-蛋白酶体通路介导的胞质泛素化蛋白降解起到一定调控作用[11, 18],尤其是当细胞中蛋白酶体活性受限后,泛素-溶酶体信号通路可被激活以补偿、维持细胞中蛋白质的稳态[19]。泛素化是由泛素激活酶(E1)、泛素结合酶(E2)和泛素连接酶(E3)介导的,将泛素蛋白结合到底物蛋白特定位点的一种翻译后修饰过程,可以控制细胞中蛋白质的半衰期和表达水平[20-21]。泛素化不仅可以作为信号传递过程中的重要调节蛋白,调控包括转录调控、细胞应答及新陈代谢等多种细胞活动,还可以直接识别并标记内质网中错误折叠的蛋白质,使其被有效降解[22-23]。Mercier和Gaye[24]在1982年研究发现, 乳蛋白的合成主要在粗面内质网的核糖体开始,然后由信号肽引导进入内质网中,并在内质网和高尔基体内进行磷酸化和糖基化等化学修饰,再由分泌泡转运至上皮细胞顶膜,最后通过胞吐方式释放到腺泡腔。VPS28基因可能通过泛素-溶酶体和泛素-蛋白酶体信号通路影响核糖体的合成过程和内质网、高尔基体的加工过程,进而影响奶牛乳腺上皮细胞中乳蛋白的合成。Liu和Zhang[14]等指出,血液中游离的氨基酸、脂肪酸、甘油等物质通过主动或被动运输进入乳腺上皮细胞,为保证进入细胞内的物质正确,这些物质需要通过ESCRTs的分选。综合以上研究推测,泛素化信号通路在调控奶牛乳腺上皮细胞合成乳蛋白和乳脂的作用机制相同。
为验证VPS28基因可能通过泛素-溶酶体和泛素-蛋白酶体信号通路影响核糖体中乳蛋白的合成,本研究首先利用RNAi干扰技术对BMECs中的VPS28基因进行有效敲降,然后检测酪蛋白、核糖体和蛋白酶体等相关基因的表达水平以及泛素蛋白的表达水平和蛋白酶体活性。结果发现,敲降VPS28基因后可显著改变细胞中酪蛋白和核糖体相关基因的表达水平,并可显著提高泛素蛋白的表达水平和降低蛋白酶体的活性,与前期研究结果相同[14, 25],这表明VPS28基因被敲降后可以通过下调蛋白酶体活性而提高泛素蛋白在细胞内的蓄积,这也提示了VPS28基因可能是通过泛素化影响了蛋白酶体活性,进而调控核糖体中乳蛋白的合成。为验证这一结果,本研究利用溶酶体抑制剂和蛋白酶体抑制剂抑制BMECs中溶酶体和蛋白酶体的活性,再次检测酪蛋白和核糖体相关基因的表达,结果发现了相同的趋势,说明VPS28基因确实可以通过泛素-溶酶体和泛素-蛋白酶体信号通路影响BMECs中乳蛋白的合成。
为进一步确定VPS28基因通过泛素化作用对BMECs中乳蛋白合成的调控水平及调控机制,本研究利用iTRAQ技术对敲降前后的BMECs进行了比较蛋白质组学分析。GO分析表明,下调蛋白主要集中在核糖体翻译、细胞质翻译和转运等过程中,而上调蛋白主要分布在细胞膜、细胞质以及细胞外,具有多种结合活性。KEGG分析表明,差异表达蛋白富集在核糖体、溶酶体、剪接体、内质网的蛋白质加工、加压素调控的水重吸收和RNA转运等相关通路中,其中,下调蛋白主要富集在核糖体和溶酶体。核糖体不仅是细胞中翻译蛋白的主要场所,还积极参与了蛋白质的折叠过程,对于各种功能性蛋白质的合成至关重要[26]。溶酶体是细胞中酸性的单层膜细胞器,不仅含有多种水解酶,用于降解细胞中的膜蛋白质、脂类、核酸和多糖等物质,还参与了细胞的胞吞、胞吐和外泌体分泌等过程[27-29]。当细胞中溶酶体的功能受到抑制可导致线粒体应激,由此引发细胞中蛋白质失衡,同时显著降低细胞的代谢活性[19],此外,有研究表明,溶酶体可以代偿性发挥蛋白酶体的泛素化胞质蛋白的降解功能,维持细胞中的蛋白质平衡和内环境的稳态,因此,溶酶体可能在游离氨基酸向乳腺上皮细胞转运、乳蛋白合成和顶浆分泌过程中均起到重要调节和决定性作用。RNA剪接是真核生物基因表达调控的重要环节之一,是“中心法则”的关键步骤之一[30-31],而负责执行这一过程的是细胞核内一个巨大的且高度动态变化的分子机器—剪接体(spliceosome)。剪接体在真核生物进化中极为保守,对于真核生物维持正常的生命活动至关重要。一个基因转录出的前体mRNA可以通过RNA剪接形成若干种mRNA[32],极大地丰富真核生物蛋白质组的多样性,其在乳蛋白的功能性蛋白质合成中起到重要作用。因此,本研究表明,敲降VPS28基因可能通过下调核糖体、溶酶体和剪接体相关蛋白的表达,减少乳蛋白的合成和分泌。
加压素主要调控细胞中水的重吸收,可以调控细胞器及细胞膜的通透性[33],参与乳蛋白的分泌及胞内运输等过程。RNA转运指RNA的核浆穿梭过程,RNA不同的亚细胞定位决定了其不同的生物学功能[34-35]。研究表明,RNA转运参与了mRNA翻译、蛋白合成和基因调控等多种生物学过程[36],因此该通路与乳蛋白的合成调控密切相关。内质网是由单层膜构成的囊状、泡状和管状系统[37-38]。内质网的蛋白质加工是影响内质网中蛋白质折叠的途径[39-40],正确折叠的蛋白质被包装成运输囊泡,转运至高尔基体,错误折叠的蛋白质则被泛素分子标记,进一步转运到溶酶体或蛋白酶体进行降解[23]。因此,本研究在敲降VPS28基因后,发现BMECs中加压素介导水分重吸收、内质网中蛋白质加工、RNA转运及泛素化介导蛋白质降解等通路的相关蛋白表达上调,与前人研究结果一致。但VP28基因对细胞中线粒体活性及细胞中所有代谢产物的具体影响还需要后续深入研究。
4 结论本研究以VPS28基因为奶牛乳蛋白性状的重要候选基因,采用RNAi干扰技术和iTRAQ比较蛋白质组学进行分析,敲降VPS28基因后发现酪蛋白、核糖体和蛋白酶体等相关基因及泛素蛋白表达水平显著上调,蛋白酶体活性下调;抑制蛋白酶体后酪蛋白相关基因发生同样变化趋势;进一步采用iTRAQ比较蛋白质组学分析发现,差异表达蛋白主要富集在核糖体、溶酶体、剪接体等通路中,表明敲降VPS28基因可以通过泛素化信号通路影响BMECs中乳蛋白的合成,本研究结果也为奶牛产奶性状的分子育种研究提供了理论基础。
| [1] |
ZENKER H E, TEODOROWICZ M, EWAZ A, et al. Binding of CML-modified as well as heat-glycated β-lactoglobulin to receptors for AGEs is determined by charge and hydrophobicity[J]. Int J Mol Sci, 2020, 21(12): 4567. DOI:10.3390/ijms21124567 |
| [2] |
FRANZOI M, NIERO G, VISENTIN G, et al. Variation of Detailed protein composition of cow milk predicted from a large database of mid-infrared spectra[J]. Animals(Basel), 2019, 9(4): 176. |
| [3] |
OSORIO J S, LOHAKARE J, BIONAZ M. Biosynthesis of milk fat, protein, andlactose: roles of transcriptional and posttranscriptional regulation[J]. Physiol Genomics, 2016, 48(4): 231-256. DOI:10.1152/physiolgenomics.00016.2015 |
| [4] |
汪东阳, 谢月琴, 罗君谊, 等. 调控乳蛋白合成信号通路的研究进展[J]. 中国奶牛, 2020(5): 6-11. WANG D Y, XIE Y Q, LUO J Y, et al. Research progress in regulating the signal pathway of milk protein synthesis[J]. China Dairy Cattle, 2020(5): 6-11. (in Chinese) |
| [5] |
MEIJER A J, LORIN S, BLOMMAART E F, et al. Regulation of autophagy by amino acids and mTOR-dependent signal transduction[J]. Amino Acids, 2015, 47(10): 2037-2063. DOI:10.1007/s00726-014-1765-4 |
| [6] |
CAI W T, LI C, LIU S L, et al. Genome wide identification of novel long non-coding RNAs and their potential associations with milk proteins in Chinese Holstein Cows[J]. Front Genet, 2018, 9: 281. DOI:10.3389/fgene.2018.00281 |
| [7] |
SCHMIDT O, TEIS D. The ESCRT machinery[J]. Curr Biol, 2012, 22(4): R116-R120. DOI:10.1016/j.cub.2012.01.028 |
| [8] |
SAKSENA S, SUN J, CHU T, et al. ESCRTing proteins in the endocytic pathway[J]. Trends Biochem Sci, 2007, 32(12): 561-573. DOI:10.1016/j.tibs.2007.09.010 |
| [9] |
RAIBORG C, STENMARK H. The ESCRT machinery in endosomal sorting of ubiquitylated membrane proteins[J]. Nature, 2009, 458(7237): 445-452. DOI:10.1038/nature07961 |
| [10] |
KATZMANN D J, BABST M, EMR S D. Ubiquitin-dependent sorting into the multivesicular body pathway requires the function of a conserved endosomal protein sorting complex, ESCRT-I[J]. Cell, 2001, 106(2): 145-155. DOI:10.1016/S0092-8674(01)00434-2 |
| [11] |
NANDI D, TAHILIANI P, KUMAR A, et al. The ubiquitin-proteasome system[J]. J Biosci, 2006, 31(1): 137-155. DOI:10.1007/BF02705243 |
| [12] |
KOLBEHDARI D, WANG Z, GRANT J R, et al. A whole genome scan to map QTL for milk production traits and somatic cell score in Canadian Holstein bulls[J]. J Anim Breed Genet, 2009, 126(3): 216-227. DOI:10.1111/j.1439-0388.2008.00793.x |
| [13] |
JIANG L, LIU X, YANG J, et al. Targeted resequencing of GWAS loci reveals novel genetic variants for milk production traits[J]. BMC Genomics, 2014, 15(1): 1105. DOI:10.1186/1471-2164-15-1105 |
| [14] |
LIU L, ZHANG Q. Identification and functional analysis of candidate gene VPS28 for milk fat in bovine mammary epithelial cells[J]. Biochem Biophys Res Commun, 2019, 510(4): 606-613. DOI:10.1016/j.bbrc.2019.01.016 |
| [15] |
JIANG L, LIU J F, SUN D X, et al. Genome wide association studies for milk production traits in Chinese Holstein population[J]. PLoS One, 2010, 5(10): e13661. DOI:10.1371/journal.pone.0013661 |
| [16] |
刘莉莉, 郭爱伟, 吴培福, 等. 敲降VPS28基因对中国荷斯坦奶牛乳脂合成的调控[J]. 遗传, 2018, 40(12): 1092-1100. LIU L L, GUO A W, WU P F, et al. Regulation of VPS28 gene knockdown on the milk fat synthesis in Chinese Holstein dairy[J]. Hereditas, 2018, 40(12): 1092-1100. (in Chinese) |
| [17] |
丁亚群, 丁宁, 谢深民, 等. Vps28基因敲除小鼠模型的构建及其对泌乳和免疫性状影响的研究[J]. 生物技术通报, 2022, 38(3): 164-172. DING Y Q, DING N, XIE S M, et al. Construction of Vps28 knock-out mice and model study of the impact onlactation and immune traits[J]. Biotechnology Bulletin, 2022, 38(3): 164-172. (in Chinese) |
| [18] |
CLAGUE M J, URBÉ S. Ubiquitin: samemolecule, different degradation pathways[J]. Cell, 2010, 143(5): 682-685. DOI:10.1016/j.cell.2010.11.012 |
| [19] |
SULKSHANE P, RAM J, GLICKMAN M H. Ubiquitination of intramitochondrial proteins: implications for metabolic adaptability[J]. Biomolecules, 2020, 10(11): 1559. DOI:10.3390/biom10111559 |
| [20] |
DOUGHERTY S E, MADUKA A O, INADA T, et al. Expanding role of ubiquitin in translational control[J]. Int J Mol Sci, 2020, 21(3): 1151. DOI:10.3390/ijms21031151 |
| [21] |
RUSTEN T E, STENMARK H. How do ESCRT proteins control autophagy?[J]. J Cell Sci, 2009, 122(Pt13): 2179-2183. |
| [22] |
DA FONSECA P C A, HE J, MORRIS E P. Molecular model of the human 26S proteasome[J]. Mol Cell, 2012, 46(1): 54-66. DOI:10.1016/j.molcel.2012.03.026 |
| [23] |
HIRSCH C, GAUSS R, HORN S C, et al. The ubiquitylation machinery of the endoplasmic reticulum[J]. Nature, 2009, 458(7237): 453-460. DOI:10.1038/nature07962 |
| [24] |
MERCIER J C, GAYE P. Early events in secretion of main milk proteins: occurrence of precursors[J]. J Dairy Sci, 1982, 65(2): 299-316. DOI:10.3168/jds.S0022-0302(82)82192-9 |
| [25] |
LIU L, ZHANG Q. Comparative proteome analysis reveals VPS28 regulates milk fat synthesis through ubiquitylation in bovine mammary epithelial cells[J]. PeerJ, 2020, 8: e9542. DOI:10.7717/peerj.9542 |
| [26] |
HASHIMOTO S, SUGIYAMA T, YAMAZAKI R, et al. Identification of a novel trigger complex that facilitates ribosome-associated quality control in mammalian cells[J]. Sci Rep, 2020, 10(1): 3422. DOI:10.1038/s41598-020-60241-w |
| [27] |
FUJIWARA Y, KIKUCHI H, AIZAWA S, et al. Direct uptake and degradation of DNA by lysosomes[J]. Autophagy, 2013, 9(8): 1167-1171. DOI:10.4161/auto.24880 |
| [28] |
INTRA J, PAVESI G, HORNER D S. Phylogenetic analyses suggest multiple changes of substrate specificity within the glycosyl hydrolase 20 family[J]. BMC Evol Biol, 2008, 8: 214. DOI:10.1186/1471-2148-8-214 |
| [29] |
PURUSHOTHAMAN B, LEE J, HONG S, et al. Multifunctional TPP-PEG-biotin self-assembled nanoparticle drug delivery-based combination therapeutic approach for co-targeting of GRP78 and lysosome[J]. J Nanobiotechnol, 2020, 18(1): 102. DOI:10.1186/s12951-020-00661-y |
| [30] |
COSTA P R, ACENCIO M L, LEMKE N. Cooperative RNA polymerase molecules behavior on a stochastic sequence-dependent model for transcription elongation[J]. PLoS One, 2013, 8(2): e57328. DOI:10.1371/journal.pone.0057328 |
| [31] |
QI H D, LIN Y, REN Q P, et al. RNA splicing of FLC modulates the transition to flowering[J]. Front Plant Sci, 2019, 10: 1625. DOI:10.3389/fpls.2019.01625 |
| [32] |
BUSETTO V, BARBOSA I, BASQUIN J, et al. Structural and functional insights into CWC27/CWC22 heterodimer linking the exon junction complex to spliceosomes[J]. Nucleic Acids Res, 2020, 48(10): 5670-5683. DOI:10.1093/nar/gkaa267 |
| [33] |
KHOYNEZHAD A, CELIS R, HASHEMZADEH M, et al. Current status of thoracic endografting and its adjunctive pharmacology[J]. Expert Opin Pharmaco, 2009, 10(17): 2871-2881. DOI:10.1517/14656560903277194 |
| [34] |
DAYIE K T. Key labeling technologies to tackle sizeable problems in RNA structural biology[J]. Int J Mol Sci, 2008, 9(7): 1214-1240. DOI:10.3390/ijms9071214 |
| [35] |
SONG H B, LIU D C, DONG S W, et al. Epitranscriptomics and epiproteomics in cancer drug resistance: therapeutic implications[J]. Signal Transduct Target Ther, 2020, 5: 193. DOI:10.1038/s41392-020-00300-w |
| [36] |
PAANANEN J, FORTINO V. Anomics perspective on drug target discovery platforms[J]. Brief Bioinform, 2020, 21(6): 1937-1953. DOI:10.1093/bib/bbz122 |
| [37] |
WANG P W, HUSSEY P J. NETWORKED 3B: a novel protein in the actin cytoskeleton-endoplasmic reticulum interaction[J]. J Exp Bot, 2017, 68(7): 1441-1450. DOI:10.1093/jxb/erx047 |
| [38] |
CHEN Y H, TENG X, HU Z J, et al. Hydrogen sulfide attenuated sepsis-induced myocardial dysfunction through TLR4 pathway and endoplasmic reticulum stress[J]. Front Physiol, 2021, 12: 653601. DOI:10.3389/fphys.2021.653601 |
| [39] |
MARZBAN H, DEL BIGIO M R, ALIZADEH J, et al. Cellular commitment in the developing cerebellum[J]. Front Cell Neurosci, 2014, 8: 450. |
| [40] |
AMIN-WETZEL N, NEIDHARDT L, YAN Y H, et al. Unstructured regions in IRE1α specify BiP-mediated destabilisation of the luminal domain dimer and repression of the UPR[J]. eLife, 2019, 8: e50793. DOI:10.7554/eLife.50793 |
(编辑 郭云雁)



