脂肪间充质干细胞(adipose-derived stem cells, ADSCs)是成体干细胞的一种,因其具有自我更新速度快、能够稳定增殖、具备多项分化潜能的特点[1],在体细胞核移植中可作为理想的供体细胞[2],并可作为临床上较为理想的干细胞治疗的细胞来源,因此有关其维持细胞干性功能的研究有非常重要的意义[3-4]。体细胞核移植(somatic cell nuclear transfer, SCNT)又称体细胞克隆,指利用显微操作技术培育出基因型与供体体细胞相同的克隆动物[5]。但是,目前SCNT技术在实际应用中仍然存在一些障碍,主要表现为几乎所有物种的克隆效率较低、克隆胚胎发育异常[6-8],以及克隆动物出生后出现免疫缺陷甚至早期死亡等现象[9]。其中SCNT胚胎的不完全重编程是导致克隆效率低的一个重要原因[10],而表观遗传修饰在其中有着决定性作用。
表观遗传机制指在不改变DNA序列的情况下,使基因发生遗传性变化并最终影响细胞表型的机制,包括DNA甲基化、组蛋白修饰、非编码RNA调控等[11],其中组蛋白修饰发挥着重要的调控作用,包括乙酰化、甲基化、磷酸化和泛素化等[12],不正常的表观遗传修饰会导致胚胎发育异常甚至胚胎死亡[13-15],研究指出供体细胞基因组的组蛋白H3K9甲基化修饰的改变是影响SCNT重编程的主要因素[16]。近年来,小分子抑制剂被普遍用于改善SCNT胚胎的表观遗传重编程[17]。小分子抑制剂毛壳素是组蛋白H3K9甲基化转移酶SUV39H1/2和G9A的特异性化学抑制剂[18],它会竞争性结合SUV39H1/2[19],与某些关键残基发生反应,下调H3K9 me2和H3K9 me3的表达[20-21],以此增强表观遗传重编程和自噬活性来提高核移植胚胎的克隆效率[22]。在人和哺乳动物中,NANOG、OCT4和SOX2被认为是调控干细胞多能性的3个重要的转录因子[23-25],并且已经被证实在胚胎发生过程中具有重要的调控作用[26]。但是, 目前对这几个基因的研究主要集中在维持干细胞干性和胚胎发育中的作用,而对其在体细胞核移植和表观遗传重编程过程中的研究鲜有报道。
本研究采用不同浓度的毛壳素处理gADSCs并检测对其生长活性的影响,筛选最适处理浓度和时间;通过检测适宜浓度毛壳素处理gADSCs后H3K9 me2和H3K9 me3的甲基化修饰相关酶转录表达水平,并检测H3K9 me2、H3K9 me3蛋白水平的变化,以研究毛壳素对细胞甲基化修饰的影响;通过检测药物处理gADSCs后胚胎多潜能相关基因转录水平,观察表观遗传修饰对胚胎发育多潜能性的影响。试验结果为研究组蛋白甲基化在山羊体细胞核移植过程中的分子机制及其对胚胎发育的影响提供试验依据。
1 材料与方法 1.1 试验材料1.1.1 试验细胞 本试验所用的gADSCs采自内蒙古自治区鄂托克旗阿尔巴斯绒山羊,为本实验室保存,且将其体外传代至第5代进行试验。
1.1.2 主要试剂 DMEM/F-12、PBS、标准胎牛血清(FBS)、胰蛋白酶均购自BI公司;RNAiso Plus、Prime ScriptTMRT Master Mix、RNA反转录试剂盒均购自TaKaRa公司;组蛋白提取试剂盒购自Epigentek公司;Tween-20、5×蛋白上样缓冲液、甲醇、12%SDS溶液、蛋白Marker、ECL显色液、PVDF膜、BCA蛋白定量检测试剂盒、TBS、电泳缓冲液、转膜液均购自康为世纪公司;明胶购自Sigma公司;CCK-8试剂盒购自上海翊圣生物有限公司;一抗H3K9 me2、H3K9 me3购自景杰生物公司;二抗购自美国Proteintech公司;毛壳素购自上海Selleck公司。
1.2 试验方法1.2.1 gADSCs细胞培养及药物处理 将连续培养的第5代gADSCs接种于含20% FBS的DMEM/F-12培养液的培养皿中,置于37 ℃、5% CO2和适宜湿度环境下培养,待细胞汇度达到60%左右,分别添加浓度为0、20、30和50 nmol·L-1的毛壳素处理24和48 h。
1.2.2 实时定量荧光PCR 收集不同浓度及不同时间毛壳素处理的细胞,用RNAiso Plus裂解液进行裂解,提取RNA并进行反转录获得cDNA。随后进行实时荧光定量PCR,对目的基因的mRNA水平表达变化情况进行检测。按照反转录试剂盒说明书配置20 μL反应体系:10 μL的SYBR Premix Ex Taq Ⅱ,0.4 μL的PCR Forward Primer,0.4 μL的PCR Reverse Primer,2 μL的RT reaction solution,7.2 μL的RNase Free dH2O;反应条件为: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 40个循环,对每个样品进行3次重复。GAPDH作内参对其进行标准化, 运用2-△△Ct方法计算目的基因的相对表达量。引物信息见表 1。
|
|
表 1 引物信息 Table 1 Primer information |
1.2.3 药物处理细胞活性检测 利用CCK-8法检测不同浓度梯度及时间梯度毛壳素处理的gADSCs的活性,试剂使用方法及活力计算按说明书进行。即在96孔板中接种不同浓度毛壳素处理的细胞悬液(每孔100 μL),在培养箱中预培养相应的时间后,向每孔加入10 μL CCK-8溶液,孵育2 h后,用酶标仪测量450 nm处吸光度值。
1.2.4 组蛋白的提取 取对数生长期的gADSCs细胞接种于培养皿中,细胞贴壁后,用毛壳素处理细胞,收集细胞于无酶管中,加入1 mL 1×预裂解液,吹打20~50次。10 000 r·min-1,4 ℃离心1 min,移去上清。用裂解液重悬浮细胞,在冰上孵育30 min。12 000 r·min-1,4 ℃离心5 min,转移上清至新管。每毫升上清加入0.3 mL Balance-DTT buffer。测定量蛋白后于-20 ℃保存(-80 ℃长久保存),避免反复冻融。
1.2.5 蛋白质印迹法(Western blot)检测 根据所需依次配制相应大小的分离胶和浓缩胶,向对应的胶孔中加入5 μL蛋白Marker和20 ng待测蛋白样品(根据之前测得的蛋白浓度确定体积),然后依次以90 V/30 min,120 V/90 min的条件进行电泳。电泳后,根据目的条带大小,切掉多余的胶,在转膜缓冲液中进行电转。电转结束后用TBST脱洗10 min,再用5%的脱脂奶粉封闭1 h。然后进行一抗、二抗孵育。孵育完成后,经ECL发光显色,用全自动化学发光成像系统进行曝光,对待测蛋白表达量的变化进行分析。
1.2.6 细胞免疫荧光检测 细胞药物处理前1 d将细胞爬片至小圆片上,待细胞融合至60%时,加药处理,然后用PBS清洗3次后加入4%多聚甲醛室温固定20 min。PBS清洗3次,加入0.1% Triton X-100冰上孵育15 min。PBS清洗3次后加入含有5% BSA的PBS封闭1 h。PBS清洗3次后用含有5% BSA的PBS按1∶300稀释H3K9 me3、H3K9 me2抗体后在4 ℃过夜孵育。PBS清洗3次, 避光加入PBS按1∶500配制的FITC荧光标记二抗, 在室温避光孵育1 h,PBS清洗3次,加入DAPI荧光染核, 在室温孵育10 min。PBS清洗3次后封片,激光共聚焦显微镜下观察。
1.3 数据分析本研究采用SPSS软件对试验数据进行独立样本T检验分析,P<0.05为差异显著,P>0.05为差异不显著,P<0.01为差异极显著,每组试验独立重复3次。图片使用Image J进行灰度分析。
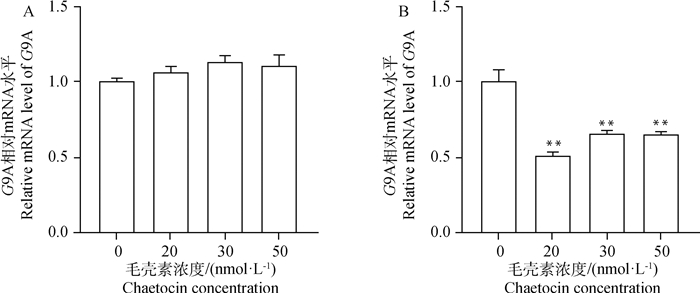
2 结果 2.1 毛壳素处理gADSCs最适浓度及时间的筛选为了选取出在对细胞活性影响最低的情况下且最大程度抑制gADSCs甲基化的毛壳素处理浓度及处理时间,本试验设置浓度梯度为0、20、30、50 nmol·L-1的毛壳素对gADSCs分别进行24和48 h的药物处理,采用CCK-8法检测不同处理组细胞的细胞活性,设定没有药物处理的细胞活性为100%。结果显示(图 1),20、30、50 nmol·L-1毛壳素处理gADSCs 24 h的细胞活性分别为85%、82%、66%,处理48 h的细胞活性分别为78%、55%、45%。实时定量PCR检测不同浓度和处理时间的毛壳素处理gADSCs的组蛋白甲基化转移酶G9A的转录,结果显示(图 2),20、30、50 nmol·L-1毛壳素处理gADSCs 24 h时,G9A的表达升高,分别为对照组的1.05倍、1.10倍、1.08倍;处理gADSCs 48 h时,G9A的表达极显著降低,分别为对照组的0.52倍、0.67倍、0.66倍(P<0.01)。综上结果,20 nmol·L-1毛壳素处理gADSCs 48 h为最适处理条件。

|
A. 毛壳素处理gADSCs 24 h;B. 毛壳素处理gADSCs 48 h。*.P<0.05;**. P<0.01,下同 A. gADSCs treated with chaetocin for 24 h; B. gADSCs treated with chaetocin for 48 h. *.P < 0.05;**. P < 0.01, the same as below 图 1 不同浓度毛壳素处理gADSCs 24和48 h的细胞活性 Fig. 1 Cell viability of gADSCs treated with different concentrations of chaetocin for 24 and 48 h |
|
A. 毛壳素处理gADSCs 24 h;B. 毛壳素处理gADSCs 48 h A. gADSCs treated with chaetocin for 24 h; B. gADSCs treated with chaetocin for 48 h 图 2 实时定量PCR检测不同浓度及时间毛壳素处理下G9A基因在gADSCs中的表达 Fig. 2 Real-time quantitative PCR to detect the expression of G9A gene in gADSCs under different concentrations and time of chaetocin treatment |
2.2.1 毛壳素处理gADSCs对组蛋白H3K9 me2和H3K9 me3相关酶的影响 通过qRT-PCR检测毛壳素处理gADSCs对组蛋白H3K9 me2和H3K9 me3相关酶的mRNA水平的影响。结果显示(图 3),处理组的H3K9 me2甲基化转移酶EHMT1、EHMT2和H3K9 me3甲基化转移酶SUV39H1、SUV39H2与未处理组相比显著降低,分别是对照组的0.59、0.65、0.34、0.36倍;H3K9 me2去甲基化酶KDM3A、KDM3B和H3K9 me3去甲基化酶KDM4B、KDM4D与未处理组相比表达量显著增高,分别是对照组的1.13、1.34、1.45、1.36倍。综上结果显示,适宜浓度毛壳素处理gADSCs后H3K9 me2和H3K9 me3相关甲基化酶表达显著降低,去甲基化酶表达显著升高。

|
图 3 H3K9 me2和H3K9 me3相关酶表达的实时定量PCR检测 Fig. 3 The real-time quantitative PCR detection of expression of H3K9 me2 and H3K9 me3 related enzymes |
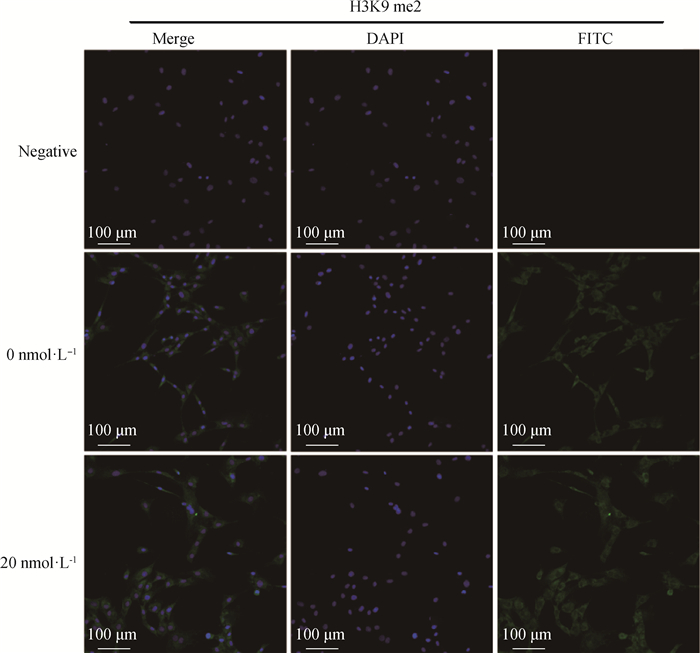
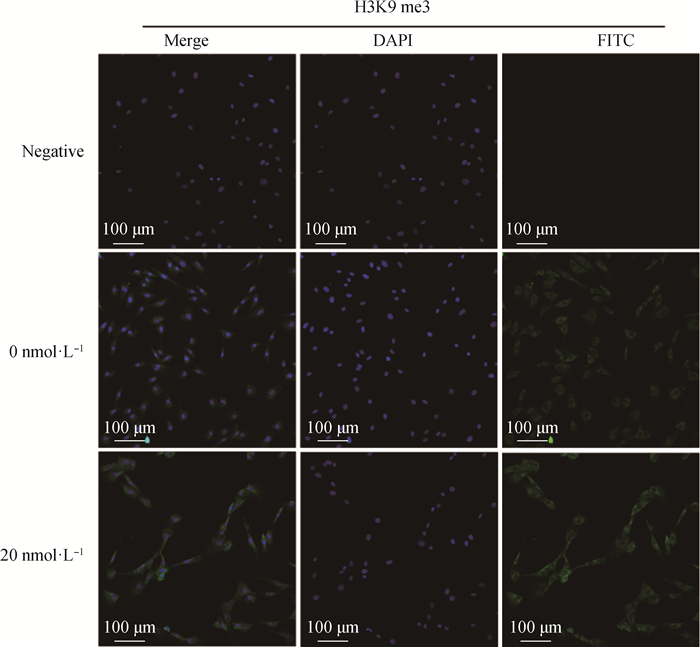
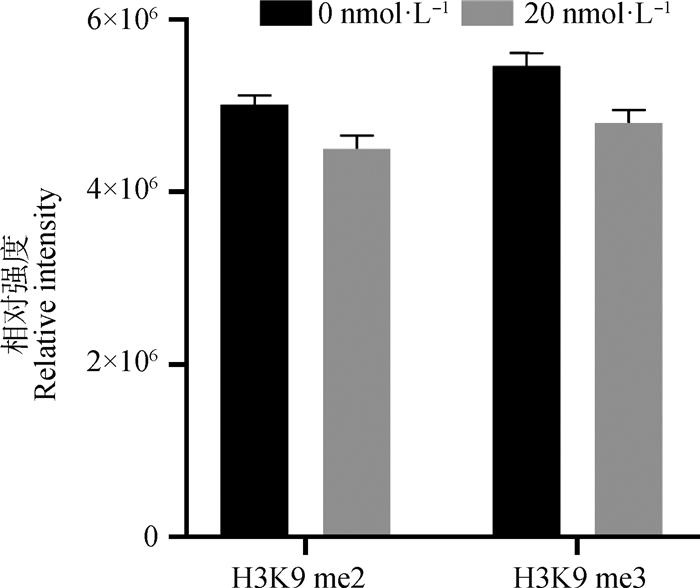
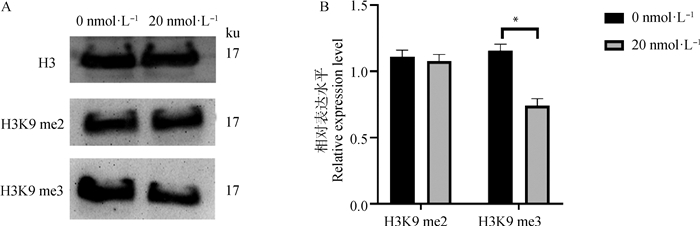
2.2.2 毛壳素处理gADSCs对H3K9 me2和H3K9 me3蛋白表达水平的影响 采用细胞免疫荧光技术检测了未处理组与处理组的gADSCs中H3K9 me2和H3K9 me3的表达情况(图 4~6)。结果显示,药物处理组细胞H3K9 me2和H3K9 me3免疫荧光强度均低于0 nmol·L-1对照组,但是影响不显著。提取毛壳素处理组的gADSCs的组蛋白,并用蛋白质印迹法检测细胞中组蛋白H3K9 me2和H3K9 me3的蛋白表达量,与未处理组进行对比分析(图 7)。结果显示,与未处理组相比,处理组的gADSCs中H3K9 me2水平无明显变化(P>0.05),而H3K9 me3水平显著性降低(P<0.05)。
|
图 4 毛壳素处理组和对照组细胞中组蛋白H3K9 me2的荧光强度比较 Fig. 4 Comparison of the fluorescence intensity of histone H3K9 me2 in gADSCs of the chaetocin-treated group and the control group |
|
图 5 毛壳素处理组和对照组细胞中组蛋白H3K9 me3的荧光强度比较 Fig. 5 Comparison of the fluorescence intensity of histone H3K9 me3 in gADSCs of the chaetocin-treated group and the control group |
|
图 6 H3K9 me2和H3K9 me3在绒山羊gADSCs中的荧光强度统计 Fig. 6 Fluorescence intensity statistics of H3K9 me2 and H3K9 me3 in gADSCs |
|
图 7 毛壳素处理对gADSCs组蛋白H3K9 me2和H3K9 me3表达的影响 Fig. 7 Effects of chaetocin treatment on the expression of histones H3K9 me2 and H3K9 me3 in gADSCs |
通过qRT-PCR检测毛壳素对gADSCs多潜能性基因SOX2、OCT4、NANOG的相对转录水平的影响。结果显示(图 8),处理组细胞多潜能性基因的表达有不同程度的上调。其中OCT4表达升高,是对照组的1.26倍;而NANOG表达量显著增高,是对照组的1.75倍(P<0.05);SOX2表达量极显著增高,是对照组的6.91倍(P<0.01)。

|
图 8 SOX2、OCT4、NANOG基因在gADSCs中的表达变化 Fig. 8 Expression changes of SOX2, OCT4, NANOG genes in gADSCs |
gADSCs是体细胞克隆技术中优良的核供体细胞,利用相关药物调控表观遗传修饰可以促进表观遗传重编程,有利于SCNT胚胎的发育。本研究通过利用组蛋白H3K9甲基化转移酶特异性化学抑制剂毛壳素处理gADSCs,观察gADSCs组蛋白甲基化修饰水平以及多潜能基因表达的改变,以便为提高绒山羊体细胞核移植的效率提供试验依据。
在研究毛壳素对猪成纤维细胞的影响中发现,20 nmol·L-1毛壳素处理24 h,对细胞生长活性影响最小[27],并且对甲基转移酶产生抑制作用,随着处理浓度的增加,毛壳素对甲基转移酶基因的抑制作用增强,但高浓度的毛壳素会引起细胞的大量死亡[28]。本试验结果证明,当药物处理浓度达50 nmol·L-1时,细胞生长活性明显降低,且20 nmol·L-1毛壳素处理gADSCs 48 h时对细胞活性影响较小,组蛋白甲基化转移酶G9A的表达显著降低,为最适处理浓度和时间。毛壳素处理猪SCNT胚胎后,H3K9 me3蛋白表达水平显著降低,甲基转移酶Suv39H1和Suv39H2的转录表达水平也显著降低[29],H3K9 me3在SCNT胚胎中的异常表达是表观遗传重编程和合子基因组激活(ZGA)的主要表观遗传障碍,导致小鼠和人类胚胎发育失败[30-32]。本试验通过实时定量荧光PCR检测毛壳素处理gADSCs后H3K9 me2和H3K9 me3相关酶的表达,发现不仅H3K9 me3甲基化转移酶G9A、Suv39H1、Suv39H2表达水平降低,而且去甲基化转移酶KDM4B、KDM4D表达水平显著升高,结果表明毛壳素对gADSCs组蛋白甲基化水平的降低作用可能是下调相关的甲基化转移酶表达以及上调特异性的去甲基化酶表达的协同机制下实现的[33-34]。H3K9 me2是H3K9 me3形成所必需的[35],但H3K9 me3的降低是否与H3K9 me2e有关还不清楚,所以为了进一步探究毛壳素处理后对细胞内H3K9 me2的影响,继续检测了H3K9 me2相关的甲基化转移酶EHMT1、EHMT2以及去甲基化转移酶KDM3A、KDM3B表达。结果表明,H3K9 me2相关的甲基化转移酶的表达与对照组相比均显著降低,去甲基化酶的表达显著升高。试验结果说明,毛壳素处理细胞后不仅能降低H3K9 me3相关甲基化酶的表达水平,还能影响H3K9 me3形成过程中H3K9 me2相关甲基化酶和去甲基化酶转录水平的表达,所以毛壳素对组蛋白甲基化相关酶表达的改变不仅仅单一作用在一种甲基化水平上,也可能对其形成过程中单甲基化、二甲基化、三甲基化都产生影响,共同调控组蛋白甲基化水平的改变。之后又对gADSCs中H3K9 me2和H3K9 me3的蛋白水平进行了研究,结果表明H3K9 me3的表达明显降低,但H3K9 me2表达没有明显变化,所以推测可能毛壳素处理细胞后只影响了H3K9 me2转化为H3K9 me3后的蛋白水平的表达,也可能因为表观遗传修饰是动态平衡的,转录水平基因的表达改变后又因其他位点的甲基化修饰或是其他修饰的协同作用造成甲基化水平的恢复,具体影响蛋白水平的机制有待深入研究。
高水平的H3K9 me3主要抑制SCNT胚胎的发育[36],而SOX2、OCT4、NANOG又是维持胚胎干细胞多能性的关键转录因子[37-38],但是目前对于表观遗传修饰对多能性基因影响的研究较少,所以本研究又对处理组进行了SOX2、OCT4、NANOG基因转录水平的检测,试验结果表明毛壳素处理gADSCs不仅可以降低细胞组蛋白甲基化水平,而且能提高胚胎干细胞多潜能相关基因的表达,其中处理组SOX2的表达极显著升高,SOX2在早期胚胎的生殖细胞、内细胞团、多能干细胞以及癌细胞中均可表达,它不仅能够调控组织细胞的增殖、决定细胞分化方向,还可以维持干细胞的全能性,它的缺失会导致胚胎停止发育[39-40],所以推测,高甲基化对胚胎发育的抑制作用可能是影响了SOX2的表达,从而抑制了核移植后重构胚的生长发育。
综合本研究结果证明,适当条件的毛壳素处理体细胞核移植供体细胞gADSCs,将有效降低组蛋白甲基化水平,改变表观遗传修饰作用,影响胚胎干细胞多潜能调控因子转录表达水平,有望提高SCNT胚胎重编程的效率。
4 结论本研究以绒山羊ADSCs为试验材料,结果显示浓度为20 nmol·L-1的毛壳素处理细胞48 h可以显著降低gADSCs组蛋白H3K9 me2和H3K9 me3相关甲基化酶的表达,提高去甲基化相关酶的表达;毛壳素处理gADSCs对H3K9 me2蛋白水平影响不显著,但有效降低H3K9 me3蛋白水平的表达。毛壳素处理细胞后,gADSCs细胞中维持胚胎干细胞多潜能性和成体细胞重编程的相关基因SOX2的表达显著升高,OCT4、NANOG表达也相对增高。
| [1] |
YOU H J, HAN S K. Cell therapy for wound healing[J]. J Korean Med Sci, 2014, 29(3): 311-319. DOI:10.3346/jkms.2014.29.3.311 |
| [2] |
BAKSH D, SONG L, TUAN R S. Adult mesenchymal stem cells: characterization, differentiation, and application in cell and gene therapy[J]. J Cell Mol Med, 2004, 8(3): 301-316. DOI:10.1111/j.1582-4934.2004.tb00320.x |
| [3] |
刘贤俊, 刘海亮, 景波, 等. 人不同组织间充质干细胞免疫调控能力的比较[J]. 同济大学学报: 医学版, 2017, 38(6): 12-17. LIU X J, LIU H L, JING B, et al. Comparison of immune regulatory properties of human mesenchymal stem cells derived from different tissues[J]. Journal of Tongji University: Medical Science, 2017, 38(6): 12-17. (in Chinese) |
| [4] |
祁旻龙, 朱良, CIERVO Y, 等. 过表达趋化因子受体2促进脂肪间充质干细胞对皮肤损伤的修复[J]. 同济大学学报: 医学版, 2018, 39(5): 23-27, 33. QI M L, ZHU L, CIERVO Y, et al. Overexpression of chemokine receptor type 2 in adipose mesenchymal stem cells promotes cutaneous wound healing[J]. Journal of Tongji University: Medical Science, 2018, 39(5): 23-27, 33. (in Chinese) |
| [5] |
杨旭琼, 吴珍芳, 李紫聪. 哺乳动物体细胞核移植表观遗传重编程研究进展[J]. 遗传, 2019, 41(12): 1099-1109. YANG X Q, WU Z F, LI Z C. Advances in epigenetic reprogramming of somatic cells nuclear transfer in mammals[J]. Hereditas (Beijing), 2019, 41(12): 1099-1109. (in Chinese) |
| [6] |
OGURA A, INOUE K, WAKAYAMA T. Recent advancements in cloning by somatic cell nuclear transfer[J]. Philos Trans Roy Soc B Biol Sci, 2013, 368(1609): 20110329. DOI:10.1098/rstb.2011.0329 |
| [7] |
GOUVEIA C, HUYSER C, EGLI D, et al. Lessons learned from somatic cell nuclear transfer[J]. Int J Mol Sci, 2020, 21(7): 2314. DOI:10.3390/ijms21072314 |
| [8] |
SRIRATTANA K, KANEDA M, PARNPAI R. Strategies to improve the efficiency of somatic cell nuclear transfer[J]. Int J Mol Sci, 2022, 23(4): 1969. DOI:10.3390/ijms23041969 |
| [9] |
LOI P, IUSO D, CZERNIK M, et al. A new, dynamic era for somatic cell nuclear transfer?[J]. Trends Biotechnol, 2016, 34(10): 791-797. DOI:10.1016/j.tibtech.2016.03.008 |
| [10] |
PEAT J R, REIK W. Incomplete methylation reprogramming in SCNT embryos[J]. Nat Genet, 2012, 44(9): 965-966. DOI:10.1038/ng.2393 |
| [11] |
ATLASI Y, STUNNENBERG H G. The interplay of epigenetic marks during stem cell differentiation and development[J]. Nat Rev Genet, 2017, 18(11): 643-658. DOI:10.1038/nrg.2017.57 |
| [12] |
DIXON J R, SELVARAJ S, YUE F. Topological domains in mammalian genomes identified by analysis of chromatin interactions[J]. Nature, 2012, 485(7398): 376-380. DOI:10.1038/nature11082 |
| [13] |
WANG J L, ZHANG M, ZHANG Y, et al. The histone demethylase JMJD2C is stage-specifically expressed in preimplantation mouse embryos and is required for embryonic development[J]. Biol Reprod, 2010, 82(1): 105-111. DOI:10.1095/biolreprod.109.078055 |
| [14] |
VARGHES B, DEL GAUDIO N, COBELLIS G, et al. KDM4 involvement in breast cancer and possible therapeutic approaches[J]. Front Oncol, 2021, 11: 750315. DOI:10.3389/fonc.2021.750315 |
| [15] |
ROSS P J, SAMPAIO R V. Epigenetic remodeling in preimplantation embryos: cows are not big mice[J]. Anim Reprod, 2018, 15(3): 204-214. DOI:10.21451/1984-3143-AR2018-0068 |
| [16] |
MATOBA S, LIU Y T, LU F L, et al. Embryonic development following somatic cell nuclear transfer impeded by persisting histone methylation[J]. Cell, 2014, 159(4): 884-895. DOI:10.1016/j.cell.2014.09.055 |
| [17] |
ZHANG Y M, GAO E E, WANG Q Q, et al. Effects of histone methyltransferase inhibitor chaetocin on histone H3K9 methylation of cultured ovine somatic cells and development of preimplantation cloned embryos[J]. Reprod Toxicol, 2018, 79: 124-131. DOI:10.1016/j.reprotox.2018.06.006 |
| [18] |
JAFARPOUR F, GHAZVINI ZADEGAN F, OSTADHOSSEINI S, et al. siRNA inhibition and not chemical inhibition of Suv39 h1/2 enhances pre-implantation embryonic development of bovine somatic cell nuclear transfer embryos[J]. PLoS One, 2020, 15(6): e0233880. DOI:10.1371/journal.pone.0233880 |
| [19] |
GREINER D, BONALDI T, ESKELAND R, et al. Identification of a specific inhibitor of the histone methyltransferase SU(VAR)3-9[J]. Nat Chem Biol, 2005, 1(3): 143-145. DOI:10.1038/nchembio721 |
| [20] |
SOUFI A, DONAHUE G, ZARET K S. Facilitators and impediments of the pluripotency reprogramming factors' initial engagement with the genome[J]. Cell, 2012, 151(5): 994-1004. DOI:10.1016/j.cell.2012.09.045 |
| [21] |
KREIMER A, ASHUACH T, INOUE F, et al. Massively parallel reporter perturbation assays uncover temporal regulatory architecture during neural differentiation[J]. Nat Commun, 2022, 13(1): 1504. DOI:10.1038/s41467-022-28659-0 |
| [22] |
STILLMAN B. Histone modifications: insights into their influence on gene expression[J]. Cell, 2018, 175(1): 6-9. DOI:10.1016/j.cell.2018.08.032 |
| [23] |
PAN G J, THOMSON J A. Nanog and transcriptional networks in embryonic stem cell pluripotency[J]. Cell Res, 2007, 17(1): 42-49. DOI:10.1038/sj.cr.7310125 |
| [24] |
AVILION A A, NICOLIS S K, PEVNY L H, et al. Multipotent cell lineages in early mouse development depend on SOX2 function[J]. Genes Dev, 2003, 17(1): 126-140. DOI:10.1101/gad.224503 |
| [25] |
CHEN J, CHEN X L, LI M, et al. Hierarchical Oct4 binding in concert with primed epigenetic rearrangements during somatic cell reprogramming[J]. Cell Rep, 2016, 14(6): 1540-1554. DOI:10.1016/j.celrep.2016.01.013 |
| [26] |
LOH Y H, WU Q, CHEW J L, et al. The Oct4 and Nanog transcription network regulates pluripotency in mouse embryonic stem cells[J]. Nat Genet, 2006, 38(4): 431-440. DOI:10.1038/ng1760 |
| [27] |
李海艳. 毛壳素对德保黑猪核移植胚胎体外发育潜能影响的初步研究[D]. 南宁: 广西大学, 2017. LI H Y. Effects of chaetocin on the in vitro development of Debao black porcine somatic cell nuclear transfer embryos[D]. Nanning: Guangxi University, 2017. (in Chinese) |
| [28] |
王婷, 李海艳, 刘晨, 等. 毛壳素对猪核移植胚胎体外发育潜能的影响研究[J]. 中国兽医科学, 2021, 51(6): 792-797. WANG T, LI H Y, LIU C, et al. Effects of chaetocin treatment on in vitro developmental potential of nuclear transfer embryos of pig[J]. Chinese Veterinary Science, 2021, 51(6): 792-797. (in Chinese) |
| [29] |
JEONG P S, SIM B W, PARK S H, et al. Chaetocin improves pig cloning efficiency by enhancing epigenetic reprogramming and autophagic activity[J]. Int J Mol Sci, 2020, 21(14): 4836. DOI:10.3390/ijms21144836 |
| [30] |
EILERTSEN K J, POWER R A, HARKINS L L, et al. Targeting cellular memory to reprogram the epigenome, restore potential, and improve somatic cell nuclear transfer[J]. Anim Reprod Sci, 2007, 98(1-2): 129-146. DOI:10.1016/j.anireprosci.2006.10.019 |
| [31] |
CHEN P R, REDEL B K, KERNS K C, et al. Challenges and considerations during in vitro production of porcine embryos[J]. Cells, 2021, 10(10): 2770. DOI:10.3390/cells10102770 |
| [32] |
JEONG P S, YANG H J, PARK S H, et al. Combined chaetocin/trichostatin a treatment improves the epigenetic modification and developmental competence of porcine somatic cell nuclear transfer embryos[J]. Front Cell Dev Biol, 2021, 9: 709574. DOI:10.3389/fcell.2021.709574 |
| [33] |
CHUNG Y G, MATOBA S, LIU Y T, et al. Histone demethylase expression enhances human somatic cell nuclear transfer efficiency and promotes derivation of pluripotent stem cells[J]. Cell Stem Cell, 2015, 17(6): 758-766. DOI:10.1016/j.stem.2015.10.001 |
| [34] |
WENG X G, CAI M M, ZHAN Y T, et al. Improvement in the in vitro development of cloned pig embryos after kdm4a overexpression and an H3K9 me3 methyltransferase inhibitor treatment[J]. Theriogenology, 2020, 146: 162-170. DOI:10.1016/j.theriogenology.2019.11.027 |
| [35] |
SAMPAIO R V, SANGALLI J R, DE BEM T H C, et al. Catalytic inhibition of H3K9 me2 writers disturbs epigenetic marks during bovine nuclear reprogramming[J]. Sci Rep, 2020, 10(1): 11493. DOI:10.1038/s41598-020-67733-9 |
| [36] |
RUAN Z Y, ZHAO X, LI Z D, et al. Effect of sex differences in donor foetal fibroblast on the early development and DNA methylation status of buffalo (Bubalus bubalis) nuclear transfer embryos[J]. Reprod Domest Anim, 2019, 54(1): 11-22. DOI:10.1111/rda.13286 |
| [37] |
YU J Y, VODYANIK M A, SMUGA-OTTO K, et al. Induced pluripotent stem cell lines derived from human somatic cells[J]. Science, 2007, 318(5858): 1917-1920. DOI:10.1126/science.1151526 |
| [38] |
LEWITZKY M, YAMANAKA S. Reprogramming somatic cells towards pluripotency by defined factors[J]. Curr Opin Biotechnol, 2007, 18(5): 467-473. DOI:10.1016/j.copbio.2007.09.007 |
| [39] |
HUSSENET T, DALI S, EXINGER J, et al. SOX2 is an oncogene activated by recurrent 3q26.3 amplifications in human lung squamous cell carcinomas[J]. PLoS One, 2010, 5(1): e8960. DOI:10.1371/journal.pone.0008960 |
| [40] |
WILBERTZ T, WAGNER P, PETERSEN K, et al. SOX2 gene amplification and protein overexpression are associated with better outcome in squamous cell lung cancer[J]. Mod Pathol, 2011, 24(7): 944-953. DOI:10.1038/modpathol.2011.49 |
(编辑 郭云雁)



