2. 中国农业大学动物医学院, 北京 100193;
3. 北京大学第三医院实验动物中心, 北京 100191
2. College of Veterinary Medicine, China Agricultural University, Beijing 100193, China;
3. Animal Experiment Center, Peking University Third Hospital, Beijing 100191, China
沙门菌病(Salmonellosis)是由沙门菌感染引起的人与多种动物共患的传染病,对养殖业造成严重的危害。此外,由于人群感染率的不断上升也使得沙门菌病成为影响全球的公共卫生问题。由于O和H抗原的多样性导致沙门菌拥有丰富的血清型,目前已经超过2 600余种[1]。肠道沙门菌肠道亚种中的鼠伤寒沙门菌(Salmonella Typhimurium)和肠炎沙门菌(Salmonella Enteritidis)是引起人畜沙门菌病的两大血清型[2-3]。鼠伤寒沙门菌是引起食源性疾病最常见血清型,可导致人的急性胃肠炎,且能够引起小鼠发生全身伤寒性疾病[4]。据文献记载,沙门菌导致的食物中毒约占58.05%,鼠伤寒沙门菌和肠炎沙门菌各分别21.59%和16.81%[5]。
抗菌肽(antimicrobial peptides, AMPs)广泛分布于动物体内各种黏液和肠道黏液中,在病原入侵时可以被诱导表达,对细菌、病毒、真菌等微生物具有杀灭的作用[6-8]。据报道,AMPs对抵抗沙门菌在巨噬细胞内生存、胃肠道持续定殖以及感染也有着至关重要的作用[9-10]。前期研究发现,由sapABCDF操纵子编码的sap(ABC)转运蛋白与沙门菌的毒力相关[11-12]。ABC转运蛋白在真核和原核生物细胞中广泛存在,底物是离子、氨基酸、多糖、多肽等物质,许多细菌ABC转运蛋白被证实与细菌耐药性有关[13-15]。为了进一步研究沙门菌转运蛋白SapC的生物学功能,本研究使用λ-同源重组技术构建鼠伤寒沙门菌sapC基因缺失株;对sapC基因缺失株的生长特性、多黏菌素B敏感性、酸敏感性、生物被膜形成能力、胞内增殖以及小鼠体内毒力进行研究,系统分析了sapC基因的功能,为阐释沙门菌致病机制奠定基础。
1 材料与方法 1.1 主要试验材料鼠伤寒沙门菌S. Typhimurium ATCC 14028s,质粒pKD46、pKD3、pCP20及互补质粒pBR322均由本实验室保存;鼠源巨噬细胞J774A.1,由中国农业大学吴清民教授实验室馈赠;BALB/c小鼠购自北京维通利华实验动物有限公司;PCR Mixture、T4连接酶、DNA Marker、限制性内切酶、凝胶回收试剂盒等均购自北京擎科生物技术有限公司。
1.2 引物设计与合成根据鼠伤寒沙门菌基因组序列,借助Primer 5.0设计3对引物。引物F1、F2由两部分组成,划线部分用于定位sapC序列,未划线部分与氯霉素抗性基因(cat)同源,最终扩增出带有sapC同源臂的氯霉素抗性序列。引物F3、F4与sapC基因同源且位于F1、F2外侧,用于缺失基因sapC的鉴定。引物F5、F6用于sapC基因扩增和互补菌株构建。上述引物均由上海生工生物工程技术服务有限公司合成(表 1)。
|
|
表 1 PCR扩增所用引物 Table 1 The primer sequences for PCR amplification |
使用引物F1和F2扩增质粒pKD3上的氯霉素基因片段,胶回收后溶于ddH2O中;将携带有pKD46的S. Typhimurium ATCC 14028s培养至OD600 nm 0.4~0.6时加入阿拉伯糖使其终浓度为30 mmol·L-1,30 ℃诱导1 h后冰浴30 min,并用预冷的去离子水洗涤3次,最后将获得的感受态细胞溶于去离子水。将带有sapC同源臂的氯霉素基因电击转化入上步所得感受态细胞中,复苏后涂于氯霉素平板,培养18~24 h,挑取单菌落振荡培养2 h,经PCR鉴定为阳性的菌液于42 ℃消除热敏质粒pKD46,最后通过氨苄抗性平板筛选和PCR鉴定获得一次重组菌株。将质粒pCP20电转进一次重组菌株中,用以消除氯霉素基因,同样经热激消除质粒,再次筛选后获得sapC基因缺失菌株SMΔsapC,并进行PCR鉴定。
利用质粒pBR322构建sapC克隆载体,继而电转到菌株SMΔsapC中,经PCR鉴定为阳性的菌株命名为SMΔsapC: : sapC,即为互补菌株[16]。
1.4 重组菌生长特性观察为测定SM、SMΔsapC和SMΔsapC: : sapC三种沙门菌株的生长特征,取单菌落于10 mL LB液体培养基中,37 ℃ 200 r·min-1震荡培养,在第2、4、6、8、10、12 h分别取样检测OD600 nm值,绘制3种菌株的生长曲线。
1.5 重组菌对多黏菌素B敏感性试验挑取SM、SMΔsapC和SMΔsapC: : sapC单菌落接种于10 mL LB培养基培养至OD600 nm=0.4~0.6,4 ℃保存备用。将各菌液稀释至106 CFU·mL-1,取100 μL加入到96孔板中,并加入100 μL 0.3 μg·mL-1的多黏菌素B,混匀后,放入37 ℃恒温箱孵育1 h后计数,计算各菌株的存活率。
1.6 重组菌酸敏感性试验使用生理盐水调整SM、SMΔsapC和SMΔsapC: : sapC菌液浓度为1×108 CFU·mL-1;各菌液平均分成两管,其中一管离心后,加入新鲜的LPM培养基(pH为3)重悬菌体,放置37 ℃恒温箱1 h后取出计数;另一管离心后先加入pH为5.5的LPM培养基适应1 h,离心后再加入pH为3的LPM培养基,放置37 ℃恒温箱1 h后取出计数,分析各菌株存活率。
1.7 重组菌生物被膜形成能力检测取SM、SMΔsapC和SMΔsapC: : sapC菌液以1%接入TSB液体培养基并培养至OD600 nm=0.5,以200 μL·孔-1加入96孔板中,置于37 ℃ 48 h后弃去菌液,PBS洗涤3次,加入200 μL甲醇固定15 min后弃去上清,用0.1%结晶紫室温染色5 min后冲洗;再加33%冰醋酸溶解结晶紫,测量OD630 nm值,根据ODc临界值判定生物被膜黏附强度,ODc为OD空白孔平均值加3倍标准差,OD≤ODc为不黏附,ODc<OD≤2ODc为弱黏附,2ODc<OD≤4ODc为中等黏附,OD>4ODc为强黏附。
1.8 重组菌胞内存活能力的测定将生长状态良好的鼠源巨噬细胞J774A.1,以1×105·孔-1铺于24孔板;用培养至OD600 nm 0.4~ 0.6的SM、SMΔsapC和SMΔsapC: : sapC细菌以MOI=10感染J774A.1细胞,孵育1 h后弃上清,加入含20 μg·mL-1庆大霉素的DMEM杀死游离的细菌。分别在感染后1、4、8、12、16、24 h加入1% Tritoxn-100,并对释放出的细菌平板计数,绘制各菌株在胞内的增殖曲线。
1.9 小鼠体内毒力测定沙门菌为胞内寄生病原菌,在机体内增殖存留时间及脏器载菌量,是评价毒力的重要指标。购买20只BALB/c小鼠,每组10只,分别以104剂量腹腔注射SM、SMΔsapC菌液,逐日观察记录小鼠死亡日期和数量。接种后7 d,剖杀SMΔsapC组3只小鼠,同对照组第7日死亡的小鼠,分别进行脾和肝的载菌量测定。
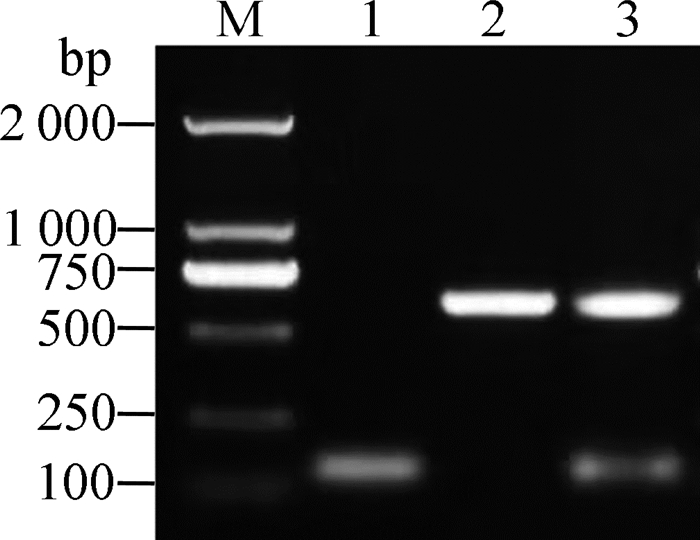
2 结果 2.1 sapC基因缺失株和互补菌株的构建与鉴定使用同源臂引物F1、F2,扩增质粒pKD3中的氯霉素基因序列,并电转进携带质粒pKD46的亲本菌中,经热激消除质粒pKD46;再电转入质粒pCP20敲除氯霉素基因,获得重组菌株SMΔsapC,PCR鉴定结果如图 1,基因缺失株SMΔsapC扩增条带为160 bp,SM扩增条带为891 bp,互补菌株扩增出两条目的条带(160和891 bp),与预期相符,表明成功构建了sapC基因缺失菌株和互补菌株。
|
M.DL5000;1.sapC基因缺失株;2.亲本菌株;3.互补菌株 M.DL5000: 1.sapC gene deleted strain; 2.Wild strain; 3. Supplemented strain 图 1 重组菌的PCR鉴定 Fig. 1 Identification of recombinant strains by PCR |
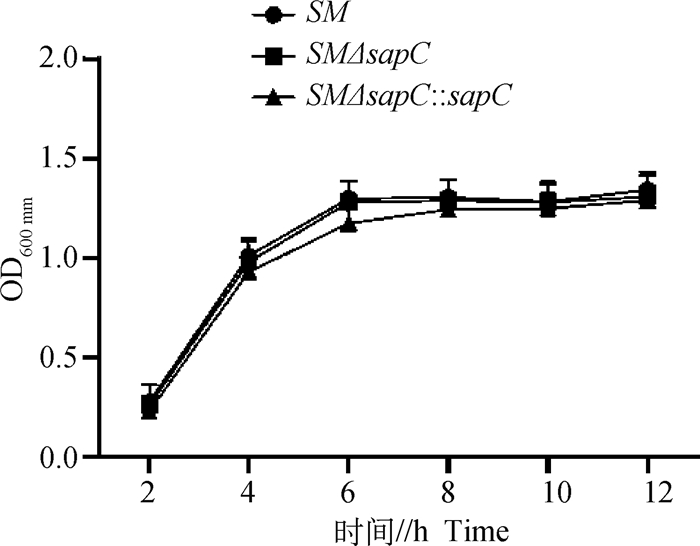
取LB中培养不同时间的SM、SMΔsapC和SMΔsapC: : sapC菌液测定OD600 nm,绘制生长曲线。结果如图 2所示,各菌株总体生长无明显差异,说明sapC基因不影响沙门菌的生长。
|
图 2 基因缺失株SM△sapC、互补菌株SM△sapC: : sapC与亲本株SM的生长特性 Fig. 2 Growth culves of SM△sapC, SM and SM△sapC: : sapC |
使用终浓度为0.15 μg·mL-1多黏菌素B作用各菌株1 h后,活菌计数。结果如图 3所示,在多黏菌素B作用下sapC基因缺失株可以正常增殖,且活菌数与亲本菌株相比差异显著。说明sapC基因对鼠伤寒沙门菌的多黏菌素B耐受性起到负调控作用。

|
与SM组相比,*.P < 0.05 Compared with SM group, *. P < 0.05 图 3 重组菌多黏菌素B敏感性试验结果 Fig. 3 Polymyxin B sensitivity test of recombinant strains |
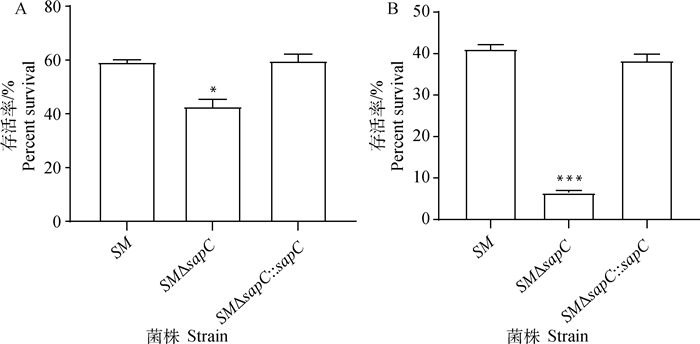
将各重组菌进行酸适应和酸性未适应两种处理。酸适应是将各菌株首先暴露于pH为5.5的LPM液体培养基,然后暴露于pH为3的LPM液体培养基中;酸性未适应是在相同条件下将各菌液在原培养基培养后直接暴露于pH为3的LPM培养基。两种情况下分别计算存活率。结果如图 4所示,在酸适应情况下,SMΔsapC的存活率为40%左右,与SM相比差异显著;在酸性未适应情况下,SMΔsapC的存活率仅为6%左右,与SM相比差异极显著,互补株酸耐受能力得到回复。由此表明sapC基因的缺失可以明显降低鼠伤寒沙门菌的耐酸能力。
|
A.酸适应处理(经pH5.5过渡至pH3培养);B.酸性未适应处理(直接入pH3培养)。与SM组相比, *. P < 0.05;***. P < 0.001 A. Acid adaptation (with pH transition from 5.5 to 3); B.Acid inadaptation (directly cultured at pH 3). Compared with SM group, *. P < 0.05; ***. P < 0.001 图 4 重组菌酸敏感性试验结果 Fig. 4 Acid sensitivity test results of recombinant strains |
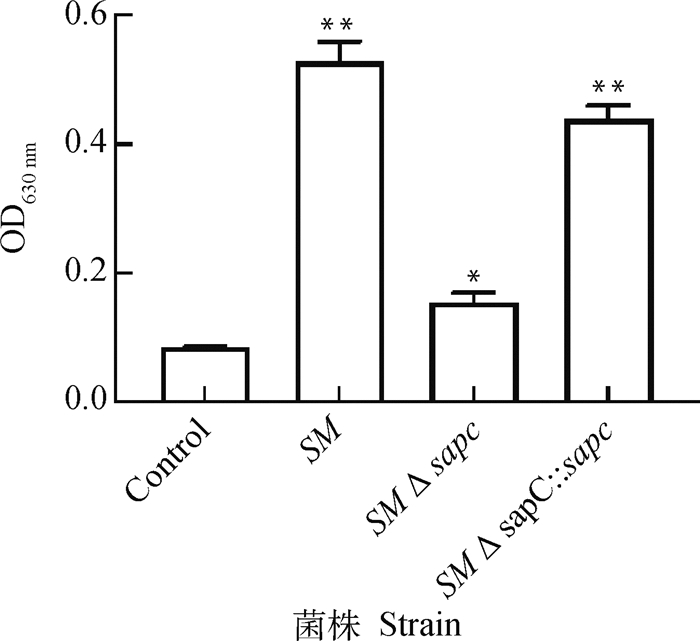
借助结晶紫染色法定量分析菌株SM、SMΔsapC和SMΔsapC: : sapC的生物被膜形成能力,结果表明,亲本菌株SM和SMΔsapC: : sapC为强黏附,SMΔsapC为弱黏附,基因缺失菌株SMΔsapC的生物被膜形成量仅为亲本菌株的30%(图 5)。表明sapC基因参与沙门菌的生物被膜形成。
|
与对照组相比, *. P < 0.05;**. P < 0.01 Compared with the control group, *. P < 0.05; **. P < 0.01 图 5 重组菌生物被膜形成能力分析 Fig. 5 Analysis of biofilm formation abilities of recombinant strains |
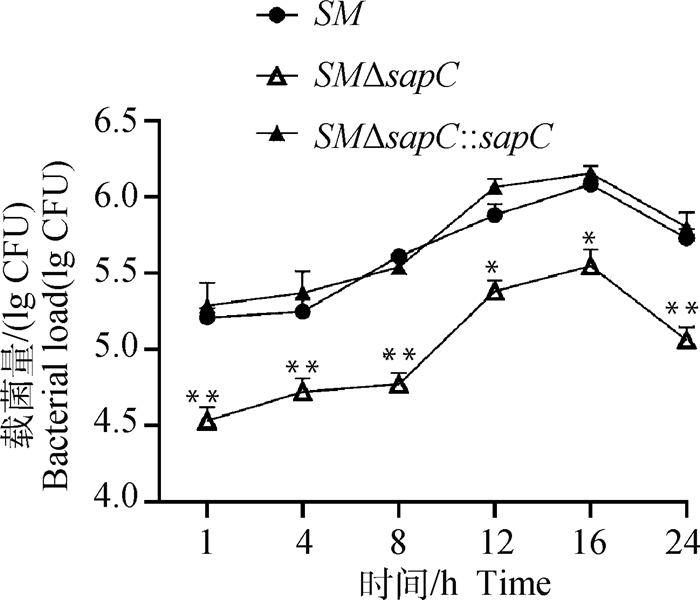
将各菌株以MOI=10感染巨噬细胞,并在不同时间点取样,对胞内沙门菌平板计数。结果如图 6所示,SMΔsapC胞内增殖能力显著低于亲本菌株;在感染后的1和24 h,基因缺失菌株SMΔsapC的胞内载菌量降低了约10倍,sapC互补株的胞内定殖能力回复。表明sapC能够降低沙门菌的胞内存活能力。
|
与SM组相比, *. P < 0.05;**. P < 0.01 Compared with SM group, *. P < 0.05; **. P < 0.01 图 6 重组菌胞内增殖能力测定结果 Fig. 6 Intracellular viability tests of recombinant strains |
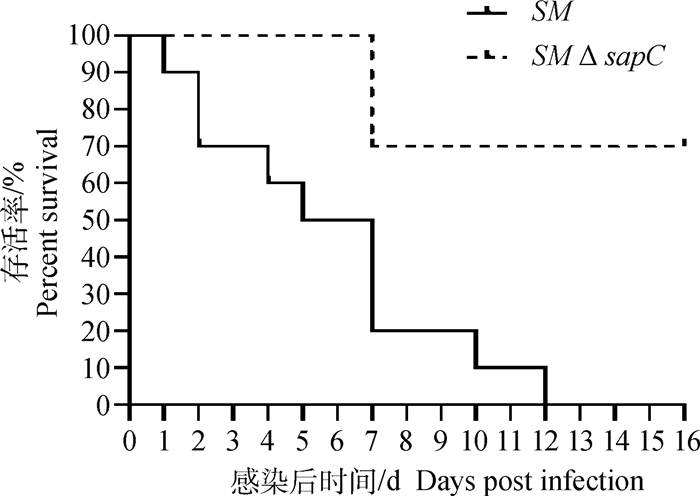
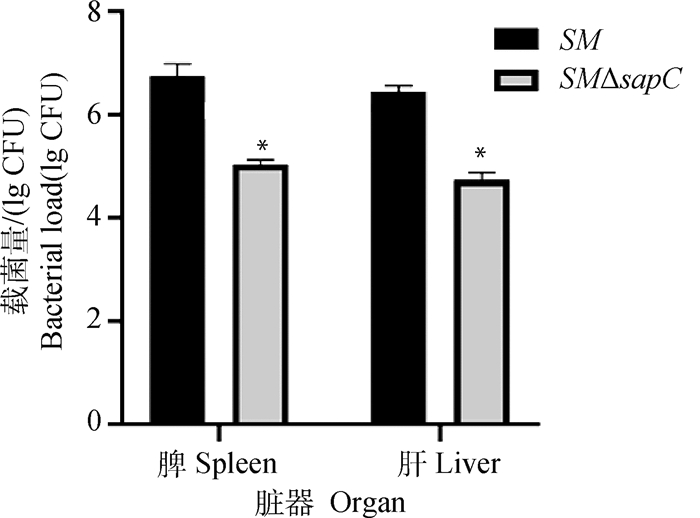
取亲本菌株SM和基因缺失菌株SMΔsapC,进行小鼠试验,感染后连续记录小鼠死亡数量。如图 7所示,接种SM的小鼠在12 d内全部死亡,而接种SMΔsapC的小鼠16 d内的存活率为100%(注:SMΔsapC接种组,第7日剖杀3只小鼠,用于脏器载菌量测定);接种7 d时,SMΔsapC组小鼠脾载菌量比SM组小鼠载菌量低近2个数量级(图 8)。表明sapC基因的缺失降低了沙门菌的毒力。
|
与SM组相比, *. P < 0.05 Compared with SM group, *. P < 0.05 图 7 小鼠死亡曲线 Fig. 7 Survival curves of mice |
|
Bacterial loads in mice spleen and liver of recombinant strains 图 8 重组菌小鼠体内载菌量测定 |
作为兼性胞内寄生菌,沙门菌可通过生成内噬体的形式保护其在细胞内的增殖和游走[17-18]。沙门菌能引起多种动物发病,导致宿主局部和全身感染,还能导致隐性和持续性感染,这给该病的防治带来很大难度[19-20]。基因工程的方法能弱化菌株毒力的同时保持较好的侵袭力,这为沙门菌病的防控提供了方向[21-22]。因此研究沙门菌的致病机制,可为沙门菌病提供可能的防控策略。本文对鼠伤寒沙门菌ABC转运膜蛋白SapC与其毒力的关系进行研究,探索该蛋白在沙门菌致病中的作用,为沙门菌病新型疫苗的研究奠定基础。
为了分析sapC基因的生物学功能,本研究利用Red同源重组技术构建了sapC基因缺失菌株。结果发现,多黏菌素B刺激下,缺失株的活菌数较亲本菌株增加,说明sapC对鼠伤寒沙门菌抗菌肽耐受起到负调控作用。但其酸性耐受能力、生物被膜形成能力较亲本菌株显著降低。细胞感染试验和小鼠体内毒力试验更加说明了sapC基因缺失菌株的胞内存活能力及其毒力均有所降低,推测sapC参与鼠伤寒沙门菌的致病过程。
生物被膜(biofilm,BF)是由细菌在生长过程中分泌的多糖基质、纤维蛋白、脂质蛋白、DNA等将菌体包裹形成的大量细菌聚集膜样物[23-24]。BF是帮助病原菌抵御外来药物和宿主免疫系统杀伤的有效物理屏障,对细菌的存活具有重要意义[25]。沙门菌在生长和感染过程中也能在多种附着物表面产生BF,这是细菌能在恶劣环境下生存甚至导致持续感染的一方面原因[26-27]。本研究发现sapC基因缺失能够抑制鼠伤寒沙门菌BF的形成。
沙门菌在胞内最常见的恶劣条件之一是酸胁迫,因此感知和响应酸胁迫的能力对其生存至关重要[28]。沙门菌通过复杂的酸存活系统响应酸性pH变化,统称为耐酸反应(ATR)[26]。当鼠伤寒沙门菌在亚致死pH(pH 4.5~5.5)下体外生长一代后,沙门菌便获得在极端酸性条件下(pH 3)存活的能力[29-30]。本研究发现,在酸适应情况下,SM下sapC的存活率为40%左右,与SM相比差异显著;在酸性未适应情况下,SMΔsapC的存活率仅为6%左右,与SM相比差异极显著,由此表明SapC能显著降低鼠伤寒沙门菌在酸性环境中的存活能力。体内毒力试验显示,注射鼠伤寒沙门菌亲本株SM的小鼠在12 d内全部死亡,而注射基因缺失株SM△sapC的小鼠存活率为100%。此外,注射基因缺失菌株的小鼠肝和脾的细菌载量都明显低于亲本菌株。
4 结论利用Red同源重组技术构建了S. Typhimurium sapC基因缺失菌株SMΔsapC和互补株SMΔsapC: : sapC,对其生物学功能进行了研究。结果表明,sapC基因缺失不影响沙门菌的正常生长,但能显著降低其酸性耐受能力、生物被膜形成能力,并能降低鼠伤寒沙门菌胞内存活能力及毒力。综上所述,sapC基因与鼠伤寒沙门菌的致病性密切相关,有望作为新型弱毒疫苗研究的候选因子。
| [1] |
FUCHE F J, SOW O, SIMON R, et al. Salmonella Serogroup C: current status of vaccines and why they are needed[J]. Clin Vaccine Immunol, 2016, 23(9): 737-745. DOI:10.1128/CVI.00243-16 |
| [2] |
ANSARI N, YAZDIAN-ROBATI R, SHAHDORDIZADEH M, et al. Aptasensors for quantitative detection of Salmonella Typhimurium[J]. Anal Biochem, 2017, 533: 18-25. DOI:10.1016/j.ab.2017.06.008 |
| [3] |
GARAI P, GNANADHAS D P, CHAKRAVORTTY D. Salmonella enterica serovars Typhimurium and Typhi as model organisms: revealing paradigm of host-pathogen interactions[J]. Virulence, 2012, 3(4): 377-388. DOI:10.4161/viru.21087 |
| [4] |
SUN H H, WAN Y P, DU P C, et al. The epidemiology of monophasic Salmonella Typhimurium[J]. Foodborne Pathog Dis, 2020, 17(2): 87-97. DOI:10.1089/fpd.2019.2676 |
| [5] |
QI X L, LI P, XU X G, et al. Epidemiological and molecular investigations on Salmonella responsible for gastrointestinal infections in the Southwest of Shanghai from 1998 to 2017[J]. Front Microbiol, 2019, 10: 2025. DOI:10.3389/fmicb.2019.02025 |
| [6] |
HANCOCK R E W, SCOTT M G. The role of antimicrobial peptides in animal defenses[J]. Proc Natl Acad Sci U S A, 2000, 97(16): 8856-8861. DOI:10.1073/pnas.97.16.8856 |
| [7] |
DE CESARE C B, CRISTY S A, GARSIN D A, et al. Antimicrobial peptides: a new frontier in antifungal therapy[J]. mBio, 2020, 11(6): e02123-20. |
| [8] |
GALLO R L, MURAKAMI M, OHTAKE T, et al. Biology and clinical relevance of naturally occurring antimicrobial peptides[J]. J Allergy ClinImmunol, 2002, 110(6): 823-831. DOI:10.1067/mai.2002.129801 |
| [9] |
BADER M W, NAVARRE W W, SHIAU W, et al. Regulation of Salmonella typhimurium virulence gene expression by cationic antimicrobial peptides[J]. Mol Microbiol, 2003, 50(1): 219-230. DOI:10.1046/j.1365-2958.2003.03675.x |
| [10] |
BRODSKY I E, ERNST R K, MILLER S I, et al. mig-14 is a Salmonella gene that plays a role in bacterial resistance to antimicrobial peptides[J]. J Bacteriol, 2002, 184(12): 3203-3213. DOI:10.1128/JB.184.12.3203-3213.2002 |
| [11] |
RODAS P I, CONTRERAS I, MORA G C. Salmonella enterica serovar Typhi has a 4. 1 kb genetic island inserted within the sapABCDF operon that causes loss of resistance to the antimicrobial peptide protamine[J]. J Antimicrob Chemother, 2010, 65(8): 1624-1630. DOI:10.1093/jac/dkq197 |
| [12] |
PARRA-LOPEZ C, NAER M T, GROISMAN E A. Molecular genetic analysis of a locus required for resistance to antimicrobial peptides in Salmonella typhimurium[J]. EMBO J, 1993, 12(11): 4053-4062. DOI:10.1002/j.1460-2075.1993.tb06089.x |
| [13] |
ROEPE P D, WEI L Y, HOFFMAN M M, et al. Altered drug translocation mediated by the MDR protein: direct, indirect, or both?[J]. J Bioenerg Biomembr, 1996, 28(6): 541-555. DOI:10.1007/BF02110444 |
| [14] |
KRAUSS S, ZIPPERER A, WIRTZ S, et al. Secretion of and self-resistance to the novel fibupeptide antimicrobial Lugdunin by distinct ABC transporters in Staphylococcus lugdunensis[J]. Antimicrob Agents Chemother, 2020, 65(1): e01734-20. |
| [15] |
YAMAGISHI A, NAKANO S, YAMASAKI S, et al. An efflux inhibitor of the MacAB pump in Salmonella enterica serovar typhimurium[J]. Microbiol Immunol, 2020, 64(3): 182-188. DOI:10.1111/1348-0421.12765 |
| [16] |
曹莉, 孙路, 孔梓安, 等. 肠炎沙门氏菌yjeA基因缺失株的构建及其生物学功能测定[J]. 北京农学院学报, 2020, 35(2): 16-20. CAO L, SUN L, KONG Z A, et al. Construction and characterization of yjeA gene deletion strain of Salmonella enteritidis[J]. Journal of Beijing University of Agriculture, 2020, 35(2): 16-20. (in Chinese) |
| [17] |
ARANDA C M A, SWANSON J A, LOOMIS W P, et al. Salmonella Typhimurium activates virulence gene transcription within acidified macrophage phagosomes[J]. Proc Natl Acad Sci U S A, 1992, 89(21): 10079-10083. DOI:10.1073/pnas.89.21.10079 |
| [18] |
GUTIÉRREZ S, FISCHER J, GANESAN R, et al. Salmonella Typhimurium impairs glycolysis-mediated acidification of phagosomes to evade macrophage defense[J]. PLoS Pathog, 2021, 17(9): e1009943. DOI:10.1371/journal.ppat.1009943 |
| [19] |
YUKI K E, EVA M M, RICHER E, et al. Suppression of hepcidin expression and iron overload mediate Salmonella susceptibility in Ankyrin 1 ENU-induced mutant[J]. PLoS One, 2013, 8(2): e55331. DOI:10.1371/journal.pone.0055331 |
| [20] |
PAUDYAL N, PAN H, WU B B, et al. Persistent asymptomatic human infections by Salmonella enterica serovar Newport in China[J]. mSphere, 2020, 5(3): e00163-20. |
| [21] |
COBBOLD R N, RICE D H, DAVIS M A, et al. Long-term persistence of multi-drug-resistant Salmonella enterica serovar Newport in two dairy herds[J]. J Am Vet Med Assoc, 2006, 228(4): 585-591. DOI:10.2460/javma.228.4.585 |
| [22] |
ZHAO X X, DAI Q L, ZHU D K, et al. Recombinant attenuated Salmonella Typhimurium with heterologous expression of the Salmonella Choleraesuis O-polysaccharide: high immunogenicity and protection[J]. Sci Rep, 2017, 7(1): 7127. DOI:10.1038/s41598-017-07689-5 |
| [23] |
KUMAR A, ALAM A, RANI M, et al. Biofilms: survival and defense strategy for pathogens[J]. Int J Med Microbiol, 2017, 307(8): 481-489. DOI:10.1016/j.ijmm.2017.09.016 |
| [24] |
DULA S, AJAYEOBA T A, IJABADENIYI O A. Bacterial biofilm formation on stainless steel in the food processing environment and its health implications[J]. Folia Microbiol (Praha), 2021, 66(3): 293-302. DOI:10.1007/s12223-021-00864-2 |
| [25] |
CHEBOTAR'I V, KONCHAKOVA E D, EVTEEVA N I. Neutrophil dependent breakdown of biofilms formed by Staphylococcus aureus[J]. Zh Mikrobiol Epidemiol Immunobiol, 2012(1): 10-15. |
| [26] |
MANGALAPPALLI-ILLATHU A K, KORBER D R. Adaptive resistance and differential protein expression of Salmonella enterica serovar Enteritidis biofilms exposed to benzalkonium chloride[J]. Antimicrob Agents Chemother, 2006, 50(11): 3588-3596. DOI:10.1128/AAC.00573-06 |
| [27] |
CHIN K C J, TAYLOR T D, HEBRARD M, et al. Transcriptomic study of Salmonella enterica subspecies enterica serovar Typhi biofilm[J]. BMC Genomics, 2017, 18: 836. DOI:10.1186/s12864-017-4212-6 |
| [28] |
HU S F, YU Y G, LV Z Q, et al. Proteomics study unveils ROS balance in acid-adapted Salmonella Enteritidis[J]. Food Microbiol, 2020, 92: 103585. DOI:10.1016/j.fm.2020.103585 |
| [29] |
JIAO S S, ZHANG H J, LIAO M J, et al. Investigation of the potential direct and cross protection effects of sublethal injured Salmonella Typhimurium induced by radio frequency heating stress[J]. Food Res Int, 2021, 150: 110789. DOI:10.1016/j.foodres.2021.110789 |
| [30] |
ARVIZU-MEDRANO S M, ESCARTÍN E F. Effect of acid shock with hydrochloric, citric, and lactic acids on the survival and growth of Salmonella typhi and Salmonella typhimurium in acidified media[J]. J Food Prot, 2005, 68(10): 2047-2053. DOI:10.4315/0362-028X-68.10.2047 |
(编辑 白永平)



