2. 国家非洲猪瘟区域实验室(广州), 广州 510642;
3. 华南农业大学兽医学院广东省临床重大疾病综合防控重点实验室, 广州 510642;
4. 华南农业大学国家生猪种业工程技术研究中心, 广州 510642
2. African Swine Fever Regional Laboratory of China (Guangzhou), Guangzhou 510642, China;
3. Key Laboratory of Comprehensive Prevention and Control for Severe Clinical Animal Diseases of Guangdong Province, College of Veterinary Medicine, South China Agricultural University, Guangzhou 510642, China;
4. National Engineering Research Center for Breeding Swine Industry, South China Agricultural University, Guangzhou 510642, China
非洲猪瘟(African swine fever,ASF)是一种具有传染性和高致病性的病毒性疾病,感染野猪和家猪,致死率可达100%[1-2]。2018年,我国首次报道了ASF疫情[3],此后疫情的蔓延给我国养猪业造成巨大损失。ASF的病原为非洲猪瘟病毒(African swine fever virus,ASFV),属于非洲猪瘟病毒科,基因组为170~194 kb的双链DNA,编码151~167个开放阅读框(ORFs)[4-5]。ASFV编码的许多蛋白质可参与病毒入侵、复制、组装、释放及免疫逃逸等过程,但由于其基因组庞大,目前,仍有约一半的ASFV基因缺乏相关已知或可预测的功能信息[5]。
p30蛋白是ASFV重要的结构蛋白[6],由病毒基因CP204L编码。在病毒感染宿主细胞早期p30大量表达,并且具有良好的抗原性,常作为血清学诊断抗原[7-8]。此外,p30蛋白在非洲猪瘟病毒粒子进入宿主细胞中发挥重要作用,抗p30蛋白的抗体对病毒内化有一定的抑制作用[9],这提示p30蛋白参与ASFV的内化过程,但具体作用机制尚不清楚[10-11]。目前,对于p30蛋白的认知有限,需要对其进行深入研究,以阐明其在ASFV感染宿主细胞过程中的作用机制。
当下没有商业化疫苗和安全有效的药物用于ASF的预防和治疗,对于疫病防控只能依赖严格的生物安全措施[12]。因此,尽早对疑似感染动物进行确诊有利于及时隔离和剔除传染源,防止疫情扩散。本研究制备了1株针对ASFV GZ201801毒株p30蛋白的单克隆抗体,利用生物信息学分析和截短表达等方式对其抗原表位区域进行初步鉴定,同时应用噬菌体十二肽库筛选并鉴定出与该单克隆抗体特异性结合的表位多肽,为ASFV感染早期血清学诊断试剂的研发和p30蛋白功能的研究奠定基础。
1 材料与方法 1.1 试验材料ASFV毒株GZ201801(GenBank:MT496893.1) 由本实验室分离,保存于华南农业大学动物生物安全三级实验室;猪肺泡巨噬细胞(PAMs)、罗猴胎肾细胞(MA-104)、pET-32a原核表达载体、pEGFP-C1真核表达载体由本实验室保存。pCzn1原核表达载体、Top10菌株、Arctic-Express表达菌、骨髓瘤细胞SP2/0购自南京钟鼎生物技术有限公司,BALB/c小鼠购自扬州大学,p30表位多肽由上海强耀生物科技公司完成合成及纯化。
1.2 酶和相关试剂Trans1-T1化学感受态细胞购自TransGen公司,Rosetta(DE3)感受态细胞购自TIANGEN公司,NdeⅠ和XbaⅠ等限制性内切酶购自TaKaRa公司,KOD高保真酶购自TOYOBO公司,琼脂糖胶回收试剂盒、质粒小提试剂盒购自Omega公司,10×DNA Loading Buffer购自Vazyme公司,Star Marker D2000 Plus购自Genstar公司,1640培养基、DMEM培养基、胎牛血清(FBS)、胰酶、灭菌PBS、Lipofectamine 3000转染试剂、Protein Marker、T4 DNA连接酶、山羊抗鼠IgG(H+L)Alexa Fluor Plus 555和山羊抗鼠IgG-HRP二抗购自Thermo Scientific公司,IRDye®800CW山羊抗鼠IgG二抗购自LI-COR公司,HiFi DNA同源重组酶购自NEB公司,弗氏佐剂购自Sigma公司,0.22 μm无菌滤器和透析袋购自Millipore公司,Ni-IDA亲和层析胶购自Novagen公司,E. coli ER2738和噬菌体展示随机12肽库试剂盒购自NEB公司,HRP-羊抗猪IgG购自KPL公司,TMB显色液购自天根生化科技有限公司,ASFV阴性血清由本实验室制备并通过法国ID-VET ASFV iELISA抗体检测试剂盒检测确认,非洲猪瘟标准阳性血清购自中国兽医药品监察所,其他试剂均为国产分析纯。
1.3 p30蛋白原核表达及纯化参考非洲猪瘟病毒GZ201801毒株的CP204L基因序列,采用基于PAS(PCR-based Accurate Synthesis)的方法进行密码子优化,由南京钟鼎生物技术有限公司进行基因合成。将该基因连入载体pCzn1的NdeⅠ和XbaⅠ酶切位点之间,将获得的重组质粒pCzn1-p30转入Top10克隆菌株,挑取阳性克隆子进行测序鉴定。将鉴定无误的重组质粒转化至Arctic Express大肠杆菌中,使用异丙基-β-D-硫代半乳糖苷(IPTG)诱导表达目的蛋白。优化表达条件,将诱导条件调整至20 ℃,经分析目标蛋白主要以包涵体形式表达。通过变复性的方式,重溶目标蛋白,Ni柱亲和纯化获得p30蛋白。
1.4 杂交瘤细胞制备及亚细胞克隆使用p30蛋白作为免疫原,选取5只BALB/c小鼠,将p30蛋白与弗氏佐剂混合免疫,按皮下注射50 μg·只-1进行免疫。2~3周加强免疫1次,3次免疫后采血检测,通过间接酶联免疫吸附试验(iELISA)确定其抗血清效价。具体操作方法:用PBS将纯化后的p30蛋白稀释为1 μg·mL-1,100 μL·孔-14 ℃包被过夜;弃去包被液,洗板3次,每孔加入200 μL封闭液,37 ℃孵育1 h;抗血清按1∶500倍比稀释,每孔100 μL,37 ℃孵育1 h;取出酶标板,弃液,洗板3次,向每孔中加入100 μL山羊抗鼠IgG-HRP二抗(1∶20 000),37 ℃孵育1 h;每孔加入100 μL TMB显色液,37 ℃显色15 min;每孔加入100 μL 1 mol·L-1 HCL溶液终止反应;立即在酶标仪450 nm波长读数,将OD值大于设定的阴性对照OD值的2.1倍的孔对应的稀释度,定为该样品的效价。
细胞融合前1周,用含10%FBS DMEM培养基扩大培养SP2/0骨髓瘤细胞。挑选3次免疫后血清ELISA效价在1∶50 000以上的小鼠,在融合前3 d终免,腹腔注射抗原100 μg;无菌取其脾,收集脾细胞并用红细胞裂解液去除红细胞;混合骨髓瘤细胞和脾细胞,使骨髓瘤细胞与脾细胞数量比为1∶2,随后使用电融合的方式进行细胞融合;将融合的细胞铺到96孔板中,每孔100 μL,然后将细胞培养板置于5% CO2培养箱中,37 ℃培养7~10 d进行融合筛选。融合筛选采用iELISA的方法(如上所述),根据iELISA结果判断阳性孔(样品孔OD值/阴性孔OD值≥2.1则判定为阳性孔),阳性孔细胞采用有限倍数稀释法进行2~3次亚细胞克隆,检测到100%阳性后,挑出单克隆孔扩大培养定株,获得单克隆细胞株。
1.5 单克隆细胞株筛选将ELISA筛选获得的单克隆细胞株进行间接免疫荧光试验(IFA)鉴定。试验方法:首先将PAMs铺于96孔细胞培养板中,20% FBS 1640培养基,5% CO2,37 ℃培养过夜;ASFV GZ201801株以MOI=0.1接种PAMs,感染24 h后,用4%多聚甲醛固定,用0.1% Triton-100透膜,并用5% BSA封闭;以单克隆细胞株上清作为一抗,37 ℃孵育2 h;山羊抗鼠IgG(H+L)Alexa Fluor TM Plus 555二抗孵育45~60 min后在荧光显微镜下观察试验结果。上述活病毒操作均在华南农业大学动物生物安全三级实验室中进行。
1.6 单克隆抗体特异性鉴定单克隆抗体特异性通过IFA和免疫印迹(Western blot)试验鉴定。IFA试验步骤同“1.5”,Western blot试验步骤:ASFV GZ201801毒株以MOI=0.1感染PAMs,分别在接毒后0、12、24和48 h裂解细胞收获蛋白样品;将样品进行SDS-PAGE,随后转移至聚偏二氟乙烯(PVDF)膜上;PVDF膜用5%脱脂乳室温封闭1 h,一抗为制备的p30单克隆抗体,4 ℃孵育过夜,二抗为IRDye®800CW山羊抗鼠IgG,室温避光孵育1 h,利用Odyssey双色激光成像系统观察试验结果。
1.7 p30蛋白抗原表位鉴定利用抗原表位预测工具(IEDB)对p30蛋白进行B细胞线性表位预测,根据预测结果对CP204L基因进行截短,并构建重组EGFP绿色荧光标签的真核表达质粒,引物如表 1所示。将质粒转染MA-104细胞,转染24~48 h后,分别进行IFA和Western blot试验筛选抗原表位。随后,将筛选到的p30蛋白的抗原表位区域进行原核表达(pET-32a载体)和Ni柱纯化,获得纯化蛋白,通过Western blot和iELISA验证其与p30单克隆抗体和非洲猪瘟阴性、阳性血清的反应原性。
|
|
表 1 引物与序列 Table 1 Primers and sequences |
采用固相生物淘选的方法,利用噬菌体十二肽库对靶分子p30单克隆抗体进行4轮淘选;前3轮生物淘选程序:噬菌体的淘选、淘选产物的滴度测定、淘选产物的扩增、扩增产物的滴度4个主要步骤,第4轮仅进行洗脱,不进行扩增。淘选的主要步骤按照《噬菌体表面展示肽库快速筛选肽配体使用手册》进行操作。将阳性噬菌体单克隆的PCR产物送上海生物工程有限公司测序(引物F-M13:5′-TCACCTCGAAAGCAAGCTGA-3′;引物R-M13:5′-CCCTCATAGTTAGCGTAACG-3′),运用生物学软件MegAlign(Version 11.0)进行比对分析,筛选获得与p30蛋白氨基酸序列同源性高的十二肽序列,并在此基础上设计合成多肽。
1.9 ELISA鉴定合成多肽与p30单克隆抗体及ASFV标准血清的反应原性多肽以碳酸盐缓冲液稀释至0.2 μg每孔包被至酶标板,每孔100 μL,4 ℃孵育过夜,并设置空白对照与阴性小鼠血清对照,p30单克隆抗体(初始浓度为4 mg·mL-1) 以PBST进行10-3~10-5倍稀释,小鼠阴性血清10-3倍稀释,ASFV阴、阳性血清进行1∶40、1∶80、1∶160、1∶320稀释,其他操作流程同“1.4”。
1.10 ASFV不同基因型毒株间p30蛋白氨基酸同源性比较分析通过NCBI(https://www.ncbi.nlm.nih.gov/gene)下载获取22种不同基因型ASFV毒株的p30蛋白氨基酸序列,并利用MegAlign(Version 11.0)进行同源比对,分析上述鉴定的表位多肽氨基酸位点的保守性。
2 结果 2.1 p30蛋白原核表达与纯化pCzn1-p30重组质粒经双酶切鉴定,得到符合CP204L基因片段大小的条带(图 1A)。利用IPTG诱导蛋白表达,经12% SDS-PAGE检测,目标蛋白主要存在于沉淀中(图 1B)。
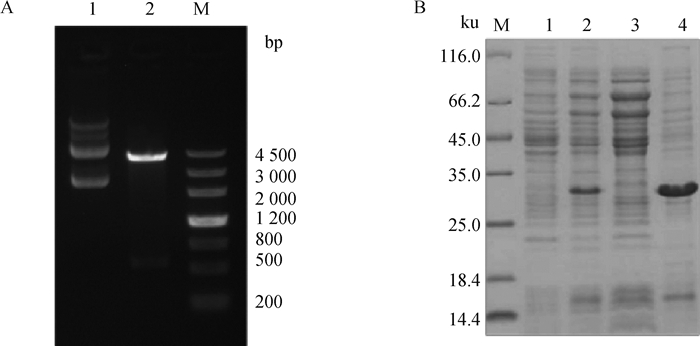
|
A. pCzn1-p30重组质粒双酶切鉴定(M. DNA相对分子质量标准;1.酶切前质粒;2.酶切后质粒);B.蛋白表达鉴定(M. 蛋白质相对分子质量标准;1.未诱导;2.诱导后;3.诱导破碎后上清;4.诱导破碎后沉淀) A. Identification of pCZN1-p30 recombinant plasmid by double enzyme digestion (M. DNA marker; 1. Pre-digestion plasmid; 2. Post-digestion plasmid); B. Identification of protein expression (M. Protein marker; 1. Uninduced; 2. Induced; 3. Induced crushing supernatant; 4. Induced crushing precipitation) 图 1 p30蛋白原核表达 Fig. 1 Prokaryotic expression of p30 protein |
包涵体经过变复性的方式重溶目标蛋白,通过Ni柱亲和纯化,将纯化后的蛋白进行12% SDS-PAGE和Western blot鉴定,结果表明成功获得纯化的p30蛋白(图 2A、B)。
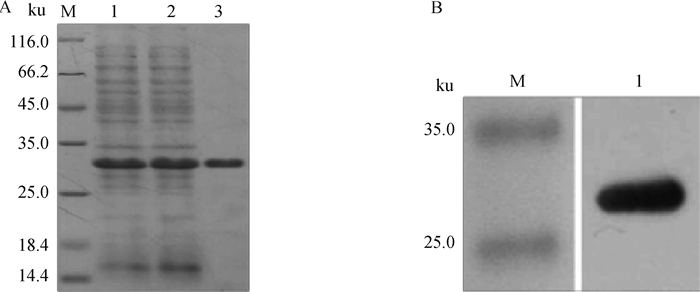
|
A. 纯化蛋白的SDS-PAGE分析(M. 蛋白质相对分子质量标准;1.破碎后处理样品;2.流出液;3.洗脱液);B. 纯化蛋白的Western blot分析(M. 蛋白质相对分子质量标准;1.纯化后样品) A. SDS-PAGE analysis of purified protein (M. Protein marker; 1. Post-crushing samples; 2. Effluent; 3. Eluent); B. Analysis of purified protein by Western blot analysis (M. Protein marker; 1. Purified samples) 图 2 p30蛋白原核纯化 Fig. 2 Purification of p30 protein |
杂交瘤细胞经过亚细胞克隆共获得6F11G6、8H7D11、8H7F6、8H7F8和8H7G4共5株单克隆细胞株,但IFA鉴定结果(结果略)显示仅6F11G6单克隆细胞株具有较好的试验效果,故选择6F11G6单克隆株进行单抗制备及后续的表位鉴定试验。将Protein A琼脂糖凝胶介质装入亲和纯化层析柱,将6F11G6细胞上清缓慢上样,待抗体结合后用甘氨酸洗脱缓冲液洗脱,立即在PBS中4 ℃透析过夜,即得到纯化单克隆抗体。
2.3 单克隆抗体特异性鉴定Western blot鉴定结果:与未接种病毒的PAMs相比,接种了ASFV的细胞在12、24和48 h后检测到特异性的p30条带,且随接毒时间的增加,p30蛋白的表达量增加(图 3A),表明该单克隆抗体具有特异性识别p30蛋白的功能。IFA鉴定结果:在病毒接种组有特异性红色荧光(TXRED),而非接毒对照组(Mock)无荧光(图 3B),表明其特异性良好,可用于后续试验。
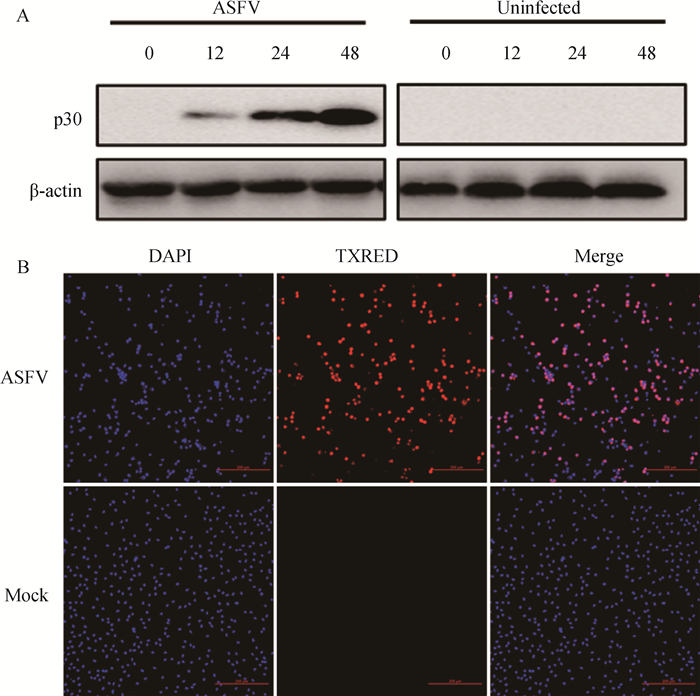
|
A. Western blot鉴定单克隆抗体特异性,ASFV表示接毒组,分别于接毒0、12、24和48 h后收集样品,uninfected表示未接毒细胞对照组,同样于0、12、24和48 h收集样品,使用β-actin作为内参蛋白;B. IFA鉴定单克隆抗体特异性,ASFV表示接毒组,Mock表示未接毒组,蓝色荧光(DAPI)代表细胞核,红色荧光(TXRED)代表p30单抗识别的p30蛋白,Merge代表两种荧光的重合图像,bar=200 μm A. Western blot was used to identify the specificity of monoclonal antibodies. ASFV represented the exposed group, samples were collected at 0, 12, 24 and 48 h after post-infection, while uninfected represented the control group, samples were also collected at 0, 12, 24 and 48 h, using β-actin as internal reference protein; B. IFA identifies the specificity of monoclonal antibodies, ASFV represents the exposed group, Mock represents the unexposed group, blue fluorescence (DAPI) represents the nucleus, red fluorescence (TXRED) represents the p30 mAb recognition of p30 protein, and Merge represents the overlap image of the two fluorescence types, bar=200 μm 图 3 单克隆抗体特异性鉴定 Fig. 3 Specific identification of the monoclonal antibody |
2.4.1 p30蛋白B细胞线性抗原表位预测与截短表达 依据氨基酸亲水性、表面可及性和抗原性对p30蛋白的抗原表位进行预测(图 4A),共预测到7个可能的抗原表位(黄色区域)。根据预测结果将p30蛋白截短表达为13个短肽(p30-1~p30-13),即分别从N端和C端依次增加预测的抗原表位数量,直至将完整的p30蛋白氨基酸序列覆盖(图 4B)。
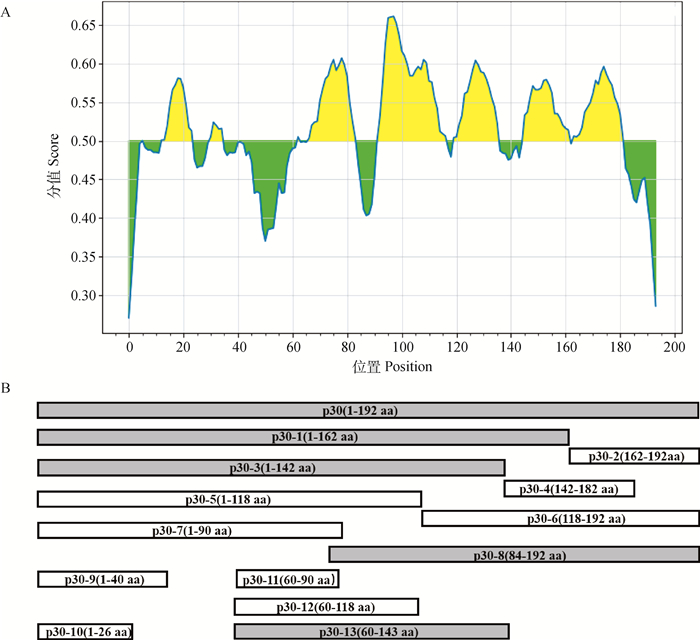
|
A. p30蛋白B细胞抗原表位预测,黄色区域为预测的抗原表位区域;B. p30蛋白截短方式示意图,用p30-1~13表示各截短肽段 A. Predicting B cell epitopes of p30 protein, and the yellow region is the predicted epitope region; B. Schematic diagram of p30 protein truncation method, using p30-1-13 to represent each truncated peptide segment 图 4 p30蛋白B细胞线性抗原表位预测与截短表达 Fig. 4 Prediction the B cells linear epitopes and truncation of p30 protein |
2.4.2 抗原表位的IFA筛选结果 将绿色荧光标签的各截短肽段重组真核表达质粒与阳性对照(绿色荧光标签的完整p30蛋白质粒)、阴性对照(绿色荧光标签空载体质粒)转染MA-104细胞,随后利用制备的单克隆抗体进行IFA试验。各组均出现绿色荧光(FITC),表明重组蛋白与短肽都能成功表达;p30、p30-1、p30-3、p30-8和p30-13出现红色荧光(TXRED),并且与绿色荧光共定位,其他短肽以及阴性对照组均未出现红色荧光(图 5)。因此短肽p30-1、p30-3、p30-8和p30-13能被p30单克隆抗体识别。

|
图 5 抗原表位筛选IFA结果(bar=200 μm) Fig. 5 The result of antigenic epitopes screening by IFA (bar=200 μm) |
2.4.3 抗原表位的Western blot筛选结果 将上述质粒转染MA-104细胞,然后利用p30单克隆抗体进行Western blot试验。Western blot结果与IFA结果一致,即p30、p30-1、p30-3、p30-8和p30-13出现符合预期大小的蛋白条带,表明短肽p30-1、p30-3、p30-8和p30-13能与p30单克隆抗体结合(图 6),其共同的氨基酸序列为84M-K142,推测该段序列为本研究制备的p30蛋白单克隆抗体识别的抗原表位区域。
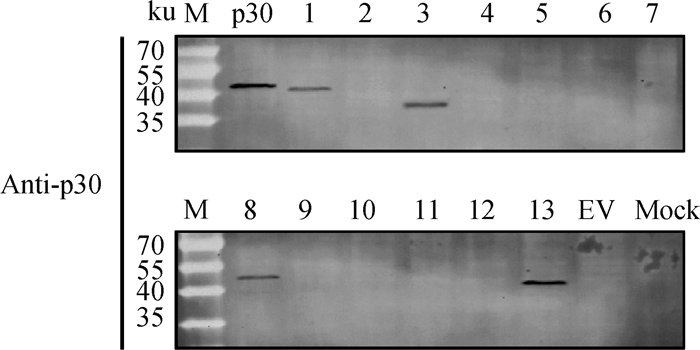
|
M. 蛋白质相对分子质量标准;p30.转染完整EGFP-p30表达质粒;1~13. 转染截短突变体EGFP-p30-1~13表达质粒;EV. 转染空载体pEGFP-C1质粒;MOCK. 未转染质粒的MA-104细胞对照组 M. Protein marker; p30. Complete EGFP-p30 expression plasmid was transfected; 1-13. Transfection of truncated mutant EGFP-p30-1-13 expression plasmid; EV. Transfected empty vector pEGFP-C1 plasmid; MOCK. MA-104 cell control without plasmid transfection 图 6 抗原表位筛选Western blot结果 Fig. 6 The result of antigenic epitopes screening by Western blot |
2.4.4 抗原表位(84M-K142)验证 进一步应用p30单克隆抗体和ASFV标准阳性血清,通过Western blot(图 7A)和iELISA(图 7B)对p30(84M-K142)特异性进行验证,结果表明84-142位氨基酸序列组成的p30截短蛋白能被p30单克隆抗体和ASFV标准阳性血清特异性识别,提示该段序列是本研究制备的mAb所识别的p30蛋白抗原表位区域。
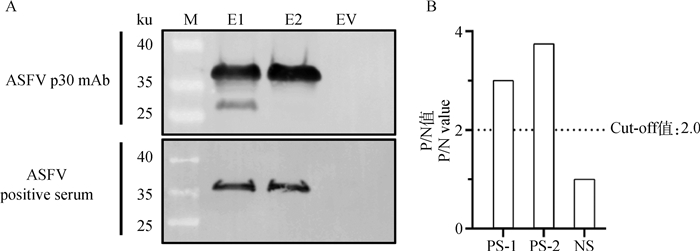
|
A. 抗原表位Western blot验证,分别使用p30蛋白mAb和ASFV阳性血清进行验证[M. 蛋白质相对分子质量标准;E1. p30(84M-K142)蛋白洗脱液1;E2. p30(84M-K142)蛋白洗脱液2;EV. pET-32a空载体原核表达对照组];B. 抗原表位iELISA验证(PS. ASFV标准阳性血清;NS. ASFV阴性血清) A. Verification of the epitopes by Western blot, the p30 protein mAb and ASFV positive serum were used for validation (M. Protein marker; E1. p30 (84M-K142) protein eluent 1;E2. p30 (84M-K142) protein eluent 2; EV. The control group of pET-32a empty vector prokaryotic expression); B. Verification of the epitopes by iELISA (PS. positive serum of ASFV; NS. negative serum of ASFV) 图 7 抗原表位验证 Fig. 7 Verification of the epitopes |
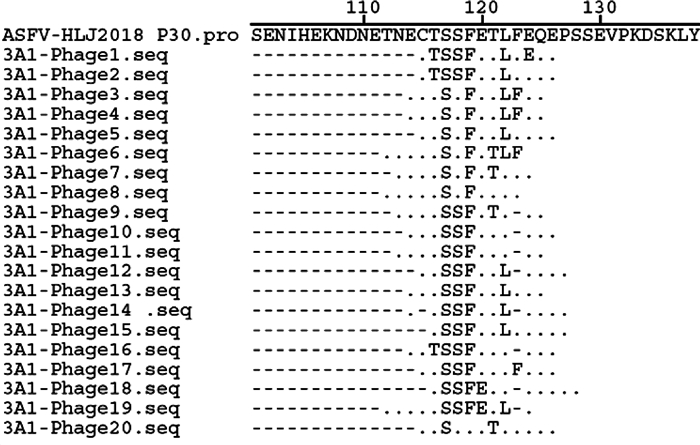
将与p30单克隆抗体特异性亲和的噬菌体PCR产物送公司测序,测序结果与ASFV-HLJ毒株p30蛋白氨基酸序列进行比对,结果提示, 该单克隆抗体对应表位分布于116TSSFETLFE124之间(图 8)。在此基础上,前后延长合成两段多肽(Peptide-TN和Peptide-CT)进行活性分析,多肽序列见表 2。
|
图 8 p30单克隆抗体特异性亲和噬菌体测序分析 Fig. 8 Sequencing analysis of p30 monoclonal antibody specific affinity phage |
|
|
表 2 合成肽信息 Table 2 Information of the synthetic peptide |
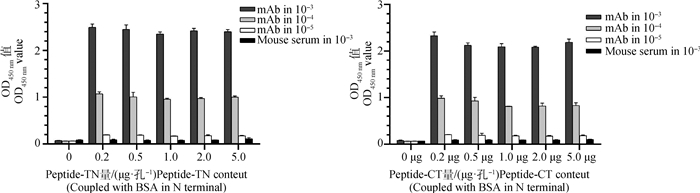
分别将合成多肽作为包被抗原与p30单克隆抗体和ASFV标准血清进行结合力验证,ELISA结果显示:两条多肽在0.2~5.0 μg·孔-1的包被区间与单克隆抗体具有相似的结合力,随着单克隆抗体稀释度增加,结合能力下降,而阴性小鼠血清与多肽几乎不反应(图 9);且与ASFV标准阳性血清具有特异性结合反应(表 3)。
|
图 9 多肽与p30单克隆抗体结合能力 Fig. 9 Binding activities of the synthetic peptides to p30 monoclonal antibody |
|
|
表 3 合成多肽与ASFV标准血清的结合能力 Table 3 Binding activities of the synthetic peptides to ASFV standard serum |
ASFV p30蛋白氨基酸序列比对结果显示:116TSSFETLFE124多肽序列在ASFV 22种基因型毒株间保守(表 4)。
|
|
表 4 ASFV不同基因型毒株间p30蛋白氨基酸同源性比较分析 Table 4 Comparative analysis of amino acid homology of p30 protein among ASFV genotypes |
非洲猪瘟是危害养猪业的重大疫病,仅根据感染猪的临床症状很难将ASF与猪瘟(CSF)、高致病性猪繁殖与呼吸综合征(HP-PRRS)和猪丹毒等具有相似临床症状的疫病进行区分[13],因此实验室诊断对于ASF的鉴定至关重要。目前,常用的实验室诊断方法主要包括红细胞吸附试验(HAD)、聚合酶链式反应(PCR)、荧光定量PCR(qPCR)、iELISA等,其中,qPCR因其灵敏度和准确性较高而被广泛使用[14],但其缺点是对仪器设备的要求较高。血清学诊断是常用的诊断检测方法,因为它操作简单,成本相对较低,对专用设备的要求也较低,缺点是灵敏度的特异性不如qPCR。但当猪被减毒或低毒力毒株感染时,血清学诊断可能是检测被感染动物的最佳方法[13]。有研究报道2020年我国家猪中已出现自然产生的低毒力非洲猪瘟病毒[15],所以研发出特异性强灵敏度高的血清学诊断试剂具有十分重要的意义。OIE推荐的非洲猪瘟的血清学检测方法是ELISA,目前商业化的ELISA试剂盒主要基于蛋白p30、p54、p72和pp62等,其中p30蛋白被认为最适合用于免疫吸附试验[13, 16]。因此,评估p30蛋白的抗原表位特征,尤其是保守表位特征,将有助于发展和改进ASF诊断和监测的血清学检测方法。
在p30蛋白抗原表位研究方面,Murgia等[17]针对BA71V毒株的p30蛋白表位鉴定结果表明免疫优势区位于111-130位氨基酸覆盖的寡肽。Petrovan等[7]的研究初步鉴定了p30的61-93位氨基酸之间的表位,该区域也被ASFV感染猪的血清识别,提示该区域是具有重要免疫功能的表位[18]。同时也鉴定了p30蛋白C端部分中的一个大多肽片段(120-204 aa)与高度柔性和潜在无序倾向区域(91-143 aa)部分重叠可能是p30蛋白的抗原表位区域,该结果与本研究的试验结果相似。Wu等[10]开发了1组21株p30蛋白的单克隆抗体,通过iELISA进行了主要表位定位,确定了4个独特的抗原表位区域,分别为61-90 aa、96-115 aa、116-125 aa和146-160 aa,其中后两个抗原区域在不同ASFV毒株中高度保守。本研究通过IFA、Western blot和iELISA初步确定了本实验室制备的1株针对ASFV GZ201801毒株p30蛋白的mAb所识别的抗原表位区域为84M-K142,并通过噬菌体展示技术对抗原表位进行定位。
噬菌体展示技术由Smith等[19]建立于1985年,迄今以其操作简便与高效筛选的优势被广泛应用于生物医学领域,如分子成像、疫苗研发、抗体制备及纳米技术等领域[20]。同时,利用噬菌体展示文库,与靶分子特异性结合的多肽配体被高效筛选与鉴定,为进一步发展疾病诊断与治疗策略提供思路。本研究应用噬菌体十二肽展示文库成功筛获得高频克隆展示序列,这些序列集中分布于ASFV GZ201801毒株p30蛋白的116TSSFETLFE124区域。在此分析基础上设计合成十四肽,iELISA结果表明两条合成多肽112TNECTSSFETLFEQ125和115CTSSFETLFEQEPS128均与制备的p30单克隆抗体具有高度亲和活性。综合氨基酸比对分析结果提示116TSSFETLFE124为针对p30蛋白的一处保守B细胞线性表位核心区域,该结果在本研究使用截短蛋白筛选基础上,进一步缩小了表位分布范围。
4 结论本研究制备了1株非洲猪瘟病毒GZ201801毒株p30蛋白的单克隆抗体,同时利用蛋白截短表达的方式鉴定了84M-K142是其识别的抗原表位区域,进一步通过噬菌体展示技术确定了一个保守表位(116TSSFETLFE124),丰富了p30蛋白的抗原表位信息,为进一步研究p30蛋白抗原核心基序及发展ASFV血清学诊断材料奠定基础。
| [1] |
DIXON L K, STAHL K, JORI F, et al. African swine fever epidemiology and control[J]. Annu Rev Anim Biosci, 2020, 8: 221-246. DOI:10.1146/annurev-animal-021419-083741 |
| [2] |
GALINDO I, ALONSO C. African swine fever virus: a review[J]. Viruses, 2017, 9(5): 103. DOI:10.3390/v9050103 |
| [3] |
GE S Q, LI J M, FAN X X, et al. Molecular characterization of African swine fever virus, China, 2018[J]. Emerg Infect Dis, 2018, 24(11): 2131-2133. DOI:10.3201/eid2411.181274 |
| [4] |
KARGER A, PÉREZ-NU'ÑEZ D, URQUIZA J, et al. An update on African swine fever virology[J]. Viruses, 2019, 11(9): 864. DOI:10.3390/v11090864 |
| [5] |
ALEJO A, MATAMOROS T, GUERRA M, et al. A Proteomic atlas of the African swine fever virus particle[J]. J Virol, 2018, 92(23): e01293-18. |
| [6] |
JIA N, OU Y W, PEJSAK Z, et al. Roles of African swine fever virus structural proteins in viral infection[J]. J Vet Res, 2017, 61(2): 135-143. DOI:10.1515/jvetres-2017-0017 |
| [7] |
PETROVAN V, YUAN F F, LI Y H, et al. Development and characterization of monoclonal antibodies against p30 protein of African swine fever virus[J]. Virus Res, 2019, 269: 197632. DOI:10.1016/j.virusres.2019.05.010 |
| [8] |
于学祥, 陈晓雨, 李栋凡, 等. 非洲猪瘟病毒无标签p30-ELISA抗体检测方法的建立及应用[J]. 畜牧兽医学报, 2022, 53(5): 1517-1526. YU X X, CHEN X Y, LI D F, et al. Establishment and application of an indirect ELISA antibody detection method based on African swine fever virus tag-free p30 protein[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(5): 1517-1526. (in Chinese) |
| [9] |
NEILAN J G, ZSAK L, LU Z, et al. Neutralizing antibodies to African swine fever virus proteins p30, p54, and p72 are not sufficient for antibody-mediated protection[J]. Virology, 2004, 319(2): 337-342. DOI:10.1016/j.virol.2003.11.011 |
| [10] |
WU P, LOWE A D, RODRÍGUEZ Y Y, et al. Antigenic regions of African swine fever virus phosphoprotein P30[J]. Transbound Emerg Dis, 2020, 67(5): 1942-4953. |
| [11] |
ANDRÉS G. African swine fever virus gets undressed: new insights on the entry pathway[J]. J Virol, 2017, 91(4): e01906-16. |
| [12] |
BLOME S, FRANZKE K, BEER M. African swine fever-a review of current knowledge[J]. Virus Res, 2020, 287: 198099. DOI:10.1016/j.virusres.2020.198099 |
| [13] |
QIU Z L, LI Z Y, YAN Q H, et al. Development of diagnostic tests provides technical support for the control of African swine fever[J]. Vaccines (Basel), 2021, 9(4): 343. DOI:10.3390/vaccines9040343 |
| [14] |
杨莎莎, 苗雨润, 薄宗义, 等. 非洲猪瘟病毒p30单克隆抗体的制备和鉴定[J]. 中国兽医科学, 2020, 50(8): 976-981. YANG S S, MIAO Y R, BO Z Y, et al. Generation and characterization of monoclonal antibodies against p30 protein of African swine fever virus[J]. Chinese Veterinary Science, 2020, 50(8): 976-981. (in Chinese) |
| [15] |
SUN E C, ZHANG Z J, WANG Z L, et al. Emergence and prevalence of naturally occurring lower virulent African swine fever viruses in domestic pigs in China in 2020[J]. Sci China Life Sci, 2021, 64(5): 752-765. DOI:10.1007/s11427-021-1904-4 |
| [16] |
徐黎晖, 迟立超, 王雨佳, 等. 非洲猪瘟病毒p30蛋白的真核表达及间接ELISA抗体检测方法的建立[J]. 中国预防兽医学报, 2021, 43(4): 382-387. XU L H, CHI L C, WANG Y J, et al. Eukaryotic expression of p30 gene of African swine fever virus and establishment of indirect ELISA for antibody detection[J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(4): 382-387. (in Chinese) |
| [17] |
MURGIA M V, MOGLER M, CERTOMA A, et al. Evaluation of an African swine fever (ASF) vaccine strategy incorporating priming with an alphavirus-expressed antigen followed by boosting with attenuated ASF virus[J]. Arch Virol, 2019, 164(2): 359-370. DOI:10.1007/s00705-018-4071-8 |
| [18] |
吕璐, 钟秋萍, 于婉琪, 等. 非洲猪瘟病毒p30蛋白单克隆抗体的制备及鉴定[J/OL]. 中国动物传染病学报, 2020: 1-10. (2020-11-25). http://kns.cnki.net/kcms/detail/31.2031.s.20201125.1039.016.html. LÜ L, ZHONG Q P, YU W Q, et al. Immunological characterization of monoclonal antibodies against African swine fever virus p30[J/OL]. Chinese Journal of Animal Infectious Diseases, 2020: 1-10. (2020-11-25). http://kns.cnki.net/kcms/detail/31.2031.s.20201125.1039.016.html. (in Chinese) |
| [19] |
SMITH G P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985, 228(4705): 1315-1317. DOI:10.1126/science.4001944 |
| [20] |
BRATKOVIČ T. Progress in phage display: evolution of the technique and its applications[J]. Cell Mol Life Sci, 2010, 67(5): 749-767. DOI:10.1007/s00018-009-0192-2 |
(编辑 白永平)



