随着养殖业的现代化发展,集约化养殖渐渐替代了传统的养殖方法[1],如何降低料重比、提高饲料利用率并发挥其最大经济效益是养殖者追求的目标。目前,我国养猪生产主要采用自由采食和分餐饲喂两种方法进行养殖[2]。自由采食是指始终保证料槽内有充足的饲料,猪可以根据自身意愿随时采食,这种饲喂方式可实现猪的快速增重,有效降低人工劳动成本,并且符合动物福利要求,但也会不可避免地造成胴体偏肥、饲料利用率低等负面效果。分餐饲喂是指在兼顾猪增重的情况下,规定每天进食的次数和时间,以期达到提高饲料利用率、降低料重比、改善肉品质等目的[3]。
研究表明,与自由采食生长猪相比,每天饲喂两次的猪平均日增重和平均日采食量都有下降的趋势,但料重比要低于自由采食的猪[4-5]。Le Naou等[6]研究却认为,每天饲喂两次会增加血浆葡萄糖、胰岛素的波动,可能会增加猪的生长效率和速度,并且与频繁进食相比,大量但不频繁的进食并不会改变组织代谢能力,也不会改变组织成分和机体的脂肪含量。有研究发现,不同的饲喂方式会改变猪的肝转录,通过影响生长猪的脂质代谢来改变其生长性能[7-8]。目前,大多数报道主要集中在不同饲喂频率对猪生长和代谢的影响方面,如Schneider等[9]研究每日饲喂2次或6次对猪生长性能的影响,陈祥文等[10]则研究每日饲喂3次和自由采食对猪生长的影响。但这些对不同饲喂频率的研究,很少有对单次采食的时间有所限制,对限时采食方面相关的报道也较少。有研究表明,与自由采食的小鼠相比,限时采食可以减少小鼠肝中蔗糖诱导的脂肪累积,改变肝脂质代谢[11]。Allee等[12]的研究也表明,给予猪每天2 h时间采食,其脂肪组织中脂肪酸含量明显高于自由采食组。这提示,限时采食可能通过影响肝的脂质代谢进而影响机体的整体代谢,但限时采食如何影响机体整体代谢尚不完全清楚。
肝作为动物体内的代谢中枢,转录组和代谢组联合分析可解析猪的肝代谢对不同采食模式的响应,同时,结合血清生化相关指标,有助于了解限时采食对机体整体代谢的影响。因此,本研究采用一日三次饲喂并限制每次采食时间的方式,比较限时采食与自由采食模式下猪的生长性能和肝代谢差异,旨在揭示限时采食影响生长猪代谢的机制,为猪的高效生产提供理论指导。
1 材料与方法 1.1 试验动物与设计本试验在南京农业大学动物科技学院动物房进行。试验选取体重相近((56.29±1.93)kg)的杜×长×大三元杂交生长猪12头,采用单因子完全随机设计,分为对照组(CON组)和限时采食组(TRF组),每组6个重复,每个重复1头猪。对照组每天饲喂1次,自由采食(给足饲料);限时采食组每天饲喂3次(分别在07:00、12:00、17:00),每次采食时间为1 h,期间饲喂足量饲料。试验期21 d,期间所有试验猪自由饮水,每天记录采食量,每周记录增重情况并统计生长性能。
1.2 样品采集试验结束当天,颈静脉采集10 mL血液,在4 ℃下3 500 r·min-1离心10 min,取上层血清,-20 ℃保存,用于血清生化指标的测定。通过颈静脉注射4%戊巴比妥钠溶液(40 mg·kg-1)将猪麻醉并安乐死。剖开腹腔迅速分离各个脏器,在肝的右叶中央部位采集样品,分装在2 mL冻存管中,置于液氮中保存,用于转录组和代谢组分析。
1.3 血清生化指标测定天冬氨酸转氨酶(AST)、丙氨酸转氨酶(ALT)、总蛋白(TP)、白蛋白(ALB)、球蛋白(GLOB)、碱性磷酸酶(ALP)、γ-谷氨酰转肽酶(GGT)、总胆汁酸(TBA)、谷氨酸脱氢酶(GLDH)、肌酸激酶(CK)、乳酸脱氢酶(LDH)、葡萄糖(GLU)、酰基载体蛋白(tACP)、总胆固醇(CHO)、高密度脂蛋白胆固醇(HDL-C)、低密度脂蛋白白胆固醇(LDL-C)、甘油三酯(TRIG)、载脂蛋白A(APOA)均使用全自动生化分析仪(奥林巴斯AU400)测定。
1.4 肝代谢产物的测定取50 mg肝组织样本,加入800 μL 80%的甲醇,涡旋30 s;60 Hz研磨90 s,4 ℃超声30 min;-40 ℃静置1 h,涡旋30 s,4 ℃静置0.5 h。置于4 ℃离心机中,12 000 r·min-1离心15 min。取尽上清液,于-40 ℃静置1 h。置于4 ℃离心机中,12 000 r·min-1离心15 min。取200 μL上清液,加入5 μL内标(140 μg·mL-1,二氯苯丙氨酸),于进样小瓶中待液相色谱-质谱(liquid chromatography-mass spectrometry, LC-MS)检测分析。色谱柱:C18色谱柱(Hyper gold C18 (100×2.1 mm,1.9 μm));流动相:水+5%乙腈+0.1%甲酸(A),水+5%乙腈+0.1%甲酸(B);柱温:40 ℃;流速:0.3 mL·min-1;进样量:4 μL。
1.5 RNA的提取、测序和分析取0.2 g肝组织样品,使用购自美国Invitrogen公司的TRIzol试剂盒,按照说明书操作流程提取RNA。总RNA样品的纯度、浓度和完整性经检测合格后,用带有Oligo(dT)的磁珠富集真核生物mRNA;加入缓冲破碎液将mRNA进行随机打断;以mRNA为模板,用六碱基随机引物合成第一条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA聚合酶I合成第二条cDNA链,利用AMPure XP beads纯化cDNA;纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后用AMPure XP beads进行片段大小选择;最后通过PCR富集得到cDNA文库。库检合格后,不同文库按照目标下机数据量进行汇集,用Illumina平台进行测序。测得的结果每个样品Q30的值大于85%,都可以进行接下来的数据分析。以上有关文库的构建、测序以及原始数据初步处理等操作均由北京百迈克生物技术有限公司完成。
1.6 数据分析与统计所有数据以“平均值±标准误”形式表示,使用SPSS 21.0软件进行统计分析。采用独立样本t检验对两组生长性能和血清指标数据进行显著性分析,P < 0.05表示差异显著,0.05≤P < 0.1表示有差异趋势。
对预处理得到的代谢组数据,通过在线分析工具(www.metaboanalyst.ca)以识别代谢途径和进行代谢物组富集分析,并采用偏最小二乘法-判别分析(partial least squares discrimination analysis, PLS-DA)处理,按照筛选变异权重参数值(variable importance inprojection, VIP)>1和P < 0.05作为标准,筛选出差异代谢物。对于差异代谢物的富集采用超几何检验方法。
对于转录组预处理所得的数据,按照差异倍数(FC>1.5或<0.67, P < 0.05) 作为标准,筛选出差异显著的基因。再将筛选出来的差异显著基因进行基因本体(gene ontology, GO)功能分析和京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes, KEGG)通路富集。
2 结果 2.1 生长性能由表 1可知,TRF组猪平均日增重显著高于对照组(P < 0.05),TRF组的料重比显著低于对照组(P < 0.05),两组间平均日采食量没有显著差异。
|
|
表 1 限时采食组与对照组中猪的生长性能(n=6) Table 1 The growth performance of pigs in TRF and CON groups (n=6) |
如表 2所示,与对照组相比,TRF组猪血清中丙氨酸转氨酶和肌酸激酶的活性有升高的趋势(P < 0.1),而总胆汁酸含量有降低趋势(P < 0.1),两组间其他生化指标浓度无显著差异。
|
|
表 2 限时采食组与对照组中生长猪血清生化指标 Table 2 The serum parameters of pigs in TRF and CON groups |
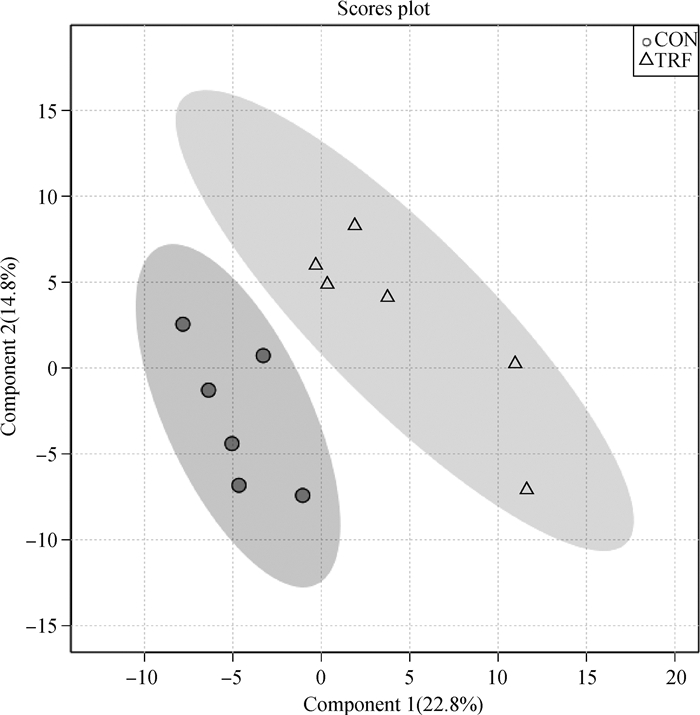
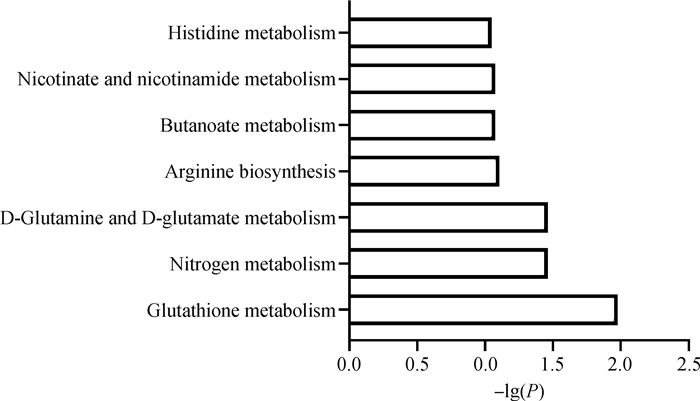
将各组数据与标准质谱数据库进行比对,共确定了247种肝代谢化合物,通过PLS-DA模型对代谢产物归一化数据进行多元变量分析(图 1),发现限时采食组和对照组的肝代谢产物的区分较大,22.8%的差异可由横坐标PC1来解释,14.8%的差异可由纵坐标PC2来解释。按照特定的筛选条件(VIP>1.0,P < 0.10),两组间的差异代谢物见表 3,与对照组相比,限时采食显著增加了猪肝组织中N-乙酰亮氨酸和13Z二十二烯酰胺的浓度,同时降低了3种氨基酸(N6, N6, N6-三甲基-L-赖氨酸、焦谷氨酸和L-谷氨酸)、顺丁烯二酸以及L-乙酰基肉碱、黄嘌呤、对乙酰氨基酚、癸酰胺、烟酰胺的浓度。将筛选得出的差异代谢物进行更深入的代谢通路富集分析,如图 2所示,限时采食组中有关谷胱甘肽代谢、氮代谢和D-谷氨酰胺与D-谷氨酸代谢等途径被显著影响。
|
图 1 限时采食组与对照组猪肝组织代谢组谱最小二乘法判别分析 Fig. 1 Partial least squares discriminant analysis of pig liver metabolites in TRF and CON groups |
|
|
表 3 限时采食组与对照组相比肝中的显著差异代谢物 Table 3 Significant differential metabolites in livers of pigs in TRF and CON groups |
|
图 2 限时采食组与对照组猪肝组织中差异代谢物通路分析 Fig. 2 Pathway analysis of differential metabolites in livers of pigs in TRF and CON groups |
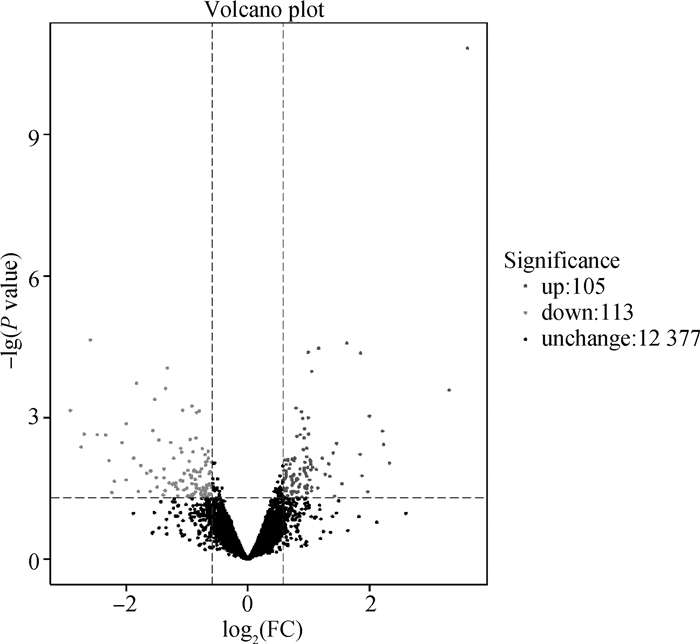
对转录组数据按照FC>1.5或 < 0.67,P < 0.05作为筛选标准,如图 3显示,与对照组相比,限时采食显著改变了肝组织中218个基因的表达,其中有105个基因表达上调,113个基因表达显著下调。在这些差异表达基因中,与脂肪酸代谢相关的基因FABP1和ACAA2在TRF组的表达量显著上升,而APOA4、ELOVL2和CPT1A的表达显著下调;此外,氨基酸代谢相关基因GLUL在限时采食组中的表达显著增高,而SDS、PC和MTR的表达被显著抑制;此外,限时采食也影响参与碳水化合物代谢的基因表达,显著上调ENPP3的表达,下调MTHFR的表达(表 4)。
|
图 3 限时采食对生长猪肝组织转录表达谱的影响 Fig. 3 Effect of time-restricted feeding on the transcriptional profiles in the liver of growing pigs |
|
|
表 4 限时采食组的猪肝组织中脂肪酸代谢、氨基酸代谢和碳水化合物代谢相关基因与对照组组表达比较 Table 4 Differential expressed genes involved in pathways relating to fatty acid metabolism, amino acid metabolism and carbohydrate metabolism in the liver of pigs in TRF group compared with CON group |
将筛选所得的差异基因进行KEGG富集分析发现(表 5),与对照组相比,TRF组中氨基酸生物合成,D-精氨酸和D-鸟氨酸代谢,脂肪酸代谢,精氨酸生物合成,碳代谢,花生四烯酸代谢,缬氨酸、亮氨酸和异亮氨酸的生物合成,乙醛酸和二羧酸代谢等途径被显著影响(P < 0.05)。
|
|
表 5 限时采食组与对照组生长猪肝组织中差异表达基因KEGG富集分析 Table 5 KEGG pathway enrichment analysis of the differentially expressed genes in the liver between TRF and CON groups |
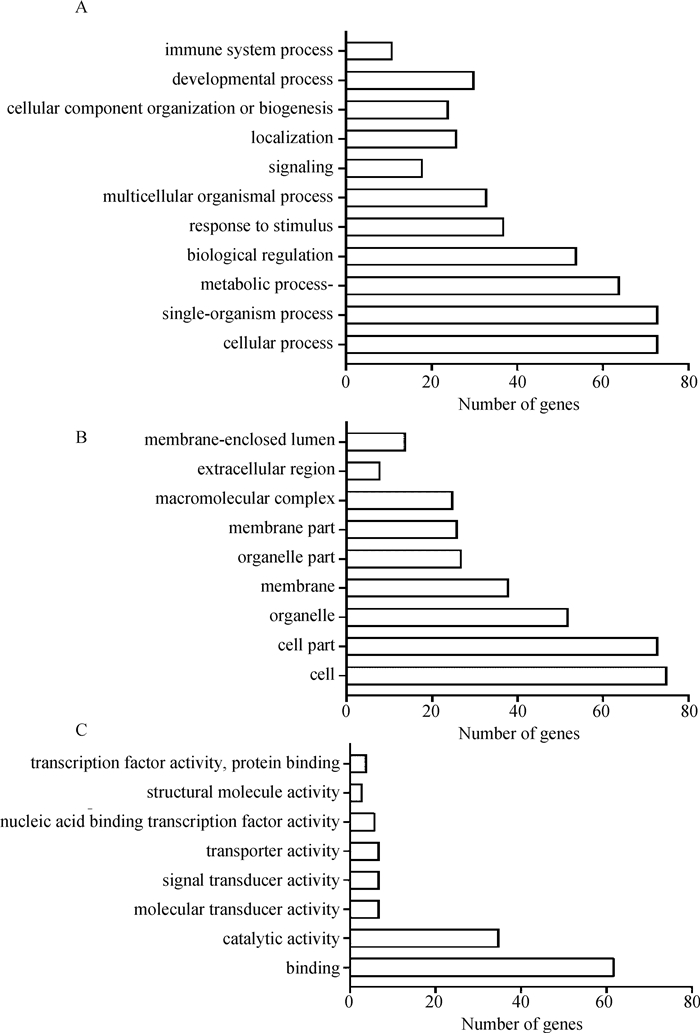
对差异基因进行GO类别分析和途径富集分析发现(图 4),在GO类别的生物进程水平中,与对照组相比,TRF组的差异基因大多集中在细胞过程、单生物过程、生物调节和代谢过程中;在GO类别的细胞组成位置中,TRF组的差异基因主要在细胞器、细胞膜和高分子络合物等位置发挥作用;而在GO类别的分子功能方面,TRF组的差异表达基因主要参与结合和催化活性等方面。
|
图 4 限时采食组与对照组相比差异基因富集在生物进程(A)、细胞组成(B)和分子功能(C)的GO功能注释 Fig. 4 GO term enrichment analysis of the biological processes (A), cellular-component (B), and molecular function (C) of differentially expressed genes in the livers of pigs in TRF group compared with the CON group |
本试验比较了两种不同饲喂模式对猪生长性能、血清生化指标和肝代谢的影响,发现分餐限时采食组猪的增重更高,料重比更低,这与Colpoys等[13]研究结果一致。然而,也有研究表明,减少饲喂次数限制饲喂时间可以减少猪的体重但不影响采食量[14]。结果不一致可能与日粮组成、猪的品种或日龄不同有关。限时采食虽然未显著影响采食量,但是显著降低了料重比,说明限时采食可以改善饲料转化率。在血清生化指标中,试验组主要影响丙氨酸转氨酶和肌酸激酶的活性以及总胆汁酸的浓度。丙氨酸转氨酶是一种反映机体氮代谢状况的生化参数[15],也是动物体内重要的转氨酶,影响多种氨基酸的代谢,本试验结果表明,限时饲喂可以提高丙氨酸转氨酶的活性进而提高生长猪对氨基酸的利用。血清中的总胆汁酸是由胆固醇在肝中代谢而来[16],限时采食组猪的血清总胆汁酸含量较对照组低,在一定程度上说明肝对胆固醇的代谢存在差异。这些结果表明,限时采食可以改变猪机体氨基酸和脂质的代谢。Le Naou等[6]发现,每天更少的摄食频率可以增加饲料向体重的转换,并且未观察到限时采食对甘油三酯浓度的影响,这与本试验结果一致。
本试验联合转录组学和代谢组学分析研究了限时采食对猪肝代谢的影响。与对照组相比,限时采食组猪中发现218个差异表达基因,这些差异基因存在于不同的生物学过程中。肝是脂肪酸主要的合成场所,因此肝是研究脂质代谢的重要组织[17]。KEGG通路显示,限时采食显著影响了脂肪酸代谢通路和脂肪酸伸长的通路。本试验中TRF组的ACAA2基因和FABP1基因的表达量较高,分别是对照组的1.81倍和1.68倍。ACAA2是乙酰辅酶A酰基转移酶两种异构体中的一种,该酶通过催化脂肪β-氧化途径的最后一步参与线粒体脂肪酸的延伸和降解[18-19]。ACAA2普遍存在于动物体内,催化游离胆固醇和长链脂肪酸合成酯化胆固醇,进而调节肝脂质代谢[20-21]。肝脂肪酸结合蛋白(FABP1) 是脂质结合蛋白超级家族中的一员,对长链脂肪酸表现出高亲和力,并在脂质的代谢和细胞内运输中发挥作用[22-23]。有研究表明,肝脂肪酸结合蛋白对花生四烯酸的摄取具有高亲和力,它可以将花生四烯酸磷脂化,使之合成为花生四烯酰乙醇酰胺(AEA)和2-花生四烯酰甘油(2-AG)[24]。FABP1基因参与了花生四烯酸的代谢,这也与差异代谢物通路分析中的丁酸代谢通路被显著影响对应。以上结果提示,限时饲喂可能会改变肝脂肪酸的含量,改变肝中脂肪的合成。
有研究表明,不同饲喂频率可影响生长猪对蛋白质和氨基酸的利用率[25]。本研究中,KEGG通路显示,限时采食显著影响氨基酸的生物合成和精氨酸及鸟氨酸的代谢。此外,代谢组学分析显示, 限时采食显著降低肝中三甲基-L-赖氨酸、焦谷氨酸和L-谷氨酸的浓度。这可能与TRF组上调基因GLUL有关。谷氨酰胺合成酶基因(GLUL)是合成谷氨酰胺的关键基因,谷氨酰胺是体内氨转移的重要载体[26],促进组织中谷氨酰胺的合成是提高机体氮利用率的有效途径[27],在包括肝在内的大部分组织中存在[28-29],细胞外谷氨酰胺的摄取可有效诱导肝细胞肿胀[30-31]。有研究指出,谷氨酰胺可以通过钠依赖性转运系统集中摄入到肝细胞中[32],通过诱导细胞肿胀和体积调节离子通量的介导来抑制自噬蛋白的水解。该结果表明,TRF组猪可以增加谷氨酰胺合成酶的活性来降低肝中部分氨基酸的含量[33],提高其他靶组织谷氨酰胺的合成,促进机体的生长。
4 结论本研究表明,限时采食可以影响肝中的氨基酸代谢,改变脂肪酸和蛋白质在肝中的代谢,调控生长猪对营养物质的利用,改善生长性能。
| [1] |
林学梅. 猪养殖发展前景和具体方法[J]. 中国动物保健, 2021, 23(5): 74-75. LIN X M. Development prospect and specific methods of pig breeding[J]. China Animal Health, 2021, 23(5): 74-75. DOI:10.3969/j.issn.1008-4754.2021.05.051 (in Chinese) |
| [2] |
郭永清, 赵宇飞, 张小宇. 不同饲养方式及密度对猪生长性能及肉品质的影响[J]. 饲料研究, 2019, 42(8): 120-123. GUO Y Q, ZHAO Y F, ZHANG X Y. Effect of different feeding modes and densities on growth performance and meat quality of pigs[J]. Feed Research, 2019, 42(8): 120-123. (in Chinese) |
| [3] |
杨玉芬, 乔建国. 猪自由采食和限制饲喂的利与弊[J]. 畜禽业, 2000(9): 25. YANG Y F, QIAO J G. Advantages and disadvantages of free feeding and restricted feeding of pigs[J]. Livestock and Poultry Industry, 2000(9): 25. DOI:10.3969/j.issn.1008-0414.2000.09.015 (in Chinese) |
| [4] |
LIU J B, CAI X, XIONG H, et al. Effects of feeding frequency on meat quality traits and Longissimus muscle proteome in finishing pigs[J]. J Anim Physiol Anim Nutr (Berl), 2017, 101(6): 1175-1184. DOI:10.1111/jpn.12636 |
| [5] |
SCHNEIDER J D, TOKACH M D, GOODBAND R D, et al. Effects of restricted feed intake on finishing pigs weighing between 68 and 114 kilograms fed twice or 6 times daily[J]. J Anim Sci, 2011, 89(10): 3326-3333. DOI:10.2527/jas.2010-3154 |
| [6] |
LE NAOU T, LE FLOC'H N, LOUVEAU I, et al. Meal frequency changes the basal and time-course profiles of plasma nutrient concentrations and affects feed efficiency in young growing pigs[J]. J Anim Sci, 2014, 92(5): 2008-2016. DOI:10.2527/jas.2013-7505 |
| [7] |
ZHANG H, LIU J J, ZHANG X P, et al. Transcriptomic responses in the livers and jejunal mucosa of pigs under different feeding frequencies[J]. Animals (Basel), 2019, 9(9): 675. |
| [8] |
YAN H L, CAO S C, LI Y, et al. Reduced meal frequency alleviates high-fat diet-induced lipid accumulation and inflammation in adipose tissue of pigs under the circumstance of fixed feed allowance[J]. Eur J Nutr, 2020, 59(2): 595-608. DOI:10.1007/s00394-019-01928-3 |
| [9] |
SCHNEIDER J D, TOKACH M D, DRITZ S S, et al. Effects of feeding schedule on body condition, aggressiveness, and reproductive failure in group-housed sows,[J]. J Anim Sci, 2007, 85(12): 3462-3469. DOI:10.2527/jas.2007-0345 |
| [10] |
陈祥文, 王忠, 黄春玲. 自由采食与分餐饲喂两种饲喂方法对猪生长性能的影响[J]. 今日养猪业, 2011(4): 37-38. CHEN X W, WANG Z, HUANG C L. Effects of free feeding and divided feeding on growth performance of pigs[J]. Pigs Today, 2011(4): 37-38. DOI:10.3969/j.issn.1673-8977.2011.04.015 (in Chinese) |
| [11] |
SUN S M, HANZAWA F, UMEKI M, et al. Time-restricted feeding suppresses excess sucrose-induced plasma and liver lipid accumulation in rats[J]. PLoS One, 2018, 13(8): e0201261. DOI:10.1371/journal.pone.0201261 |
| [12] |
ALLEE G L, ROMSOS D R, LEVEILLE G A, et al. Metabolic adaptation induced by meal-eating in the pig[J]. J Nutr, 1972, 102(9): 1115-1122. DOI:10.1093/jn/102.9.1115 |
| [13] |
COLPOYS J D, JOHNSON A K, GABLER N K. Daily feeding regimen impacts pig growth and behavior[J]. Physiol Behav, 2016, 159: 27-32. DOI:10.1016/j.physbeh.2016.03.003 |
| [14] |
CHIANG J Y L, FERRELL J M. Bile acid metabolism in liver pathobiology[J]. Gene Expr, 2018, 18(2): 71-87. DOI:10.3727/105221618X15156018385515 |
| [15] |
LIU Y Y, KONG X F, JIANG G L, et al. Effects of dietary protein/energy ratio on growth performance, carcass trait, meat quality, and plasma metabolites in pigs of different genotypes[J]. J Anim Sci Biotechnol, 2015, 6(1): 36. DOI:10.1186/s40104-015-0036-x |
| [16] |
KELLEY J J, TSAI A C. Effect of pectin, gum arabic and agar on cholesterol absorption, synthesis, and turnover in rats[J]. J Nutr, 1978, 108(4): 630-639. DOI:10.1093/jn/108.4.630 |
| [17] |
SODHI S S, SONG K D, GHOSH M, et al. Comparative transcriptomic analysis by RNA-seq to discern differential expression of genes in liver and muscle tissues of adult Berkshire and Jeju Native Pig[J]. Gene, 2014, 546(2): 233-242. DOI:10.1016/j.gene.2014.06.005 |
| [18] |
ZHANG Y N, WANG Y J, WANG X Y, et al. Acetyl-coenzyme A acyltransferase 2 promote the differentiation of sheep precursor adipocytes into adipocytes[J]. J Cell Biochem, 2019, 120(5): 8021-8031. DOI:10.1002/jcb.28080 |
| [19] |
LI Y L, LIU X X, NIU L, et al. Proteomics analysis reveals an important role for the PPAR signaling pathway in DBDCT-induced hepatotoxicity mechanisms[J]. Molecules, 2017, 22(7): 1113. DOI:10.3390/molecules22071113 |
| [20] |
许瑞霞, 高磊, 赵伟利, 等. FABP4基因在阿勒泰羊尾脂沉积与代谢模型中的表达变化规律[J]. 遗传, 2015, 37(2): 174-182. XU R X, GAO L, ZHAO W L, et al. Analysis of FABP4 expression pattern in rump fat deposition and metabolism of Altay sheep[J]. Hereditas (Beijing), 2015, 37(2): 174-182. DOI:10.3760/cma.j.issn.1003-9406.2015.02.034 (in Chinese) |
| [21] |
HUANG H, MCINTOSH A L, MARTIN G G, et al. FABP1:A novel hepatic endocannabinoid and cannabinoid binding protein[J]. Biochemistry, 2016, 55(37): 5243-5255. DOI:10.1021/acs.biochem.6b00446 |
| [22] |
WANG Q, LI H, LI N, et al. Tissue expression and association with fatness traits of liver fatty acid binding protein gene in chicken[J]. Poult Sci, 2006, 85(11): 1890-1895. DOI:10.1093/ps/85.11.1890 |
| [23] |
MARTIN G G, CHUNG S, LANDROCK D, et al. FABP-1 gene ablation impacts brain endocannabinoid system in male mice[J]. J Neurochem, 2016, 138(3): 407-422. DOI:10.1111/jnc.13664 |
| [24] |
CHASTANET F, PAHM A A, PEDERSEN C, et al. Effect of feeding schedule on apparent energy and amino acid digestibility by growing pigs[J]. Anim Feed Sci Technol, 2007, 132(1-2): 94-102. DOI:10.1016/j.anifeedsci.2006.03.012 |
| [25] |
BOTT A J, SHEN J L, TONELLI C, et al. Glutamine anabolism plays a critical role in pancreatic cancer by coupling carbon and nitrogen metabolism[J]. Cell Rep, 2019, 29(5): 1287-1298. DOI:10.1016/j.celrep.2019.09.056 |
| [26] |
ZHOU Y, EID T, HASSEL B, et al. Novel aspects of glutamine synthetase in ammonia homeostasis[J]. Neurochem Int, 2020, 140: 104809. DOI:10.1016/j.neuint.2020.104809 |
| [27] |
HÄUSSINGER D, SCHLIESS F. Glutamine metabolism and signaling in the liver[J]. Front Biosci, 2007, 12: 371-391. DOI:10.2741/2070 |
| [28] |
GRIFFIN J W D, LIU Y, BRADSHAW P C, et al. In silico preliminary association of ammonia metabolism genes GLS, CPS1, and GLUL with risk of alzheimer's disease, major depressive disorder, and type 2 diabetes[J]. J Mol Neurosci, 2018, 64(3): 385-396. DOI:10.1007/s12031-018-1035-0 |
| [29] |
HÄUSSINGER D, LANG F. Cell volume in the regulation of hepatic function: A mechanism for metabolic control[J]. Biochim Biophys Acta, 1991, 1071(4): 331-350. DOI:10.1016/0304-4157(91)90001-D |
| [30] |
HÄUSSINGER D, GRAF D, WEIERGRÄBER O H. Glutamine and cell signaling in liver[J]. J Nutr, 2001, 131(9): 2509S-2514S. DOI:10.1093/jn/131.9.2509S |
| [31] |
HALLBRUCKER C, VOM DAHL S V, LANG F, et al. Control of hepatic proteolysis by amino acids[J]. Eur J Biochem, 1991, 197(3): 717-724. DOI:10.1111/j.1432-1033.1991.tb15963.x |
| [32] |
ST-PIERRE M V, KULLAK-UBLICK G A, HAGENBUCH B, et al. Transport of bile acids in hepatic and non-hepatic tissues[J]. J Exp Biol, 2001, 204(10): 1673-1686. DOI:10.1242/jeb.204.10.1673 |
| [33] |
HÄUSSINGER D, KUBITZ R, REINEHR R, et al. Molecular aspects of medicine: from experimental to clinical hepatology[J]. Mol Aspects Med, 2004, 25(3): 221-360. DOI:10.1016/j.mam.2004.02.001 |
(编辑 范子娟)



