哺乳动物的精子发生是个非常复杂的过程[1],分为3个阶段:首先,原始生殖细胞进行有丝分裂;其次,精母细胞经历两次连续的分裂过程产生单倍体精子细胞;最后,精子细胞的核、顶体、细胞质等亚细胞器结构发生重塑,最终逐渐发育成熟[1-2]。附睾是位于睾丸表面的供精子成熟和储存的长曲小管,它包括3个独立但密切联系的部分:假复层柱状上皮(主细胞、基细胞、亮细胞等)、腔内环境和精子[3]。腔内环境通过直接分泌或囊泡运输介导上皮细胞和精子之间蛋白质、小分子代谢物的转移和交换[4]。根据解剖学规定,附睾分为头、体和尾部3个区域[5],头部和体部是精子成熟的地方,尾部主要发挥存储精子的作用[6-7]。精子进入附睾之前,虽然形态结构和染色质已基本成熟,但不具备识别、结合卵细胞的能力[8]。精子发生和成熟是精子和复杂的外部环境之间的相互作用所驱动的,附睾对于睾丸后精子成熟极其重要[9]。在相同且适宜的培养基中,附睾尾部精子可以进行直线运动,而附睾头部精子则表现出微弱摆动,极少有直线运动的个体。未成熟精子会影响早期胚胎的发生,且降低卵母细胞受精率,有研究表明,使用附睾头部精子受精的卵母细胞在后续发育中将产生严重缺陷。在现代农业生产中,绵羊具有极大的经济价值。因此,了解绵羊附睾的结构和功能,阐明精子在附睾头、体和尾部迁移过程中蛋白质表达量的变化,对探索精子成熟机制和调节雄性生育能力具有指导意义。
生物信息学的发展为寻找精子成熟相关蛋白质的种类及功能分析提供了条件。近年来,蛋白质组学技术已成功应用于人类[10]、猪[11]、水牛[12]、猫[13]和小鼠[14]等不同物种的精子研究上。随着液相色谱和质谱技术的发展,蛋白质组学已经在生物学领域取得了长足的进步,并产生了大量的原始数据和研究材料,为探索潜在机制和发现具有临床意义的潜在生物标志物打开了另一扇窗。大分子物质穿过核膜的双向运输是真核细胞的特点[15],核孔复合体(NPC)是细胞核膜上沟通核质与胞质的开口,由内外两层膜的局部融合所形成,它能调节核质运输,是参与转录、翻译和细胞周期等过程的关键结构之一[16]。精子在附睾中不断成熟的过程离不开核质物质转运[17],因此,通过蛋白质组学的方法找到相关的差异表达蛋白是阐明问题的关键[18]。
本研究采用TMT标记定量蛋白质组学的方法,通过肽TMT标记、高pH反相色谱柱分级分离和LC-MS/MS分析,明晰绵羊附睾头、体和尾部精子的差异与特点。鉴定出616个差异表达蛋白,通过蛋白质免疫印迹、免疫荧光和流式细胞术验证精子成熟、核质转运的相关蛋白,并进一步探究其作用机制。
1 材料与方法 1.1 主要材料从日照市屠宰场获得性成熟(12月龄)雄性湖羊的附睾样品,把附睾按解剖结构分为头、体和尾部3个区域,依次提取精子。聚丙烯酰胺凝胶、PVDF膜、胎牛血清、Hoechst 33342和Triton-X均购自北京索莱宝公司;抗体均购自武汉Abcolnol公司;蛋白质组学部分所用试剂、仪器均由上海拜谱生物科技有限公司提供。
1.2 测序与质控9例绵羊精子细胞,3组样本(头、体、尾),每组3个生物学重复。从每个样品中取出100 μg肽,并使用高pH反相色谱柱(PierceTM High pH Reversed-Phase Peptide Fractionation Kit, Thermo Fisher)对干燥的肽进行处理。从每个样品中取出适量的肽,并使用Easy nLC 1200色谱系统(Thermo Scientific)进行色谱分离。将得到的LC-MS/MS原始文件导入Proteome Discoverer 2.4软件,用于对数据库进行数据解释和蛋白质鉴定(https://www.uniprot.org)。初始搜索设置在10 ppm的前体质量窗口,修饰组如下:固定修饰:氨基甲酰甲基(C)、TMT10plex(K)、TMT10plex(N-term);可变修饰:Oxidation(M)和Acetyl(Protein N-term)。使用质量控制后的数据进行后续分析。
1.3 差异表达蛋白质分析使用Microsoft Excel和R软件对质量控制后的数据进行分析。差异表达蛋白的筛选标准为Fold change>1.2或<0.83,P<0.05。进行GO、KEGG注释和富集分析,以及针对多个测试的FDR校正。富集的GO和KEGG通路在P<0.05具有统计学意义。
1.4 蛋白质免疫印迹检测蛋白质表达量:通过十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)分离蛋白质,然后转移到聚偏二氟乙烯膜(polyvinylidene fluoride)上。5%胎牛血清白蛋白(BSA)室温孵育封闭2 h,一抗孵育PVDF膜过夜,4 ℃。HRP标记的二抗孵育PVDF膜2 h,曝光,使用Image J进行灰度值统计分析。
1.5 流式细胞术使用流式细胞仪检测精子FITC水平。1)制备适当浓度的精子细胞悬液,0.1% Triton-X工作液细胞膜穿孔30 min。2)5% BSA封闭30 min。3)一抗孵育90 min,37 ℃。4)FITC标记的二抗孵育1 h,避光37 ℃。5)使用流式细胞仪检测样品。
1.6 免疫荧光免疫荧光定位蛋白质表达部位:1)制备适当浓度的精子细胞悬液,取10 μL精子悬液滴在载玻片上,室温静置10 min。2)4%多聚甲醛固定30 min。3) 0.1% Triton-X工作液细胞膜穿孔30 min。4)5% BSA封闭30 min。5)一抗孵育过夜,4 ℃。6)FITC标记的二抗孵育1 h,37 ℃。7)Hoechst 33342用于核染色,5 min。8)成像系统(Carl Zeiss AG, German)拍照并进行后续分析。
2 结果 2.1 附睾头、体和尾部精子的全蛋白结果本研究使用TMT标记定量蛋白质组学分析绵羊附睾头、体和尾部3个区域的精子之间蛋白质表达量的变化。通过LC-MS/MS分析,从22 841个唯一性肽中鉴定到2 691种蛋白质。GO注释分析结果如下,生物过程结果表明,精子表达蛋白主要以细胞过程(cellular process,966)、代谢过程(metabolic process,677)、生物调节(biological regulation,195)为主;分子功能结果表明,精子表达蛋白主要以结合(binding,1 105)、催化活性(catalytic activity,858)、结构分子活性(structural molecule activity,114)、ATP酶(ATPase,110)为主;细胞组分结果表明,精子表达蛋白主要以细胞解剖实体(cellular anatomical entity,542)、含蛋白质复合物(protein-containing complex,319)为主(图 1)。KEGG注释通路[19]主要为代谢途径(metabolic pathways)、氧化磷酸化(oxidative phosphorylation)、产热(thermogenesis)、碳代谢(carbon metabolism)、内质网中的蛋白质加工(protein processing in endoplasmic reticulum)、亨延顿病(Huntington disease)和帕金森病(Parkinson disease)等(图 2)。
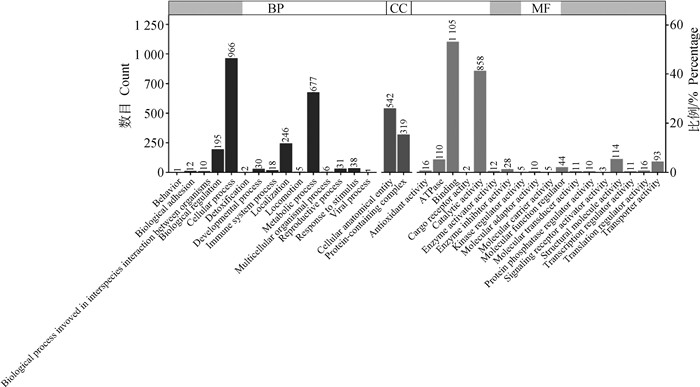
|
图 1 附睾头、体和尾部精子的蛋白质组GO分析 Fig. 1 GO analysis of sperm from caput, corpus and cauda in epididymis |
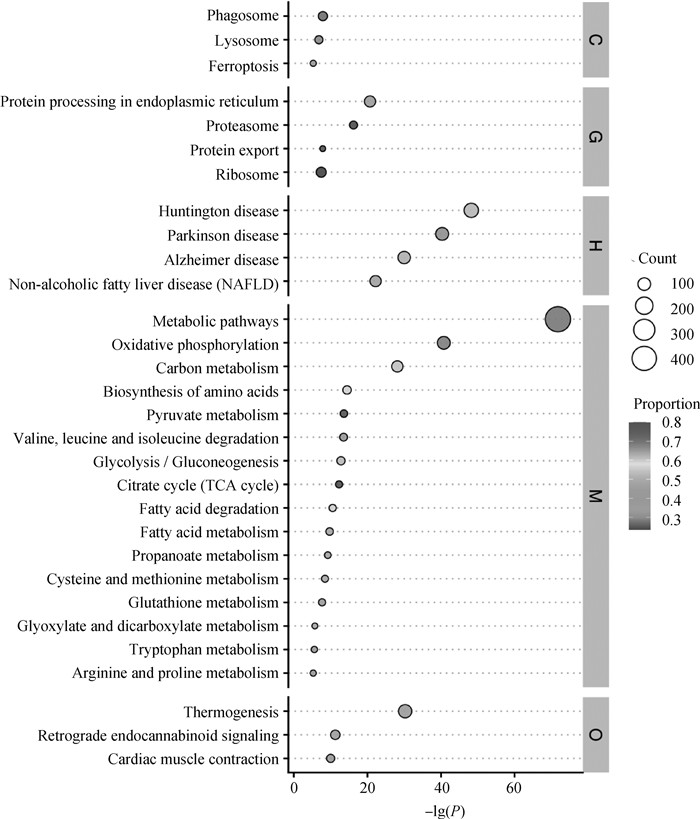
|
图 2 附睾头、体和尾部精子的蛋白质组KEGG通路富集分析气泡图 Fig. 2 KEGG pathway enrichment analysis bubble chart of sperm from caput, corpus and cauda in epididymis |
经T-test检验,在组间差异比较的阈值为P<0.05和Fold change>1.2或<0.83时,鉴定出差异表达蛋白616个。其中,尾vs.头组鉴定出309个差异表达蛋白(上调:213个,下调:96个);尾vs.体组鉴定出167个差异表达蛋白(上调:107个,下调:60个);体vs.头组鉴定出140个差异表达蛋白(上调:88个,下调:52个)。本研究锁定入核载体蛋白组成亚基—KPNA4,作为候选蛋白并后续验证。KPNA4在尾vs.头组中差异表达倍数为1.95,在尾vs.体组中差异表达倍数为1.45,均显著。Venn图如图 3所示。
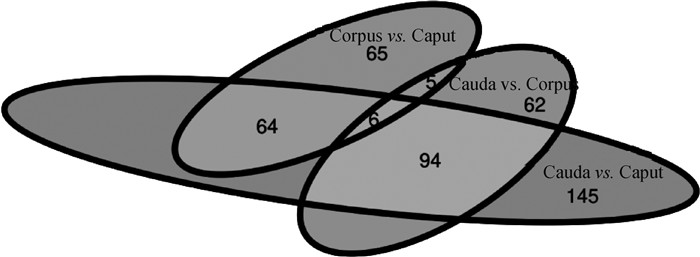
|
图 3 尾vs.头、尾vs. 体和体vs.头3组差异表达蛋白质的集合关系 Fig. 3 The collective relationship of the 3 groups (cauda vs. caput group, cauda vs. corpus group, corpus vs. caput group) of differentially expressed proteins |
对附睾头、体和尾部精子进行蛋白质免疫印迹试验,精子在附睾移行过程中,KPNA4表达量呈上升趋势(图 4),精子样本分组为:附睾头部组(caput)、附睾体部组(corpus)和附睾尾部组(cauda)。
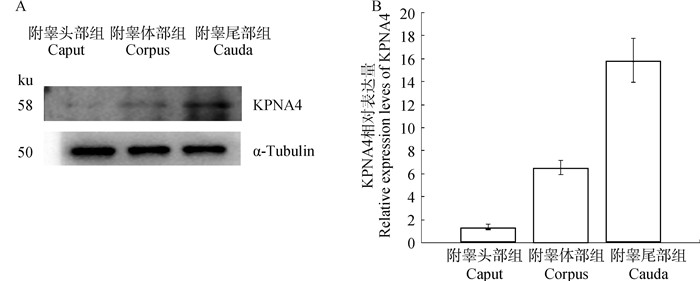
|
A. 蛋白质免疫印迹结果图,KPNA4为58 ku;α-Tubulin为50 ku,内参蛋白。B. 蛋白质相对表达量结果,使用Image J分析[20] A. Western blot, KPNA4 is 58 ku; α-Tubulin is 50 ku, internal reference protein. B. Analysis of relative expression levels using Image J [20] 图 4 Western blot结果 Fig. 4 Western blot result |
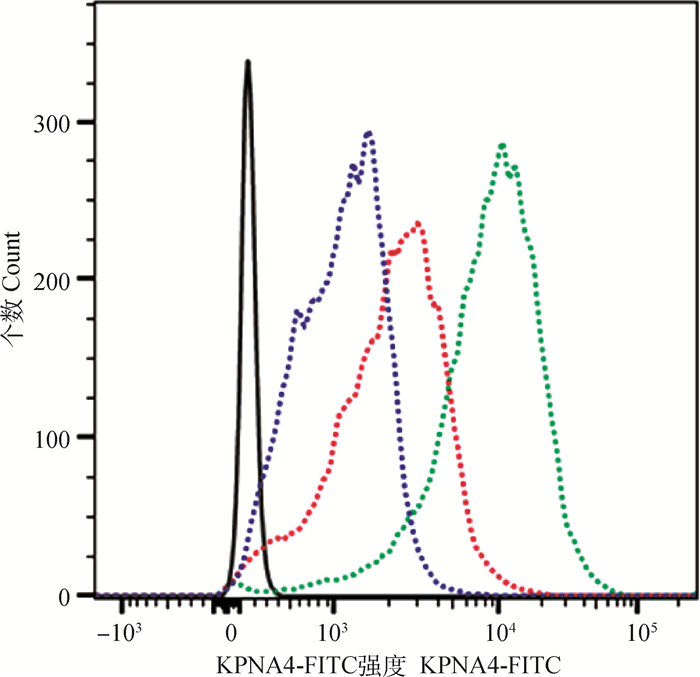
对附睾头、体和尾部精子进行流式细胞术试验,FITC强度梯度增高,因此,KPNA4的表达量在3个区域的精子中呈上升趋势(图 5),精子样本分组为:附睾头部组(caput)、附睾体部组(corpus)和附睾尾部组(cauda)。
|
纵坐标为精子数,横坐标为FITC强度。蓝色虚线. 附睾头部的精子;红色虚线. 附睾体部的精子;绿色虚线.附睾尾部的精子;黑色实线.阴性对照 The ordinate is the number of sperm, and the abscissa is the FITC intensity. Blue dotted line. Sperm in the caput of the epididymis; red dotted line. Sperm in the corpus of the epididymis; green dotted line. Sperm in the cauda of the epididymis; black solid line. Negative control 图 5 KPNA4流式细胞术结果 Fig. 5 KPNA4 flow cytometry results |
KPNA4作为入核载体蛋白应主要分布于核仁或细胞质,免疫荧光结果与预测相吻合:显示其定位于精子头部,尾部少有表达。附睾头、体和尾部3个区域的精子均定位于精子头部(图 6)。精子样本分组为:附睾头部组(caput)、附睾体部组(corpus)和附睾尾部组(cauda)。KPNA4呈绿色,Hoechst 33342用于核染呈蓝色,Merge为KPNA4-FITC和Hoechst融合图。

|
KPNA4. 绿色荧光;Hoechst. 核染;Merge.KPNA4-FITC和Hoechst融合图 KPNA4. Green fluorescence; Hoechst. Nuclear staining; Merge. KPNA4 and Hoechst merge image 图 6 免疫荧光结果 Fig. 6 Immunofluorescence results |
蛋白质组学是对生物系统中全部蛋白质的研究,正在成为一门数据丰富的科学,蛋白质的序列和结构在公共数据库中被全面编译,这些不断更新的数据库除了提供各种生物的氨基酸序列和相关基因外,还为蛋白质组学比较分析、蛋白质注释、多重比对计算等提供了重要的信息。随着串联质谱(MS)技术的发展,蛋白质表达和翻译后修饰(PTMs)可以在全球范围内的各种生物系统中进行研究。蛋白质的翻译后修饰广泛存在,从而改变其结构和功能,因此数据信息不能简单地依靠基因组或转录组的测序结果——蛋白质组学是基因组学和转录组学的补充与进步。近年来,生物信息学飞速地发展,利用计算机强大的计算能力解决了生物试验中出现的大量数据,并在其中发现了更多的生物规律和特征[21]。有大量文献证明精子在附睾中进一步成熟,但详细的机制、基因和蛋白质尚不清楚。蛋白质组学的发展为解决这一问题提供了可能。本研究使用TMT标定定量分析来构建绵羊头、体和尾部3个区域精子的蛋白质组谱。成功鉴定出了307、167、140个差异表达蛋白,虽然富集的通路多与代谢相关,但发现了位于精子头部的入核载体蛋白亚基KPNA4[22],且差异倍数排名靠前。KPNA4的表达水平通过流式细胞术和蛋白质印迹验证,结果与测序数据一致,排除测序结果假阳性的可能。
精子发生过程中,顶体位于凝聚核的一侧,染色体主要由鱼精蛋白而非组蛋白组成,尽管精子在睾丸中已呈现出特有形状,但其功能并不成熟。精子成熟伴随着精子活力和能量代谢的增强,蛋白质、脂质和小RNA等物质的含量均发生变化,附睾通过离子运输、囊泡分泌等途径为精子成熟提供合适的环境。核质转运是哺乳动物配子发生的驱动因素[23]。核孔复合体(NPC)是嵌入双层核膜并介导细胞核和细胞质之间的分子运输的巨大蛋白质,允许小于40 ku的分子被动扩散,蛋白质、mRNA等大分子物质则需主动运输[24-25]。Ran作为重要分子,调节货物受体复合物的组装和解体,在核内主要是RanGTP形式,在核外主要为RanGDP形式[26]。需要入核的蛋白质与核定位信号(NLS)结合[27],被受体蛋白importin-α识别,后者识别并结合importin-β形成三聚体复合物[28],通过核孔蛋白上的多个苯丙氨酸-甘氨酸(FG)重复序列,该复合物中的importin-β与NPC发生相互作用,这使得该复合物能够转移到核质中[29-30]。在小鼠中,目前已知的importin-α有7种[31-32](impα1、impα2、impα3、impα4、impα6、impα7、impα8),不同亚型之间的区别为NLS结合特异性[33]。有研究表明,KPNA4基因敲除的雄性小鼠比对照组小鼠生育力低,产仔数少,顶体反应受到损害。体外受精和精子活力测定表明,来自KPNA4缺失小鼠的精子质量和活力显著降低[34-35]。精子成熟和发生过程伴随着大量物质的合成、代谢与运输[31],KPNA4作为入核运输蛋白,在此过程中行使着重要功能。
4 结论通过对绵羊附睾头、体和尾部精子的蛋白质组学分析,本研究确定了表达量提高的KPNA4可能是参与精子成熟过程的相关蛋白,与物质的核质跨膜运输有关。本研究从分子层面展示了精子在附睾运输过程中蛋白质的动态变化,挖掘可能调控精子成熟的生物学机制,并为后续研究提供了丰富的资源和依据。
| [1] |
BRIZ M D, BONET S, PINART B, et al. Comparative study of boar sperm coming from the caput, corpus, and cauda regions of the epididymis[J]. J Androl, 1995, 16(2): 175-188. |
| [2] |
CHOCU S, CALVEL P, ROLLAND A D, et al. Spermatogenesis in mammals: proteomic insights[J]. Syst Biol Reprod Med, 2012, 58(4): 179-190. DOI:10.3109/19396368.2012.691943 |
| [3] |
JAMES E R, CARRELL D T, ASTON K I, et al. The role of the epididymis and the contribution of epididymosomes to mammalian reproduction[J]. Int J Mol Sci, 2020, 21(15): 5377. DOI:10.3390/ijms21155377 |
| [4] |
OZKOCER S E, KONAC E. The current perspective on genetic and epigenetic factors in sperm maturation in the epididymis[J]. Andrologia, 2021, 53(3): e13989. |
| [5] |
LIU J M, YAO T, WENG X X, et al. Antioxidant properties and transcriptome of cauda epididymis with different levels of fertility in Hu lambs[J]. Theriogenology, 2022, 182: 85-95. DOI:10.1016/j.theriogenology.2022.01.035 |
| [6] |
NIXON B, DE IULIIS G N, HART H M, et al. Proteomic profiling of mouse epididymosomes reveals their contributions to post-testicular sperm maturation[J]. Mol Cell Proteomics, 2019, 18(S1): S91-S108. |
| [7] |
TINGARI M. The fine structure of the epithelial lining of the epididymis of the camel (Camelus dromedarius) with special reference to regional differences[J]. J Anat, 1989, 165: 201-204. |
| [8] |
CHU C, ZHANG Y L, YU L, et al. Epididymal small non-coding RNA studies: progress over the past decade[J]. Andrology, 2019, 7(5): 681-689. |
| [9] |
ZHAO W S, AHMED S, LIU J X, et al. Comparative iTRAQ proteomics identified proteins associated with sperm maturation between Yak and Cattleyak epididymis[J]. BMC Vet Res, 2021, 17(1): 255. DOI:10.1186/s12917-021-02907-9 |
| [10] |
NOWICKA-BAUER K, KURPISZ M. Current knowledge of the human sperm proteome[J]. Exp Rev Proteomics, 2013, 10(6): 591-605. DOI:10.1586/14789450.2013.846221 |
| [11] |
WEBER A, ARGENTI L E, DE SOUZA A P B, et al. Ready for the journey: A comparative proteome profiling of porcine cauda epididymal fluid and spermatozoa[J]. Cell Tissue Res, 2020, 379(2): 389-405. DOI:10.1007/s00441-019-03080-0 |
| [12] |
ZHANG P F, HUANG Y L, FU Q, et al. Integrated analysis of phosphoproteome and ubiquitylome in epididymal sperm of buffalo (Bubalus bubalis)[J]. Mol Reprod Dev, 2021, 88(1): 15-33. DOI:10.1002/mrd.23432 |
| [13] |
ROWLISON T, CLELAND T P, OTTINGER M A, et al. Novel proteomic profiling of epididymal extracellular vesicles in the domestic Cat reveals proteins related to sequential sperm maturation with differences observed between normospermic and teratospermic individuals[J]. Mol Cell Proteomics, 2020, 19(12): 2090-2104. DOI:10.1074/mcp.RA120.002251 |
| [14] |
SUN Z L, LI S J, YU Y X, et al. Alterations in epididymal proteomics and antioxidant activity of mice exposed to fluoride[J]. Arch Toxicol, 2018, 92(1): 169-180. DOI:10.1007/s00204-017-2054-2 |
| [15] |
HAMPOELZ B, ANDRES-PONS A, KASTRITIS P, et al. Structure and assembly of the nuclear pore complex[J]. Annu Rev Biophys, 2019, 48: 515-536. DOI:10.1146/annurev-biophys-052118-115308 |
| [16] |
SAKUMA S, D'ANGELO M A. The roles of the nuclear pore complex in cellular dysfunction, aging and disease[J]. Semin Cell Dev Biol, 2017, 68: 72-84. DOI:10.1016/j.semcdb.2017.05.006 |
| [17] |
PACI G, CARIA J, LEMKE E A. Cargo transport through the nuclear pore complex at a glance[J]. J Cell Sci, 2021, 134(2): jcs247874. DOI:10.1242/jcs.247874 |
| [18] |
SKERRETT-BYRNE D A, ANDERSON A L, HULSE L, et al. Proteomic analysis of koala (phascolarctos cinereus) spermatozoa and prostatic bodies[J]. Proteomics, 2021, 21(19): 2100067. DOI:10.1002/pmic.202100067 |
| [19] |
KANEHISA M, SATO Y, KAWASHIMA M. KEGG mapping tools for uncovering hidden features in biological data[J]. Protein Sci, 2022, 31(1): 47-53. DOI:10.1002/pro.4172 |
| [20] |
SCHROEDER A B, DOBSON E T A, RUEDEN C T, et al. The ImageJ ecosystem: Open-source software for image visualization, processing, and analysis[J]. Protein Sci, 2021, 30(1): 234-249. DOI:10.1002/pro.3993 |
| [21] |
TANG B H, PAN Z X, YIN K, et al. Recent advances of deep learning in bioinformatics and computational biology[J]. Front Genet, 2019, 10: 214. DOI:10.3389/fgene.2019.00214 |
| [22] |
PING X Y, CHENG Y L, BAO J, et al. KPNA4 is involved in cataract formation via the nuclear import of p53[J]. Gene, 2021, 786: 145621. DOI:10.1016/j.gene.2021.145621 |
| [23] |
ITMAN C, MIYAMOTO Y, YOUNG J, et al. Nucleocytoplasmic transport as a driver of mammalian gametogenesis[J]. Semin Cell Dev Biol, 2009, 20(5): 607-619. DOI:10.1016/j.semcdb.2009.05.002 |
| [24] |
ALBER F, DOKUDOVSKAYA S, VEENHOFF L M, et al. The molecular architecture of the nuclear pore complex[J]. Nature, 2007, 450(7170): 695-701. DOI:10.1038/nature06405 |
| [25] |
WÄLDE S, KEHLENBACH R H. The Part and the Whole: functions of nucleoporins in nucleocytoplasmic transport[J]. Trends Cell Biol, 2010, 20(8): 461-469. DOI:10.1016/j.tcb.2010.05.001 |
| [26] |
NEMERGUT M E, MIZZEN C A, STUKENBERG T, et al. Chromatin docking and exchange activity enhancement of RCC1 by histones H2A and H2B[J]. Science, 2001, 292(5521): 1540-1543. DOI:10.1126/science.292.5521.1540 |
| [27] |
KALITA J, KAPINOS L E, LIM R Y H. On the asymmetric partitioning of nucleocytoplasmic transport-recent insights and open questions[J]. J Cell Sci, 2021, 134(7): jcs240382. DOI:10.1242/jcs.240382 |
| [28] |
ROBBINS J, DILWORTHT S M, LASKEY R A, et al. Two interdependent basic domains in nucleoplasmin nuclear targeting sequence: identification of a class of bipartite nuclear targeting sequence[J]. Cell, 1991, 64(3): 615-623. DOI:10.1016/0092-8674(91)90245-T |
| [29] |
LINDSAY M E, PLAFKER K, SMITH A E, et al. Npap60/Nup50 is a tri-stable switch that stimulates importin-α: β-mediated nuclear protein import[J]. Cell, 2002, 110(3): 349-360. DOI:10.1016/S0092-8674(02)00836-X |
| [30] |
GILCHRIST D, MYKYTKA B, REXACH M. Accelerating the rate of disassembly of karyopherin cargo complexes[J]. J Biol Chem, 2002, 277(20): 18161-18172. DOI:10.1074/jbc.M112306200 |
| [31] |
MAJOR A T, WHILEY P A F, LOVELAND K L. Expression of nucleocytoplasmic transport machinery: clues to regulation of spermatogenic development[J]. Biochim Biophys Acta Biomembr, 2011, 1813(9): 1668-1688. DOI:10.1016/j.bbamcr.2011.03.008 |
| [32] |
HU J J, WANG F C, YUAN Y, et al. Novel importin-α family member Kpna7 is required for normal fertility and fecundity in the mouse[J]. J Biol Chem, 2010, 285(43): 33113-33122. DOI:10.1074/jbc.M110.117044 |
| [33] |
MIYAMOTO Y, BOAG P R, HIME G R, et al. Regulated nucleocytoplasmic transport during gametogenesis[J]. Biochim Biophys Acta Gene Regul Mech, 2012, 1819(6): 616-630. DOI:10.1016/j.bbagrm.2012.01.015 |
| [34] |
MIYAMOTO Y, SASAKI M, MIYATA H, et al. Genetic loss of importin α4 causes abnormal sperm morphology and impacts on male fertility in mouse[J]. FASEB J, 2020, 34(12): 16224-16242. DOI:10.1096/fj.202000768RR |
| [35] |
YOUNG J C, LY-HUYNH J D, LESCESEN H, et al. The nuclear import factor importin α4 can protect against oxidative stress[J]. Biochim Biophys Acta Mol Cell Res, 2013, 1833(10): 2348-2356. DOI:10.1016/j.bbamcr.2013.06.007 |
(编辑 郭云雁)



