2. 宁夏回族自治区反刍动物分子细胞育种重点实验室, 银川 750021
2. Key Laboratory of Ruminant Molecular Cell Breeding, Ningxia Hui Autonomous Region, Yinchuan 750021, China
奶牛乳房炎是由病原微生物感染乳腺组织所产生的一种炎症性疾病,其发病率高,治愈率低,易复发,治疗代价昂贵,可造成产奶量下降、乳品质降低,以至于被迫过早淘汰奶牛,给现代奶业造成了极大的经济损失[1-2]。近年来,国内外研究人员在奶牛乳房炎的分子调控机制方面做了大量研究工作,发现乳房炎的发生、发展过程及易感性和抗病性均受多个基因组成的基因网络的调控,其中NF-κB信号通路是重要的调节途径[3-4]。但是,这些基因或信号通路对奶牛乳房炎的分子调控机制尚不完全清楚,因此阻碍了奶牛乳房炎抗性分子育种工作的有效开展。
lncRNA是一种长度超过200个核苷酸的内源性非编码RNA分子,在包括机体免疫在内的多种生物学过程中发挥重要作用[5]。lncRNA的特殊结构和亚细胞定位使其能够与多种分子相互作用,通过转录、表观修饰和转录后3个水平发挥生物学作用[6-7]。例如lncRNA可以作为miRNA的分子海绵,竞争性地吸附与mRNA结合的miRNA,以减弱miRNA对靶mRNA表达的抑制,从而促进了靶基因的表达[8-9]。差异表达lncRNA可以调节人和模式动物炎症性疾病过程,但是,不同物种间lncRNA的保守性较低,且靶向调节的基因序列在不同物种间也存在差异,这使得人和模式动物上的研究结果很难借鉴并直接应用于奶牛[10-11]。因此,lncRNA在奶牛乳房炎中的作用机制仍然需要深入研究。目前,在奶牛的炎症性疾病研究中,已在肺炎、关节炎和乳房炎等多种疾病中鉴定出一些差异表达lncRNAs,其功能主要富集于NF-κB、TNF、MAPK、Rap1、Jak-STAT、mTOR、PI3K-Akt和TGF-β等信号通路,参与调节炎症反应、应激反应、物质代谢、血管生成、细胞分化、趋化因子活性和细胞凋亡等多个生物学过程[12-15]。目前,仅有4个lncRNAs(lncRNA H19、lncRNA TUB、lncRNA XIST和LRRC75A-AS1)在奶牛乳腺炎调控机制方面进行了作用机制研究[16-19]。
前期本课题组通过乳房炎乳腺组织lncRNA测序分析获得了差异表达lncRNA BCL2(数据未发表),提示它可能在奶牛乳房炎中有重要功能。为探讨lncRNA BCL2的功能,本研究采用生物信息学和lncRNA超表达等技术研究lncRNA BCL2对炎症性奶牛乳腺上皮细胞中的炎症因子和凋亡相关基因表达的影响,为奶牛乳腺炎调控研究提供基础。
1 材料与方法 1.1 材料1.1.1 组织与细胞 本试验分别采集处于泌乳盛期的36月龄健康和患有临床乳腺炎的2胎次荷斯坦奶牛的乳腺组织,液氮冷冻保存;所采用的乳腺上皮细胞为实验室鉴定和冻存的奶牛乳腺上皮细胞[20]。
1.1.2 主要试剂与仪器 DMEM/F12培养基购于Hyclone;胎牛血清(FBS)购于BI;荧光定量试剂盒(货号为MF787-01)购于诺唯赞生物科技有限公司;逆转录试剂盒(货号为RR047A)和TRIzol购于TaKaRa;X-tremeGENE HP转染试剂(货号为06366236001)购于Roche;Opti-MEM购于广州锐博生物有限公司。多功能酶标仪、普通PCR仪、电泳凝胶成像仪和CFX 96 Touch荧光定量PCR仪均为Bio-Rad公司产品。
1.2 试验方法1.2.1 lncRNA BCL2的生物信息学分析 在本课题组前期对奶牛乳腺组织转录组测序分析中(数据未发表),获得了序列长度为242 bp的lncRNA TCONS-00019437。根据生物信息学分析,发现该lncRNA可能通过竞争性结合多个miRNAs,从而促进其靶基因BCL2的表达,故将该lncRNA命名为lncRNA BCL2。为研究lncRNA BCL2的功能,本研究利用RNAhybrid软件预测分析lncRNA BCL2可能海绵吸附的miRNAs;利用Targetscan和miRwalk预测相关miRNA靶基因;利用Cluster Profiler软件包(R-3.2.4)对靶基因进行KEGG的功能富集分析。
1.2.2 乳腺上皮细胞体外培养及炎症诱导 将乳腺上皮细胞解冻后,使用10%胎牛血清的DMEM/F12培养基,在37℃、5% CO2浓度和100%饱和湿度的条件下,接种培养于6孔细胞培养板中。待细胞融合至60%~70%时,利用终浓度为50 ng·μL-1的LPS诱导细胞,分别在诱导的0 (对照组)、3和6 h收集细胞,用于RNA提取。
1.2.3 lncRNA BCL2载体构建及鉴定 根据lncRNA BCL2序列,由上海生工生物工程有限公司合成两端分别带有BamH I和EcoR I酶切位点的lncRNA BCL2片段,经双酶切和纯化回收后,与pcDNA3.1超表达载体连接,通过重组子筛选等步骤获得lncRNA BCL2_pcDNA3.1超表达重组载体,使用BamH I酶和Avr II酶双酶切重组载体,并利用测序技术检测目标片段的序列,并与转录组测序结果进行序列比对,以验证重组载体的可靠性。
1.2.4 细胞转染及超表达效率检测 待细胞生长至60%~70%融合度时,根据罗氏转染试剂说明书进行细胞转染。每个转染组3个重复,每孔500ng,以pcDNA3.1(+)空质粒转染组为对照组,lncRNA BCL2_pcDNA3.1重组质粒转染组为处理组。加完样后十字方向摇匀放入恒温培养箱中。48h后,在收细胞前6 h加入50 ng·μL-1 LPS诱导细胞产生炎症,随后在LPS诱导6h后收集细胞,提取RNA。通过qPCR检测lncRNA BCL2的超表达效率。
1.2.5 RNA提取及RT-PCR 本试验用TRIzol法,按照TRIzol试剂说明书,分别提取奶牛乳腺组织和LPS诱导0、3和6 h的乳腺上皮细胞的总RNA,多功能酶标仪检测RNA的浓度与纯度。以1 000ng经检测合格RNA为模板,按照逆转录试剂盒说明书分别进行逆转录试验,获得奶牛乳腺组织和LPS诱导0、3和6h的乳腺上皮细胞的cDNA。
1.2.6 引物设计 根据测序获得的lncRNA BCL2序列以及GenBank公布的相关基因的mRNA序列,采用Primer 5.0软件设计了lncRNA BCL2、细胞凋亡相关基因(BAX、BCL2、BAD、CASP3和CASP9)、炎症相关基因(NF-κB/p65、NF-κB/p50、IL-1β、IL-6和IL-8)及内参引物(GAPDH和RPS18),如表 1所示。所有引物的扩增效率均在95%~105%之间。引物由通用生物系统(安徽)有限公司合成,超纯水溶解、备用。
|
|
表 1 qPCR引物序列 Table 1 The primer sequences used for qPCR |
1.2.7 qPCR检测基因表达量 分别以LPS诱导0、3和6 h的细胞和乳腺炎组织cDNA为模板,采用qPCR技术检测lncRNA BCL2的表达量;分别以转染lncRNA BCL2_pcDNA3.1重组质粒和pcDNA3.1(+)空质粒的乳腺上皮细胞的cDNA为模板,采用qPCR技术检测炎症相关基因(IL-1β、IL-6、IL-8、NF-κB/p65和NF-κB/p50)和凋亡相关基因(BAX、BCL2、BAD、CASP3和CASP9)的表达量。qPCR反应体系为20 μL:2×M5 HiPer SYBR Premix EsTaq (withTli RNaseH) 10 μL,上、下游引物各0.8 μL,cDNA模板1.0 μL,灭菌去离子水至7.4 μL。qPCR反应程序:95℃预变性30 s;95℃ 5 s,60℃ 30 s,共45个循环;95℃ 10 s,65℃ 5 s。每个检测指标设置3个复孔。
1.2.8 统计分析 qPCR结果以GAPDH和RPS18为内参,采用2-ΔΔCt方法计算基因的相对表达量,采用SPSS 25.0软件进行组间的差异显著性检验,所有数据用“均值±标准差”表示。*P<0.05表示差异显著;**P<0.01、***P<0.001和****P<0.000 1表示差异极显著。
2 结果 2.1 lncRNA BCL2的生物信息学分析为探究lncRNA BCL2的生物学功能,本研究采用生物信息学方法预测了lncRNA BCL2海绵吸附的miRNAs及其靶基因的功能富集,结果表明,lncRNA BCL2可能海绵吸附54个miRNAs(图 1),其中bta-miR-2436-3p、bta-miR-2894、bta-miR-129-5p、bta-miR-940和bta-miR-2888共同调控靶基因BCL2;Bta-miR-129-5p、bta-miR-2888、bta-miR-2436-3p、bta-miR-1777a、bta-miR-574、bta-miR-664a、bta-miR-671和bta-miR-2382-5p共同调控IGF2。这些miRNAs靶基因功能富集结果表明,lncRNA BCL2可能通过上述miRNAs以及NF-κB、CAMP和PI3K-Akt等细胞炎症和凋亡相关的信号通路而参与细胞炎症与凋亡过程(图 2)。
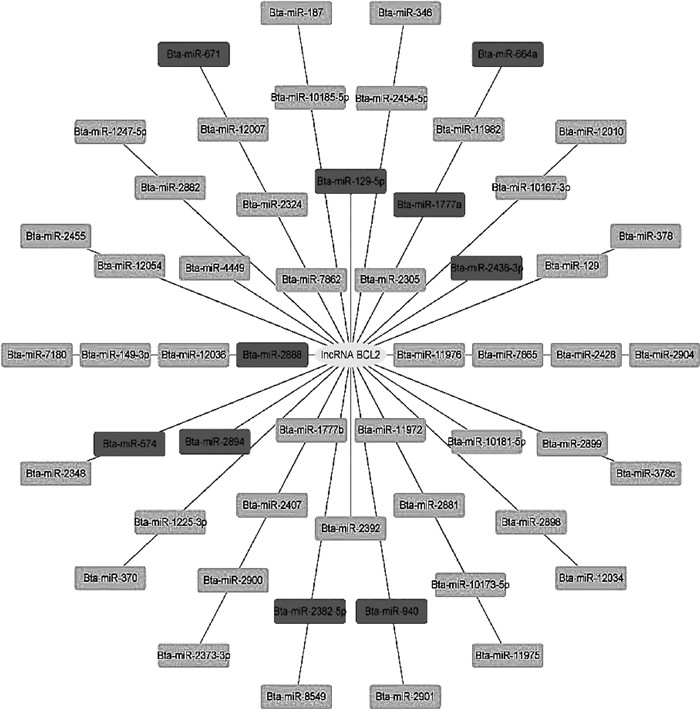
|
图 1 lncRNA BCL2海绵吸附的miRNAs Fig. 1 miRNAs adsorbed by lncRNA BCL2 sponge |

|
图 2 lncRNA BLC2相关的mRNA所富集的信号通路 Fig. 2 Signaling pathway enriched by lncRNA BCL2 related mRNA |
为探究lncRNA BCL2在奶牛乳腺炎中的表达情况,本研究采用RT-qPCR技术检测了lncRNA BCL2在临床乳腺炎奶牛乳腺组织(图 3A)及LPS诱导的乳腺上皮细胞(图 3B)中的表达量,结果表明,与对照组相比,lncRNA BCL2在临床乳腺炎奶牛的乳腺组织及LPS诱导炎症的细胞中的表达量均显著下调(P<0.05),且在LPS刺激乳腺上皮细胞后6 h时lncRNA BCL2的下调幅度最大。因此,本试验以50 ng·μL-1 LPS诱导6 h的乳腺上皮细胞为模型开展后续试验。
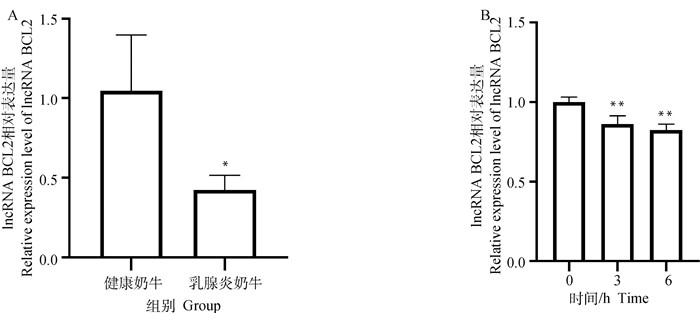
|
A. lncRNA BCL2在健康奶牛(对照组)和临床乳腺炎奶牛的乳腺组织中的表达量;B. lncRNA BCL2在50 ng·μL-1 LPS诱导乳腺上皮细胞0、3和6 h的表达量。*. P<0.05,**. P<0.01。下同 A. The expression of lncRNA BCL2 in mammary tissues of healthy dairy cows (control group) and clinical mastitis cows; B. The expression of lncRNA BCL2 in mammary epithelial cells induced with 50 ng·μL-1 LPS at 0, 3 and 6 h. *. P<0.05, **. P<0.01. The same as below 图 3 lncRNA BCL2在奶牛乳腺组织及乳腺上皮细胞中的表达量 Fig. 3 The expression of lncRNA BCL2 in cow mammary tissues and mammary epithelial cells |
本研究采用双酶切、琼脂糖凝胶电泳和测序进行lncRNA BCL2_pcDNA3.1超表达载体的鉴定,结果显示,lncRNA BCL2_pcDNA3.1超表达载体构建成功(图 4A、4B和4C),可用于下一步试验。此外,采用qPCR技术对lncRNA BCL2超表达效率进行检测,结果表明,与对照组相比,超表达lncRNA BCL2载体转染组的表达量极显著上升27.002 5倍(图 5),其超表达的细胞可用于后续相关基因表达量的检测。
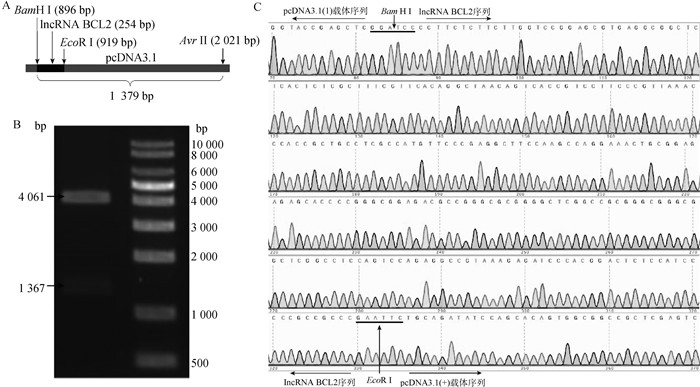
|
A. lncRNA BCL2 _pcDNA3.1超表达载体载体策略,插入lncRNA BCL2的酶切位点为BamH I/EcoR I;B. 超表达重组载体双酶切鉴定,双酶切鉴定使用的酶为BamH I/Avr II,酶切片段(1 367 bp)=插入lncRNA BCL2片段(242 bp)+载体片段(1 125 bp);C. 超表达重组载体测序峰图,lncRNA BCL2序列位于88~329 bp A. The schematic of lncRNA BCL2 _pcDNA3.1 overexpression vector construction. The enzyme digestion sites of lncRNA BCL2 were BamH I/EcoR I; B. The identification of overexpression recombinant vectors by double enzyme digestion. Double digestion assay was performed to identify the enzyme with BamH I / Avr II, the digested fragment(1 367 bp)=Insertion of lncRNA BCL2 fragments(242 bp)+ Vector fragments(1 125 bp); C. The sequencing peak plots of the overexpression recombinant vectors. The lncRNA BCL2 sequence located in 88~329 bp 图 4 lncRNA BCL2_pcDNA3.1超表达重组载体的构建与鉴定 Fig. 4 Construction and identification of lncRNA BCL2 overexpression recombinant vector |
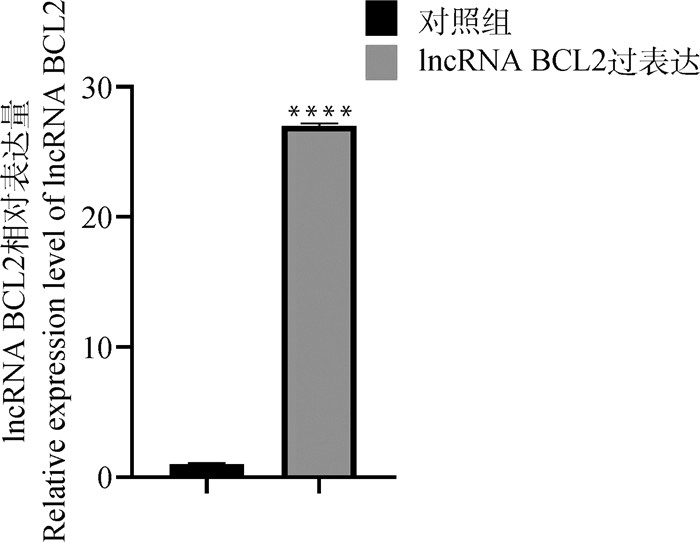
|
****. P<0.000 1 图 5 lncRNA BCL2超表达效率的检测结果 Fig. 5 Detection of lncRNA BCL2 overexpression efficiency |
为探究lncRNA BCL2对乳腺上皮细胞凋亡的作用,在乳腺上皮细胞超表达lncRNA BCL2的情况下,采用qPCR技术检测了凋亡相关基因的表达情况,结果表明,与对照组相比,lncRNA BCL2超表达组的细胞凋亡相关基因CASP3、CASP9和BAD的相对表达量均显著下调(P<0.05,图 6A、6B和6C);BAX基因的相对表达量变化差异不显著(P>0.05,图 6D),BCL2基因的表达量极显著上升(P<0.001,图 6E),且BCL2与BAX的比值大于2(比值越大抑制细胞凋亡能力越强)。上述结果表明,超表达的lncRNA BCL2可能通过抑制凋亡相关基因,从而抑制乳腺上皮细胞凋亡。
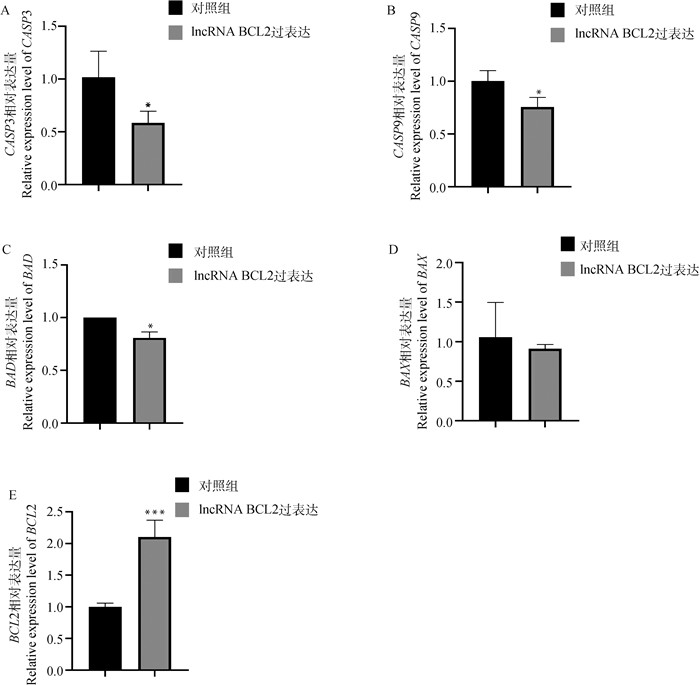
|
A. CASP3基因;B. CASP9基因;C. BAD基因;D. BAX基因;E. BCL2基因。***. P<0.001。下同 A. CASP3 gene; B. CASP9 gene; C. BAD gene; D. BAX gene; E. BCL2 gene. ***. P<0.001. The same as below 图 6 超表达lncRNA BCL2对细胞凋亡相关基因mRNA表达的影响 Fig. 6 Effect of lncRNA BCL2 overexpression on the mRNA expression of apoptosis-related genes |
为探究lncRNA BCL2在乳腺上皮细胞炎症中的作用,本研究检测了超表达lncRNA BCL2的乳腺上皮细胞情况中细胞炎症相关基因的表达量情况,结果表明,与对照组相比,lncRNA BCL2超表达组的炎症因子IL-6基因的表达量差异不显著(P>0.05,图 7A),而炎症信号通路关键基因NF-κB(p65/p50)和炎症因子IL-1β与IL-8的表达量均显著上调(P<0.05,图 7B~E),表明lncRNA BCL2可能通过上调NF-κB表达量进而上调IL-1β与IL-8基因的表达,进而加重细胞炎症反应。
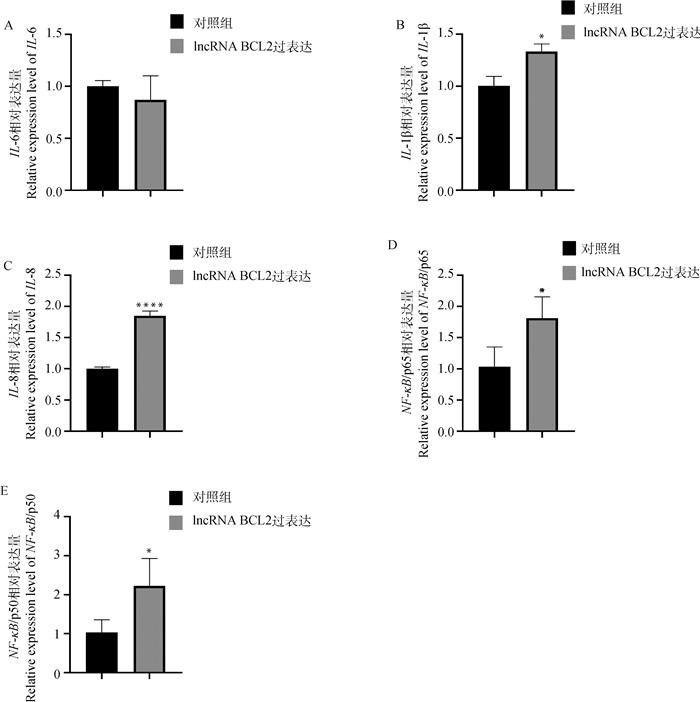
|
A. IL-6;B. IL-1β;C. IL-8;D. NF-κB/p65;E. NF-κB/p50 图 7 超表达lncRNA BCL2对乳腺上皮细胞炎症相关mRNA表达的影响 Fig. 7 Effect of lncRNA BCL2 overexpression on the expression of inflammation related mRNA in mammary epithelial cells |
lncRNA作为分子调控的关键因子,已被证实在多数炎症性疾病中发挥作用。Zhang等[21]研究表明,沉默lncRNA XIST导致海绵吸附的miR- 370-3p的表达上调,从而抑制JAK/STAT和NF-κB通路以减轻LPS诱导的人胚肺细胞炎症损伤。在模式动物的炎症性疾病中,研究人员发现lncRNA TUG1通过靶向miR-9-5p从而抑制NF-κB/p50的表达,最终缓解了LPS诱导的小胶质细胞(BV2)炎症[22]。LncRNA MALAT1可下调miR-199b以激活IKKbeta/NF-κB通路,促进LPS诱导的BV2中TNF-α和IL-1β分泌[23]。此外,lncRNA在奶牛乳腺上皮细胞炎症中的分子机制研究非常少。Li等[24]研究表明,lncRNA H19的过度表达能够激活NF-κB信号通路,并促进炎症因子(包括TNF-α、IL-6、CXCL2和CCL5)的表达,从而影响了LPS诱导MAC-T细胞的免疫反应;LncRNA H19还可以通过调控TGF-β1导致乳腺细胞纤维化[19];超表达的lncRNA XIST抑制了NF-κB炎症通路的激活,进而抑制了NLRP3炎症小体和促炎性细胞因子(TNFα、IL-1β和IL-6)的产生,缓解了细胞炎症反应[16];LRRC75A-AS1可以通过LRRC75A调节紧密连接蛋白的表达,从而影响炎症的发展[18];敲除lncRNA TUB会加剧MAC-T炎症反应[17]。但是,迄今尚未见lncRNA BCL2在奶牛乳房炎上的研究报道。本研究发现,在LPS诱导炎症的乳腺上皮细胞中,超表达lncRNA BCL2会刺激炎症信号通路激活,从而导致炎症因子释放,可能加剧奶牛乳房的炎症趋势。
为探索lncRNA BCL2的功能,本研究发现超表达lncRNA BCL2可上调NF-κB/p50和NF-κB/p65以及炎症因子(IL-1β和IL-8)的表达。由现有研究报道可知,NF-κB/p50和NF-κB/p65均是NF-κB家族的一员,均在促炎过程中发挥着重要作用[25-26]。NF-κB主要以p65(RelA)和p50亚基的异二聚体形式存在,其中p65具有转活域,而p50主要参与DNA结合[27]。在奶牛乳腺上皮细胞中NF-κB/p65的高表达有效地激活了炎症因子IL-8的表达,从而导致细胞炎症加剧[28]。在视网膜Müller细胞中,炎症因子IL-1β也可通过NF-κB/p50通路激活IL-6促进剂的活性[29]。该结果与本研究中NF-κB/p50、NF-κB/p65以及炎症因子(IL-1β和IL-8)在表达量变化趋势上是一致的。结合上述文献结果,可以看出乳腺上皮细胞lncRNA BCL2的表达量上调可促使NF-κB基因mRNA表达量上调,进而激活炎症因子基因IL-1β和IL-8的mRNA表达上调,加剧乳腺上皮细胞炎症趋势。
lncRNA在细胞凋亡中也发挥着重要的作用。例如在巨噬细胞和内皮细胞中lncRNA GAS5超表达可以显著促进细胞的凋亡[30]。低表达的lncRNA TUG1通过上调其靶点AURKA从而抑制上皮卵巢癌细胞的增殖和凋亡[31];在血管平滑肌肉细胞和人静脉内皮细胞中lncRNA TUG1通过海绵吸附miR-148b促进IGF2基因的蛋白质表达,从而调控细胞的凋亡[32]。在炎症刺激的奶牛乳腺上皮细胞中,高表达的lncRNA XIST可有效抑制细胞的凋亡率[16]。但是,迄今尚未见lncRNA BCL2参与乳腺上皮细胞凋亡的研究报道。本研究发现,牛乳腺上皮细胞内超表达lncRNA BCL2可促进BCL2基因的表达。现有文献表明,BCL2不仅是抑制细胞凋亡的关键蛋白,还在炎症性疾病中发挥重要的作用[33-34]。在SFTSV引起的炎症发生过程中,BCL2的激活导致细胞溶质释放并触发NLRP3刺激性激活,进一步加剧炎症反应[35]。Faustin等[36]研究表明,BCL2以浓度依赖性抑制NLRP1引起CASP1的激活,从而激活NLRP3导致炎症反应的加剧。因此推测,lncRNA BCL2不仅可以调控乳腺上皮细胞的凋亡,又可以调控乳腺上皮细胞的炎症反应。
此外,本研究还发现,若在奶牛乳腺上皮细胞内超表达lncRNA BCL2,则促进凋亡相关基因(CASP3、CASP9和BAD)表达量均显著下调。有研究表明,CASP3、CASP9、BAX和BAD是凋亡关键蛋白,与BCL2关系密不可分,这些蛋白均是促进凋亡的相关蛋白[37-39]。由生物信息学分析可知,在lncRNA BCL2海绵吸附的miRNAs中,bta-miR-129-5p、bta-miR-2436-3p和bta-miR-2888等可能直接靶向BCL2,进而参与调控乳腺上皮细胞炎症和凋亡的生物学过程。此外,还预测出多个miRNAs靶向IGF2而参与细胞凋亡的生物学过程;Bta-miR-2382-5p可能靶向NLRP3参与乳腺上皮细胞炎症反应过程(图 8)。本试验中的lncRNA BCL2和相关mRNA的表达量趋势与生物信息学分析结果一致,因此,推测lncRNA BCL2可能通过lncRNA BCL2-miRNA-BCL2(NLRP1、CASP1、NLRP3)网络调控奶牛乳腺上皮细胞的炎症以及凋亡(图 8),但仍需进一步试验验证,方可得到lncRNA BCL2确切的调控机制。
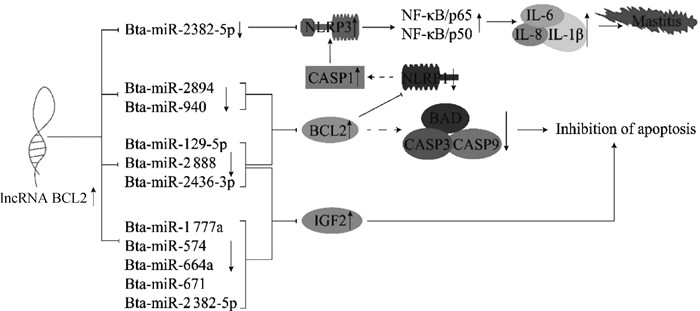
|
图 8 lncRNA BCL2对奶牛乳腺上皮细胞炎症及凋亡机制的预测分析 Fig. 8 Prediction of lncRNA BCL2 on inflammation and apoptosis of dairy cow mammary epithelial cells |
lncRNA BCL2在乳房炎奶牛的乳腺组织和LPS诱导的乳腺上皮细胞炎症中的表达量均显著下调;超表达lncRNA BCL2可显著下调促进细胞凋亡相关基因(BAD、CASP3和CASP9)的表达,并促进抑制细胞凋亡关键基因BCL2和炎症因子(IL-1β和IL-8)及其炎症信号通路(NF-κB/p65和NF-κB/p50)mRNA的表达。综合上述结果,可以得到,在奶牛乳腺炎过程中,lncRNA BCL2的表达量下调,减弱了其对IL-1β、IL-8和NF-κB表达上调的作用,从而缓解了乳腺炎症;同时,lncRNA BCL2表达量下降不仅减弱了其下调CASP3和CASP9基因表达的作用,还减弱了其上调BCL2基因表达的作用,从而可能促进了乳腺上皮细胞凋亡。该研究结果可为lncRNA BCL2在奶牛乳房炎中的深入研究奠定基础。
| [1] |
HUGHES K, WATSON C J. The mammary microenvironment in mastitis in humans, dairy ruminants, rabbits and rodents: a one health focus[J]. J Mammary Gland Biol Neoplasia, 2018, 23(1-2): 27-41. DOI:10.1007/s10911-018-9395-1 |
| [2] |
SAH K, KARKI P, SHRESTHA R D, et al. MILK Symposium review: improving control of mastitis in dairy animals in Nepal[J]. J Dairy Sci, 2020, 103(11): 9740-9747. DOI:10.3168/jds.2020-18314 |
| [3] |
AKHTAR M, GUO S, GUO Y F, et al. Upregulated-gene expression of pro-inflammatory cytokines (TNF-α, IL-1β and IL-6) via TLRs following NF-κB and MAPKs in bovine mastitis[J]. Acta Trop, 2020, 207: 105458. DOI:10.1016/j.actatropica.2020.105458 |
| [4] |
WU J, LI L, SUN Y, et al. Altered molecular expression of the TLR4/NF-κB signaling pathway in mammary tissue of Chinese Holstein cattle with mastitis[J]. PLoS One, 2015, 10(2): e0118458. DOI:10.1371/journal.pone.0118458 |
| [5] |
CHEN J, AO L F, YANG J. Long non-coding RNAs in diseases related to inflammation and immunity[J]. Ann Transl Med, 2019, 7(18): 494. DOI:10.21037/atm.2019.08.37 |
| [6] |
HADJICHARALAMBOUS M R, LINDSAY M A. Long non-coding RNAs and the innate immune response[J]. Noncoding RNA, 2019, 5(2): 34. |
| [7] |
SCACALOSSI K R, VAN SOLINGEN C, MOORE K J. Long non-coding RNAs regulating macrophage functions in homeostasis and disease[J]. Vascul Pharmacol, 2019, 114: 122-130. DOI:10.1016/j.vph.2018.02.011 |
| [8] |
LI Y, CHEN D, GAO X, et al. LncRNA NEAT1 regulates cell viability and invasion in esophageal squamous cell carcinoma through the miR-129/CTBP2 Axis[J]. Dis Markers, 2017, 2017: 5314649. |
| [9] |
LI Y H, ZHANG S S, ZHANG C X, et al. LncRNA MEG3 inhibits the inflammatory response of ankylosing spondylitis by targeting miR-146a[J]. Mol Cell Biochem, 2020, 466(1-2): 17-24. DOI:10.1007/s11010-019-03681-x |
| [10] |
NITSCHE A, STADLER P F. Evolutionary clues in lncRNAs[J]. Wiley Interdiscip Rev RNA, 2017, 8(1): e1376. DOI:10.1002/wrna.1376 |
| [11] |
PARDINI B, DRAGOMIR M P. SCIRT lncRNA blocks the shot of breast cancer cells self-renewal mechanism[J]. Cancer Res, 2021, 81(3): 535-536. DOI:10.1158/0008-5472.CAN-20-3903 |
| [12] |
CHEN Y, JING H Y, CHEN M Y, et al. Transcriptional profiling of exosomes derived from Staphylococcus aureus-infected bovine mammary epithelial cell line MAC-T by RNA-seq analysis[J]. Oxid Med Cell Longev, 2021, 2021: 8460355. |
| [13] |
ÖZDEMIR S, ALTUN S. Genome-wide analysis of mRNAs and lncRNAs in Mycoplasma bovis infected and non-infected bovine mammary gland tissues[J]. Mol Cell Probes, 2020, 50: 101512. DOI:10.1016/j.mcp.2020.101512 |
| [14] |
YANG B, JIAO B L, GE W, et al. Transcriptome sequencing to detect the potential role of long non-coding RNAs in bovine mammary gland during the dry and lactation period[J]. BMC Genomics, 2018, 19(1): 605. DOI:10.1186/s12864-018-4974-5 |
| [15] |
杨兵. 奶牛乳腺差异表达长链非编码RNA的筛选、鉴定及其功能研究[D]. 杨凌: 西北农林科技大学, 2019. YANG B. Screening, identification and functional studies of long non-coding RNAs differentially expressed in mammary gland of dairy cows[D]. Yangling: Northwest A & F University, 2019. (in Chinese) |
| [16] |
MA M R, PEI Y F, WANG X X, et al. LncRNA XIST mediates bovine mammary epithelial cell inflammatory response via NF-κB/NLRP3 inflammasome pathway[J]. Cell Prolif, 2019, 52(1): e12525. DOI:10.1111/cpr.12525 |
| [17] |
WANG H, WANG X X, LI X R, et al. A novel long non-coding RNA regulates the immune response in MAC-T cells and contributes to bovine mastitis[J]. FEBS J, 2019, 286(9): 1780-1795. DOI:10.1111/febs.14783 |
| [18] |
WANG X X, WANG H, ZHANG R Q, et al. LRRC75A antisense lncRNA1 knockout attenuates inflammatory responses of bovine mammary epithelial cells[J]. Int J Biol Sci, 2020, 16(2): 251-263. DOI:10.7150/ijbs.38214 |
| [19] |
YANG W, LI X Z, QI S P, et al. lncRNA H19 is involved in TGF-β1-induced epithelial to mesenchymal transition in bovine epithelial cells through PI3K/AKT signaling pathway[J]. PeerJ, 2017, 5: e3950. DOI:10.7717/peerj.3950 |
| [20] |
HUYNH H T, ROBITAILLE G, TURNER J D. Establishment of bovine mammary epithelial cells (MAC-T): an in vitro model for bovine lactation[J]. Exp Cell Res, 1991, 197(2): 191-199. DOI:10.1016/0014-4827(91)90422-Q |
| [21] |
ZHANG Y N, ZHU Y Y, GAO G S, et al. Knockdown XIST alleviates LPS-induced WI-38 cell apoptosis and inflammation injury via targeting miR-370-3p/TLR4 in acute pneumonia[J]. Cell Biochem Funct, 2019, 37(5): 348-358. DOI:10.1002/cbf.3392 |
| [22] |
YUE P J, JING L J, ZHAO X Y, et al. Down-regulation of taurine-up-regulated gene 1 attenuates inflammation by sponging miR-9-5p via targeting NF-κB1/p50 in multiple sclerosis[J]. Life Sci, 2019, 233: 116731. DOI:10.1016/j.lfs.2019.116731 |
| [23] |
ZHU W, MEN X L. Negative feedback of NF-κB signaling by long noncoding RNA MALAT1 controls lipopolysaccharide-induced inflammation injury in human lung fibroblasts WI-38[J]. J Cell Biochem, 2020, 121(2): 1945-1952. DOI:10.1002/jcb.29429 |
| [24] |
LI X Z, WANG H, ZHANG Y F, et al. Overexpression of lncRNA H19 changes basic characteristics and affects immune response of bovine mammary epithelial cells[J]. PeerJ, 2019, 7: e6715. DOI:10.7717/peerj.6715 |
| [25] |
CHAN C, LI L W, MCCALl C E, et al. Endotoxin tolerance disrupts chromatin remodeling and NF-κB transactivation at the IL-1β promoter[J]. J Immunol, 2005, 175(1): 461-468. DOI:10.4049/jimmunol.175.1.461 |
| [26] |
GIRIDHARAN S, SRINIVASAN M. Mechanisms of NF-κB p65 and strategies for therapeutic manipulation[J]. J Inflamm Res, 2018, 11: 407-419. DOI:10.2147/JIR.S140188 |
| [27] |
ROLOVA T, DHUNGANA H, KORHONEN P, et al. Deletion of nuclear factor kappa B p50 subunit decreases inflammatory response and mildly protects neurons from transient forebrain ischemia-induced damage[J]. Aging Dis, 2016, 7(4): 450-465. DOI:10.14336/AD.2015.1123 |
| [28] |
LIU S Z, SHI X M, BAUER I, et al. Lingual antimicrobial peptide and IL-8 expression are oppositely regulated by the antagonistic effects of NF-κB p65 and C/EBPβ in mammary epithelial cells[J]. Mol Immunol, 2011, 48(6-7): 895-908. DOI:10.1016/j.molimm.2010.12.018 |
| [29] |
LIU X F, YE F, XIONG H B, et al. IL-1β induces IL-6 production in retinal Müller cells predominantly through the activation of p38 MAPK/NF-κB signaling pathway[J]. Exp Cell Res, 2015, 331(1): 223-231. DOI:10.1016/j.yexcr.2014.08.040 |
| [30] |
CHEN L, YANG W J, GUO Y J, et al. Exosomal lncRNA GAS5 regulates the apoptosis of macrophages and vascular endothelial cells in atherosclerosis[J]. PLoS One, 2017, 12(9): e0185406. DOI:10.1371/journal.pone.0185406 |
| [31] |
LI T H, CHEN Y, ZHANG J J, et al. LncRNA TUG1 promotes cells proliferation and inhibits cells apoptosis through regulating AURKA in epithelial ovarian cancer cells[J]. Medicine (Baltimore), 2018, 97(36): e12131. DOI:10.1097/MD.0000000000012131 |
| [32] |
WU X G, ZHENG X H, CHENG J, et al. LncRNA TUG1 regulates proliferation and apoptosis by regulating miR-148b/IGF2 axis in ox-LDL-stimulated VSMC and HUVEC[J]. Life Sci, 2020, 243: 117287. DOI:10.1016/j.lfs.2020.117287 |
| [33] |
ROBERTS A W. Therapeutic development and current uses of BCL-2 inhibition[J]. Hematology Am Soc Hematol Educ Program, 2020, 2020(1): 1-9. DOI:10.1182/hematology.2020000154 |
| [34] |
SIDDIQUI W A, AHAD A, AHSAN H. The mystery of BCL2 family: Bcl-2 proteins and apoptosis: an update[J]. Arch Toxicol, 2015, 89(3): 289-317. DOI:10.1007/s00204-014-1448-7 |
| [35] |
LI S F, LI H, ZHANG Y L, et al. SFTSV infection induces BAK/BAX-dependent mitochondrial DNA release to trigger NLRP3 inflammasome activation[J]. Cell Rep, 2020, 30(13): 4370-4385.e7. DOI:10.1016/j.celrep.2020.02.105 |
| [36] |
FAUSTIN B, CHEN Y, ZHAI D Y, et al. Mechanism of Bcl-2 and Bcl-XL inhibition of NLRP1 inflammasome: loop domain-dependent suppression of ATP binding and oligomerization[J]. Proc Natl Acad Sci U S A, 2009, 106(10): 3935-3940. DOI:10.1073/pnas.0809414106 |
| [37] |
LIU X M, ZHAO H B, JIN Q, et al. Resveratrol induces apoptosis and inhibits adipogenesis by stimulating the SIRT1-AMPKα-FOXO1 signalling pathway in bovine intramuscular adipocytes[J]. Mol Cell Biochem, 2018, 439(1-2): 213-223. DOI:10.1007/s11010-017-3149-z |
| [38] |
HU Q L, CUI X J, TAO L, et al. Staphylococcus aureus induces apoptosis in primary bovine mammary epithelial cells through Fas-FADD death receptor-linked caspase-8 signaling[J]. DNA Cell Biol, 2014, 33(6): 388-397. DOI:10.1089/dna.2013.2195 |
| [39] |
FU Y R, JIN Y C, ZHAO Y, et al. Zearalenone induces apoptosis in bovine mammary epithelial cells by activating endoplasmic reticulum stress[J]. J Dairy Sci, 2019, 102(11): 10543-10553. DOI:10.3168/jds.2018-16216 |
(编辑 范子娟)



