2. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
气候环境在物种的长期遗传进化过程中扮演重要的角色,它是物种遗传进化的重要驱动力[1]。气候环境是影响物种空间分布的决定性因素,气候的变化可能会改变物种的分布格局和生物的遗传多样性[2],如对拟南芥[3]、松树[4]、绵羊[5]、人类[6]等的研究发现,气候环境的差异会使各个物种的群体之间甚至个体之间产生表型差异和遗传变异。探索气候环境介导的遗传标记是进化生物学的重要内容之一,它有可能揭示当地物种适应性进化的遗传机理,为应对将来的气候变化提供理论基础。因此,在基因组水平上研究动植物对气候环境的适应性,在保护物种的多样性和维持生物圈的平衡等方面具有重要意义。
近年来,随着分子生物学、遗传学、生物信息学等重要学科的不断发展,对家畜基因组学的研究有了许多重要成果,例如在山羊[7]、绵羊[8]、猪[9]、牛[10]、马[11]等家畜的起源进化[12]、重要性状基因挖掘、环境适应性等方面的研究都有了突破性的进展。对家畜的环境适应性研究,起初主要聚焦于群体遗传学的方法,根据环境变量将物种的群体或个体进行简单区分,然后基于群体遗传分化水平(Fst)进行比较分析,来筛选出与环境变量相关的遗传标记或基因。这样的方法仅能定性地对环境变量进行简单区分,无法直接确定遗传标记与环境变量间的有效关联,容易产生假阳性的结果[13-14]。新近发展起来的景观基因组学方法主要依托于遗传环境关联分析(genetic-environment association, GEA)来解析环境适应性的遗传机制[15]。景观基因组学可以直接整合遗传变异数据和环境变量数据,通过logistic回归模型、一般线性模型等方法直接分析遗传变异与环境变量的关联性,不仅可以挖掘出与环境变量相关联的特异性位点,还能找出关键的环境驱动因素[16]。伴随着景观基因组学的广泛应用,研究者们开始综合利用群体遗传学和景观基因组学的方法对动植物的环境适应性进行深入研究[17-18],有效地结合群体遗传学对群体结构、遗传分化历史研究的优势和景观基因组学能更准确挖掘环境适应性相关联位点的优势,在许多物种的研究中发现了一大批与环境适应性显著关联的功能位点,对山羊的环境适应性研究有重要的指导和借鉴意义。
根据考古学和遗传学的研究发现,现代家养山羊的驯化大约起源于11 000年前的新月沃土地区[7],而Bezoar (Capra aegagrus)被认为是家养山羊主要的祖先[19-20]。山羊在被驯化后,跟随着人类的迁移,迅速在亚洲地区扩散,进而迁移到欧洲、非洲、美洲及大洋洲[21]。经过万年的迁移进化,如今家山羊已遍布于世界各地,并逐渐分化形成了适应于当地气候环境的独特品种。从北欧高纬度的极寒地区到非洲低纬度的炎热地区,从北非干旱的沙漠地区到东南亚的湿润地区,从低海拔的平原地区到低温低氧的高原地区,在这些极端环境条件下都存在独特地方山羊品种的生存繁衍[22],可见山羊对极端的气候环境条件具有极强的适应能力[23]。另一方面,山羊作为重要的家畜之一,其产绒、产奶、产肉等重要经济性状极大地满足了人类对物质生活资料的需求,在农业、经济、文化等领域都成为了人类社会的重要部分。研究报道,环境的温度和湿度过高会造成家畜的生产力下降、死亡率上升等问题,给畜牧产业造成经济损失[24]。因此,对山羊气候适应性的深入研究,也可能为一些重要的经济性状、抗病性状等优良性状的保护和利用提供理论支撑[25],促进山羊产业的不断发展。本文重点立足于基因组学,从不同技术方法的角度切入,阐述了当前在家畜气候适应性研究中所应用的主要研究方法。针对山羊的环境适应性研究,以5个方面的环境适应性研究为分类单元,对近年来的最新研究进行了综述,初步概括了山羊对不同气候条件适应性的关键候选基因。
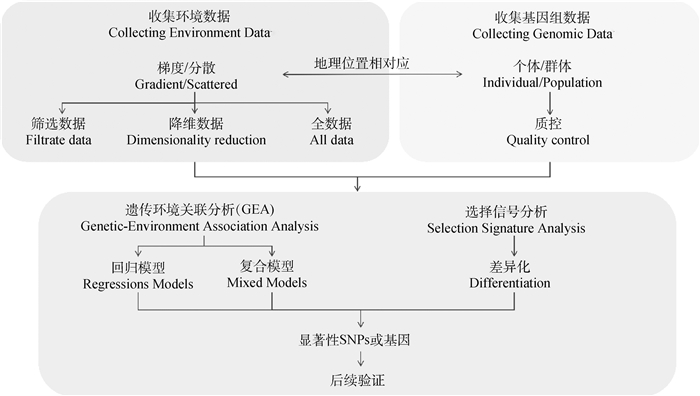
1 环境适应性研究方法随着全球气候监测技术和数据管理等相关部门的发展,目前世界上已出现了许多成熟的气候数据库,如:国家生态科学数据中心(National Ecosystem Science Data Center, NESDC)[26]、中国科学院资源环境科学与数据中心(Resource and Environment Science and Data Center)[27]、WorldClim[28]等数据库,这些数据库整理记录了详细的气候数据,这些数据时间跨度大、覆盖范围广、气候变量多样、分辨率高,为深入探索动植物气候环境长期适应性的遗传机制提供了必要的前提。另一方面,分子生物技术和全基因组测序技术也在持续进步发展,新一代的测序技术具有高通量、高效率、低成本、高分辨率等优点[29],许多动植物都已完成了基因组序列的测定[30],为揭示动植物环境适应性的分子遗传机制奠定了坚实的基础。在此背景下,遗传环境关联分析开始逐渐发展起来,用以识别与特定环境条件密切相关的遗传变异[31]。景观基因组学是一个新兴的研究领域,GEA是景观基因组学的核心,它整合了景观遗传学和群体基因组学的工具[32],以识别形成当今适应性遗传变异的环境因素和驱动本地适应的遗传变异[33] (图 1)。表 1展现了环境适应性研究常用的分析方法和软件。
|
图 1 基于景观基因组学进行环境适应性研究的策略 Fig. 1 Strategies for environmental adaptability research based on landscape genomics |
|
|
表 1 环境适应性研究常用的分析方法和软件 Table 1 Analysis methods and softwares commonly used in environmental adaptability research |
在对各类物种的环境适应性研究中,常以环境因素作为区分标准,将所研究样本分组进行比较分析,例如高海拔与低海拔的群体或干旱地区与湿润地区的群体。在统计分类样本的过程中,不同类型的环境变量会作为分类变量引入参数或非参数检验。以环境因素为标准确定样本分组后,许多研究常通过选择信号分析方法检测目标群体中受到相应环境因素驱动而产生的遗传变异位点[34]。常用的选择信号检测方法包括基于核苷酸多态性的方法(如θπ)、基于群体分化的方法(如Fst、XP-EHH)、基于基因频率的方法(如Tajima’s D、ZHp)和基于连锁不平衡的方法(如EHH、iHS)等[35-36]。Liu等[11]根据海拔数据将我国的多个地方马群体分为高、低两组,并结合Fst、θπ和ZHp 3种选择信号方法进行分析,鉴定到EPAS1基因受到强烈的选择,对藏马的高原适应具有重要作用。Yang等[37]利用多个选择信号检测方法,对根据海拔和降水量数据分组的绵羊群体进行分析,筛选到多个涉及高原适应性和干旱适应性的候选基因。重要的是,许多选择信号分析方法可以通过方向性的区分来挖掘与环境因素的某一方向相适应的基因,这可以为候选基因受驱动的方向提供直接的证据。如Gheyas等[38]对鸡的环境适应性研究中,探索了所用环境变量的高、低两个方向的变异位点,鉴定出适应于环境变量两个方向的候选基因。
1.2 logistic回归模型Logistic回归可以检验环境因素是否影响等位基因或单位点基因型。需要注意的是,由于logistic回归只能考虑两种状态(等位基因或位点基因型的存在或缺失),没有明确的方法来处理杂合子个体位点中出现的3种或更多的基因型状态。因此,该类分析必须整理数据集的格式(即0, 1, 2格式),以描述每个等位基因或位点基因型的存在或缺失,适用于从不同的生存环境或沿环境梯度取样的个体[39]。
空间分析方法(spatial analysis method,SAM)[40-41]是采用logistic回归模型研究与环境相关的局部适应性特征,以及测量环境和分子数据集的空间自相关性的常用方法[42]。但需要注意的是,该分析方法忽略了群体结构的影响,这可能在一定程度上对结果的准确性造成干扰。因此,在SAM基础上,新开发的Samβada软件将中性遗传结构引入其中,可进一步量化基因型的空间自相关水平,有效降低了假阳性结果的概率[42]。同时,相较于其他方法(如潜在因素混合模型,latent factor mixed models,LFMM),该方法的运算速度更快,尤其在应用其内置的拆分组合模块后更为明显,这对处理大型数据文件非常具有优势[43]。另一方面,该方法可直接在结果中反馈显著性位点关联的环境变量,这为寻找家畜适应性过程中的环境驱动因素提供了直观有力的证据。如Lv等[16]对绵羊气候适应性的研究中,通过SAM方法确定了最佳模型中月降水量、日照百分比和月降水量≥0.1 mm天数这3个变量对绵羊地方环境适应的驱动作用。
1.3 一般线性模型一般线性模型框架可以扩展到具有多元响应变量的模型,以解释适应性状的多基因结构[44]。其中比较常用的方法是典型相关分析(canonical correlation analysis,CCA)[45],该方法可以找到两组变量,即多个位点(解释变量)和多个环境因素(响应变量)间最大相关的线性组合。如Mosca等[46]利用CCA分析方法对意大利山区4种针叶林的种群结构及其与地理环境间的关联进行了研究,发现了多个与冬季降水量、季节性最低温等环境变量存在显著关联的SNP,解释了针叶林对低温等环境的适应性。另一种常用的方法,冗余分析(redundancy analysis,RDA)可以构建和检测不同复杂性的模型,包括基于中性遗传结构或空间效应的结果,也被称为部分RDA(pRDA)[47]。Lasky等[48]就利用pRDA对拟南芥多变量气候和多变量遗传变异之间的相关性进行了评估,并通过控制空间效应确定了环境适应性的候选SNP。Mdladla等[49]同样利用RDA对南非本地山羊群体的地方适应性进行研究,发现了一些适应于当地炎热干旱环境的候选SNP。
1.4 潜在因素混合模型潜在因素混合模型(LFMM)是Frichot等[50]提出的一种基于群体遗传学、生态建模和统计分析方法构建的新算法,以筛选基因组的局部适应性特征。将中性遗传结构(即可能的祖先群体数量)作为一个潜在因子引入其中的分析方法。其优点是可以同时估计环境因素和群体遗传结构对等位基因频率的影响,通过评估种群历史和地理隔离模式的随机效应,来有效降低关联分析结果中的假阳性概率。分析中所用到的种群结构潜在因子(K值)一般通过对目标群体的基因组数据进行主成分分析和Admixture分析来确定[51]。此外,该方法所用的环境数据需尽可能通过线性组合的方式来总结呈现,如采用主成分分析或类似方法将第一主成分作为新的环境变量引入分析[52-53]。
2 山羊环境适应性研究进展 2.1 高海拔适应性研究位于我国西南部的青藏高原地区,是全世界平均海拔最高、面积范围最大的高原,平均海拔在4 000 m以上。高海拔的特殊地理环境导致其氧分压仅为海平面的60%左右[54],由此产生的缺氧对人和动物的新陈代谢造成了严重的影响[55],容易引发多种高原性疾病[56]。另一方面,高原地区昼夜温差大、紫外线强等特殊气候条件也对高原生物的生存繁殖提出严峻的挑战。因此,高海拔适应性的遗传机制解析是近年来备受关注的课题,这对了解高原特殊环境下的适应和缺氧性相关疾病的诊断治疗具有重要的意义(表 2)[57-58]。
|
|
表 2 主要山羊群体的气候环境适应性研究 Table 2 Study on climatic and environmental adaptability of major goat populations |
高原地区的家畜包括藏牛、藏羊、藏马、藏犬等,它们为高原地区的居民提供了重要的物质生活材料和交通运输工具,经过长期的适应性进化,它们在表型特征上与低海拔地区的家畜相比,表现出鲜明的特点,比如皮毛长而浓密,毛色较深等[59]。Wang等[60]在对我国8个山羊群体的研究中发现了与高海拔适应性相关的候选基因,其中包括具有调控氧运输和缺氧反应的基因(CDK2、SOCS2、NOXA1、ENPEP),以及具有调控毛发长度(fibroblast growth factor 5, FGF5)和表皮颜色(KIT ligand, KITLG)功能的基因,并认为这些基因与山羊适应高原的低氧寒冷环境密切相关。由此可见,藏山羊毛发的独特表型特征是其适应高原特殊环境的一个重要表现,主要体现在对低温和强紫外线的适应能力。除此之外,高原地区低氧分压的环境特点对山羊生存繁殖的影响更为强烈。在Guo等[61]的研究中,利用6个地方山羊品种(包括2个高海拔地区的藏山羊品种)的全基因组重测序数据,通过Fst和ZHp的选择信号检测方法,筛选出了与缺氧适应(endothelial PAS domain protein 1, EPAS1)相关的候选基因。Song等[62]利用6个山羊群体的外显子测序数据,对高、低海拔地区的两组山羊群体进行了选择信号分析,筛选到了339个与高海拔适应性相关的基因,通过富集分析发现这些基因主要参与心血管系统的相关过程,并发现EPAS1的错义突变对西藏本土山羊的高原适应有重要意义,在先前分别对人类[63]、马[11]、犬[64]等的研究中均鉴定到该基因;并且在对西藏地区的马、绵羊、山羊、牛、猪和犬的基因组协同进化分析中进一步证明了EPAS1基因在西藏高海拔适应性的主效作用。此外,PTPRJ、C10orf67等基因也被鉴定与山羊高海拔适应性相关[65]。
2.2 热适应性研究高温环境可以直接或间接影响家畜的生存生长。气温升高可以导致多数地区的牧草质量和数量的变化,同时伴随温度升高而来的是家畜对虫害疾病的易感性增加[66],这些因素都可能会间接引起家畜生理或行为的变化,成为家畜适应高温环境的驱动力之一。另一方面,家畜正常的体温是由新陈代谢产生的热量与机体热量损失之间的平衡来协调控制的[67],当外界环境温度过高,家畜体内的内源性热量无法及时消散时,热应激便会发生[68]。热应激状态会直接影响家畜的饲料摄入量、个体生长发育及产奶量等,甚至会引发疾病[69],从而给农业生产带来巨大的损失(表 2)。因此,山羊热适应性的研究对保护农业生产有重要的意义,近年来也逐渐成为家畜环境适应性的研究热点之一。
山羊能够通过表皮颜色[70]和生理变化,如较高的呼吸速率、出汗率和呼吸量来应对高温环境[71],这是在温度因素的直接驱动下形成的适应性表现。Benjelloun等[72]利用摩洛哥地区3个地方山羊品种的全基因组测序(WGS)数据探索山羊的遗传结构、连锁不平衡和选择标记,使用基于群体分化的XP-CLR方法鉴定到了一些与毛色、体型、新陈代谢和呼吸系统相关的基因,并认为这些受选择的功能性基因有利于山羊对当地热、干旱环境的适应。山羊除了通过毛色和生理变化来实现对热环境的适应以外,个体的生长发育和形态变化也对热环境的适应有所贡献。Kim等[73]利用非洲地区地方山羊、绵羊群体和5个外来山羊群体的芯片数据,通过Fst和iHS两种选择信号检测方法对热带干旱地区和寒冷潮湿地区的群体进行分析,识别出了几个受到积极选择的候选区域,并鉴定到一些与热适应性和表型特征相关的基因,包括热耐受性(FGF2、GNAI3、PLCB1),体型和发育(BMP2、BMP4、GJA3、GJB2),能量和代谢(MYH、TRHDE、ALDH1A3),神经和自身免疫反应(GRIA1、IL2、IL7、IL21、IL1R1)方面的基因。在Brito等[74]对山羊的研究中也同样鉴定出了热耐受性相关的GNAL3基因。Bertolini等[75]利用来自全球多个地区的山羊芯片数据,通过Samβada的遗传环境关联分析方法和Fst选择信号方法,发现HOX基因家族及其附近区域的Pre-B-Cell Leukemia Homeobox 1 (PBX1)基因通过调节山羊身体和组织的形态发育实现对热环境的适应。此外,还发现GPR37L1、INS等基因通过参与胰岛素分泌和糖代谢等过程影响山羊的热应激。
在热带地区,除了高温环境对山羊的生理和表型会产生直接的影响外,高温环境下生存的各类病虫害威胁同样不容小觑,其中威胁最大的就是锥虫病[76]。锥虫病在非洲、亚洲和南美洲的低纬度热带地区家畜中非常普遍,而山羊更是深受影响,每年在非洲地区约有60%的山羊会受到锥虫病的侵扰[77]。面对锥虫病的威胁,Serranito等[51]利用44个非洲山羊品种的芯片数据,结合采采蝇(锥虫病传播的主要媒介)传播锥虫病的强度和当地的温度、湿度数据,通过LFMM潜在混合模型方法,鉴定出一组与锥虫病耐受性相关的候选基因(DIS3L2、COPS7B、PD5A、UBE2K、UBR1、AKAP6和MKL2)。因此,对山羊热适应性的基因挖掘和机制解析不仅应关注高温环境对山羊生理状态的直接影响因素,也应深入挖掘由高温引起的一系列连锁反应对山羊生存生长产生的间接影响因素,这也是山羊热适应能力的重要部分。
2.3 冷适应性研究温度变化是影响家畜生存和繁殖的关键气候因素之一,过冷的环境往往会直接影响新生羔羊的生存生长,容易导致羔羊的出生体重低、死亡率高等情况[78]。家畜皮毛的密度、厚度和长度可直接影响御寒的效果,更加密集和更长的毛发可有效降低体内热量的消散,从而适应寒冷的环境[79]。Wang等[80]利用192只内蒙古绒山羊的芯片数据,对内蒙古绒山羊的绒纤维长度、纤维直径和羊绒产量进行了全基因组关联研究,发现了4个与羊绒特征显著相关的SNPs位点,并注释到FGF12、SEMA3D、EVPL和SX5基因参与毛囊发育过程。这些参与调控羊绒表型特性的基因也可能是山羊对寒冷环境适应的重要功能基因。Burren等[81]利用10个瑞士地方山羊品种的芯片数据,通过分析发现了25个重要的基因组区域,其中包含与表皮颜色(MITF、KIT、ASIP)、生长(IGF2、IGF2R、HRAS、FGFR3)等表型相关的基因,并推测这些基因是为了适应瑞士高山寒冷的气候环境而受到选择。另一方面,哺乳动物脂肪组织也被认为是维持自身热量的关键因素,在低温环境下它们可以通过高比例非饱和脂肪酸的脂肪组织来保障体内的热量水平[82]。尤其是机体内的棕色脂肪组织(Bat) [83],在冷环境的刺激下可以增强脂质新陈代谢释放出用以维持能量平衡的热量[84-85]。有研究者通过对山羊样本进行RNA-Seq分析,鉴定出CTNNB、PFDN5和EIF3M为从棕色脂肪组织(BAT)到白色脂肪组织(WAT)过渡的候选基因[86]。因此,脂肪的转化利用能力也许是山羊应对寒冷环境的一个关键(表 2)。通过结合山羊脂肪含量相关数据进行全基因组关联分析(GWAS)也是探索山羊寒冷适应性相关候选基因的一个有效途径,但目前针对山羊的相关研究仍存在不足。
寒冷的气候环境作为驱动因素在山羊的长期驯化过程中,不仅对山羊的表型特征产生影响,也使山羊在新陈代谢等方面表现出对寒冷环境的适应性。2017年,Li等[87]使用中国内蒙古和辽宁地区地方山羊品种的重测序数据,通过基于遗传分化(Fst)和多态性水平的交叉分析方法,鉴定出一些有利于山羊适应寒冷干燥环境的候选基因,包括内分泌调节相关的基因(ADCY4、ROCK1、ACNA1C、OXTR等)以及脂代谢和皮肤发育等相关的基因。
2.4 干旱适应性研究在干旱的环境条件下,水源的短缺往往会导致植被匮乏等一系列问题[88],此时能量和营养代谢对该环境下的家畜就变得至关重要。此外,干旱的环境地区通常也伴随着高强的太阳照射、强紫外线辐射以及炎热的特点,这些因素也会对家畜的生殖生理过程产生直接的影响。Mwacharo等[5]利用来自埃及沙漠地区5个绵羊群体的芯片数据,鉴定到了172个与干旱适应性相关的候选基因,主要涉及新陈代谢、应激反应,神经内分泌系统,免疫反应的激活,生殖器官功能,体型的大小和形态、表皮的色素沉积和角化等生物过程(表 2)。而在对山羊的研究中,Mdladla等[49]利用来自亚热带湿润、干旱和沙漠地区239只南非地方山羊个体的芯片数据,通过遗传环境关联分析(RDA、SAM、LFMM)方法确定了478个重要SNPs位点上的基因,并发现受选择的基因主要参与内分泌调节、细胞生长发育、新陈代谢和免疫力等相关通路,推测是南非山羊群体适应当地炎热、干旱环境的结果。由此可见,山羊对于干旱环境的适应性是一个涉及多方面的复杂过程。而其中各个生理过程之间的联系和最关键的候选基因还有待进一步研究挖掘。
2.5 综合气候适应性研究气候环境作为驱动物种进化的关键力量之一,除了在某些特殊环境地区拥有某一代表性的极端气候变量(如沙漠地区的极低降水量、热带地区的极高温度等)以外,大多数地区气候环境都是由多种气候变量协同产生的结果。一些气候数据库已提供多个时间点的全球气候数据,如日平均温度、日最高温度、降水量、蒸汽压等多种气候变量,通过结合这些多变量的环境数据和基因组数据进行分析,不仅可以了解家畜对当地环境的适应性遗传特征,还可以为家畜空间分布的历史驯化过程研究提供重要证据。利用主成分分析(PCA)等方法对多变量的环境数据进行降维处理,再与遗传数据进行关联分析,即可了解物种内对多变量综合气候适应性的遗传变异。如Cao等[25]通过对地方绵羊群体的气候适应性和野羊的基因渗入研究发现,绵羊中与多变量综合气候适应性相关的基因主要参与肿瘤与细胞的发育和增殖、神经系统发育;能量代谢和内分泌系统的调节及免疫与炎症反应等过程,其中还包括来源于野羊基因渗入的基因。而在对山羊的研究中,Cortellari等[89]通过遗传环境关联分析方法(LFMM、Samβada)对意大利33个地方山羊种群的基因组数据进行分析,发现了62个与温度和湿度相关的基因,并进一步注释到它们主要参与山羊的生长、昼夜节律调节、个体发育和炎症反应过程。此外,他们还对关键SNP基因型频率的未来变化进行了预测,这在对山羊气候适应性的研究中是个新思路。因此,通过利用丰富多样的气候环境数据对山羊的综合气候适应性进行探究,是揭示山羊应对气候变化的分子遗传机制的有效途径(表 2)。
3 展望景观基因组学是将基因组学、遗传学、生态学等多个领域结合起来用以探索生物基因组与气候环境之间关联的一门新近发展起来的学科。测序技术的成熟,使得在多个体或多群体内筛选单核苷酸多态性的成本大幅降低,准确性显著提高。与此同时,大批的气候环境数据集也在持续更新完善,并越来越多地允许公开访问,这些因素都有利于推动景观基因组学的快速进步发展。然而, 目前已有的景观基因组学研究多集中在植物和昆虫等领域,针对山羊乃至针对家畜领域的相关研究仍处于相对落后的水平,这为今后对山羊遗传资源的评价、保护和利用工作带来了全新的机遇和挑战。
山羊经过漫长的遗传驯化过程,如今已成功地适应了沙漠、高原、山区、热带地区等不同的生存环境,而其他家畜物种很少能在如此多样的环境中健康成长。除此之外,山羊的产肉、产奶、产绒等优良经济性状为人类社会的发展提供了丰富的物质生活材料。这也使得山羊成为全世界范围内的重要家畜之一,也使山羊的遗传资源挖掘工作显得尤为重要。在对山羊环境适应性的研究中,随着环境数据的丰富和测序技术的进步,逐步从起初简单定性的单一环境变量的适应性分析过渡到如今多解释变量和多响应变量的精准关联分析。这使得对家畜气候适应性相关候选基因的挖掘变得更加简单、高效,同时能够直接找出与遗传变异位点相关联的具体环境因素,这为解析家畜的气候适应性分子机制提供了重要的直接证据。
然而在现有的研究中,面对筛选到的众多气候适应性相关的候选基因,在其后续的功能验证过程中往往存在困难甚至难以实现。推测其可能的原因包括以下两点:1)山羊的气候适应能力多是一个通过多方面生物过程调控的复杂性状,仅针对某单一方面的功能基因进行验证难以体现对相应气候的适应能力;2)气候变量的条件难以在验证试验中实现准确的复制。因此,对气候适应性候选基因后续的功能验证研究仍有待进行技术和方法的创新。此外,针对某一气候适应性相关的多个候选基因间的内在关联也尚不明确,在研究中仍有待进一步深入分析挖掘。只有详细地了解山羊环境适应性的分子遗传机制,才能充分地发挥山羊遗传资源的价值,为山羊的育种和繁殖工作提供坚实的理论依据。深入开展山羊气候适应性研究工作,不仅可以揭示自然驱动下山羊适应性进化的历史进程,还能为许多环境因素导致的疾病提供重要基础资料,对保护和利用山羊的遗传资源,保护山羊品种的多样性,提高山羊产业的产能和效率具有重大意义。
| [1] |
ERWIN D H. Climate as a driver of evolutionary change[J]. Curr Biol, 2009, 19(14): R575-R583. DOI:10.1016/j.cub.2009.05.047 |
| [2] |
GARCIA R A, CABEZA M, RAHBEK C, et al. Multiple dimensions of climate change and their implications for biodiversity[J]. Science, 2014, 344(6183): 1247579. DOI:10.1126/science.1247579 |
| [3] |
TERÉS J, BUSOMS S, MARTÍN L P, et al. Soil carbonate drives local adaptation in Arabidopsis thaliana[J]. Plant Cell Environ, 2019, 42(8): 2384-2398. DOI:10.1111/pce.13567 |
| [4] |
TYRMI J S, VUOSKU J, ACOSTA J J, et al. Genomics of Clinal local adaptation in Pinus sylvestris under continuous environmental and spatial genetic setting[J]. G3 (Bethesda), 2020, 10(8): 2683-2696. DOI:10.1534/g3.120.401285 |
| [5] |
MWACHARO J M, KIM E S, ELBELTAGY A R, et al. Genomic footprints of dryland stress adaptation in Egyptian fat-tail sheep and their divergence from East African and western Asia cohorts[J]. Sci Rep, 2017, 7(1): 17647. DOI:10.1038/s41598-017-17775-3 |
| [6] |
WITT K E, HUERTA-SÁNCHEZ E. Convergent evolution in human and domesticate adaptation to high-altitude environments[J]. Philos Trans R Soc Lond B Biol Sci, 2019, 374(1777): 20180235. DOI:10.1098/rstb.2018.0235 |
| [7] |
DALY K G, DELSER P M, MULLIN V E, et al. Ancient goat genomes reveal mosaic domestication in the Fertile Crescent[J]. Science, 2018, 361(6397): 85-88. DOI:10.1126/science.aas9411 |
| [8] |
LI X, YANG J, SHEN M, et al. Whole-genome resequencing of wild and domestic sheep identifies genes associated with morphological and agronomic traits[J]. Nat Commun, 2020, 11(1): 2815. DOI:10.1038/s41467-020-16485-1 |
| [9] |
FRANTZ L A F, HAILE J, LIN A T, et al. Ancient pigs reveal a near-complete genomic turnover following their introduction to Europe[J]. Proc Natl Acad Sci U S A, 2019, 116(35): 17231-17238. DOI:10.1073/pnas.1901169116 |
| [10] |
QIU Q, WANG L Z, WANG K, et al. Yak whole-genome resequencing reveals domestication signatures and prehistoric population expansions[J]. Nat Commun, 2015, 6: 10283. DOI:10.1038/ncomms10283 |
| [11] |
LIU X X, ZHANG Y L, LI Y F, et al. EPAS1 gain-of-function mutation contributes to high-altitude adaptation in Tibetan horses[J]. Mol Biol Evol, 2019, 36(11): 2591-2603. DOI:10.1093/molbev/msz158 |
| [12] |
MCHUGO G P, DOVER M J, MACHUGH D E. Unlocking the origins and biology of domestic animals using ancient DNA and paleogenomics[J]. BMC Biol, 2019, 17(1): 98. DOI:10.1186/s12915-019-0724-7 |
| [13] |
EVENO E, COLLADA C, GUEVARA M A, et al. "Contrasting patterns of selection at Pinus pinaster Ait. Drought stress candidate genes as revealed by genetic differentiation analyses"[J]. Mol Biol Evol, 2008, 25(2): 417-437. DOI:10.1093/molbev/msm272 |
| [14] |
HOBAN S, KELLEY J L, LOTTERHOS K E, et al. Finding the genomic basis of local adaptation: pitfalls, practical solutions, and future directions[J]. Am Nat, 2016, 188(4): 379-397. DOI:10.1086/688018 |
| [15] |
RELLSTAB C, GUGERLI F, ECKERT A J, et al. A practical guide to environmental association analysis in landscape genomics[J]. Mol Ecol, 2015, 24(17): 4348-4370. DOI:10.1111/mec.13322 |
| [16] |
LV F H, AGHA S, KANTANEN J, et al. Adaptations to climate-mediated selective pressures in sheep[J]. Mol Biol Evol, 2014, 31(12): 3324-3343. DOI:10.1093/molbev/msu264 |
| [17] |
SORK V L. Genomic studies of local adaptation in natural plant populations[J]. J Hered, 2017, 109(1): 3-15. DOI:10.1093/jhered/esx091 |
| [18] |
FITZPATRICK M C, KELLER S R. Ecological genomics meets community-level modelling of biodiversity: mapping the genomic landscape of current and future environmental adaptation[J]. Ecol Lett, 2015, 18(1): 1-16. DOI:10.1111/ele.12376 |
| [19] |
ZEDER M A. Domestication and early agriculture in the Mediterranean Basin: origins, diffusion, and impact[J]. Proc Natl Acad Sci U S A, 2008, 105(33): 11597-11604. DOI:10.1073/pnas.0801317105 |
| [20] |
PIDANCIER N, JORDAN S, LUIKART G, et al. Evolutionary history of the genus Capra (Mammalia, Artiodactyla): discordance between mitochondrial DNA and Y-chromosome phylogenies[J]. Mol Phylogenet Evol, 2006, 40(3): 739-749. DOI:10.1016/j.ympev.2006.04.002 |
| [21] |
AMILLS M, CAPOTE J, TOSSER-KLOPP G. Goat domestication and breeding: a jigsaw of historical, biological and molecular data with missing pieces[J]. Anim Genet, 2017, 48(6): 631-644. DOI:10.1111/age.12598 |
| [22] |
GILBERT M, NICOLAS G, CINARDI G, et al. Global distribution data for cattle, buffaloes, horses, sheep, goats, pigs, chickens and ducks in 2010[J]. Sci Data, 2018, 5: 180227. DOI:10.1038/sdata.2018.227 |
| [23] |
NAIR M R R, SEJIAN V, SILPA M V, et al. Goat as the ideal climate-resilient animal model in tropical environment: revisiting advantages over other livestock species[J]. Int J Biometeorol, 2021, 65(12): 2229-2240. DOI:10.1007/s00484-021-02179-w |
| [24] |
ZHANG M H, DUNSHEA F R, WARNER R D, et al. Impacts of heat stress on meat quality and strategies for amelioration: a review[J]. Int J Biometeorol, 2020, 64(9): 1613-1628. DOI:10.1007/s00484-020-01929-6 |
| [25] |
CAO Y H, XU S S, SHEN M, et al. Historical introgression from wild relatives enhanced climatic adaptation and resistance to pneumonia in sheep[J]. Mol Biol Evol, 2021, 38(3): 838-855. DOI:10.1093/molbev/msaa236 |
| [26] |
国家生态科学数据中心[Z]. 2019. National Ecosystem Science Data Center[Z]. 2019. (in Chinese) |
| [27] |
资源环境科学与数据中心[Z]. Resource and Environment Science and Data Center[Z]. (in Chinese) |
| [28] |
WorldClim[Z].
|
| [29] |
VAN DIJK E L, AUGER H, JASZCZYSZYN Y, et al. Ten years of next-generation sequencing technology[J]. Trends Genet, 2014, 30(9): 418-426. DOI:10.1016/j.tig.2014.07.001 |
| [30] |
WORLEY K C. A golden goat genome[J]. Nat Genet, 2017, 49(4): 485-486. DOI:10.1038/ng.3824 |
| [31] |
MANEL S, HOLDEREGGER R. Ten years of landscape genetics[J]. Trends Ecol Evol, 2013, 28(10): 614-621. DOI:10.1016/j.tree.2013.05.012 |
| [32] |
MANEL S, ANDRELLO M, HENRY K, et al. Predicting genotype environmental range from genome-environment associations[J]. Mol Ecol, 2018, 27(13): 2823-2833. DOI:10.1111/mec.14723 |
| [33] |
LI X, GUO T T, MU Q, et al. Genomic and environmental determinants and their interplay underlying phenotypic plasticity[J]. Proc Natl Acad Sci U S A, 2018, 115(26): 6679-6684. DOI:10.1073/pnas.1718326115 |
| [34] |
MANEL S, PERRIER C, PRATLONG M, et al. Genomic resources and their influence on the detection of the signal of positive selection in genome scans[J]. Mol Ecol, 2016, 25(1): 170-184. DOI:10.1111/mec.13468 |
| [35] |
FOLL M, GAGGIOTTI O. A genome-scan method to identify selected loci appropriate for both dominant and codominant markers: a Bayesian perspective[J]. Genetics, 2008, 180(2): 977-993. DOI:10.1534/genetics.108.092221 |
| [36] |
BONHOMME M, CHEVALET C, SERVIN B, et al. Detecting selection in population trees: the Lewontin and Krakauer test extended[J]. Genetics, 2010, 186(1): 241-262. DOI:10.1534/genetics.110.117275 |
| [37] |
YANG J, LI W R, LV F H, et al. Whole-genome sequencing of native sheep provides insights into rapid adaptations to extreme environments[J]. Mol Biol Evol, 2016, 33(10): 2576-2592. DOI:10.1093/molbev/msw129 |
| [38] |
GHEYAS A A, VALLEJO-TRUJILLO A, KEBEDE A, et al. Integrated environmental and genomic analysis reveals the drivers of local adaptation in African indigenous chickens[J]. Mol Biol Evol, 2021, 38(10): 4268-4285. DOI:10.1093/molbev/msab156 |
| [39] |
DE MITA S, THUILLET A C, GAY L, et al. Detecting selection along environmental gradients: analysis of eight methods and their effectiveness for outbreeding and selfing populations[J]. Mol Ecol, 2013, 22(5): 1383-1399. DOI:10.1111/mec.12182 |
| [40] |
JOOST S, BONIN A, BRUFORD M W, et al. A spatial analysis method (SAM) to detect candidate loci for selection: towards a landscape genomics approach to adaptation[J]. Mol Ecol, 2007, 16(18): 3955-3969. DOI:10.1111/j.1365-294X.2007.03442.x |
| [41] |
JOOST S, KALBERMATTEN M, BONIN A. Spatial analysis method (SAM): a software tool combining molecular and environmental data to identify candidate loci for selection[J]. Mol Ecol Resour, 2008, 8(5): 957-960. DOI:10.1111/j.1755-0998.2008.02162.x |
| [42] |
DURUZ S, SEVANE N, SELMONI O, et al. Rapid identification and interpretation of gene-environment associations using the new R.SamBada landscape genomics pipeline[J]. Mol Ecol Resour, 2019, 19(5): 1355-1365. DOI:10.1111/1755-0998.13044 |
| [43] |
STUCKI S, OROZCO-TERWENGEL P, FORESTER B R, et al. High performance computation of landscape genomic models including local indicators of spatial association[J]. Mol Ecol Resour, 2017, 17(5): 1072-1089. DOI:10.1111/1755-0998.12629 |
| [44] |
OKSANEN J, BLANCHET F G, FRIENDLY M, et al. vegan: community Ecology Package. Ordination methods, diversity analysis and other functions for community and vegetation ecologists. Version 2.5-7[Z]. 2020.
|
| [45] |
BLANCO-PASTOR J L, BARRE P, KEEP T, et al. Canonical correlations reveal adaptive loci and phenotypic responses to climate in perennial ryegrass[J]. Mol Ecol Resour, 2021, 21(3): 849-870. DOI:10.1111/1755-0998.13289 |
| [46] |
MOSCA E, ECKERT A J, DI PIERRO E A, et al. The geographical and environmental determinants of genetic diversity for four alpine conifers of the European Alps[J]. Mol Ecol, 2012, 21(22): 5530-5545. DOI:10.1111/mec.12043 |
| [47] |
CAPBLANCQ T, LUU K, BLUM M, et al. Evaluation of redundancy analysis to identify signatures of local adaptation[J]. Mol Ecol Resour, 2018, 18(6): 1223-1233. DOI:10.1111/1755-0998.12906 |
| [48] |
LASKY J R, DES MARAIS D L, LOWRY D B, et al. Natural variation in abiotic stress responsive gene expression and local adaptation to climate in Arabidopsis thaliana[J]. Mol Biol Evol, 2014, 31(9): 2283-2296. DOI:10.1093/molbev/msu170 |
| [49] |
MDLADLA K, DZOMBA E F, MUCHADEYI F C. Landscape genomics and pathway analysis to understand genetic adaptation of South African indigenous goat populations[J]. Heredity (Edinb), 2018, 120(4): 369-378. DOI:10.1038/s41437-017-0044-z |
| [50] |
FRICHOT E, SCHOVILLE S D, BOUCHARD G, et al. Testing for associations between loci and environmental gradients using latent factor mixed models[J]. Mol Biol Evol, 2013, 30(7): 1687-1699. DOI:10.1093/molbev/mst063 |
| [51] |
SERRANITO B, TAURISSON-MOURET D, HARKAT S, et al. Search for selection signatures related to trypanosomosis tolerance in African goats[J]. Front Genet, 2021, 12: 715732. DOI:10.3389/fgene.2021.715732 |
| [52] |
GAIN C, FRANÇOIS O. LEA 3:factor models in population genetics and ecological genomics with R[J]. Mol Ecol Resour, 2021, 21(8): 2738-2748. DOI:10.1111/1755-0998.13366 |
| [53] |
CAYE K, JUMENTIER B, LEPEULE J, et al. LFMM 2:fast and accurate inference of gene-environment associations in genome-wide studies[J]. Mol Biol Evol, 2019, 36(4): 852-860. DOI:10.1093/molbev/msz008 |
| [54] |
BEALL C M. Two routes to functional adaptation: Tibetan and Andean high-altitude natives[J]. Proc Natl Acad Sci U S A, 2007, 104 Suppl 1(Suppl 1): 8655-8660. |
| [55] |
WEST J B. Early history of high-altitude physiology[J]. Ann N Y Acad Sci, 2016, 1365(1): 33-42. DOI:10.1111/nyas.12719 |
| [56] |
LUKS A M, SWENSON E R, BARTSCH P. Acute high-altitude sickness[J]. Eur Respir Rev, 2017, 26(143): 160096. DOI:10.1183/16000617.0096-2016 |
| [57] |
SIMONSON T S, YANG Y Z, HUFF C D, et al. Genetic evidence for high-altitude adaptation in Tibet[J]. Science, 2010, 329(5987): 72-75. DOI:10.1126/science.1189406 |
| [58] |
ZHANG W P, FAN Z X, HAN E J, et al. Hypoxia adaptations in the grey wolf (Canis lupus chanco) from Qinghai-Tibet Plateau[J]. PLoS Genet, 2014, 10(7): e1004466. DOI:10.1371/journal.pgen.1004466 |
| [59] |
张天留, 高雪, 徐凌洋, 等. 高原家养动物环境适应性的研究进展[J]. 畜牧兽医学报, 2020, 51(7): 1475-1487. ZHANG T L, GAO X, XU L Y, et al. Research progress on environment adaptation of plateau domestic animals[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(7): 1475-1487. (in Chinese) |
| [60] |
WANG X L, LIU J, ZHOU G X, et al. Whole-genome sequencing of eight goat populations for the detection of selection signatures underlying production and adaptive traits[J]. Sci Rep, 2016, 6: 38932. DOI:10.1038/srep38932 |
| [61] |
GUO J Z, TAO H X, LI P F, et al. Whole-genome sequencing reveals selection signatures associated with important traits in six goat breeds[J]. Sci Rep, 2018, 8(1): 10405. DOI:10.1038/s41598-018-28719-w |
| [62] |
SONG S, YAO N, YANG M, et al. Exome sequencing reveals genetic differentiation due to high-altitude adaptation in the Tibetan cashmere goat (Capra hircus)[J]. BMC Genomics, 2016, 17: 122. DOI:10.1186/s12864-016-2449-0 |
| [63] |
LORENZO F R, HUFF C, MYLLYMÄKI M, et al. A genetic mechanism for Tibetan high-altitude adaptation[J]. Nat Genet, 2014, 46(9): 951-956. DOI:10.1038/ng.3067 |
| [64] |
GOU X, WANG Z, LI N, et al. Whole-genome sequencing of six dog breeds from continuous altitudes reveals adaptation to high-altitude hypoxia[J]. Genome Res, 2014, 24(8): 1308-1315. DOI:10.1101/gr.171876.113 |
| [65] |
WU D D, YANG C P, WANG M S, et al. Convergent genomic signatures of high-altitude adaptation among domestic mammals[J]. Natl Sci Rev, 2020, 7(6): 952-963. DOI:10.1093/nsr/nwz213 |
| [66] |
BERMAN A. Invited review: are adaptations present to support dairy cattle productivity in warm climates?[J]. J Dairy Sci, 2011, 94(5): 2147-2158. DOI:10.3168/jds.2010-3962 |
| [67] |
BERNABUCCI U, BIFFANI S, BUGGIOTTI L, et al. The effects of heat stress in Italian Holstein dairy cattle[J]. J Dairy Sci, 2014, 97(1): 471-486. DOI:10.3168/jds.2013-6611 |
| [68] |
KOCH F, THOM U, ALBRECHT E, et al. Heat stress directly impairs gut integrity and recruits distinct immune cell populations into the bovine intestine[J]. Proc Natl Acad Sci U S A, 2019, 116(21): 10333-10338. DOI:10.1073/pnas.1820130116 |
| [69] |
SILVA P S, HOOPER H B, MANICA E, et al. Heat stress affects the expression of key genes in the placenta, placental characteristics, and efficiency of Saanen goats and the survival and growth of their kids[J]. J Dairy Sci, 2021, 104(4): 4970-4979. DOI:10.3168/jds.2020-18301 |
| [70] |
GUO J Z, ZHONG J, LI L, et al. Comparative genome analyses reveal the unique genetic composition and selection signals underlying the phenotypic characteristics of three Chinese domestic goat breeds[J]. Genet Sel Evol, 2019, 51: 70. DOI:10.1186/s12711-019-0512-4 |
| [71] |
GONZALEZ-RIVAS P A, CHAUHAN S S, HA M, et al. Effects of heat stress on animal physiology, metabolism, and meat quality: a review[J]. Meat Sci, 2020, 162: 108025. DOI:10.1016/j.meatsci.2019.108025 |
| [72] |
BENJELLOUN B, ALBERTO F J, STREETER I, et al. Characterizing neutral genomic diversity and selection signatures in indigenous populations of Moroccan goats (Capra hircus) using WGS data[J]. Front Genet, 2015, 6: 107. |
| [73] |
KIM E S, ELBELTAGY A R, ABOUL-NAGA A M, et al. Multiple genomic signatures of selection in goats and sheep indigenous to a hot arid environment[J]. Heredity (Edinb), 2016, 116(3): 255-264. DOI:10.1038/hdy.2015.94 |
| [74] |
BRITO L F, KIJAS J W, VENTURA R V, et al. Genetic diversity and signatures of selection in various goat breeds revealed by genome-wide SNP markers[J]. BMC Genomics, 2017, 18(1): 229. DOI:10.1186/s12864-017-3610-0 |
| [75] |
BERTOLINI F, SERVIN B, TALENTI A, et al. Signatures of selection and environmental adaptation across the goat genome post-domestication[J]. Genet Sel Evol, 2018, 50(1): 57. DOI:10.1186/s12711-018-0421-y |
| [76] |
GEERTS S, OSAER S, GOOSSENS B, et al. Trypanotolerance in small ruminants of sub-Saharan Africa[J]. Trends Parasitol, 2009, 25(3): 132-138. DOI:10.1016/j.pt.2008.12.004 |
| [77] |
GUTIERREZ C, CORBERA J A, MORALES M, et al. Trypanosomosis in goats: current status[J]. Ann N Y Acad Sci, 2006, 1081: 300-310. DOI:10.1196/annals.1373.040 |
| [78] |
LUO N J, WANG J, HU Y, et al. Cold and heat climatic variations reduce indigenous goat birth weight and enhance pre-weaning mortality in subtropical monsoon region of China[J]. Trop Anim Health Prod, 2020, 52(3): 1385-1394. DOI:10.1007/s11250-019-02142-3 |
| [79] |
DO PRADO PAIM T, BORGES B O, DE MELLO T L P, et al. Thermographic evaluation of climatic conditions on lambs from different genetic groups[J]. Int J Biometeorol, 2013, 57(1): 59-66. DOI:10.1007/s00484-012-0533-y |
| [80] |
WANG F H, ZHANG L, GONG G, et al. Genome-wide association study of fleece traits in Inner Mongolia Cashmere goats[J]. Anim Genet, 2021, 52(3): 375-379. DOI:10.1111/age.13053 |
| [81] |
BURREN A, NEUDITSCHKO M, SIGNER-HASLER H, et al. Genetic diversity analyses reveal first insights into breed-specific selection signatures within Swiss goat breeds[J]. Anim Genet, 2016, 47(6): 727-739. DOI:10.1111/age.12476 |
| [82] |
GUERRERO A I, ROGERS T L. From low to high latitudes: changes in fatty acid desaturation in mammalian fat tissue suggest a thermoregulatory role[J]. BMC Evol Biol, 2019, 19(1): 155. DOI:10.1186/s12862-019-1473-5 |
| [83] |
VIRTANEN K A, LIDELL M E, ORAVA J, et al. Functional brown adipose tissue in healthy adults[J]. N Engl J Med, 2009, 360(15): 1518-1525. DOI:10.1056/NEJMoa0808949 |
| [84] |
VAN MARKEN LICHTENBELT W D, VANHOMMERIG J W, SMULDERS N M, et al. Cold-activated brown adipose tissue in healthy men[J]. N Engl J Med, 2009, 360(15): 1500-1508. DOI:10.1056/NEJMoa0808718 |
| [85] |
FULLER-JACKSON J P, HENRY B A. Adipose and skeletal muscle thermogenesis: studies from large animals[J]. J Endocrinol, 2018, 237(3): R99-R115. DOI:10.1530/JOE-18-0090 |
| [86] |
WANG L J, CHEN X Y, SONG T Z, et al. Using RNA-Seq to identify reference genes of the transition from brown to white adipose tissue in goats[J]. Animals, 2020, 10(9): 1626. DOI:10.3390/ani10091626 |
| [87] |
LI X K, SU R, WAN W T, et al. Identification of selection signals by large-scale whole-genome resequencing of cashmere goats[J]. Sci Rep, 2017, 7(1): 15142. DOI:10.1038/s41598-017-15516-0 |
| [88] |
SELEIMAN M F, AL-SUHAIBANI N, ALI N, et al. Drought stress impacts on plants and different approaches to alleviate its adverse effects[J]. Plants (Basel), 2021, 10(2): 259. |
| [89] |
CORTELLARI M, BARBATO M, TALENTI A, et al. The climatic and genetic heritage of Italian goat breeds with genomic SNP data[J]. Sci Rep, 2021, 11(1): 10986. DOI:10.1038/s41598-021-89900-2 |
(编辑 郭云雁)



