2. 西北农林科技大学 农业农村部动物生物技术重点实验室,杨凌 712100
2. Key Laboratory of Animal Biotechnology of the Ministry of Agriculture and Rural Affairs, Northwest A&F University, Yangling 712100, China
千百万年来地球围绕地轴自转,形成了周期性的明/暗循环,赋予了地球表面光照时间、温度和能量等外部环境的昼夜节律性变化。这些节律性变化使地球上的生物在进化压力的长期驱动下产生了与之相适应的内在计时机制,从而导致机体大多数生理功能具有了以近似24 h为周期的节律性振荡,被称为昼夜节律[1]。生物体用以预测时间变化和调节昼夜节律的内源性计时机制则被称为生物钟[2]。作为一种重要的调控系统,生物钟与机体的生长发育等生理功能的正常运转密切相关。当生物钟受到环境、基因突变等因素干扰时,机体的生理过程也会发生紊乱,从而导致疾病的发生[3-4]。哺乳动物的生物钟系统由位于下丘脑视交叉上核(suprachiasmatic nucleus,SCN)的中枢生物钟和分布于机体几乎所有细胞、组织与器官的外周生物钟组成[5]。SCN通过接收和整合来自视网膜下丘脑束传来的光信号来调控机体中枢生物钟的运转,并通过神经和体液途径将信号信息传递给大脑其他部位以及外周组织,驱动外周生物钟的节律性振荡,从而使机体维持生理稳态[6]。
哺乳动物生物钟调控机制虽然具有一定的物种差异性,但在分子水平上其主要依赖于BMAL1、CLOCK、PERs(PER1、PER2、PER3)、CRYs(CRY1、CRY2)和RORs(RORα、RORβ)等核心生物钟基因的转录调控,几乎所有的细胞都表达这些生物钟基因,并具有产生昼夜节律性振荡的潜能,不同物种的核心生物钟调控机制在进化上具有高度保守性[7]。维甲酸相关孤儿受体α(retinoic acid-related orphan receptors alpha,RORα)作为生物钟系统的重要组成部分,在维持生物钟的正常运转过程中发挥重要作用。在生物钟的转录反馈环路中,RORα通过与BMAL1基因启动子上特定的DNA反应元件即维甲酸相关孤儿受体反应元件(RORE:由一段(G/A)GGTCA核心序列和一段6 bp富含A/T的前导序列构成)结合来激活基因转录[8-9]。同时,REV-ERBs作为转录抑制因子,通过与RORα竞争性结合RORE元件来抑制BMAL1的转录[10-11]。此外,RORα自身的转录也受到一些上游因子的调节,如DBP/NFIL3会与RORα基因启动子上的D-box元件结合来调控其转录[12-13]。反过来,DBP和NFIL3的转录也分别受到BMAL1/CLOCK和RORα的调控[14]。总之,RORα与BMAL1和CLOCK等核心生物钟基因一起形成了多个转录翻译反馈环路,共同维持着生物钟系统的正常运转。除此之外,有研究发现RORα作为脂质和脂蛋白代谢、脂肪生成和血管炎症的重要调节因子,在机体脂代谢和炎症反应过程中发挥重要调控作用[15-16]。研究表明,RORα还在细胞凋亡以及肿瘤发生过程中扮演着重要角色[17-18]。
然而,上述文献证据大多是基于模式动物小鼠来研究的,RORα蛋白的功能是否具有种属特异性尚不清楚。山羊作为人类最早驯化的家畜之一,在人类农业文明和经济发展中发挥重要作用,一直是畜牧兽医领域关注的热点。有研究报道,绒山羊的产绒性能受到昼夜节律生物钟的影响[19]。然而,山羊生物钟系统的转录调控机制仍未被完全解析。作为生物钟系统的重要组成部分,RORα基因的全长编码区(coding sequence,CDS)还未被克隆和深入研究。基于此,本研究旨在克隆山羊生物钟基因RORα的编码区CDS片段并构建其真核表达载体,进一步对山羊RORα基因进行生物信息学分析和功能的初步研究,为后续探究RORα蛋白生物学功能及其在山羊生物钟系统的转录调控机制奠定基础。
1 材料与方法 1.1 试验材料、主要试剂及仪器山羊肝组织取自1只12月龄健康的雄性西农萨能奶山羊; HEK293T细胞购于中国科学院细胞库; TRIzol、PrimeSTAR® Max DNA Polymerase、Quick CutTM EcoR I、Quick CutTM Kpn I、Quick CutTM Xho I购自日本TaKaRa公司; 反转录试剂盒购自AG生物工程有限公司; THUNDERBIRD SYBR®qPCR Mix购自日本ToYoBo公司; ClonExpress®II One Step Cloning Kit购自南京诺唯赞生物科技股份有限公司; DH5α感受态细胞、胶回收试剂盒、无内毒素质粒小提中量试剂盒、基因组DNA提取试剂盒购自北京天根生化科技有限公司; 胎牛血清、Opti-MEM培养基均购自美国Gibco公司; DMEM高糖培养基购自Hyclone公司; TurboFect Transfection Reagent购自美国Thermo Fisher公司; 双荧光素酶报告实验试剂盒购于Promega公司; BCA蛋白浓度检测试剂盒购自江苏凯基生物技术股份有限公司; 脱脂奶粉购于美国BD公司; HRP标记的鼠抗β-actin抗体购自中国三箭公司; 兔抗HA抗体购自Abcam公司; HRP共轭山羊抗兔抗体购自中国中杉金桥公司; ECL HRP底物试剂盒购自北京迪宁生物科技有限公司。实验室具有PCR扩增仪(美国Bio-Rad公司)、荧光定量系统(美国Bio-Rad公司)、电泳仪(美国Bio-Rad公司)、凝胶成像分析系统(英国Syngene公司); 核酸蛋白测定仪(美国Thermo Fisher公司)和CO2培养箱(美国Thermo Fisher公司)等仪器设备。
1.2 山羊肝组织cDNA的获取及引物设计称取20 mg山羊肝组织,使用TRIzol法提取总RNA,反转录得到cDNA。根据NCBI数据库中山羊RORα基因(NM_001285652.1)的CDS区[20],使用Primer Premier 6.0软件设计PCR引物。同时在引物上下游的5′端添加与pcDNA3.1-Puro-N-3HA载体EcoR I酶切位点处互补配对的同源序列,设计带有同源臂的引物。另外,根据NCBI数据库中山羊BMAL1基因启动子(BMAL1-Promoter)序列(NC_030822.1)与NR1D1启动子(NR1D1-Promoter)序列(NC_030826.1)(两个基因的启动子序列均含有RORE元件),使用Primer Premier 6.0软件分别设计扩增山羊BMAL1-Promoter区域和NR1D1-Promoter区域的引物。前者在引物上下游5′端添加与pGL4.10载体Kpn I酶切位点互补配对的同源序列,后者在引物上下游5′端添加与pGL4.10载体Xho I酶切位点互补配对的同源序列,设计带有同源臂的引物。引物序列信息见表 1,所有引物均交由西安擎科生物科技有限责任公司进行合成。
|
|
表 1 引物序列信息 Table 1 Primer sequence information |
1.3.1 山羊RORα基因CDS区的克隆 以山羊的cDNA为模板扩增带有同源臂的RORα基因的CDS区片段。PCR反应程序如下:在反应离心管中加入cDNA模板4 μL(50 ng · μL-1),上、下游引物各1 μL(10 μmol·L-1),2×PrimeSTAR Max Premix 25 μL,ddH2O 19 μL,建立50 μL的PCR反应体系; PCR程序为:98 ℃预变性5 min; 98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸105 s,循环35次; 72 ℃总延伸5 min。PCR产物进行琼脂糖凝胶电泳鉴定,对符合预期的条带按照DNA纯化回收试剂盒说明书操作进行纯化回收。
1.3.2 pcDNA3.1-3HA-gRORα载体的构建及鉴定 使用限制性内切酶EcoR I将pcDNA3.1-Puro-N-3HA载体进行酶切线性化处理,并通过琼脂糖凝胶电泳试验检测酶切产物,将符合预期的条带使用胶回收试剂盒回收纯化。根据ClonExpress快速重组克隆试剂盒说明书,将胶回收得到的带有同源臂的目的基因片段与酶切线性化的真核表达载体pcDNA3.1-Puro-N-3HA进行重组连接,重组反应体系:线性化载体1.5 μL,片段3.5 μL,Exnase Ⅱ 2 μL,5×CE Ⅱ Buffer 4 μL,加水至20 μL。将反应混合液在37 ℃条件下反应15 min以完成重组反应,得到重组产物。将重组产物按照转化反应操作说明转至DH5α大肠杆菌感受态细胞中,之后涂布于含氨苄霉素30 μg · mL-1的固体培养基中。12 h后挑取单克隆菌落,于37 ℃摇床培养12 h,使用无内毒素小提中量试剂盒提取质粒并测定浓度。随后使用限制性内切酶EcoR I对重组质粒进行单酶切鉴定。同时以重组质粒为模板,使用RORα基因的引物进行PCR鉴定。将酶切与PCR鉴定结果符合预期的重组质粒送至西安擎科生物有限公司进行测序鉴定。返回序列与NCBI数据库中已知序列进行比对,将比对正确的质粒样本命名为pcDNA3.1-3HA-gRORα真核表达载体。
1.3.3 重组质粒转染HEK293T细胞 复苏HEK293T细胞并进行传代,以每孔4×105个细胞接种至1个6孔板,以每皿8×105个细胞接种至6个60 mm细胞培养皿。待HEK293T细胞密度达到60%左右时进行转染。将pcDNA3.1-3HA-gRORα作为试验组,pcDNA3.1-Puro-N-3HA作为对照组。将两组质粒按照TurboFect转染试剂说明书的操作步骤分别转染细胞。6孔板中,试验组质粒和对照组质粒分别转染3孔,转染后24 h收样,用于检测山羊RORα基因在mRNA水平的表达。6个60 mm皿中,试验组质粒和对照组质粒分别转染3皿,转染48 h后收样,用于检测山羊RORα基因在蛋白水平的表达。
1.3.4 实时荧光定量PCR检测山羊RORα基因在mRNA水平的表达 转染24 h后,使用TRIzol试剂提取细胞样品总RNA,反转录合成cDNA,并稀释至5 ng·μL-1,作为模板备用。用Primer Premier 6.0设计人与山羊通用的RORα引物序列以及人的GAPDH引物序列(表 1)。使用ToYoBo的THUNDERBIRD SYBR®qPCR Mix并按说明书操作进行实时荧光定量PCR(qPCR)反应。反应体系:cDNA 5 μL,qPCR Mix 10 μL,上、下游引物各1 μL,去离子水3 μL。qPCR反应程序:95 ℃ 3 min; 95 ℃ 10 s; 60 ℃ 30 s,共40个循环; 95 ℃ 15 s,60 ℃ 15 s,95.5 ℃ 5 s。使用荧光定量系统进行反应,以人的GAPDH基因作为内参基因,并采用2-△△Ct的相对定量法对样本进行定量分析。
1.3.5 蛋白质免疫印迹检测山羊RORα基因在蛋白水平的表达 转染48 h后,提取细胞蛋白样品进行蛋白质免疫印迹(Western blotting,WB)试验。具体操作:将细胞收集至离心管中,按照RIPA裂解液操作说明处理细胞得到蛋白样品。使用BCA蛋白含量检测试剂盒测定总蛋白浓度后加入Loading Buffer,100 ℃水浴10 min后进行SDS-PAGE凝胶电泳。电泳结束后将蛋白转印至PVDF膜上; 使用10%的脱脂奶粉封闭2 h。封闭完成后,TBST溶液中洗膜4次,每次5 min。将PVDF膜置于兔抗HA抗体工作液(1∶2 000稀释)中,室温摇床孵育2 h; TBST洗膜4次,每次5 min。之后将PVDF膜置于HRP标记的山羊抗兔二抗工作液(1∶5 000稀释)中,室温摇床孵育2 h; TBST洗膜4次,每次5 min。稍晾干后,将膜置于3 mL ECL发光液中作用2 min,于暗室中曝光并保存目标蛋白结果图。将曝光后的膜取出,TBST洗膜2次,每次5 min。加入适量抗体洗脱液,室温孵育15 min,弃去洗脱液,TBST洗膜3次,每次5 min。随后再次进行封闭,操作与上述相同。封闭完成后,将PVDF膜置于HRP标记的β-actin抗体工作液(1∶5 000稀释)中,室温摇床孵育2 h; TBST洗膜4次,每次5 min。之后将膜放入ECL发光液中作用2 min,于暗室中曝光并保存目标蛋白结果图。最后使用ImageJ软件对转染组和对照组的蛋白条带分别进行灰度分析,并与β-actin蛋白进行均一化处理。
1.4 山羊RORα基因生物信息学分析利用Ensembl在线数据库(https://www.ensembl.org/index.html)对山羊RORα基因全长序列进行分析。在NCBI数据库中查找山羊和小鼠、大鼠、人、牛、绵羊、猪、马、斑马鱼等多个物种的RORα基因CDS序列(表 2)。使用生物信息学软件DNAStar对各物种RORα基因的CDS序列进行同源性比对; 应用MEGA7软件对各物种RORα基因的CDS序列进行分析并构建系统进化树; 使用在线软件SignalP-5.0(https://services.healthtech.dtu.dk/service.php?SignalP-5.0)、TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM/)[21]、ProtParam(https://web.expasy.org/protparam/)[22]、ProtScale(https://web.expasy.org/protscale/)[23]、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL(https://swissmodel.expasy.org/)对山羊RORα蛋白的理化性质、亲/疏水性、信号肽、跨膜结构、蛋白二级以及三级结构进行生物信息学预测和分析。最后,使用pyMOL软件对山羊、小鼠和人三者的RORα蛋白三维结构进行相似性比较。
|
|
表 2 各物种RORα基因CDS区序列来源 Table 2 The sources of CDS of RORα gene in different species |
使用基因组DNA提取试剂盒提取山羊肝组织的基因组DNA。以基因组DNA为模板,利用设计好的带有同源臂的引物(表 1)分别扩增山羊BMAL1-Promoter片段以及NR1D1-Promoter片段,扩增产物用1%琼脂糖凝胶进行凝胶电泳鉴定,目的条带按照胶回收试剂盒操作说明分别进行胶回收。将胶回收产物分别与Kpn I或Xho I线性化的pGL4.10载体进行同源重组连接,操作步骤同“1.3.2”。随后分别使用限制性核酸内切酶Kpn I和Xho I对连接有BMAL1-Promoter片段及连接有NR1D1-Promoter片段的重组质粒进行单酶切鉴定。同时以重组质粒为模板,使用BMAL1-Promoter和NR1D1-Promoter的扩增引物对相应的重组质粒进行PCR鉴定。将酶切及PCR鉴定结果符合预期的重组质粒送至西安擎科生物有限公司进行测序鉴定,返回序列与NCBI数据库中已知序列进行比对。将测序正确的重组质粒分别命名为pGL4.10-BMAL1-Promoter-Luc和pGL4.10-NR1D1-Promoter-Luc。
1.6 双荧光素酶报告试验将HEK293T细胞以每孔5×104个细胞接种至1个24孔板中,置于细胞培养箱中培养24 h。随后,使用TurboFect转染试剂将pcDNA3.1-3HA-gRORα真核表达载体(试验组)、pcDNA3.1-Puro-N-3HA空载体(对照组)分别与pRL-CMV海肾荧光素酶报告质粒(内参质粒)和pGL4.10-BMAL1-Promoter-Luc(或pGL4.10-NR1D1-Promoter-Luc)萤火虫荧光素酶报告质粒共转染至HEK293T细胞中,每组平行设3个重复孔。转染36 h后,按照双荧光素酶报告试验试剂盒说明收集并处理细胞样品,使用BioTek Synergy 2多功能酶标仪测定荧光值,计算相对荧光强度,测定重复3次取平均值。
1.7 数据统计分析使用GraphPad Prism 6.0软件进行统计学分析,结果以“平均值±标准误”表示,两组间样本均数比较采取t检验,*P < 0.05表示差异显著,**P < 0.01表示差异极显著。
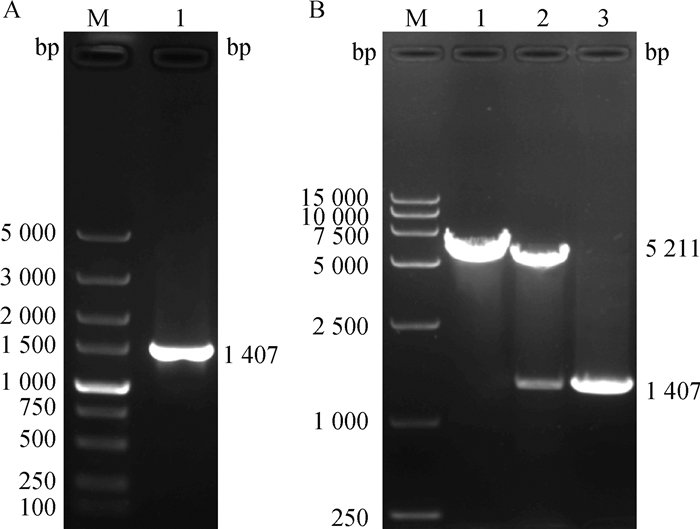
2 结果 2.1 山羊RORα基因的克隆及其真核表达载体的构建对山羊RORα基因CDS区全长序列分析后发现,山羊RORα基因共包含10个外显子,各个外显子长度在28 ~ 396 bp之间,所有内含子的剪接位点序列均符合GT-AG法则(表 3)。随后,提取了山羊肝组织的总RNA,反转录得到cDNA,以cDNA为模板使用PCR技术成功扩增获得山羊RORα基因CDS区片段,电泳结果如图 1A所示,在1 500 bp下方观察到单一条带,与预期结果一致(1 407 bp)。将扩增片段进行纯化回收后,与EcoR I酶切线性化的骨架载体pcDNA3.1-Puro-N-3HA进行同源重组连接,构建真核表达载体pcDNA3.1-3HA-gRORα。通过酶切和PCR鉴定重组载体,同时用EcoR I对空载体进行单酶切作对照,鉴定结果如图 1B所示。重组载体经过EcoR I酶切后获得两条目的条带,上方条带大小与空载体单酶切条带一致(5 211 bp),下方条带大小与PCR扩增产物条带大小一致(1 407 bp)。最后将鉴定正确的质粒进行测序,测序结果显示pcDNA3.1-3HA-gRORα中插入片段的序列与NCBI库中山羊RORα基因已知序列完全一致。以上结果表明成功获得了山羊RORα基因CDS区全长片段,且真核表达载体pcDNA3.1-3HA-gRORα构建成功。
|
|
表 3 山羊RORα基因的剪接位点 Table 3 Splice sites of the goat RORα gene |
|
A. RORα基因CDS区扩增结果:M. DNA相对分子质量标准; 1. RORα基因CDS区片段。B. pcDNA3.1-3HA-gRORα重组载体酶切鉴定结果:M. DNA相对分子质量标准; 1. 空载体单酶切产物; 2. 重组载体pcDNA3.1-3HA-gRORα单酶切产物; 3. RORα基因质粒PCR目的条带 A. Amplification result of RORα gene CDS region: M. 5 000 bp DNA Marker; 1. The fragment of RORα gene CDS region. B. Restriction digestion result of pcDNA3.1-3HA-gRORα eukaryotic expression vector: M. 15 000 bp DNA Marker; 1. Single digested product of empty vector; 2. Single digestion product of recombinant vector pcDNA3.1-3HA-gRORα; 3. The PCR target band of RORα gene 图 1 山羊RORα基因CDS扩增与pcDNA3.1-3HA-gRORα真核表达载体鉴定结果 Fig. 1 Results of goat RORα gene CDS amplification and identification of pcDNA3.1-3HA-gRORα eukaryotic expression vector |
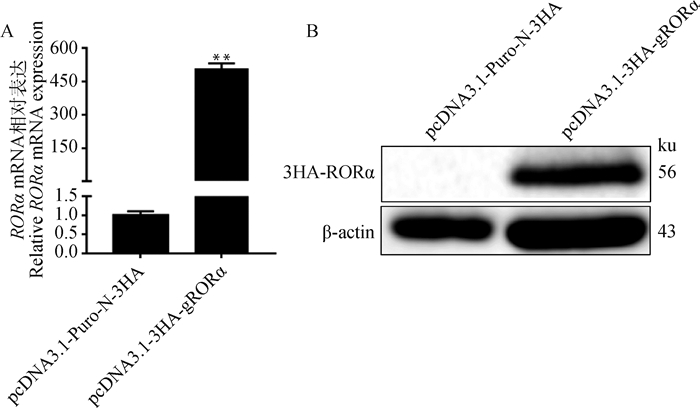
qPCR结果如图 2A所示,与对照组相比,试验组RORα基因的mRNA表达量升高约500倍(P < 0.01)。WB结果如图 2B所示,用HA抗体孵育后,试验组在56 ku处有明显的条带,而对照组在相应位置无条带。上述结果表明,pcDNA3.1-3HA-gRORα转染HEK293T细胞后山羊RORα基因在mRNA和蛋白表达水平均明显高于对照组。
|
A.山羊RORα基因mRNA水平相对表达量; B. RORα蛋白WB鉴定结果。**表示差异极显著(P < 0.01) A. Relative mRNA expression of goat RORα gene; B. WB analysis of goat RORα protein expression. ** indicating that the difference is extremely significant (P < 0.01) 图 2 qPCR及WB检测结果 Fig. 2 Results of qPCR and WB analysis |
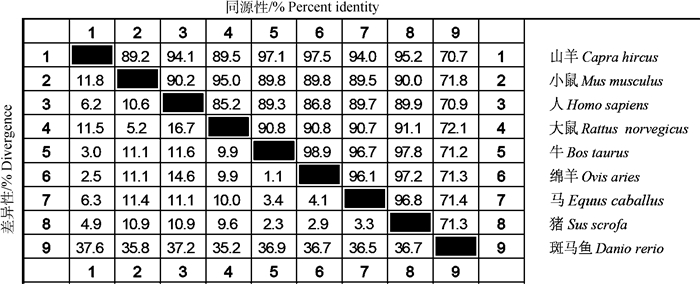
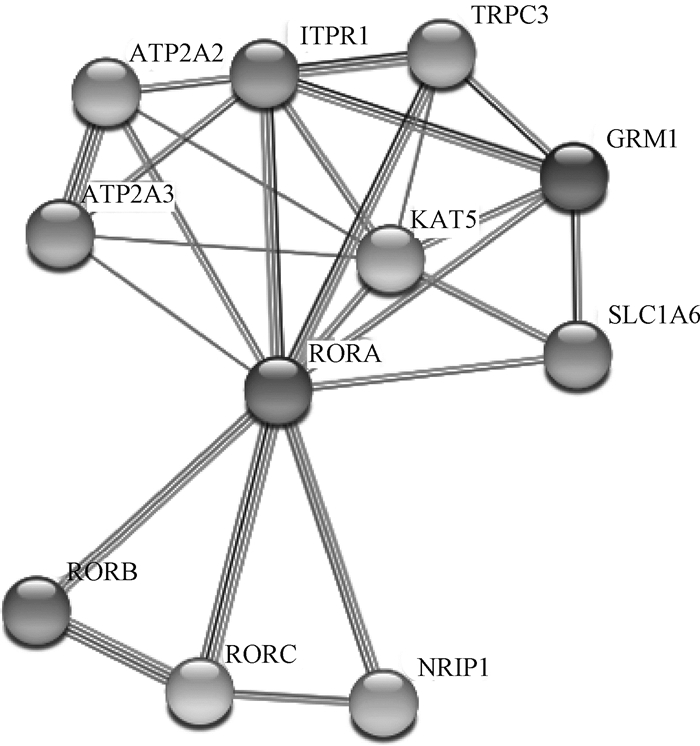
2.3.1 同源性比对及系统进化树构建 使用DNAStar软件对多个物种RORα基因CDS区进行相似性分析,结果如图 3所示。山羊RORα CDS区与小鼠、人、大鼠、牛、绵羊、马、猪和斑马鱼的RORα CDS区相似性分别为:89.2%、94.1%、89.5%、97.1%、97.5%、94.0%、95.2%、70.7%。不同物种间基因相似性较高,进化过程中具有高度保守性。使用MEGA6软件构建RORα基因的系统进化树,结果如图 4所示,山羊与绵羊的亲缘关系最近,其次是和牛、猪等其它哺乳动物,和斑马鱼亲缘关系最远。利用STRING数据库检索到RORB、RORC、NRIP1(nuclear receptor interacting protein 1)、SLC1A6(solute carrier family 1 member 6)、KAT5(lysine acetyltransferase 5)、GRM1(glutamate metabotropic receptor 1)、TRPC3(transient receptor potential cation channel subfamily C member 3)、ITPR1(inositol 1, 4, 5-trisphosphate receptor type 1)、ATP2A2(ATPase sarcoplasmic/endoplasmic reticulum Ca2+transporting 2)、ATP2A3(ATPase sarcoplasmic/endoplasmic reticulum Ca2+ transporting 3)共10个蛋白可能和RORα蛋白存在相互作用(图 5)。
|
图 3 RORα基因CDS区序列同源性比对 Fig. 3 Sequence homology alignment of RORα gene CDS region |

|
图 4 RORα基因的系统进化树 Fig. 4 Phylogenetic tree of RORα gene |
|
图 5 RORα互作蛋白预测 Fig. 5 Prediction of proteins interacting with RORα protein |
2.3.2 山羊RORα蛋白理化性质分析 利用在线工具ExPASy中的ProtParam对山羊RORα蛋白进行理化性质分析,结果显示山羊RORα蛋白理论分子质量为53 397.15,分子式为C2355H3692N644O705S34,原子总数是7 430个,等电点(pI)为6.37,属于碱性蛋白; 该蛋白含有468个氨基酸,其中亮氨酸含量最高(8.3%),色氨酸含量最低(0.6%)(表 4);在水中以280 nm预测消光系数是41 590;半衰期为30 h; 不稳定系数为38.97;脂肪族系数为78.16。
|
|
表 4 山羊RORα蛋白氨基酸组成 Table 4 Amino acids composition of goat RORα protein |
2.3.3 亲/疏水性预测 利用在线工具ExPASy对山羊RORα蛋白氨基酸序列的亲水/疏水性进行预测分析,结果如图 6所示,山羊RORα蛋白氨基酸序列最低分值为-3.578(位于第115、116位氨基酸),亲水性最强; 最高分值为2.744(位于第369位氨基酸),疏水性最强。综合来看,该蛋白整条肽链大部分属于亲水区域,由此推测山羊RORα蛋白为亲水性蛋白。
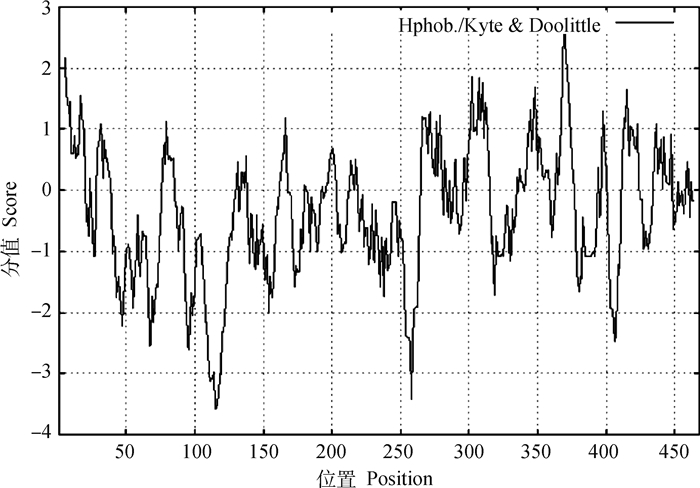
|
图 6 山羊RORα蛋白亲/疏水性预测 Fig. 6 Hydrophobicity/hydrophilicity prediction of goat RORα protein |
2.3.4 信号肽及跨膜区预测 利用在线工具SignalP-5.0对山羊RORα氨基酸进行信号肽预测,结果显示,该蛋白有12%的概率存在位于0~18氨基酸之间的信号肽,该蛋白有信号肽的概率较小。利用在线软件TMHMM Serverv.2.0对山羊RORα蛋白进行跨膜结构预测,结果显示,该蛋白不存在跨膜区。
2.3.5 山羊RORα蛋白二级结构预测 利用在线工具SOPMA对山羊RORα蛋白二级结构进行预测,结果如图 7所示,山羊RORα蛋白主要由α-螺旋、延伸链、β-转角和无规则卷曲等组成,其中α-螺旋占43.8%,延伸链占10.04%,β-转角占4.91%,无规则卷曲占41.24%。

|
h表示α-螺旋,e表示延伸链,t表示β-转角,c表示无规则卷曲 h stands for alpha helix, e stands for extended strand, t stands for beta turn, c stands for random coil 图 7 山羊RORα蛋白二级结构分析 Fig. 7 Analysis of the goat RORα protein secondary structure |
2.3.6 山羊RORα蛋白三级结构预测分析 通过在线软件SWISS-MODEL对山羊(图 8A)、小鼠(图 8B)和人(图 8C)的RORα蛋白三级结构进行预测,结果显示,3个物种的RORα蛋白均具有α-螺旋、β-转角和无规则卷曲等复杂结构。使用pyMOL软件对3个物种的蛋白模型的结构进行比对,结果显示山羊和小鼠模型的原子位置均方根偏差(root-mean-square deviation,RMSD)为0.068,山羊和人的RMSD为0.077,山羊RORα蛋白三级结构模型与小鼠和人的差异均极小。
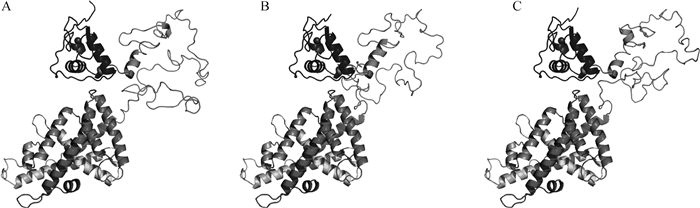
|
A.山羊RORα蛋白三级结构预测; B.小鼠RORα蛋白三级结构预测; C.人RORα蛋白三级结构预测 A. Prediction of goat RORα protein tertiary structure; B. Prediction of mouse RORα protein tertiary structure; C. Prediction of human RORα protein tertiary structure 图 8 山羊、小鼠和人RORα蛋白的三级结构预测 Fig. 8 Prediction of the tertiary structure of RORα protein in goat, mouse and human |
以山羊肝基因组DNA为模板,利用合成的BMAL1-Promoter及NR1D1-Promoter引物进行PCR扩增。结果如图 9A、9C所示,BMAL1-Promoter(862 bp)及NR1D1-Promoter(1 657 bp)PCR扩增产物条带单一,与目的片段大小一致,无非特异性条带。将克隆得到的目的条带分别与pGL4.10空载体进行重组连接,构建pGL4.10-BMAL1-Pro- moter-Luc和pGL4.10-NR1D1-Promoter-Luc重组质粒。对重组质粒分别进行PCR及酶切鉴定,结果显示pGL4.10-BMAL1-Promoter-Luc报告质粒经Kpn I单酶切后得到862和4 242 bp两个片段(图 9B),且以pGL4.10-BMAL1-Promoter-Luc为模板PCR扩增出大小约862 bp的片段。pGL4.10-NR1D1-Promoter-Luc报告质粒经Xho I单酶切后得到1 657和4 242 bp两个片段(图 9D),且以pGL4.10- NR1D1-Promoter-Luc为模板PCR扩增出大小约1 657 bp的片段。同时,两个重组质粒的测序结果也均与NCBI数据库中已知序列完全一致。上述结果表明,含有山羊BMAL1基因启动子片段的pGL4.10-BMAL1-Promoter-Luc重组质粒和含有山羊NR1D1基因启动子片段的pGL4.10-NR1D1-Promoter-Luc重组质粒构建成功。
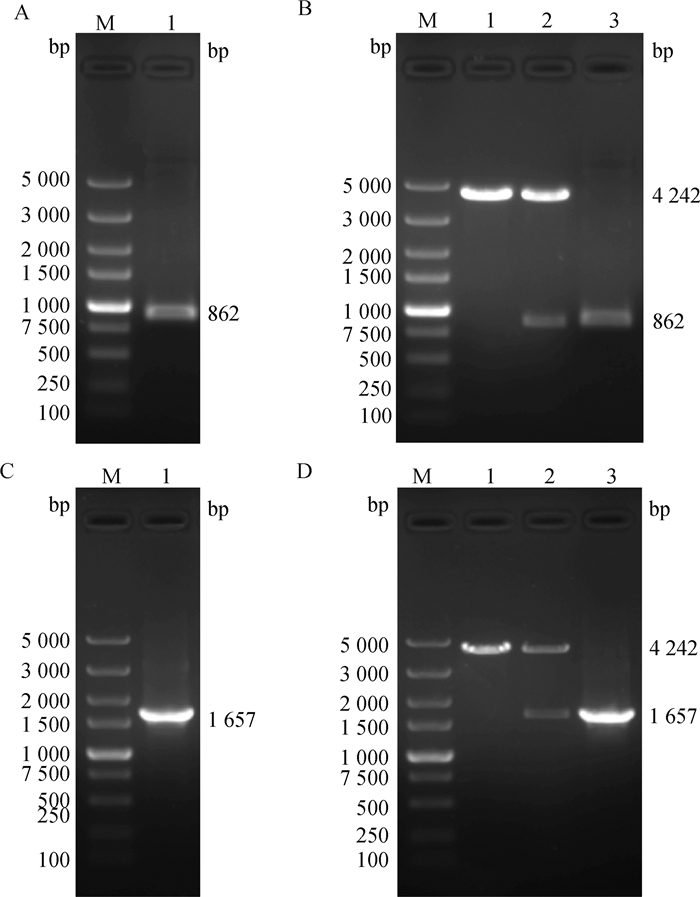
|
A. BMAL1-Promoter PCR扩增结果:M. DNA相对分子质量标准; 1. BMAL1-Promoter目的片段。B. pGL4.10-BMAL1-Promoter-Luc载体酶切及PCR鉴定结果:M. DNA相对分子质量标准; 1. 空载体单酶切产物; 2. pGL4.10-BMAL1-Promoter-Luc载体单酶切产物; 3. BMAL1-Promoter质粒PCR目的条带。C. NR1D1-Promoter PCR扩增结果:M.DNA相对分子质量标准; 1.NR1D1-Promoter目的片段。D. pGL4.10-NR1D1-Promoter-Luc载体酶切及PCR鉴定结果:M.DNA相对分子质量标准; 1. 空载体单酶切产物; 2. pGL4.10-NR1D1-Promoter-Luc载体单酶切产物; 3. NR1D1-Promoter质粒PCR目的条带 A. PCR amplification result of BMAL1-Promoter: M. 5 000 bp DNA Marker; 1. The target band of BMAL1-Promoter.B. Restriction digestion and PCR result of pGL4.10-BMAL1-Promoter-Luc vector: M. 5 000 bp DNA Marker; 1. Single digested product of empty vector; 2. Single digestion product of pGL4.10-BMAL1-Promoter-Luc vector; 3. The target band of BMAL1-Promoter. C. PCR amplification result of NR1D1-Promoter: M. 5 000 bp DNA Marker; 1. The target band of NR1D1-Promoter.D. Restriction digestion and PCR result of pGL4.10-NR1D1-Promoter-Luc vector: M. 5 000 bp DNA Marker; 1. Single digested product of empty vector; 2. Single digestion product of pGL4.10-NR1D1-Promoter-Luc vector; 3. The target band of NR1D1-Promoter 图 9 pGL4.10-BMAL1-Promoter-Luc和pGL4.10-NR1D1-Promoter-Luc质粒鉴定结果 Fig. 9 Results of identification of pGL4.10-BMAL1-Promoter-Luc and pGL4.10-NR1D1-Promoter-Luc plasmids |
使用pcDNA3.1-3HA-gRORα质粒及含有RORE元件的pGL4.10-BMAL1-Promoter-Luc和pGL4.10-NR1D1-Promoter-Luc重组质粒分别进行双荧光素酶报告试验以探究山羊RORα蛋白的功能。如图 10A所示,本研究中扩增获得的BMAL1-Promoter片段位于山羊BMAL1基因的-684 ~ +178位置(山羊BMAL1基因的转录起始位点为+1),包含3个RORE元件; 扩增获得的NR1D1-Promoter位于山羊NR1D1基因的-1 421~ +236位置(山羊NR1D1基因的转录起始位点为+1),也包含3个RORE元件。双荧光素酶报告试验结果表明,与共转染pcDNA3.1-Puro-N-3HA空质粒的对照组相比,共转染pcDNA3.1-3HA-gRORα质粒时BMAL1-Promoter(图 10B)和NR1D1-Promoter(图 10C)调控下的荧光素酶活性均极显著升高(P < 0.01)。上述结果表明,山羊RORα蛋白可以显著上调山羊生物钟基因BMAL1和NR1D1启动子的转录活性,这与小鼠等哺乳动物生物钟转录调控机制相符。
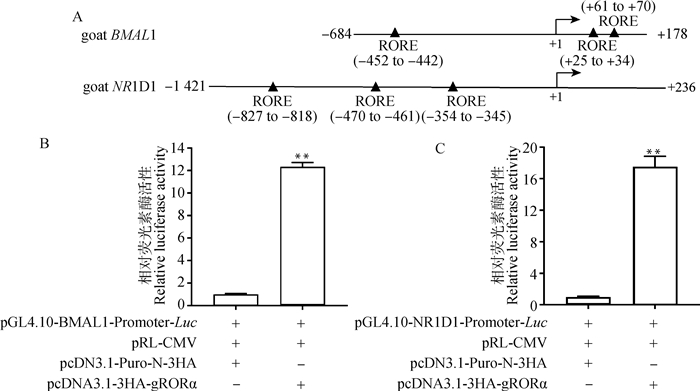
|
A.山羊BMAL1和NR1D1基因启动子区域RORE元件相对于基因转录起始位点(+1)的位置示意图; B、C. 双荧光素酶报告试验结果。**表示差异极显著(P < 0.01) A. Schematic diagram of the position of the RORE response element in the promoter region of goat BMAL1 and NR1D1 genes relative to the transcription start site (+1) of the gene; B, C. Dual-luciferase reporter assay results. ** means the difference is extremely significant (P < 0.01) 图 10 RORα蛋白对山羊BMAL1和NR1D1启动子转录活性的影响 Fig. 10 Effects of RORα protein on the transcriptional activities of goat BMAL1 and NR1D1 promoters |
昼夜节律是地球上的生命体对地球自转产生的昼夜更替现象所做出的主动适应性变化。多项研究表明,昼夜节律紊乱与睡眠障碍、代谢失调、肿瘤等多种疾病的发生密切相关[24-25]。生物体内参与生物钟发生和维持的基因被称为生物钟基因,这些基因与钟控基因共同调控哺乳动物生理、生化、行为等方面的昼夜节律性变化[26]。RORα作为生物钟系统的重要组分,在维持生物钟的正常运转过程中发挥重要作用[8-9]。然而,目前有关生物钟分子调控机制的研究大多是以小鼠为模型,关于山羊生物钟基因及山羊生物钟转录调控机制方面的研究相对较少。本课题组前期成功构建了山羊生物钟基因BMAL1、CLOCK和NR1D1的真核表达载体,发现BMAL1与NR1D1基因mRNA节律性表达于山羊睾丸间质细胞中,并证明了BMAL1基因通过调节山羊睾丸间质细胞中类固醇合成相关基因的转录来调控睾酮产生[27-29]。赵艳红等[20, 30]前期虽已克隆了内蒙古绒山羊RORα基因的CDS区,但只是部分序列(417 bp),目前尚未有克隆山羊RORα基因全长CDS序列(1 407 bp)的研究报道。基于此,本研究克隆了山羊RORα基因CDS区全长片段,构建了其真核表达载体,并对山羊RORα进行了生物信息学分析,初步探究了其在山羊生物钟系统中的作用机制,为后续进一步深入解析山羊生物钟系统的转录调控机制奠定基础。
自RORα基因在小鼠中被克隆鉴定以来,人们对其生理功能的认识不断增加。Chen等[31-32]发现,RORα基因在小鼠小脑发育中起着关键作用,RORα基因缺陷小鼠表现出共济失调和严重的小脑萎缩,其特征是浦肯野细胞明显减少,小脑颗粒细胞丢失。Dzhagalov等[33]研究表明,RORα在胸腺生成和淋巴细胞发育过程中也发挥重要作用,RORα缺失小鼠的脾和胸腺都明显较小,且成熟T和B淋巴细胞均显著减少。此外,还有大量证据表明,RORα在视网膜、大脑以及T细胞等发育过程中有着重要的调控作用[16, 34-36]。同时,许多研究发现RORα可以参与调控小鼠和人等哺乳动物的生殖系统发育以及睾酮合成。Sayed等[37]在RORα基因敲除小鼠睾丸中观察到生精不足、睾丸间质细胞数量减少以及精子形态发生变化等现象。随后,该研究又发现RORα基因突变的老年小鼠会出现生精小管萎缩以及精子顶体帽缺失和空泡化的现象[38]。Yang等[39]研究发现,褪黑素可通过RORα通路来提高雄性哺乳动物睾丸间质细胞的睾酮合成能力。另外,在绵羊的研究发现,褪黑素可通过RORα在体内和体外水平上提高青春期前卵丘细胞抗氧化能力[40]。同时,在山羊精原干细胞体外分化培养系统中,褪黑素可通过RORα通路增加睾丸间质细胞的睾酮分泌[41]。总之,上述研究结果表明RORα在哺乳动物的多个生物学过程中都扮演着重要角色。
哺乳动物的生物钟系统由位于SCN的中枢生物钟和分布于机体大部分组织器官的外周生物钟组成[5]。肝是机体中最为主要的外周生物钟器官,其在机体营养代谢中发挥重要作用,由相对均质的细胞类型组成,且易于获取[42-43]。另外,生物钟基因RORα作为一种重要的核受体,与肝脂质代谢调节密切相关,且在小鼠肝中高表达[16, 44]。据此推测,RORα基因也高表达于山羊肝中,所以本研究选取山羊肝组织来提取cDNA,进一步克隆RORα基因的CDS区全长序列。另外,本研究还对山羊RORα基因序列进行了生物信息学分析,同源性分析结果显示山羊RORα基因的CDS区序列与绵羊和牛等昼行性反刍动物的RORα基因相似性极高,说明此基因在反刍动物具有较高的保守性和同源性。另外,分析系统进化树结果发现,进化树的形态与分类学基本保持一致。
此外,本研究结果还表明山羊RORα蛋白属于一种亲水性的非跨膜蛋白。通过将山羊、小鼠和人的RORα蛋白三维结构比对后发现,三者间存在高度的相似性,均含有4个主要功能区组成的核受体结构域:N-末端(A/B)结构域、高度保守的DNA结合域(DNA binding domain,DBD)、铰链域和C-末端配体结合域。由于小鼠RORα蛋白的DBD已被证明可通过与特定的DNA反应元件(RORE元件)结合来调控基因转录,进而发挥其在生物钟系统及各项生理过程中的调控作用[8]。蛋白质结构决定其功能,所以推测山羊RORα蛋白功能与小鼠之间存在保守性,其可能也在山羊生物钟系统中发挥重要调控作用。本研究使用了双荧光素酶报告试验探究验证山羊RORα蛋白的功能,结果显示山羊RORα蛋白可以显著上调山羊BMAL1和NR1D1基因启动子的转录活性。该结果进一步证实了上述猜想,证实山羊RORα蛋白确实在山羊生物钟系统中发挥重要调控作用。另外,结合小鼠和人RORα在调控机体生理功能方面已有的研究证据推测,RORα可能在山羊的生长发育过程中也发挥重要作用,但具体功能有待于进一步的研究。
4 结论本研究克隆了山羊RORα基因的全长CDS区片段,构建了其真核表达载体,从mRNA水平和蛋白水平验证了RORα在HEK293T细胞中的过表达效果,并对该基因进行了生物信息学分析。结果表明,RORα蛋白在山羊与小鼠等哺乳动物间存在高度保守性,其可以正向调控山羊BMAL1和NR1D1基因的启动子活性,为进一步探究山羊核受体RORα的功能及山羊生物钟的转录调控机制提供了前期基础和关键材料。
| [1] |
王晗. 生物钟生物学及其研究进展[J]. 生命科学, 2015, 27(11): 1313-1319. WANG H. Circadian biology and its recent progresses[J]. Chinese Bulletin of Life Sciences, 2015, 27(11): 1313-1319. (in Chinese) |
| [2] |
邢陈, 宋伦. 昼夜节律产生和维持的调控系统[J]. 军事医学, 2017, 41(8): 698-702. XING C, SONG L. Regulation system for generation and maintenance of circadian rhythms[J]. Military Medical Sciences, 2017, 41(8): 698-702. (in Chinese) |
| [3] |
MARCHEVA B, RAMSEY K M, BUHR E D, et al. Disruption of the clock components CLOCK and BMAL1 leads to hypoinsulinaemia and diabetes[J]. Nature, 2010, 466(7306): 627-631. DOI:10.1038/nature09253 |
| [4] |
ZHANG R, LAHENS N F, BALLANCE H I, et al. A circadian gene expression atlas in mammals: implications for biology and medicine[J]. Proc Natl Acad Sci U S A, 2014, 111(45): 16219-16224. DOI:10.1073/pnas.1408886111 |
| [5] |
ASTIZ M, HEYDE I, OSTER H. Mechanisms of communication in the mammalian circadian timing system[J]. Int J Mol Sci, 2019, 20(2): 343. DOI:10.3390/ijms20020343 |
| [6] |
ROSENWASSER A M, TUREK F W. Neurobiology of circadian rhythm regulation[J]. Sleep Med Clin, 2015, 10(4): 403-412. DOI:10.1016/j.jsmc.2015.08.003 |
| [7] |
ABBOTT S M, ZEE P C. Circadian rhythms: implications for health and disease[J]. Neurol Clin, 2019, 37(3): 601-613. DOI:10.1016/j.ncl.2019.04.004 |
| [8] |
GIGUÈRE V, TINI M, FLOCK G, et al. Isoform-specific amino-terminal domains dictate DNA-binding properties of RORα, a novel family of orphan hormone nuclear receptors[J]. Genes Dev, 1994, 8(5): 538-553. DOI:10.1101/gad.8.5.538 |
| [9] |
MA H Z, KANG J, FAN W G, et al. ROR: nuclear receptor for melatonin or not?[J]. Molecules, 2021, 26(9): 2693. DOI:10.3390/molecules26092693 |
| [10] |
PILORZ V, ASTIZ M, HEINEN K O, et al. The concept of coupling in the mammalian circadian clock network[J]. J Mol Biol, 2020, 432(12): 3618-3638. DOI:10.1016/j.jmb.2019.12.037 |
| [11] |
URIZ-HUARTE A, DATE A, ANG H, et al. The transcriptional repressor REV-ERB as a novel target for disease[J]. Bioorg Med Chem Lett, 2020, 30(17): 127395. DOI:10.1016/j.bmcl.2020.127395 |
| [12] |
STRATMANN M, STADLER F, TAMANINI F, et al. Flexible phase adjustment of circadian albumin D site-binding protein (Dbp) gene expression by CRYPTOCHROME1[J]. Genes Dev, 2010, 24(12): 1317-1328. DOI:10.1101/gad.578810 |
| [13] |
UEDA H R, HAYASHI S, CHEN W B, et al. System-level identification of transcriptional circuits underlying mammalian circadian clocks[J]. Nat Genet, 2005, 37(2): 187-192. DOI:10.1038/ng1504 |
| [14] |
RIPPERGER J A, SCHIBLER U. Rhythmic CLOCK-BMAL1 binding to multiple E-box motifs drives circadian Dbp transcription and chromatin transitions[J]. Nat Genet, 2006, 38(3): 369-374. DOI:10.1038/ng1738 |
| [15] |
POURCET B, DUEZ H. Circadian Control of inflammasome pathways: implications for circadian medicine[J]. Front Immunol, 2020, 11: 1630. DOI:10.3389/fimmu.2020.01630 |
| [16] |
JETTEN A M. Retinoid-related orphan receptors (RORs): critical roles in development, immunity, circadian rhythm, and cellular metabolism[J]. Nucl Recept Signal, 2009, 7: e003. |
| [17] |
FAN J S, LV Z L, YANG G H, et al. Retinoic acid receptor-related orphan receptors: critical roles in tumorigenesis[J]. Front Immunol, 2018, 9: 1187. DOI:10.3389/fimmu.2018.01187 |
| [18] |
LI Z Q, ZHAO J, LIU H Y, et al. Melatonin inhibits apoptosis in mouse Leydig cells via the retinoic acid-related orphan nuclear receptor α/p53 pathway[J]. Life Sci, 2020, 246: 117431. DOI:10.1016/j.lfs.2020.117431 |
| [19] |
贾丽丽, 金凤, 付绍印, 等. 生物钟基因在绒山羊皮肤中表达模式的比较[J]. 中国畜牧兽医, 2015, 42(2): 251-257. JIA L L, JIN F, FU S Y, et al. Comparation of expression pattern of Clock genes in cashmere goat skin[J]. China Animal Husbandry & Veterinary Medicine, 2015, 42(2): 251-257. (in Chinese) |
| [20] |
ZHAO Y H, LIU Z H, WANG L, et al. Expression of the RORα gene in Inner Mongolian cashmere goat hair follicles[J]. Genet Mol Res, 2015, 14(1): 380-388. DOI:10.4238/2015.January.23.11 |
| [21] |
刘宗立, 陈涛, 杨丹丹, 等. 猪Nrf2基因克隆、生物信息学分析及启动子区转录活性分析[J]. 畜牧兽医学报, 2019, 50(7): 1328-1339. LIU Z L, CHEN T, YANG D D, et al. Molecular cloning, bioinformatics analysis and transcriptional activity of promoter of nuclear factor erythroid 2-related factor(Nrf2) gene in pig[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(7): 1328-1339. (in Chinese) |
| [22] |
杜鹏飞, 陈博, 高林歌, 等. 鸡HMIT基因的克隆与表达分析[J]. 畜牧兽医学报, 2020, 51(8): 1811-1822. DU P F, CHEN B, GAO L G, et al. Cloning and expression analysis of chicken HMIT gene[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(8): 1811-1822. (in Chinese) |
| [23] |
王宪军, 向华, 张焕容, 等. 山羊HABP4基因的克隆、序列分析及功能预测[J]. 畜牧兽医学报, 2021, 52(12): 3426-3438. WANG X J, XIANG H, ZHANG H R, et al. Cloning, sequence analysis and function prediction of HABP4 gene in goat[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(12): 3426-3438. DOI:10.11843/j.issn.0366-6964.2021.012.010 (in Chinese) |
| [24] |
MAURY E. Off the Clock: from circadian disruption to metabolic disease[J]. Int J Mol Sci, 2019, 20(7): 1597. DOI:10.3390/ijms20071597 |
| [25] |
YU E A, WEAVER D R. Disrupting the circadian clock: gene-specific effects on aging, cancer, and other phenotypes[J]. Aging (Albany NY), 2011, 3(5): 479-493. |
| [26] |
凡杰夫, 何颖, 王伟忠, 等. 调控生物钟的小分子化合物的研究进展[J]. 中国药房, 2021, 32(7): 890-896. FAN J F, HE Y, WANG W Z, et al. Research progress of small molecular compounds regulating biological clock[J]. China Pharmacy, 2021, 32(7): 890-896. (in Chinese) |
| [27] |
XIAO Y Y, ZHAO L J, LI W D, et al. Circadian clock gene BMAL1 controls testosterone production by regulating steroidogenesis-related gene transcription in goat Leydig cells[J]. J Cell Physiol, 2021, 236(9): 6706-6725. DOI:10.1002/jcp.30334 |
| [28] |
赵泓淙, 高登科, 江海圳, 等. 山羊生物钟基因CLOCK真核表达载体的构建和生物信息学分析[J]. 中国畜牧兽医, 2021, 48(12): 4327-4338. ZHAO H C, GAO D K, JIANG H Z, et al. Construction of eukaryotic expression vector and bioinformatics analysis of circadian CLOCK gene in goats[J]. China Animal Husbandry & Veterinary Medicine, 2021, 48(12): 4327-4338. (in Chinese) |
| [29] |
王逸群, 高登科, 赵泓淙, 等. 山羊NR1D1基因真核表达载体的构建及生物信息学分析[J]. 中国畜牧杂志. WANG Y Q, GAO D K, ZHAO H C, et al. Construction of a eukaryotic expression vector in goat NR1D1 gene and its bioinformatics analysis[J]. Chinese Journal of Animal Science. DOI:10.19556/j.0258-7033.20210825-06 (in Chinese) |
| [30] |
赵艳红, 孙永明, 刘志红, 等. 内蒙古绒山羊孤核受体RORα部分cDNA克隆及序列分析[J]. 内蒙古农业大学学报, 2010, 31(3): 1-4. ZHAO Y H, SUN Y M, LIU Z H, et al. Cloning and sequence analysis of RORα gene in inner Mongolian cashmere goat[J]. Journal of Inner Mongolia Agricultural University, 2010, 31(3): 1-4. DOI:10.3969/j.issn.1009-4458.2010.03.001 (in Chinese) |
| [31] |
CHEN C T, SCHULTZ J A, HAVEN S E, et al. Loss of RAR-related orphan receptor alpha (RORα) selectively lowers docosahexaenoic acid in developing cerebellum[J]. Prostaglandins Leukot Essent Fatty Acids, 2020, 152: 102036. DOI:10.1016/j.plefa.2019.102036 |
| [32] |
YASUI H, MATSUZAKI Y, KONNO A, et al. Global knockdown of retinoid-related orphan Receptor α in mature Purkinje cells reveals aberrant cerebellar phenotypes of Spinocerebellar ataxia[J]. Neuroscience, 2021, 462: 328-336. DOI:10.1016/j.neuroscience.2020.04.004 |
| [33] |
DZHAGALOV I, GIGUÈRE V, HE Y W. Lymphocyte development and function in the absence of retinoic acid-related orphan receptor α[J]. J Immunol, 2004, 173(5): 2952-2959. DOI:10.4049/jimmunol.173.5.2952 |
| [34] |
COOK D N, KANG H S, JETTEN A M. Retinoic acid-related orphan receptors (RORs): regulatory functions in Immunity, development, circadian rhythm, and metabolism[J]. Nucl Receptor Res, 2015, 2: 101185. |
| [35] |
FUJIEDA H, BREMNER R, MEARS A J, et al. Retinoic acid receptor-related orphan receptor α regulates a subset of cone genes during mouse retinal development[J]. J Neurochem, 2009, 108(1): 91-101. DOI:10.1111/j.1471-4159.2008.05739.x |
| [36] |
CAI Z M, ISHIBASHI T, KOZAI M, et al. ROR agonist hampers the proliferation and survival of postactivated CD8+ T cells through the downregulation of cholesterol synthesis-related genes[J]. Immunol Cell Biol, 2021, 99(3): 288-298. DOI:10.1111/imcb.12406 |
| [37] |
SAYED R K A, MOKHTAR D M, FERNÁNDEZ-ORTIZ M, et al. Retinoid-related orphan nuclear receptor alpha (RORα)-deficient mice display morphological testicular defects[J]. Lab Invest, 2019, 99(12): 1835-1849. DOI:10.1038/s41374-019-0299-5 |
| [38] |
SAYED R K A, MOKHTAR D M, FERNÁNDEZ-ORTIZ M, et al. Lack of retinoid acid receptor-related orphan receptor alpha accelerates and melatonin supplementation prevents testicular aging[J]. Aging (Albany NY), 2020, 12(13): 12648-12668. |
| [39] |
YANG M H, GUAN S Y, TAO J L, et al. Melatonin promotes male reproductive performance and increases testosterone synthesis in mammalian Leydig cells[J]. Biol Reprod, 2021, 104(6): 1322-1336. DOI:10.1093/biolre/ioab046 |
| [40] |
FANG Y, ZHANG J L, LI Y H, et al. Melatonin-induced demethylation of antioxidant genes increases antioxidant capacity through RORα in cumulus cells of prepubertal lambs[J]. Free Radic Biol Med, 2019, 131: 173-183. DOI:10.1016/j.freeradbiomed.2018.11.027 |
| [41] |
DENG S L, ZHANG Y, YU K, et al. Melatonin up-regulates the expression of the GATA-4 transcription factor and increases testosterone secretion from Leydig cells through RORα signaling in an in vitro goat spermatogonial stem cell differentiation culture system[J]. Oncotarget, 2017, 8(66): 110592-110605. DOI:10.18632/oncotarget.22855 |
| [42] |
徐晴, 黄鸣, 王晓明, 等. TF通过影响肝脏生物钟基因表达促进昼夜节律相位前移[J]. 军事医学, 2021, 45(1): 1-6. XU Q, HUANG M, WANG X M, et al. TF promotes circadian phase advancement by affecting expressions of liver circadian clock genes[J]. Military Medical Sciences, 2021, 45(1): 1-6. DOI:10.3969/j.issn.1009-0754.2021.01.001 (in Chinese) |
| [43] |
PANDA S. Circadian physiology of metabolism[J]. Science, 2016, 354(6315): 1008-1015. DOI:10.1126/science.aah4967 |
| [44] |
YANG X Y, DOWNES M, YU R T, et al. Nuclear receptor expression links the circadian clock to metabolism[J]. Cell, 2006, 126(4): 801-810. DOI:10.1016/j.cell.2006.06.050 |
(编辑 郭云雁)



