2. 山西省养殖技术试验基地,太原 030000
2. Shanxi Province Breeding Technology Test Base, Taiyuan 030000, China
共轭亚油酸(conjugated linoleic acids,CLA)是一类含有共轭双键的亚油酸异构体,主要以c9, t11-CLA和t10, c12-CLA这两种异构体为主[1]。CLA是食物中的天然成分,主要发现于反刍动物脂肪和牛奶产品中[2]。除天然来源以外,CLA还可以通过对亚油酸热异构化处理和部分氢化获得[3]。CLA具有广泛的生物学功能:调节脂类代谢,降低脂肪沉积、抗动脉粥样硬化、抗癌、抗氧化、提高免疫性能和改善骨骼健康等[4]。
边鸡主要分布在内蒙古自治区与山西省北部相毗连的长城内外一带,具有蛋重大、肉质好、适应性强、耐粗饲和抗寒等优点,是肉蛋兼用的中国地方品种。因其肉质鲜美,肉色红润而深受当地老百姓欢迎[5]。随着肉鸡体重增加,过量的腹部脂肪沉积会使胴体品质下降[6]。肉鸡胴体脂肪过多,生产中会给肉鸡加工企业带来较大的经济损失。降低鸡肉脂肪含量,生产优质鸡肉,既符合人们的健康需求,又促进了家禽产业的发展。据报道,在饲料中添加CLA可以抑制成脂分化进程和脂肪合成,下调与成脂分化和脂质合成相关基因的表达水平,从而降低体脂沉积,增加胴体瘦肉率,提高肉产品中CLA的含量,并改善肉品质[7-8]。Lavandera等[9]研究表明,在小鼠饲料中添加1% CLA能够降低FAS、ACC、SCD和SREBP-1c的基因表达水平,抑制脂肪沉积。Yeganeh等[10]在小鼠脂肪细胞中研究发现,CLA可以通过影响3T3-L1脂肪细胞中的Wnt/β-catenin通路来抑制脂肪细胞分化,从而抑制脂肪生成。Wang等[11]在猪饲粮中添加1.5% CLA并采集脂肪组织进行高通量测序,结果发现CLA可以通过调节Wnt信号通路来抑制脂肪组织中脂肪生成,调节体脂沉积。张天颖[12]用0、100、200 μmol·L-1 t10, c12-CLA处理山羊乳腺上皮细胞并进行转录组测序,共得到25 153条转录本,并通过对差异表达基因进行GO和KEGG富集分析,发现t10, c12-CLA可以抑制山羊乳腺上皮细胞脂肪酸合成以及固醇合成相关基因的表达,信号通路显著富在PPAR和AMPK通路中,从而调控脂代谢。骆娜[13]对黄羽肉鸡的胸肌组织和腹脂组织进行转录组测序,结果发现,GAPDH、LDHA、GPX1和GBE1等基因参与调控胸肌肌内脂肪沉积;FABP1、SCD和ADIPOQ等基因参与调控腹脂沉积。欧小倩[14]采集黄山黑鸡大腿肌肉组织进行转录组测序,共筛选出128个差异表达基因,其中94个基因上调,34个基因下调,对这些差异表达基因进行GO和KEGG分析,筛选出G0S2、FABP4、SCD等14个影响肌内脂肪含量的候选基因,发现12个GO条目和4个KEGG通路显著富集在脂肪代谢相关信号通路中。目前,人们在研究CLA影响动物机体脂肪沉积途径的分子机制中,对猪、羊、小鼠等的报道很多,但是对家禽相关的报道几乎没有。
本试验以边鸡为研究对象,在基础日粮中添加不同比例的CLA进行饲养试验,通过转录组测序技术筛选出与边鸡胸肌脂类代谢相关的主要调控基因,对差异表达基因进行GO功能和KEGG通路富集分析,为揭示CLA影响边鸡胸肌脂类代谢的分子作用机制提供理论依据。
1 材料与方法 1.1 试验动物及样品采集试验动物在山西省养殖技术试验基地进行饲养,选取90日龄健康边鸡180只,随机分成5组,每组3个重复,分别添加不同比例的CLA 0%(对照组)、0.5%、1.0%、1.5%和2.0%。对照组添加豆油,试验组分别在基础日粮中以等量的CLA替换豆油,添加豆油以确保各组饲料中能量相同,CLA购自青岛澳海生物有限公司(CLA含量为80%)。试验预饲期1周,正饲期6周。基础日粮参照《鸡饲养标准》(NY/T 33-2004)配制,基础日粮组成及营养成分见表 1。饲养试验结束后,每组随机选取6只边鸡进行屠宰取样,颈静脉放血法处死,采集胸肌组织,立刻投入液氮速冻,后转入-80 ℃保存。
|
|
表 1 基础日粮组成及营养成分 Table 1 Composition and nutrient levels of basal diet |
称取边鸡胸肌组织50~100 mg,采用Trizol法提取组织总RNA。用Agilent 2100生物分析仪进行总RNA品质检测。用带有Oligo(dT)的磁珠富集真核生物mRNA;加入破碎缓冲液将mRNA随机打断;以mRNA为模板,用六碱基随机引物合成第一条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA polymerase I合成第二条cDNA链;纯化cDNA再进行末端修复、加A尾并连接测序接头,进行片段大小选择;最后通过PCR富集得到cDNA文库。库检合格后,用Illumina平台进行测序,由北京百迈客生物科技有限公司完成文库构建及测序。
1.3 转录组生物信息学分析将原始数据(Raw Data)去除含有接头的Reads和低质量的Reads(含N比例>10%的Reads和质量值Q≤10的碱基数占整条Read的50%以上的Reads)得到高质量的Clean Data。使用HISAT2软件[15]将Clean Data与指定的参考基因组进行序列比对。基于所选参考基因组序列,使用StringTie软件[16]与原有的基因组注释进行比较,挖掘新基因;根据基因在不同样品中的表达量进行差异表达分析。
1.4 差异表达基因(DEGs)分析StringTie通过最大流量算法, 采用FPKM(Fragments Per Kilobase of transcript per Million fragments mapped)作为衡量基因表达水平的指标,FPKM是将Map到基因的Fragments数除以Map到Genome的所有Read数(以Million为单位)与RNA的长度(以KB为单位)。使用DESeq2软件[17]进行差异表达分析。在DEGs检测过程中,将Fold Change≥1.5且P value < 0.05作为筛选标准。差异倍数(Fold Change)表示两样品间表达量的比值。
1.5 GO和KEGG功能注释和富集分析利用GO(Gene Ontology database)数据库和KEGG(The database of Kyoto Encyclopedia of Genes and Genomes)数据库对DEGs进行GO功能分类和信号通路富集分析。
1.6 实时荧光定量PCR(qPCR)验证为保证转录组测序结果的准确性,随机选取9个DEGs进行qPCR验证。选择β-actin作为内参基因,用Oligo 7软件设计引物,交由华大基因合成,引物序列见表 2。选择TB GreenⓇ Premix Ex TaqTM Ⅱ试剂盒(Code:RR820A)进行qPCR验证,反应总体系10 μL:TB Green Premix Ex Taq Ⅱ 5.0 μL,上、下游引物各0.2 μL,cDNA 1.0 μL,ddH2O 3.6 μL。反应条件:95 ℃,30 s;95 ℃ 5 s,58 ℃ 30 s,45个循环;65 ℃ 5 s,95 ℃ 30 s。使用2-ΔΔCt法计算目的基因的相对表达量。
|
|
表 2 实时荧光定量PCR引物 Table 2 Primers for quantitative real-time PCR |
通过百迈客云平台BMK Cloud对添加不同比例CLA的边鸡胸肌样本进行转录组测序,共得到68.97 Gb Clean Data,各样品Clean Data均达到5.36 Gb。GC碱基含量在52.36%~56.56%之间;Q20碱基百分比≥96.84%;Q30碱基百分比≥92.30%(表 3)。上述结果表明,转录组测序数据质量高,可用于后续试验进一步分析。
|
|
表 3 测序数据统计表 Table 3 The statistical table of sequencing data |
采用HISAT2软件将过滤后各样品的Clean Reads与指定的参考基因组进行序列比对。结果显示,各样品转录组测序的总片段数分别在35~72百万对之间。各样品的Reads与参考基因组的比对效率在77.72%~85.81%之间;比对到参考基因组唯一位置的Reads数目及在Clean Reads中占的比例在72.15%~82.61%;比对到参考基因组多处位置的Reads数目及在Clean Reads中占的比例在2.85%~6.58%之间;比对到参考基因组正链的Reads数目比例在38.78%~42.82%;比对到参考基因组负链的Reads数目比例在38.89%~42.95%。上述结果表明测序数据符合试验要求,可以进行后续分析(表 4)。
|
|
表 4 转录组测序数据与参考基因组比对结果统计表 Table 4 Statistics of comparison results between transcriptome sequencing data and reference genome |
用所选参考基因组序列与原有的基因组注释信息进行比较,共发掘3 550个新基因。使用DIAMOND软件[18]将发掘的新基因与GO、KEGG等数据库进行序列比对,共有783个新基因得到功能注释。皮尔逊相关系数r(Pearson’s Correlation Coefficient)是生物学重复相关性的评估指标,r2越接近1,说明两个重复样品相关性越强(图 1)。从图 1中看出,不同试验组中的2个重复样品的r2均大于0.9,说明样品重复性较好,试验可靠性高。
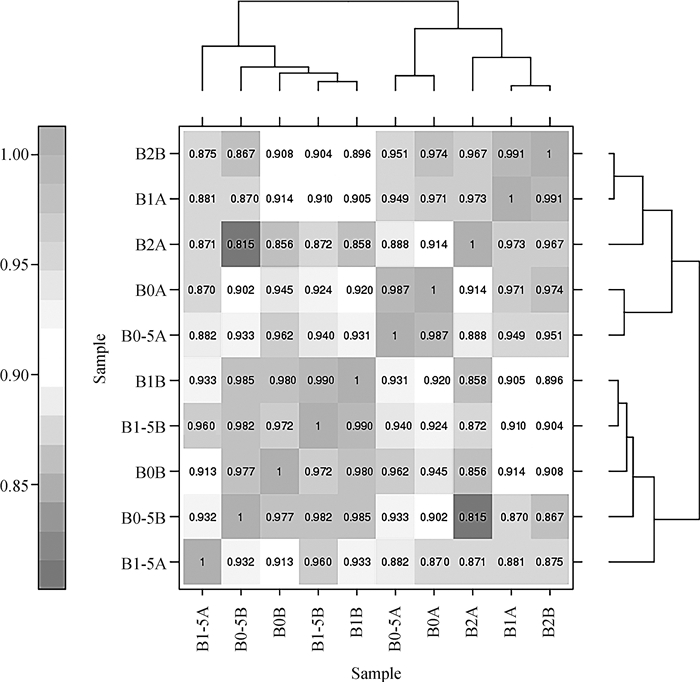
|
图 1 不同样品间的皮尔逊相关分析 Fig. 1 Pearson correlation analysis between different samples |
使用DESeq2软件对转录组测序数据进行差异表达分析,将Fold Change≥1.5且P value < 0.05作为筛选标准。结果共筛选出DEGs 1 229个,其中上调基因594个,下调基因635个(表 5)。
|
|
表 5 差异表达基因数目统计表 Table 5 Statistical of the DEGs number |
通过火山图(volcano plot)可以快速地查看基因在两组样品中表达水平的差异,以及差异的统计学显著性(图 2)。火山图横坐标绝对值越大,说明表达量在两样品间的表达量倍数差异越大;纵坐标值越大,表明差异表达越显著,筛选得到的DEGs越可靠。
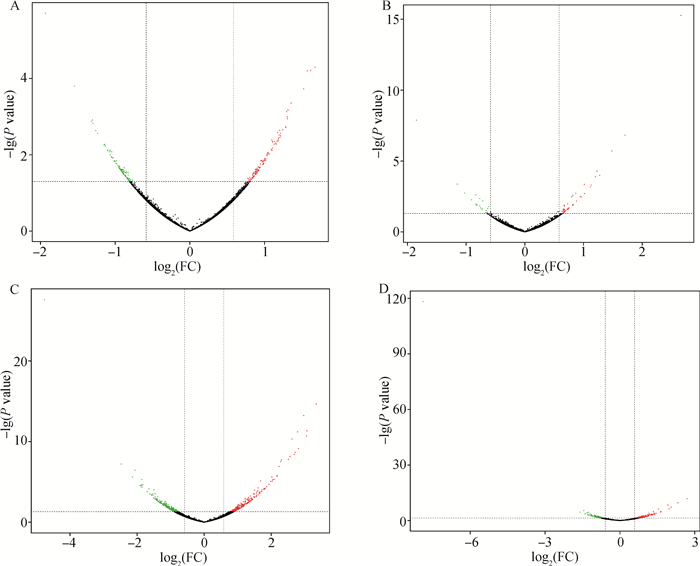
|
A. 0 vs.0-5;B. 0 vs.1;C. 0 vs.1-5;D. 0 vs.2(下同)。图中每一个点表示一个基因,绿点代表下调差异表达基因,红点代表上调差异表达基因,黑点代表非差异表达基因 A. 0 vs.0-5; B. 0 vs.1; C. 0 vs.1-5; D. 0 vs.2 (the same as below). Each point in the figure represents a gene, the green dots represent down-regulation of DEGs, red dots represent up-regulation of DEGs, black dots represent non-DEGs 图 2 对照组与不同处理组间差异表达基因火山图 Fig. 2 Volcano plot of DEGs between the control group and different treatment groups |
运用GO数据库进行功能富集分析。结果显示(图 3),共有1 045个DEGs被GO数据库注释。DEGs在生物学过程中主要参与细胞过程(cellular process)、单一生物过程(single-organism process)、生物调节(biological regulation)和代谢过程(metabolic process)等功能;在分子功能中主要参与结合(binding)和催化活性(catalytic activity)等功能;在细胞组分中主要参与细胞(cell)、细胞部分(cell part)、细胞器官(organelle)和膜(membrane)等功能。
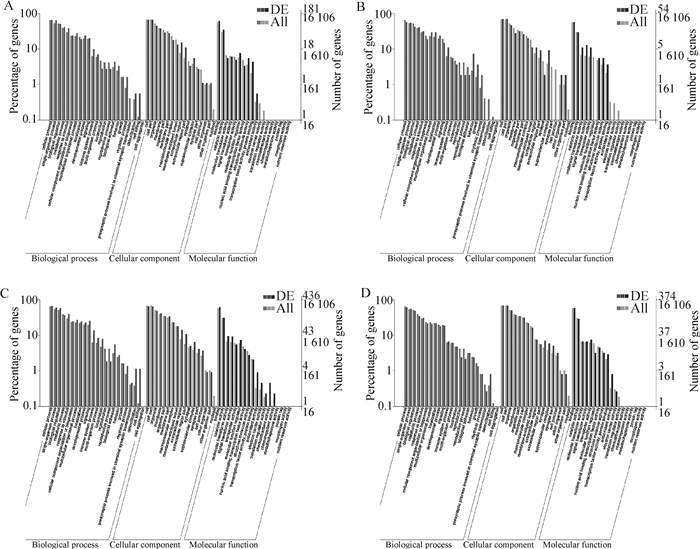
|
横坐标为GO功能分类; 纵坐标左边为基因数目所占百分比, 右边为基因数目 The abscissa is the GO function classification; The left side of the ordinate is the percentage of the number of genes, the number of genes on the right 图 3 差异表达基因GO功能分类统计图 Fig. 3 GO function classification statistics map of DEGs |
运用KEGG数据库进行差异表达基因的通路富集分析。结果显示,共有989个DEGs被KEGG注释。这些DEGs共富集到389条信号通路,选取每组显著性q value值最小的前20个通路作散点图(图 4)。从图中可以看出,添加0.5% CLA时,DEGs显著富集在ECM-受体相互作用(ECM-receptor interaction)和黏着斑(focal adhesion)等通路中;添加1% CLA时,DEGs显著富集在吞噬体(phagosome)、细胞凋亡(apoptosis)和ECM-受体相互作用(ECM-receptor interaction)等通路中;添加1.5% CLA时,DEGs显著富集在吞噬体(phagosome)、肌动蛋白细胞骨架调节(regulation of actin cytoskeleton)和细胞因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction)等通路中;添加2% CLA时,DEGs在信号通路中不显著富集。
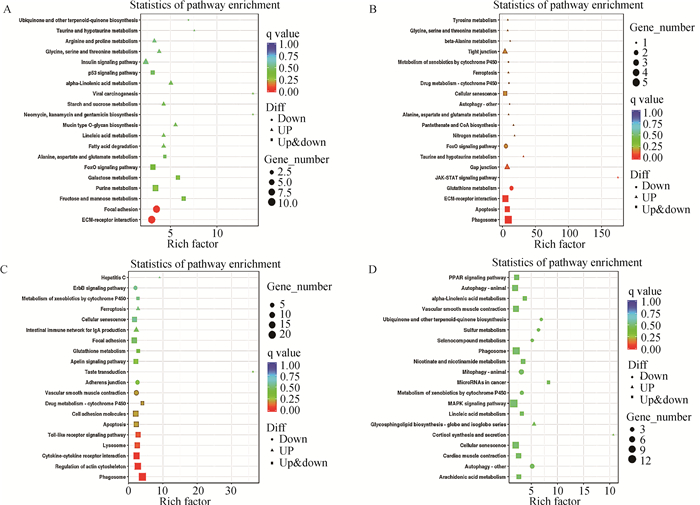
|
纵坐标表示通路名称,横坐标为富集因子。圆圈颜色代表q value值,圆圈的大小表示通路中富集的基因数目 The ordinate represents the name of the pathway, the abscissa represents the enrichment factor. The color of the circle represents the q value, the size of the circle indicates the number of genes enriched in the pathway 图 4 差异表达基因KEGG通路富集散点图 Fig. 4 KEGG pathway scatter plot of DEGs enrichment |
通过GO和KEGG功能注释分析,共筛选出18个与边鸡胸肌脂类代谢相关的DEGs(表 6)。从表 6可以看出,在基础日粮中添加1.5% CLA时有8个基因的表达量与对照组的表达量相比显著上调(P<0.05),添加0.5%、1%和2%的CLA时基因表达量与对照组相比差异不显著。添加0.5% CLA时有5个基因的表达量与对照组相比显著上调(P<0.05),添加1%、1.5%和2%的CLA时基因表达量与对照组相比差异不显著。有3个基因添加2%的CLA时表达量与对照组相比显著下调(P<0.05),添加0.5%、1%和1.5%的CLA时差异不显著。有2个基因添加1.5%的CLA时表达量与对照组相比显著下调(P<0.05),添加0.5%、1%和2%的CLA时差异不显著。
|
|
表 6 边鸡胸肌中与脂类代谢相关的差异表达基因 Table 6 DEGs related to lipid metabolism in breast muscle of Bian chicken |
为进一步验证转录组测序结果的准确性,随机选取9个DEGs进行qPCR验证(图 5)。结果表明,这9个基因的mRNA表达量变化趋势与转录组测序结果一致,说明本试验转录组测序结果准确性好。
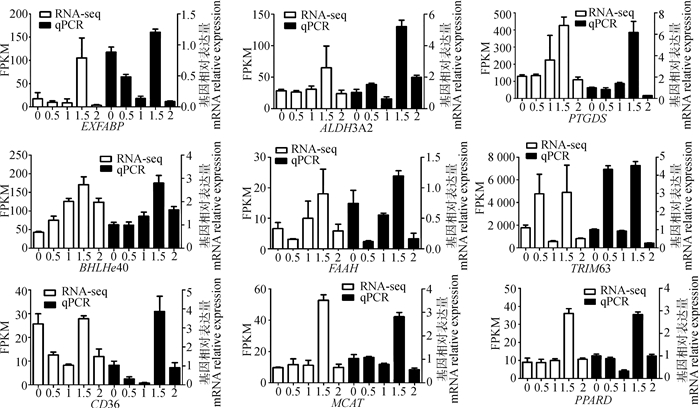
|
横坐标表示5个试验组 The abscissa represents 5 test groups 图 5 差异表达基因测序结果和qPCR验证 Fig. 5 DEGs sequencing results and qPCR validation |
转录组测序是利用高通量测序技术将细胞或组织中全部或部分mRNA和非编码RNA进行测序分析的技术[19]。CLA具有调节脂类代谢的功能,通过调控参与脂类代谢的相关基因,降低脂肪沉积[20]。本研究通过转录组测序,共鉴定出3 550个新基因,其中783个新基因得到功能注释。各试验组中的2个重复样品间的皮尔逊相关系数大于0.9,表明试验结果可靠。使用DESeq2软件对测序数据进行差异性分析,共筛选出DEGs 1 229个,其中上调基因594个,下调基因635个。随机选取9个DEGs进行qPCR验证,其基因表达量变化趋势与转录组测序结果一致,表明转录组测序结果可靠。
通过对DEGs的筛选,共筛选出18个主要参与边鸡胸肌脂类代谢的DEGs。试验组基础日粮中添加CLA有13个DEGs表达量上调,PPARD、DGAT2、PTGDS、MCAT、FAAH、APOA1、ALDH3A2、PLTP、FABP3、FOXO1、UCP3、INSIG1和DHCR24已被发现参与调控脂肪代谢。1.5% CLA试验组中有5个基因(MCAT、APOA1、PTGDS、ALDH3A2和PLTP)的表达量差异极显著。MCAT是脂肪酸合成酶(FAS)Ⅱ脂肪酸代谢途径中FAS复合体组成酶之一,在脂肪酸合成途径中起装载作用[21]。有研究表明,MCAT参与了脂肪酸代谢过程,被认为是脂肪酸代谢途径的结合点[22-23]。APOA1是高密度脂蛋白(HDL)的主要载脂蛋白,占蛋白总量的60%~70%[24]。载脂蛋白是一种具有脂类转运功能的血浆蛋白质,在脂质代谢和脂质运输等过程中发挥重要的做作用[25]。PTGDS是一种谷胱甘肽非依赖性的前列腺素合成酶,能够催化前列腺素H2(PGH2)转化为前列腺素D2(PGD2),前列腺素是脂肪酸代谢的终末产物,参与机体正常生理过程[26]。ALDH3A2是醛脱氢酶家族成员之一,催化长链脂肪醛氧化为脂肪酸,调控脂质代谢[27]。PLTP介导脂蛋白之间的磷脂转运,参加脂质和脂蛋白代谢[28]。0.5% CLA试验组中有3个基因(FABP3、FOXO1和UCP3)的表达量差异极显著。FABP3属于脂肪酸结合蛋白家族,主要调节脂肪酸的吸收和代谢[29]。FOXO1是叉头蛋白框O亚族(FOXO)成员之一,可以降低脂肪酸氧化,调控脂质代谢[30]。UCP3是解偶联蛋白(UCPs)家族成员之一,能够调节脂肪酸代谢,运输脂肪酸和脂质过氧化物[31]。Ribot等[32]研究发现,饲料中添加1% CLA使UCP3 mRNA表达水平升高,调节脂肪酸代谢,降低脂肪沉积。本试验结果表明,基础日粮中添加CLA使一些主要调控脂类代谢的基因表达量上调,5个基因在1.5% CLA试验组表达量极显著上调,已发现这5个基因主要参与脂肪酸合成,在脂肪酸的合成和代谢途径中起装载和结合点作用,参与脂质运输、催化前列腺素合成、催化长链脂肪醛氧化为脂肪酸以及调节脂蛋白代谢;3个基因在0.5% CLA试验组表达量上调且差异极显著,已发现这3个基因主要参与脂肪酸的吸收、氧化和运输。本试验结果发现,基础日粮中添加1.5%和0.5% CLA可能会对胸肌脂类代谢调控起主要作用,具体调控机理有待进一步研究。从DEGs的调控功能发现,1.5% CLA可能调控边鸡胸肌脂肪酸的富集。试验组基础日粮中添加CLA有5个DEGs表达量下调,PDK4、FABP4、ACACB、MEF2C和CMYA5已被发现参与调控脂肪代谢。2% CLA试验组中FABP4的基因表达量差异极显著,FABP4属于脂肪酸结合蛋白家族,主要在脂肪细胞中表达,负责脂肪酸的合成和运输[33]。O’Reilly等[34]研究发现,给小鼠饲喂含CLA的饲料能够降低FABP4表达水平,从而降低脂肪沉积,减少肥胖发生。本试验结果发现,在基础日粮中添加2%的CLA可极显著降低FABP4基因表达,从而影响胸肌脂肪酸代谢,具体调控机理有待进一步研究。本研究对DEGs的筛选发现各个试验组中出现不同的差异显著的DEGs,揭示了基础日粮中添加CLA影响边鸡胸肌DEGs表达水平,进而调控脂肪代谢,为今后相关研究提供理论依据。
对DEGs的GO功能富集分析,发现各试验组边鸡胸肌组织中DEGs主要集中在生物学过程的细胞过程、单一生物过程、生物调节和代谢过程,其次集中在分子功能和细胞组分。GO功能富集分析发现,DEGs可能主要在生物学过程中参与调控脂类代谢,其调控机制有待研究。对各试验组差异表达基因的KEGG通路富集分析发现,各试验组DEGs所富集的信号通路不同,表明基础日粮中CLA含量影响ECM-受体相互作用、黏着斑、吞噬体、细胞凋亡、肌动蛋白细胞骨架调节和细胞因子-细胞因子受体相互作用等通路功能的实现,可能参与脂类代谢调控,其作用机制有待研究。
4 结论本试验探究了在边鸡基础日粮中添加CLA对胸肌脂类代谢影响的研究,利用转录组测序技术筛选出18个与边鸡胸肌脂类代谢相关的差异表达基因,其中MCAT、APOA1、PTGDS、ALDH3A2、PLTP、FABP3、FOXO1、UCP3和FABP4可能在调节边鸡胸肌脂肪代谢过程中发挥了重要作用,为今后揭示CLA调节边鸡胸肌脂类代谢的分子作用机制提供了理论依据。
| [1] |
WANG S H, WANG W W, ZHANG H J, et al. Conjugated linoleic acid regulates lipid metabolism through the expression of selected hepatic genes in laying hens[J]. Poult Sci, 2019, 98(10): 4632-4639. DOI:10.3382/ps/pez161 |
| [2] |
司倩倩, 刘斌, 史雪萍, 等. 共轭亚油酸的生理功能及其在动物生产中的应用[J]. 饲料博览, 2015(1): 26-30. SI Q Q, LIU B, SHI X P, et al. Physiological functions of conjugated linoleic acid and application in animal production[J]. Feed Review, 2015(1): 26-30. DOI:10.3969/j.issn.1001-0084.2015.01.008 (in Chinese) |
| [3] |
杨媚, 马杰, 杨泰, 等. 共轭亚油酸的生物学功能及其在动物生产中的应用[J]. 中国畜牧兽医, 2019, 46(11): 3216-3224. YANG M, MA J, YANG T, et al. Biological function of conjugated linoleic acid and its application in animal production[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(11): 3216-3224. (in Chinese) |
| [4] |
IBRAHIM K S, EL-SAYED E M. Dietary conjugated linoleic acid and medium-chain triglycerides for obesity management[J]. J Biosci, 2021, 46(1): 12. DOI:10.1007/s12038-020-00133-3 |
| [5] |
张俊珍, 李彩娥, 张蒙, 等. 催乳素及其受体在右玉边鸡卵巢中的表达及与就巢性的相关性研究[J]. 中国畜牧杂志, 2020, 56(4): 51-55. ZHANG J Z, LI C E, ZHANG M, et al. Expression of PRL and PRLR in ovary of Youyu Bian chicken and the correlation with broodiness[J]. Chinese Journal of Animal Science, 2020, 56(4): 51-55. (in Chinese) |
| [6] |
FU C Y, ZHANG Y, WANG W B, et al. Supplementing conjugated linoleic acid (CLA) in breeder hens diet increased CLA incorporation in liver and alters hepatic lipid metabolism in chick offspring[J]. Br J Nutr, 2021, 4: 1-41. |
| [7] |
伏春燕, 张燕, 魏祥法, 等. 共轭亚油酸降低脂肪沉积的分子机制研究进展[J]. 动物营养学报, 2019, 31(8): 3456-3462. FU C Y, ZHANG Y, WEI X F, et al. Advances in molecular mechanisms of conjugated linoleic acid-reduced fat deposition[J]. Chinese Journal of Animal Nutrition, 2019, 31(8): 3456-3462. (in Chinese) |
| [8] |
SHEN W, MCINTOSH M K. Nutrient regulation: conjugated linoleic acid's inflammatory and browning properties in adipose tissue[J]. Annu Rev Nutr, 2016, 36: 183-210. DOI:10.1146/annurev-nutr-071715-050924 |
| [9] |
LAVANDERA J, GERSTNER C D, SAÍN J, et al. Maternal conjugated linoleic acid modulates TAG metabolism in adult rat offspring[J]. Br J Nutr, 2017, 118(11): 906-913. DOI:10.1017/S0007114517003002 |
| [10] |
YEGANEH A, TAYLOR C G, POOLE J, et al. Trans10, cis12 conjugated linoleic acid inhibits 3T3-L1 adipocyte adipogenesis by elevating β-catenin levels[J]. Biochim Biophys Acta, 2016, 1861(4): 363-370. DOI:10.1016/j.bbalip.2016.01.004 |
| [11] |
WANG Q, QI R L, LIU H, et al. Effects of conjugated linoleic acid supplementation on the expression profile of miRNAs in porcine adipose tissue[J]. Genes (Basel), 2017, 8(10): 271. DOI:10.3390/genes8100271 |
| [12] |
张天颖. 反10、顺12共轭亚油酸(Trans 10, Cis 12-CLA)对奶山羊乳腺上皮细胞脂代谢的调控作用研究[D]. 杨凌: 西北农林科技大学, 2018. ZHANG T Y. Regulation mechanism of Trans 10, Cis 12 conjugated linoleic acid (T10C12-CLA) in lipid metabolism of mammary epithelial cells on dairy goat[D]. Yangling: Northwest A&F University, 2018. (in Chinese) |
| [13] |
骆娜. 鸡肌内脂肪和腹脂差异沉积的分子机理解析[D]. 北京: 中国农业科学院, 2021. LUO N. Molecular mechanism of differential deposition of intramuscular fat and abdominal fat in chicken[D]. Beijing: Chinese Academy of Agricultural Sciences, 2021. (in Chinese) |
| [14] |
欧小倩. G0S2基因对黄山黑鸡肌内脂肪调控机制及冷鲜鸡肉保鲜技术的研究[D]. 合肥: 合肥工业大学, 2019. OU X Q. Study on regulation mechanism of GOS2 gene on intramuscular fat of Huangshan black chicken and the preservation technology of chilled chicken[D]. Hefei: Hefei University of Technology, 2019. (in Chinese) |
| [15] |
KIM D, LANGMEAD B, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nat Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317 |
| [16] |
PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290-295. DOI:10.1038/nbt.3122 |
| [17] |
LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biol, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 |
| [18] |
BUCHFINK B, XIE C, HUSON D H. Fast and sensitive protein alignment using DIAMOND[J]. Nat Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [19] |
崔凯, 吴伟伟, 刁其玉. 转录组测序技术的研究和应用进展[J]. 生物技术通报, 2019, 35(7): 1-9. CUI K, WU W W, DIAO Q Y. Application and research progress on transcriptomics[J]. Biotechnology Bulletin, 2019, 35(7): 1-9. (in Chinese) |
| [20] |
刘丽娜, 缪锦来, 郑洲. 共轭亚油酸的生理功能综述[J]. 食品安全质量检测学报, 2020, 11(8): 2552-2557. LIU L N, MIAO J L, ZHENG Z. Review on the physiological function of conjugated linoleic acid[J]. Journal of Food Safety & Quality, 2020, 11(8): 2552-2557. (in Chinese) |
| [21] |
王建勇. 油茶种子脂肪酸代谢过程7个关键酶基因的克隆与功能研究[D]. 长沙: 中南林业科技大学, 2014. WANG J Y. The study on cloning and function of seven genes involved in the fatty acid metabolism of Camellia oleifera seeds[D]. Changsha: Central South University of Forestry and Technology, 2014. (in Chinese) |
| [22] |
HONG S K, KIM K H, KIM E E. Cloning, purification, crystallization and preliminary X-ray crystallographic analysis of MCAT from Staphylococcus aureus[J]. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2010, 66(1): 20-22. DOI:10.1107/S1744309109045989 |
| [23] |
HONG S K, KIM K H, PARK J K, et al. New design platform for malonyl-CoA-acyl carrier protein transacylase[J]. FEBS Lett, 2010, 584(6): 1240-1244. DOI:10.1016/j.febslet.2010.02.038 |
| [24] |
刘培彬, 叶子璐, 蔡潭溪, 等. 脂蛋白质组学研究进展[J]. 生物化学与生物物理进展, 2014, 41(12): 1197-1206. LIU P B, YE Z L, CAI T X, et al. The research advances in lipoproteomics[J]. Progress in Biochemistry and Biophysics, 2014, 41(12): 1197-1206. (in Chinese) |
| [25] |
夏斌, 刘天宇, 孙忠东. 载脂蛋白与急性心肌梗死的相关性研究进展[J]. 医学综述, 2021, 27(17): 3394-3400. XIA B, LIU T Y, SUN Z D. Research progress in relationship between apolipoprotein and acute myocardial infarction[J]. Medical Recapitulate, 2021, 27(17): 3394-3400. DOI:10.3969/j.issn.1006-2084.2021.17.012 (in Chinese) |
| [26] |
陈志贤, 何凯, 黄煜, 等. PTGDS在前列腺癌中的表达及其与AR的相关性研究[J]. 临床泌尿外科杂志, 2021, 36(5): 371-376, 381. CHEN Z X, HE K, HUANG Y, et al. Expression of PTGDS in prostate cancer and its correlation with AR[J]. Journal of Clinical Urology, 2021, 36(5): 371-376, 381. (in Chinese) |
| [27] |
RAJESHWARI M, KARTHI S, SINGH R, et al. Novel ALDH3A2 mutations in structural and functional domains of FALDH causing diverse clinical phenotypes in Sjögren-Larsson syndrome patients[J]. Hum Mutat, 2021, 42(8): 1015-1029. DOI:10.1002/humu.24236 |
| [28] |
王瑾. 开心散调节脂质代谢紊乱发挥抗抑郁作用及其关键靶点的研究[D]. 太原: 山西中医药大学, 2020. WANG J. KXS regulate lipid metabolic disorders and study its key targets to play antidepressant effect[D]. Taiyuan: Shanxi University of Traditional Chinese Medicine, 2020. (in Chinese) |
| [29] |
BI Y L, YUAN X Y, ZHU P F, et al. A novel long noncoding RNA, ENSGALG00000021686, regulates the intracellular transport of fatty acids by targeting the FABP3 gene in chicken[J]. Biochem Biophys Res Commun, 2020, 528(4): 706-712. DOI:10.1016/j.bbrc.2020.05.133 |
| [30] |
LIU X J, QIAO A J, KE Y J, et al. FoxO1 represses LXRα-mediated transcriptional activity of SREBP-1c promoter in HepG2 cells[J]. FEBS Lett, 2010, 584(20): 4330-4334. DOI:10.1016/j.febslet.2010.09.027 |
| [31] |
范一萍, 王彦芳, 陶聪. UCP3基因在巴马猪和藏猪脂肪组织中的表达和甲基化分析[J]. 中国畜牧兽医, 2019, 46(12): 3627-3634. FAN Y P, WANG Y F, TAO C. Expression and methylation analysis of UCP3 gene in adipose tissue of Bama and Tibetan pigs[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(12): 3627-3634. (in Chinese) |
| [32] |
RIBOT J, PORTILLO M P, PICÓ C, et al. Effects of trans-10, cis-12 conjugated linoleic acid on the expression of uncoupling proteins in hamsters fed an atherogenic diet[J]. Br J Nutr, 2007, 97(6): 1074-1082. DOI:10.1017/S0007114507682932 |
| [33] |
公绪同. 肌内脂肪沉积基因PPARγ2、FABP4、DGAT1在牛肌卫星细胞中过表达对脂肪沉积影响的比较[D]. 杨凌: 西北农林科技大学, 2020. GONG X T. Comparison of overexpression of PPARγ2, FABP4 and DGAT1 in bovine muscle satellite cells[D]. Yangling: Northwest A&F University, 2020. (in Chinese) |
| [34] |
O'REILLY M E, LENIGHAN Y M, DILLON E, et al. Conjugated linoleic acid and alpha linolenic acid improve cholesterol homeostasis in obesity by modulating distinct hepatic protein pathways[J]. Mol Nutr Food Res, 2020, 64(7): 1900599. DOI:10.1002/mnfr.201900599 |
(编辑 郭云雁)



