2. 青海省高原家畜遗传资源保护与创新利用重点实验室,西宁 810016;
3. 西北农林科技大学动物科技学院,杨凌 712100;
4. 青海省格尔木市畜牧兽医站,格尔木 816000;
5. 青海省海西州农业技术推广服务中心,德令哈 817099
2. Key Laboratory of Plateau Livestock Genetic Resources Protection and Innovative Utilization, Xining 810016, China;
3. College of Animal Science and Technology, Northwest A&F University, Yangling 712100, China;
4. Golmud Animal Husbandry and Veterinary Station in Qinghai, Golmud 816000, China;
5. Haixi Agricultural Technology Extension Service Center in Qinghai, Delingha 817099, China
牦牛(Bos grunniens)是青藏高原及其毗邻高地十分重要的种质资源,为高海拔游牧民族提供主要的生产、生活资料和经济支撑[1-2]。当前,国家畜禽遗传资源委员会认定的我国牦牛品种(遗传资源)有20个,其中地方品种(遗传资源)18个,培育品种2个[3]。青海省地处“世界屋脊”青藏高原东北部,牦牛遗传资源十分丰富,拥有牦牛500多万头,包括4个地方品种(即青海高原牦牛、环湖牦牛、雪多牦牛和玉树牦牛)、2个培育品种(即大通牦牛和阿什旦牦牛)和少量的野牦牛[3]。线粒体基因组(mitochondrial genome,mitogenome)是一种环状DNA分子,进化速率快、母系遗传、不易发生重组且多态性丰富,故被认为是开展动物母系遗传多样性、群体遗传结构及分类等研究的理想工具[4-5]。牦牛线粒体基因组群体遗传研究将为更准确地揭示各品种(群体)的母系遗传多样性水平、群体结构组成及遗传关系等问题提供重要信息,为后续有效开展其遗传资源的保护和选育实践提供理论基础。当前,研究者已基于Y-SNP和Y-STR分子标记对青海的9个牦牛品种(群体)以及野牦牛的父系遗传多样性、群体遗传结构及系统发育关系等进行了较为系统的分析,表明青海牦牛父系遗传多样性丰富,由2个父系支系组成[6-9]。然而,在母系遗传研究方面,虽然先前基于mtDNA D-loop区和Cyt b等基因核苷酸序列变异对野牦牛及青海地方牦牛品种的研究较多[9-33],但基于线粒体基因组全序列变异的研究相对较少[9, 18, 34]。在野牦牛和青海牦牛线粒体基因组研究方面,Wang等[18]对包括21头青海牦牛(即大通、环湖、青海高原和玉树牦牛)在内的51头家牦牛和21头野牦牛线粒体基因组序列进行综合分析,表明野牦牛的母系遗传多样性高于家牦牛,牦牛由3个高度分化的遗传分支组成,其中2大分支包含野牦牛和家牦牛的单倍型,其余1个分支仅含有野牦牛的单倍型。最近,Wang等[34]对青海省4个牦牛品种(群体)(即雪多、环湖、祁连和玉树牦牛)共86头个体线粒体基因组进行了测定并对其遗传多样性及系统发育进行了分析,表明雪多牦牛的单倍型多样度最高(0.992±0.015),而环湖牦牛的单倍型多样度最低(0.905±0.048);揭示牦牛由3个不同的母系支系组成,且发现雪多牦牛和野牦牛少量个体共存于第3个母系分支。
综上所述,研究者已利用Y染色体分子标记较为系统地开展了野牦牛及青海地方牦牛品种(群体)的父系遗传研究[6-9]。但在其母系遗传研究中,尽管当前已有少量基于线粒体基因组全序列变异对野牦牛及青海部分牦牛品种(群体)的报道[9, 18, 34],但其所分析的各品种个体数量较少、采样点分布不多且缺乏系统性比较研究。此外,青海部分地方牦牛品种(如青海高原牦牛和玉树牦牛)的分布区与野牦牛生存地毗邻,存在家、野牦牛窜群现象,故探究各地方牦牛品种与野牦牛之间的关系也显得尤为重要。然而,已有研究均未能系统深入地反映野牦牛及青海各地方牦牛品种间的母系遗传多样性差异、分化状况、遗传关系等问题。鉴于此,本研究对野牦牛及青海4个地方牦牛品种进行线粒体基因组全序列比较和系统分析,探究其母系遗传多样性水平、分化状况、聚类关系及系统发育情况,为深入了解野牦牛及青海地方牦牛品种遗传资源状况及彼此之间的遗传关系提供基础材料,并为后续开展保种和选育研究奠定基础。
1 材料与方法 1.1 样品采集、基因组DNA提取和序列数据收集在青海省格尔木市、共和县、同德县、河南县和曲麻莱县采集青海高原牦牛、环湖牦牛、雪多牦牛、玉树牦牛颈静脉血样3、10、5和4份(表 1),共计22份血样经ACD抗凝后放入带有冰袋的保温箱中带回实验室,-80 ℃保存备用。采样前询问牧户有关牦牛群的选育历史背景信息并查阅系谱记录情况,确保样品间无亲缘关系且具有代表性。采用血液DNA提取试剂盒(北京艾莱德生物科技有限公司)提取基因组DNA,使用琼脂糖凝胶电泳和NanoDrop 2000超微量分光光度计检测基因组DNA的浓度与纯度,当样品的质量控制参数大于100 ng·μL-1,OD260nm/OD280nm比值为1.7~1.9时,表明DNA的质量较高,可用于后续基因组重测序。此外,本研究从GenBank中下载已报道的所有野牦牛和青海省4个地方牦牛品种线粒体基因组序列共142条,结合本试验中测定的22条青海牦牛线粒体基因组序列,对共计164条野牦牛和青海地方牦牛品种线粒体基因组序列进行数据综合分析(表 1)。
|
|
表 1 野牦牛及青海地方牦牛品种线粒体基因组序列信息来源 Table 1 Sources of mitogenome information of wild yak and local yak breeds in Qinghai |
1.2.1 主要试剂 DL2000 DNA Marker、2×Taq PCR MasterMix、10×Loading Buffer、核酸染料和50×TAE电泳缓冲液均购自上海宝生物工程有限公司。
1.2.2 主要仪器 迷你离心机(Super Mini Dancer)购自上海生工生物工程股份有限公司;0~2.5、0.5~10、10~100、20~200和100~1 000 μL微量移液器和PCR仪(P70)购于德国Eppendorf公司;电泳仪(DYY-12)购于北京六一仪器厂;超净工作台(PCR3A-1)购于上海艺斯高贸易有限公司;超纯水仪(密理博)购于德国默克公司;-80 ℃超低温冰柜(8525)购于美国Forma Scintific公司;凝胶成像分析系统(6300)购于上海天能仪器公司;涡旋混合器(WH-861)购于太仓华利达实验设备有限公司;电子天平(AB204-E)购于梅特勒-托利多集团;高压蒸气自动灭菌锅(LDZ-30KBS)购于上海申安公司;大数据计算机数据处理平台来源于西北农林科技大学的High-Performance Computing (HPC)。
1.3 牦牛基因组重测序及全基因组序列的组装获取将22头青海地方牦牛品种个体的基因组DNA送往北京诺禾致源科技股份有限公司进行全基因组重测序,基于Illunina NOVA 6 000测序平台进行双末端建库,reads的长度为2×150 bp。将原始测序数据Raw reads用Trimmomatic v0.36软件进行质检,去除低质量序列片段和接头序列,获得牦牛各个体的基因组clean reads。具体流程为:1)修剪掉序列双端质量得分小于20的碱基和N碱基(Leading: 20、Trailing: 20);2)自5′端以3 bp滑动窗口进行检测,平均质量值小于15的进行截断(Slidingwindow: 3:15);3)去除平均碱基质量值低于20的序列片段(Avgqual: 20);4)去除修剪后长度小于35 bp的序列片段(Minlen: 35);5)碱基质量值体系设置为phred-33(Tophred 33)。使用BWA-MEM v0.7.15软件将质控后的clean reads比对到牦牛的参考基因组(序列号: VBZB00000000, BosGru3.0)上,组装成完整的牦牛基因组序列。分别使用samtools v0.1.19和picard v2.5.0软件进行bam文件排序和序列PCR去重,用qualimap软件统计所有样本的测序深度。
1.4 牦牛线粒体基因组全序列的提取为了提高获取的线粒体基因组序列完整性,将参考线粒体基因组(登录号:NC_006380)首端和末端各添加可识别的30 bp序列,先使用Burrows-Wheeler Aligner v0.7.15软件将本研究中的22条牦牛全基因组序列与参考线粒体基因组序列进行比对,再使用samtools v0.1.19软件将比对结果转换为BAM文件,后使用基因组分析工具包GATK v3.8进行插入或缺失重新排列(InDel重组),以确定获得完整的线粒体基因组序列[35]。
1.5 数据分析用BioEdit 7.2.5软件[36]进行牦牛全线粒体基因组序列的多序列比对分析,统计序列长度和多态位点。基于Dnasp5.10[37]和Arlequin3.11软件[38]分析确定各品种(群体)的单倍型及单倍型组数目和类型,计算单倍型多样度(haplotype diversity, Hd± SD)和核苷酸多样度(nucleotide diversity, π±SD)大小,取10 000次重复在5%的水平上确定其差异显著情况,以探究各牦牛品种(群体)的遗传多样性水平及其差异。品种(群体)间的固定分化指数(fixation index, FST)也通过Arlequin3.11软件[38]计算获得,以揭示牦牛品种(群体)间的分化状况。RST值通过线性化的FST值计算获得,即RST=FST/(1-FST)。用SPSS18.0软件基于RST值进行多维尺度(multidimensional scaling, MDS)分析,以揭示各牦牛品种(群体)间的聚类情况和遗传关系。用MEGA7.0软件[39]中的邻接法(neighbor-joining, NJ)构建系统发育树,并用Network10.1软件[40]构建网络中介图(median-Joining, MJ)以揭示不同单倍型(组)间的系统发育关系。
2 结果 2.1 5个牦牛品种(群体)线粒体基因组遗传变异及遗传多样性分析经基因组重测序获得的22头青海地方牦牛品种个体的基因组测序深度在22.44X-27.36X之间,平均测序深度为23.94X,平均基因组覆盖率达98.01%。序列比对分析表明,164条全线粒体基因组序列长度在16 322~16 329 bp之间,排除15处插入或缺失位点后共检测到322个变异位点,其中单一多态位点148个,简约信息位点174个(表 2)。青海4个地方牦牛品种中共发现变异位点203个,在青海高原、玉树、环湖和雪多牦牛中分别检测到152、151、144和138个变异位点,而在野牦牛群体中检测到247个变异位点。根据线粒体基因组序列间遗传变异,共确定了115种单倍型,其中H37与H38两种单倍型为优势单倍型,均被4个青海地方牦牛品种所共享,所占个体(序列)数分别为10个和19个;有101种单倍型为稀有单倍型,均被不同牦牛品种(群体)的1个个体(序列)所拥有;其余单倍型被不同的牦牛品种(群体)所共享,如单倍型H28被环湖、青海高原、玉树牦牛所共享,H40被环湖、青海高原牦牛所共享,H56被环湖、玉树牦牛所共享。在青海高原、环湖、雪多、玉树和野牦牛品种(群体)中分别确定了30、24、26、26和22种单倍型,其中特异单倍型数依次为26、18、23、19和22种(表 2)。各品种(群体)的单倍型组分布情况见表 2,可以看出,野牦牛群体的个体(序列)分布在A、B、C、D、E、F、G和H共8种单倍型组中,频率分别为37.50%(9/24)、20.83%(5/24)、4.17%(1/24)、16.67%(4/24)、8.33%(2/24)、4.17%(1/24)、4.17%(1/24)和4.17%(1/24),而4个青海地方牦牛品种的个体(序列)分布在A、B、C、D、G和H共6种单倍型组中,所占比列分别为62.14%(87/140)、16.43%(23/140)、7.14%(10/140)、12.86%(18/140)、0.71%(1/140)和0.71%(1/140),提示E和F两种单倍型组仅存在于野牦牛群体中。值得注意的是,只有A、B和D三种单倍型组被野牦牛和青海4个地方牦牛品种所共享,所占频率分别为58.54%(96/164)、17.07%(28/164)和13.41%(22/164),而C单倍型组被除环湖牦牛外的其他品种(群体)共享,G单倍型组只被雪多牦牛和野牦牛所共享,H单倍型组只被玉树牦牛和野牦牛所共享。单倍型多样度和核苷酸多样度计算结果表明,野牦牛的单倍型多样度(0.992 8±0.014 4)和核苷酸多样度(0.003 5±0.001 8)均高于青海4个地方牦牛品种的单倍型多样度(0.973 1±0.007 7)和核苷酸多样度(0.002 5± 0.001 2)。在4个青海地方牦牛品种中,雪多牦牛的单倍型多样度最高,为0.988 5±0.012 6;环湖牦牛的单倍型多样度最低,为0.939 3± 0.027 8;而玉树牦牛与青海高原牦牛的单倍型多样度值居中,分别为0.975 8±0.018 7和0.973 0±0.016 6。青海高原、环湖、雪多、玉树牦牛的核苷酸多样度分别为0.002 9±0.001 4、0.002 1±0.001 0、0.002 1±0.001 1和0.002 7±0.001 4(表 2)。
|
|
表 2 5个牦牛品种(群体)遗传多样性指数大小 Table 2 Genetic diversity indexes of 5 yak breeds (populations) |
通过计算5个牦牛品种(群体)间的固定分化指数值(FST)大小(表 3),结果表明,野牦牛与青海高原、环湖、雪多、玉树牦牛间的FST值分别为0.009 9、0.041 2、0.023 6和-0.008 8;4个青海地方牦牛中,青海高原牦牛与环湖、雪多、玉树牦牛间的FST值分别为0.020 0、0.035 8和0.018 7,环湖牦牛与雪多、玉树牦牛间的FST值分别为0.011 2和0.033 8,而雪多牦牛和玉树牦牛之间的FST值为0.011 3。
|
|
表 3 5个牦牛品种(群体)间FST值(下三角)和RST值(上三角) Table 3 FST values (below diagonal) and RST values (above diagonal) among 5 yak breeds (populations) |
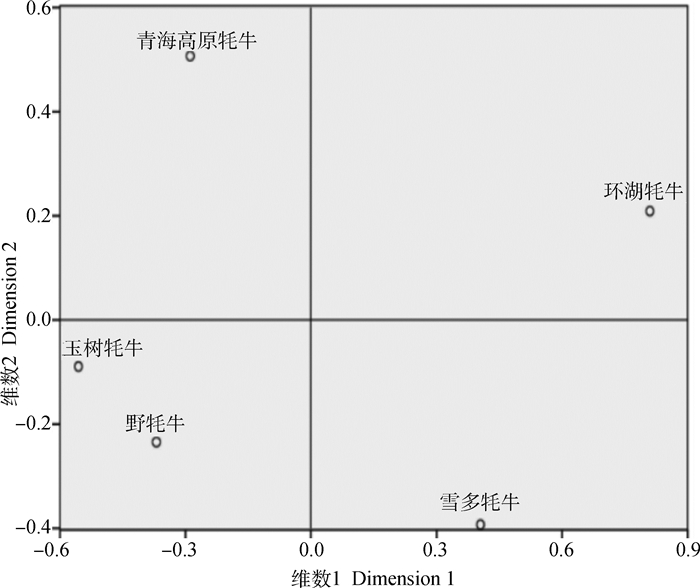
基于各牦牛品种(群体)间的RST值构建多维尺度(MDS)关系聚类图(图 1),结果显示,4个青海地方牦牛品种各自占据一个象限;在维数1中野牦牛、玉树牦牛和青海高原牦牛聚为1类,而环湖牦牛和雪多牦牛聚为1类;在维数2中青海高原牦牛与野牦牛、玉树牦牛再次被分开,而环湖牦牛与雪多牦牛被再次分开。
|
图 1 基于MDS分析构建的5个牦牛品种(群体)间聚类关系 Fig. 1 Cluster relationships among 5 yak breeds (populations) based on MDS analysis |
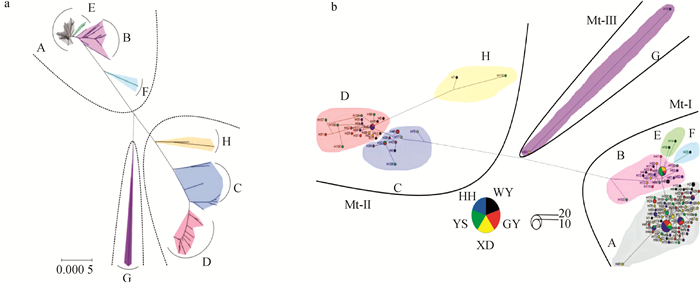
对5个牦牛品种(群体)线粒体基因组单倍型构建系统发育树(图 2),结果表明,115种单倍型分布在A、B、C、D、E、F、G和H共8种单倍型组中,分别占58.54%、17.07%、6.71%、13.41%、1.22%、0.61%、1.22%和1.22%。8种单倍型组组成了明显的3个母系遗传分支(即Mt-Ⅰ、Mt-Ⅱ和Mt-Ⅲ),其中Mt-Ⅰ支系由包含83种单倍型的A、B、E和F共4种单倍型组构成,所占比例为72.17%(83/115),由127个个体(序列)分享。Mt-Ⅱ支系包含30种单倍型,占26.09%(30/115),分属C、D和H 3种单倍型组,由35个个体(序列)分享。值得注意的是,共有2种单倍型分布在Mt-Ⅲ支系中,包括雪多牦牛和野牦牛各1个个体(序列),所占比例仅为1.74%(2/115)。
|
a. NJ法构建的系统发育图;b. MJ法构建的Network图 a. Phylogenetic tree constructed by NJ method; b. Network diagram constructed by MJ method 图 2 野牦牛和青海4个地方牦牛品种(群体)系统发育树 Fig. 2 Phylogenetic trees of wild yak and 4 local yak breeds (populations) in Qinghai |
遗传多样性是衡量一定区域范围内动物遗传资源丰富程度的重要指标之一,其对动物遗传资源的保护和育种策略的制定都具有重要指导意义[41]。在本研究中,发现5个牦牛品种(群体)均拥有其特有的单倍型,如野牦牛中确定特有单倍型22种,青海高原、环湖、雪多、玉树牦牛中各确定26、18、23和19种特有单倍型,这说明野牦牛及青海省4个地方牦牛品种均拥有特有的母系遗传信息。本研究母系遗传多样性分析表明,野牦牛线粒体基因组单倍型多样度为0.992 8±0.014 4,高于4个青海地方牦牛品种的单倍型多样度(0.973 1± 0.007 7),表明野牦牛具有更丰富的母系遗传多样性,其多样性水平高于家牦牛,这与Wang等[18]基于51条家牦牛和21条野牦牛线粒体基因组序列对家、野牦牛母系遗传多样性的研究结果吻合,也与先前基于mtDNA D-loop区、Cyt b基因序列对家、野牦牛遗传多样性的研究结果一致[9-33]。而在青海4个地方牦牛品种中,本研究结果表明雪多牦牛的单倍型多样度最高(0.988 5±0.012 6),玉树牦牛与青海高原牦牛的单倍型多样度值居中(即(0.975 8±0.018 7)和(0.973 0±0.016 6)),而环湖牦牛的单倍型多样度最低(0.939 3±0.027 8),说明雪多牦牛的母系遗传多样性在4个地方牦牛品种中最高,而环湖牦牛的遗传多样性水平最低,这与Wang等[34]基于86条线粒体基因组序列对青海省4个牦牛品种(群体)(即雪多、环湖、祁连和玉树牦牛)的遗传多样性研究结果基本吻合。总体来看,本研究中4个青海地方牦牛品种的母系遗传多样性均较高,遗传变异丰富,但相比而言,青海高原、玉树和雪多牦牛的母系遗传多样性水平相差不大,均稍高于环湖牦牛的遗传多样性。究其原因,可能与各地方牦牛品种不同的近交程度、受选择强度及群体结构组成等因素相关。
固定分化指数(FST)值为动物群体间遗传分化指数大小,可以评估不同群体间的遗传分化程度。Wright[42]研究表明,FST值在0~0.05之间说明群体间遗传分化程度很弱;在0.05~0.15之间说明群体间达到中等遗传分化;在0.15~0.25之间说明群体间遗传分化大;大于0.25时则说明群体间分化程度极大。当前,尚未见有关野牦牛及青海4个地方牦牛品种间母系遗传分化状况的研究报道。在本研究中,野牦牛与4个青海地方牦牛品种间的FST值在-0.008 8~0.041 2之间,而4个青海地方牦牛品种间的FST值在0.011 2~0.035 8之间,说明4个青海地方牦牛品种间及各品种与野牦牛群体间的FST值均在0~0.05之间,表明青海各地方牦牛品种间以及与野牦牛群体间的遗传分化均很小,分化程度很弱,该结果与马志杰[8]对青海地方牦牛品种及野牦牛父系遗传分化的研究结果一致,说明4个青海地方牦牛品种间及与野牦牛群体之间遗传分化均较小。本研究结果表明,雪多牦牛与青海高原牦牛间FST值最大(0.035 8),而与环湖牦间FST值最小,为0.011 2,提示雪多牦牛与青海高原牦牛间分化程度最大,而与环湖牦牛分化程度最小。总体来看,各牦牛品种(群体)间FST值大小存在差异,其值大小一定程度上反映了它们之间分化程度的情况。此外,本研究各牦牛品种(群体)间多维尺度(MDS)聚类关系分析显示,4个青海地方牦牛品种(即青海高原、环湖、雪多和玉树牦牛)各自占据一个象限,分别独立为1类,表明青海各地方牦牛品种间存在明显的母系遗传差异。总体来看,玉树牦牛与野牦牛聚类更近,表明它们之间遗传关系也较近且拥有更多相似的母系遗传成分,而环湖牦牛和雪多牦牛聚类较近,青海高原牦牛和玉树牦牛聚类较近,均一定程度上反映了它们彼此之间母系遗传关系的远近程度,这种品种(群体)间的聚类结果和遗传关系,与各品种(群体)间的分化程度、地理分布远近基本一致。
牦牛的起源和系统发育研究有助于揭示其遗传背景、进化历史和系统发育关系。在先前的研究报道中,基于mtDNA D-loop区、Cyt-b和COIII等基因序列片段核苷酸变异对家、野牦牛母系系统发育的研究相对较多[10-33],而利用线粒体基因组全序列变异对牦牛母系遗传支系的组成及起源研究相对较少[9, 18, 34]。在本研究中,基于线粒体基因组全序列变异对野牦牛及青海4个地方牦牛品种进行了系统发育分析,研究表明所确定的115种家、野牦牛单倍型可明显的聚为3个母系遗传分支,即Mt-Ⅰ、Mt-Ⅱ和Mt-Ⅲ,所占比例分别为72.17%、26.09%和1.74%,且3个母系分支中均有家、野牦牛个体分布,这与Wang等[18]、Wang等[34]、Ma等[9]基于线粒体全基因组变异对野牦牛及其他家牦牛品种(群体)的研究结果基本一致,说明牦牛由3个母系支系组成,以Mt-Ⅰ支系为主,推测可能有3个母系起源。
4 结论本研究结果表明,青海4个地方牦牛品种和野牦牛群体均具有丰富的母系遗传多样性;各品种(群体)拥有特有的母系遗传信息,存在一定的母系遗传差异,但彼此间遗传分化程度很弱;玉树牦牛与野牦牛亲缘关系最近,4个地方牦牛品种中雪多牦牛与环湖牦牛亲缘关系最近;野牦牛和青海家牦牛品种由3个母系支系组成,推测牦牛有3个母系起源。
| [1] |
WIENER G, HAN J L, LONG R J. The yak[M]. 2nd ed. Bangkok: Food and Agriculture Organization of the United Nations, 2003: 1-6.
|
| [2] |
贾功雪, 丁路明, 徐尚荣, 等. 青藏高原牦牛遗传资源保护和利用: 问题与展望[J]. 生态学报, 2020, 40(18): 6314-6323. JIA G X, DING L M, XU S R, et al. Conservation and utilization of yak genetic resources in Qinghai-Tibet plateau: problems and perspectives[J]. Acta Ecologica Sinica, 2020, 40(18): 6314-6323. (in Chinese) |
| [3] |
国家畜禽遗传资源委员会办公室. 关于公布《国家畜禽遗传资源品种名录》的通知[J]. 中华人民共和国农业农村部公报, 2020(6): 36. Office of National Livestock and Poultry Genetic Resources Committee. Circular on releasing the directory of national livestock & poultry genetic resources[J]. Gazette of the Ministry of Agriculture and Rural Affairs of the People's Republic of China, 2020(6): 36. (in Chinese) |
| [4] |
NABHOLZ B, MAUFFREY J F, BAZIN E, et al. Determination of mitochondrial genetic diversity in mammals[J]. Genetics, 2008, 178(1): 351-361. DOI:10.1534/genetics.107.073346 |
| [5] |
PAKENDORF B, STONEKING M. Mitochondrial DNA and human evolution[J]. Annu Rev Genom Hum Genet, 2005, 6: 165-183. DOI:10.1146/annurev.genom.6.080604.162249 |
| [6] |
LI R, WANG S Q, XU S Y, et al. Novel Y-chromosome polymorphisms in Chinese domestic yak[J]. Anim Genet, 2014, 45(3): 449-452. DOI:10.1111/age.12139 |
| [7] |
MA Z J, XIA X T, CHEN S M, et al. Identification and diversity of Y-chromosome haplotypes in Qinghai yak populations[J]. Anim Genet, 2018, 49(6): 618-622. DOI:10.1111/age.12723 |
| [8] |
马志杰. 牦牛父系遗传多样性及起源研究[D]. 杨凌: 西北农林科技大学, 2019. MA Z J. Study on the paternal genetic diversity and origin of the yak (Bos grunniens)[D]. Yangling: Northwest A&F University, 2019. (in Chinese) |
| [9] |
MA Z J, LI G Z, CHEN S M, et al. Rich maternal and paternal genetic diversity and divergent lineage composition in wild yak (Bos mutus)[J]. Anim Biotechnol. DOI:10.1080/10495398.2021.1884567 |
| [10] |
涂正超, 张亚平, 邱怀. 中国牦牛线粒体DNA多态性及遗传分化[J]. 遗传学报, 1998, 25(3): 205-212. TU Z C, ZHANG Y P, QIU H. Mitochondrial DNA polymorphism and genetic diversity in Chinese Yaks[J]. Acta Genetica Sinica, 1998, 25(3): 205-212. (in Chinese) |
| [11] |
赖松家, 王玲, 刘益平, 等. 中国部分牦牛品种线粒体DNA遗传多态性研究[J]. 遗传学报, 2005, 32(5): 463-470. LAI S J, WANG L, LIU Y P, et al. Study on mitochondrial DNA genetic polymorphism of some yak breeds in China[J]. Acta Genetica Sinica, 2005, 32(5): 463-470. (in Chinese) |
| [12] |
GUO S C, SAVOLAINEN P, SU J P, et al. Origin of mitochondrial DNA diversity of domestic yaks[J]. BMC Evol Biol, 2006, 6: 73. DOI:10.1186/1471-2148-6-73 |
| [13] |
LAI S J, CHEN S Y, LIU Y P, et al. Mitochondrial DNA sequence diversity and origin of Chinese domestic yak[J]. Anim Genet, 2007, 38(1): 77-80. DOI:10.1111/j.1365-2052.2007.01555.x |
| [14] |
郭松长, 祁得林, 陈桂华, 等. 家牦牛线粒体DNA(mtDNA)遗传多样性及其分类[J]. 生态学报, 2008, 28(9): 4286-4294. GUO S C, QI D L, CHEN G H, et al. Genetic diversity and classification of domestic yaks inferred from mitochondrial DNA sequences[J]. Acta Ecologica Sinica, 2008, 28(9): 4286-4294. DOI:10.3321/j.issn:1000-0933.2008.09.027 (in Chinese) |
| [15] |
马志杰, 钟金城, 韩建林, 等. 野牦牛(Bos grunniens mutus) mtDNA D-Loop区的遗传多样性[J]. 生态学报, 2009, 29(9): 4798-4803. MA Z J, ZHONG J C, HAN J L, et al. Genetic diversity in the mtDNA D-Loop region of wild yak (Bos grunniens mutus)[J]. Acta Ecologica Sinica, 2009, 29(9): 4798-4803. DOI:10.3321/j.issn:1000-0933.2009.09.025 (in Chinese) |
| [16] |
MA Z J, ZHONG J C, HAN J L, et al. Genetic diversity and demographic history of wild yak (Bos grunniens mutus) inferred from mtDNA D-loop sequences[J]. Afr J Biotechnol, 2010, 9(46): 7805-7810. DOI:10.5897/AJB10.1074 |
| [17] |
LI G Z, LUO J, CHEN S M, et al. Maternal genetic diversity, differentiation and phylogeny of three white yak breeds/populations in China[J]. Anim Biotechnol. DOI:10.1080/10495398.2021.1973018 |
| [18] |
WANG Z F, SHEN X, LIU B, et al. Phylogeographical analyses of domestic and wild yaks based on mitochondrial DNA: new data and reappraisal[J]. J Biogeogr, 2010, 37(12): 2332-2344. DOI:10.1111/j.1365-2699.2010.02379.x |
| [19] |
马志杰, 钟金城, 韩建林, 等. 牦牛分子遗传多样性研究进展[J]. 遗传, 2013, 35(2): 151-160. MA Z J, ZHONG J C, HAN J L, et al. Research progress on molecular genetic diversity of the yak (Bos grunniens)[J]. Hereditas (Beijing), 2013, 35(2): 151-160. (in Chinese) |
| [20] |
马志杰. 牦牛线粒体基因组研究进展[J]. 中国农业大学学报, 2014, 19(3): 154-161. MA Z J. Research progress of the yak (Bos grunniens) mitochondrial genome[J]. Journal of China Agricultural University, 2014, 19(3): 154-161. (in Chinese) |
| [21] |
成述儒, 曾玉峰, 王欣荣, 等. 甘肃境内6个牦牛群体mtDNA D-环序列遗传多样性与聚类分析[J]. 华北农学报, 2014, 29(3): 16-21. CHENG S R, ZENG Y F, WANG X R, et al. Genetic diversity and cluster analysis of six yak populations in Gansu province using mitochondrial D-loop sequence[J]. Acta Agriculturae Boreali-Sinica, 2014, 29(3): 16-21. (in Chinese) |
| [22] |
涂世英, 宋娜娜, 柴志欣, 等. 中甸牦牛mtDNA Cytb基因遗传多样性及系统进化分析[J]. 中国畜牧兽医, 2016, 43(10): 2680-2687. TU S Y, SONG N N, CHAI Z X, et al. Analysis on genetic diversity and phyletic evolution of mtDNA Cytb gene in Zhongdian Yak[J]. China Animal Husbandry & Veterinary Medicine, 2016, 43(10): 2680-2687. (in Chinese) |
| [23] |
胡丹, 钟金城, 柴志欣. 新疆塔什库尔干牦牛mtDNA Cytb基因和D-Loop区遗传多样性及系统进化分析[J]. 家畜生态学报, 2018, 39(11): 11-16. HU D, ZHONG J C, CHAI Z X. Genetic diversity and phyletic evolution on mtDNA Cytb gene and D-loop region of Taxkorgan yak[J]. Acta Ecologae Animalis Domastici, 2018, 39(11): 11-16. DOI:10.3969/j.issn.1673-1182.2018.11.003 (in Chinese) |
| [24] |
李瑞哲, 马志杰, 陈生梅, 等. 基于mtDNA D-loop序列分析探究青海雪多牦牛的遗传多样性及母系遗传背景[J]. 青海畜牧兽医杂志, 2018, 48(2): 1-3. LI R Z, MA Z J, CHEN S M, et al. Exploration on genetic diversity and matrilineal genetic background of Xueduo yak based on mtDNA D-loop sequence analysis[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 2018, 48(2): 1-3. DOI:10.3969/j.issn.1003-7950.2018.02.001 (in Chinese) |
| [25] |
季文博, 王会, 柴志欣, 等. 西藏牦牛mtDNA-Cytb及ZFY基因遗传多样性与系统进化分析[J]. 家畜生态学报, 2019, 40(11): 12-17. JI W B, WANG H, CHAI Z X, et al. Analysis on genetic diversity and phyletic evolution of mtDNA Cytb & ZFY Gene in Tibetan Yak[J]. Acta Ecologae Animalis Domastici, 2019, 40(11): 12-17. DOI:10.3969/j.issn.1673-1182.2019.11.003 (in Chinese) |
| [26] |
李静, 王莉, 道敏, 等. 喀喇昆仑-帕米尔地区牦牛mtDNA Cytb遗传多样性及系统发育分析[J]. 生物技术进展, 2019, 9(5): 509-517. LI J, WANG L, DAO M, et al. Genetic diversity and phylogenetic analysis on mtDNA Cytb in Yak of Karakorum-Pamir Region[J]. Current Biotechnology, 2019, 9(5): 509-517. (in Chinese) |
| [27] |
李静, 郭丽君, 王莉, 等. 喀喇昆仑-帕米尔地区牦牛mtDNA D-loop区遗传多样性及系统发育分析[J]. 中国畜牧兽医, 2020, 47(8): 2481-2492. LI J, GUO L J, WANG L, et al. Analyses of genetic diversity and phylogeny of mtDNA D-loop from yak in Karakoram-Pamir area[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(8): 2481-2492. (in Chinese) |
| [28] |
侯孟典, 宋仁德, 王会, 等. 玉树牦牛与四个牦牛群体线粒体DNA D-Loop区遗传多样性分析[J]. 家畜生态学报, 2020, 41(3): 21-26. HOU M D, SONG R D, WANG H, et al. Analysis on genetic diversity of mtDNA D-loop region from Yushu yak population and other four yak population[J]. Acta Ecologae Animalis Domastici, 2020, 41(3): 21-26. DOI:10.3969/j.issn.1673-1182.2020.03.004 (in Chinese) |
| [29] |
李广祯, 马志杰, 陈生梅, 等. 青海省曲麻莱牦牛的母系遗传多样性及遗传背景探究[J]. 青海大学学报, 2020, 38(1): 27-31. LI G Z, MA Z J, CHEN S M, et al. Maternal genetic diversity and genetic background of Qumalai yaks in Qinghai province[J]. Journal of Qinghai University, 2020, 38(1): 27-31. (in Chinese) |
| [30] |
王兴东, 郭宪, 吴晓云, 等. 青海高原牦牛mtDNA Cytb基因和D-loop区遗传多样性及系统进化分析[J]. 基因组学与应用生物学, 2021, 40(1): 9-17. WANG X D, GUO X, WU X Y, et al. Genetic diversity and phylogenetic analysis of mtDNA Cytb gene and D-loop region in Yak of Qinghai Plateau[J]. Genomics and Applied Biology, 2021, 40(1): 9-17. (in Chinese) |
| [31] |
李广祯, 马志杰, 陈生梅, 等. 基于mtDNA D-loop序列探究门源白牦牛与天祝白牦牛间的遗传差异[J]. 中国畜牧兽医, 2021, 48(5): 1647-1654. LI G Z, MA Z J, CHEN S M, et al. Genetic difference between Menyuan white yaks and Tianzhu white yaks based on mtDNA D-loop sequences[J]. China Animal Husbandry & Veterinary Medicine, 2021, 48(5): 1647-1654. (in Chinese) |
| [32] |
李广祯, 马志杰, 赵学军, 等. 青海省同德牦牛的母系遗传多样性及群体遗传结构分析[J]. 中国牛业科学, 2021, 47(3): 17-21. LI G Z, MA Z J, ZHAO X J, et al. Maternal genetic diversity and population genetic structure of Tongde Yak in Qinghai province[J]. China Cattle Science, 2021, 47(3): 17-21. DOI:10.3969/j.issn.1001-9111.2021.03.005 (in Chinese) |
| [33] |
陈生梅, 马志杰, 李瑞哲, 等. 青海省门源白牦牛mtDNA D-loop区遗传多样性及母系起源[J]. 青海畜牧兽医杂志, 2021, 51(2): 15-18. CHEN S M, MA Z J, LI R Z, et al. MtDNA D-loop sequence variations and maternal origin of Menyuan white yak in Qinghai province[J]. Chinese Qinghai Journal of Animal and Veterinary Sciences, 2021, 51(2): 15-18. DOI:10.3969/j.issn.1003-7950.2021.02.003 (in Chinese) |
| [34] |
WANG X D, PEI J, BAO P J, et al. Mitogenomic diversity and phylogeny analysis of yak (Bos grunniens)[J]. BMC Genomics, 2021, 22(1): 325. DOI:10.1186/s12864-021-07650-x |
| [35] |
XIA X T, ACHILLI A, LENSTRA J A, et al. Mitochondrial genomes from modern and ancient Turano-Mongolian cattle reveal an ancient diversity of taurine maternal lineages in East Asia[J]. Heredity, 2021, 126(6): 1000-1008. DOI:10.1038/s41437-021-00428-7 |
| [36] |
HALL T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucl Acids Symp Ser, 1999, 41: 95-98. |
| [37] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25: 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [38] |
EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evol Bioinform, 2007, 1: 47-50. |
| [39] |
HALL B G. Building phylogenetic trees from molecular data with MEGA[J]. Mol Biol Evol, 2013, 30(5): 1229-1235. DOI:10.1093/molbev/mst012 |
| [40] |
BANDELT H J, FORSTER P, RÖHL A. Median-joining networks for inferring intraspecific phylogenies[J]. Mol Biol Evol, 1999, 16(1): 37-48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [41] |
纪会, 官久强, 王会, 等. 亚丁牦牛和拉日马牦牛遗传多样性及遗传结构分析[J]. 草业学报, 2021, 30(5): 134-145. JI H, GUAN J Q, WANG H, et al. Genetic structure and diversity of Yading yak and Larima yak populations[J]. Acta Prataculturae Sinica, 2021, 30(5): 134-145. (in Chinese) |
| [42] |
WRIGHT S. Evolution and the genetics of populations vol.4:variability within and among natural populations[M]. Chicago: University of Chicago Press, 1978.
|
(编辑 郭云雁)



