2. 南京农业大学淮安研究院,淮安 223001;
3. 淮安市淮阴新淮种猪场,淮安 223322;
4. 江苏现代农业 (生猪) 产业体系集成创新中心,南京 210095
2. Huai 'an Academy, Nanjing Agricultural University, Huai 'an 223001, China;
3. Huaiyin Xinhuai Pig Breeding Farm of Huai 'an City, Huai 'an 223322, China;
4. Industrial Technology System Integration Innovation Center of Jiangsu Modern Agriculture (Pig), Nanjing 210095, China
滴水损失(drip loss, DL)是评价猪肌肉品质的重要经济性状,它是指在猪只屠宰后,由于肌肉组织的生理状态发生改变,肌肉束缚水能力下降,肌肉内的水分受到重力作用自然缓慢渗出而造成的重量损失[1]。过高的滴水损失不仅会使肉制品加工行业由于鲜肉重量减少而遭受直接的经济损失,而且也会影响消费者购买欲望。如何改善猪肉的品质,降低肌肉的滴水损失是养殖行业和肉制品加工行业共同面临的挑战。目前,我国受非洲猪瘟疫情影响,国内各个省份之间的猪肉供应从“调猪”向“调肉”的方向改变,这意味着未来猪胴体需经历长时间的运输才能到达消费市场,这无疑将对猪胴体滴水损失性状提出更高的要求。
滴水损失作为肉品质评价系统的重要组成部分,主要由遗传和环境因素调控[2]。早期一些研究发现,滴水损失为中低等遗传力数量性状(h2=0.08~ 0.30)[3-6]。准确的测定猪肉滴水损失性状需要对猪进行屠宰,测定代价高,并且受到屠宰工艺、测定方法、测定时间等多种因素影响,因此传统育种方法很难高效改善滴水损失性状。通过分子标记辅助选择和全基因组选择技术,鉴别影响猪肉滴水损失的主效基因和因果位点,将会加快猪肉滴水损失性状的遗传育种进程。
近年来,研究人员利用分子生物学技术手段在猪基因组中鉴别到了部分影响猪肉滴水损失的关键基因,例如Hal[7]、PRKAG3[8]和PHKG1基因[9]。但猪肉滴水损失由多基因控制,其它影响猪肉滴水损失的基因还有待进一步挖掘。随着第二代测序技术的发展和成本的降低,选择信号分析已经成为研究性状表型差异遗传机制的重要手段之一。当前,检测选择信号的方法根据计算原理的不同主要分为3大类,分别为:基于群体分化的方法,以Fst[10]方法为代表; 基于连锁不平衡的方法,以iHS[11]方法为代表; 基于等位基因频谱的方法,代表的计算方法包括Tajima’s D[12]和CLR(composite likelihood ratio)[13]等。Sun等[14]基于中小梅山猪的重测序数据,利用Fst研究方法发现了与体重相关的SPDEF和PACSIN1基因; 张威[15]利用Fst检测技术,在皖南黑猪中找到与耐粗饲、抗病性、高繁殖力特征相关的基因; 吴林慧等[16]基于54头恩施黑猪的高密度SNP芯片数据,利用iHS技术,找到了与滴水损失、精子形成、骨骼肌分化和总产仔数相关的基因; Lukić等[17]基于Turopolje猪的高密度SNP芯片数据,利用iHS技术,挖掘到了与猪血液特性相关的PEBP4基因。
苏淮猪是在新淮猪基础上再导入大白猪血统,通过横交、多世代选育而成,含淮猪血统25%,大白猪血统75% [18]。本课题组前期研究发现,苏淮猪群体滴水损失性状存在较大的变异,变异系数达到66.88%[19]。因此,本研究利用苏淮猪作为试验群体,鉴别影响猪群体滴水损失变异的关键基因。通过对高、低滴水损失组的苏淮猪群体进行80K SNP芯片分型,利用Fst法和iHS法进行选择信号检测,筛选出受到选择的SNP位点,结合对受选择的代表性SNP在苏淮猪全群中的关联性验证,并通过对选择信号周边基因进行功能注释与富集分析,以期筛选出受到选择的且与苏淮猪滴水损失性状相关的候选基因和位点,旨在为苏淮猪肉品质的改善提供理论依据。
1 材料与方法 1.1 试验动物本试验在江苏省淮安市淮阴种猪场内筛选478头饲养管理条件和日粮相同、日龄相近((237.95±2.13)d)、体质健康的苏淮育肥猪, 其中阉公猪290头,母猪188头,在淮安市金源肉品中心分4个批次屠宰。屠宰前禁食24 h, 期间保持猪只自由饮水。
1.2 试验方法1.2.1 样品采集 试验苏淮猪击晕放血后,烫毛,去头、尾、蹄和内脏(保留板油和肾),称量胴体重; 剪取猪只右耳组织样1份·头-1,放入盛有75%乙醇的2 mL离心管中,注明耳号,-20 ℃保存; 采集左半胴体倒数3~4根肋骨处的背最长肌肌肉样品200 g左右,注明耳号,用于肌肉滴水损失的测定。
1.2.2 苏淮猪滴水损失表型的测定 本试验参考国家农业部标准《NY/T 821—2019猪肌肉品质测定技术规范》,采用吊袋法进行苏淮猪滴水损失表型的测定,具体方法参考文献[19]。
1.2.3 根据滴水损失EBV挑选试验群体 本研究先利用Excel 2019软件对苏淮猪全群滴水损失表型数据进行描述性统计。再使用DMU软件计算478头苏淮猪滴水损失的EBV[20],具体模型:
| $ Y=S+B+a+c+A+e $ |
式中,Y代表滴水损失表型的观测值; S代表性别的固定效应; B代表批次的固定效应; a代表苏淮猪屠宰日龄,作为协变量; c代表苏淮猪的胴体重,作为协变量; A为个体的加性遗传效应; e为随机残差效应。
根据滴水损失EBV将478头苏淮猪按照从大到小进行排序,挑选高、低各10%的苏淮猪个体(N=48)用于后续的芯片分型和选择信号分析。
利用SPSS软件对苏淮猪试验群体内高、低EBV两组之间的育种值和滴水损失进行单因素方差分析。
1.2.4 高、低滴水损失苏淮猪基因型检测 使用天根生物科技有限公司的基因组DNA提取试剂盒提取苏淮猪耳组织样品的DNA,质检合格后,使用基于Illumina平台的GeneSeek公司开发的GGP 80K SNP芯片进行检测,获得SNP分型数据。原始基因型数据以ped和map文件存储。接着利用PLINK软件对SNP进行质量控制[21]。质控标准:剔除非常染色体上的SNP,次等位基因频率大于0.01,个体的SNP检出率大于0.9。
1.2.5 基因型数据填充 由于进行iHS分析时需要所有的SNP都分型成功,因此本研究将质控后的芯片数据里部分没有分型成功的SNP位点进行了填充,本研究对有缺失的基因型数据进行填充时使用的Beagle软件[22]。
1.2.6 选择信号检测 按照Weir和Cockerham[23]的统计方法进行高、低滴水损失苏淮猪群体间Fst值的计算,利用vcftools软件计算每个位点的Fst 值[24]。接着利用R包qqman进行绘图,来展示全基因组水平上Fst值的分布[25]。
本研究将低滴水损失的苏淮猪群体作为试验群体,使用selscan软件[26]进行iHS选择信号的计算。然后将原始的iHS值正态化处理后取绝对值,同时按从大到小的顺序进行排序。
根据Wright[27]的研究,若群体Fst值在0~0.05之间,说明各亚群间不存在分化; 若Fst值在0.05~0.15之间,为中度分化; 若Fst值在0.15~0.25之间,则为高度分化。合并选择信号分析结果后,取|iHS|值在前5%,且Fst 值≥0.15的SNP位点作为受选择位点。
1.2.7 候选基因检测及功能注释和富集分析 基于猪11.1版本参考基因组(Sscrofa 11.1)的位置信息使用Ensembl在线网站对受选择的SNP位点上、下游各50 kb的区域进行基因注释,并使用NCBI和Genecard在线网站查找相关基因的功能[28]。通过KOBAS 3.0在线数据库对候选基因进行GO和KEGG功能富集分析[29],分析内容涵盖了细胞组分(cellular component)、分子功能(molecular function)、生物学过程(biological process)。
1.2.8 影响猪滴水损失的QTL注释 根据猪11.1版本参考基因组(Sscrofa 11.1)的位置信息,利用在线网站Animal QTL Database下载影响猪滴水损失的全部QTL信息。接着,与选择信号显著候选位点进行比对。
1.2.9 受选择的代表性SNP在苏淮猪全群中的关联性验证 本研究对3个位于候选基因内含子上同时又位于QTL区域内的SNPs位点进行分型,通过一代测序进行全群分型。利用SAS 9.4软件将共有SNP位点基因型与滴水损失进行关联性分析,具体模型:
| $Y=S+B+a+c+G+e $ |
式中,Y代表滴水损失表型的观测值; S代表性别的固定效应; B代表批次的固定效应; a代表苏淮猪屠宰日龄,作为协变量; c代表苏淮猪的胴体重,作为协变量; G为个体基因型的固定效应; e为随机残差效应。以P < 0.05表示差异达到显著水平,以P < 0.01表示差异达到极显著水平。所得结果均以滴水损失表型的“平均数±标准误”表示。
2 结果 2.1 苏淮猪滴水损失表型的变异分析本研究利用Excel 2019软件对苏淮猪群体滴水损失表型数据进行描述性统计后发现, 苏淮猪群体滴水损失均值2.04%,群体内存在较大表型变异,变异系数高达76.45%。
利用SPSS软件进行单因素方差分析,结果见表 1,苏淮猪试验群体内高、低EBV两组之间的育种值和滴水损失差异均极显著(P<0.01)。
|
|
表 1 苏淮猪群体内高、低EBV组间滴水损失的比较分析 Table 1 Comparative analysis of drip loss between high and low EBV groups in Suhuai pig population |
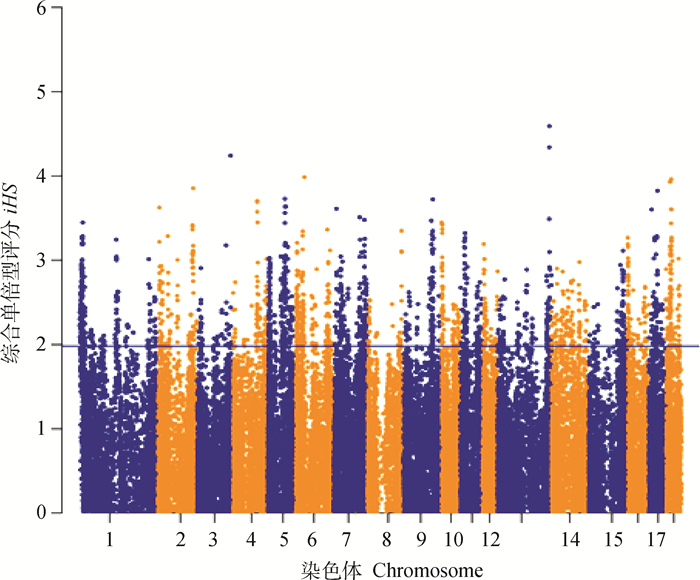
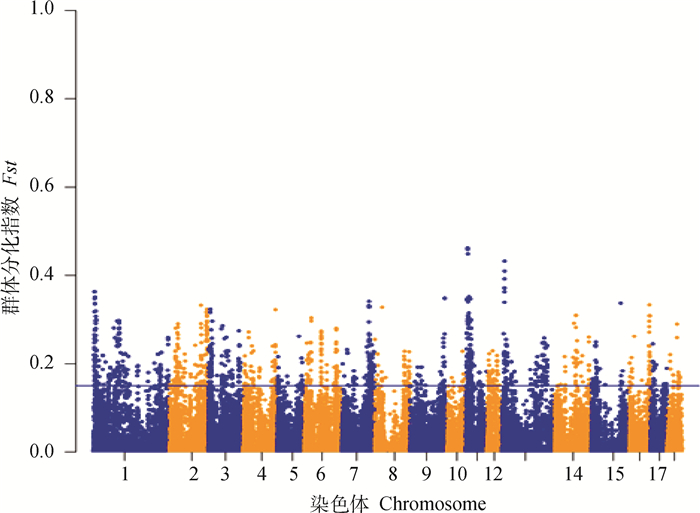
首先分别进行了基于iHS和Fst的选择信号分析。使用selcsan软件对质控后的全基因组数据进行iHS分析,共得到48 344个标记的iHS值,取绝对值后使用R语言绘图,以|iHS|值在前5%为阈值线,共有2 417个SNPs位于阈值线之上,这些SNPs主要分布在1、2、4、5、6、7、9、11、13、14、15、18号染色体上,其中|iHS|值最大的SNP位点位于13号染色体上,结果如图 1所示。接着,又使用vcftools软件对质控后的51 705个SNPs进行计算,得到了高、低滴水损失苏淮猪群体间Fst值,并利用R语言进行绘图,共有1 597个SNPs的Fst值大于0.15,这些SNPs主要分布在1、2、3、6、7、11、13号染色体上,其中Fst值最大的SNP位点位于11号染色体上,结果如图 2所示。
|
图 1 低滴水损失苏淮猪全基因组水平上的|iHS|分布 Fig. 1 Genome-wide iHS values of Suhuai pig populations with low drip loss |
|
图 2 高、低滴水损失苏淮猪组间全基因组水平上SNP的Fst分布 Fig. 2 Genome-wide Fst values of each SNP in drip loss between high and low groups of Suhuai pig populations |
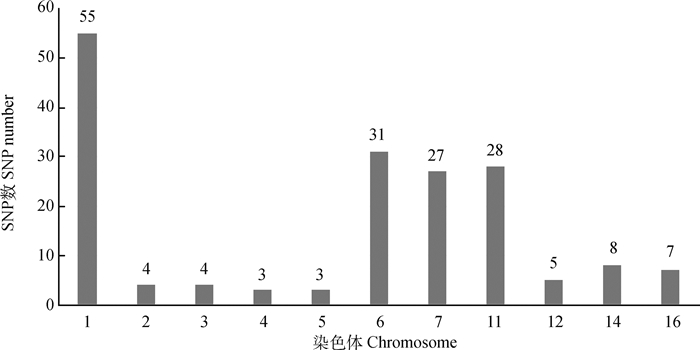
本研究基于两种选择信号的检测结果,选择iHS值在前5%,同时Fst值≥0.15的SNP位点作为达到显著水平的受选择位点,共计175个SNPs。由图 3可知,这些SNP位点分布在1、2、3、4、5、6、7、11、12、14、16号染色体上,其中主要分布在1、6、7、11号染色体上。
|
图 3 达到显著水平的SNP位点在染色体上的分布 Fig. 3 Distribution of significant SNP loci on chromosomes |
本研究根据猪11.1版本参考基因组(Sscrofa 11.1)的位置信息,使用Ensembl在线网站对达到显著水平的SNP位点进行基因注释,共获得了73个基因。由于很多基因在猪上的报道和研究不多,本研究利用NCBI和Genecard在线网站查找人类直系同源基因的功能。同时,利用KOBAS 3.0在线数据库对所有的候选基因进行富集分析。如表 2所示,通过富集分析结果发现,部分条目的生物学功能与肌肉发育以及细胞氧化应激有关。
|
|
表 2 候选基因富集分析结果 Table 2 The result of candidate genes enrichment analyses |
如表 3所示,对选择信号进行基因注释后,发现多个基因功能与肌动蛋白功能相关,如PACRG、EZR、MRTFA和LCP1基因; 其次还发现了VKORC1L1基因与细胞氧化应激相关。
|
|
表 3 与滴水损失相关的部分重要候选基因的位置和基因的功能 Table 3 Location and function of some important candidate genes related to drip loss |
本研究对苏淮猪全基因组选择信号分析鉴别的175个位点进行影响猪滴水损失的QTL注释。结果如表 4所示,通过注释发现,共有27个SNPs分别分布在15个QTLs上,这些位点涉及到了13个基因,其中rs80930355、rs340037952位点分别位于与肌动蛋白结合有关的EZR、MRTFA基因的内含子上,rs320624660位点位于与细胞氧化应激相关的VKORC1L1基因的内含子上。
|
|
表 4 苏淮猪全基因组选择信号显著位点的QTL注释结果 Table 4 QTL annotation results of significant loci for genome wide selection signals in Suhuai pigs |
本研究选择与已报道的QTL区域重合且位于候选基因上的3个SNPs(rs80930355、rs340037952和rs320624660位点)在苏淮猪全群进行基因分型。通过苏淮猪全群滴水损失与3个SNPs关联分析发现(表 5),rs340037952位点与苏淮猪群体滴水损失存在显著关联(P < 0.05),rs320624660位点与苏淮猪群体滴水损失存在极显著关联(P < 0.01)。其中,对于rs320624660位点而言,AA基因型个体的滴水损失极显著高于GG基因型个体(P < 0.01),AG基因型个体的滴水损失显著高于GG型个体(P < 0.05)。而rs80930355位点与滴水损失不存在显著关联(P>0.05)。
|
|
表 5 基因型与滴水损失的关联性分析 Table 5 Association analysis of genotypes with drip loss |
苏淮猪含有75% 的大白猪血统和25% 的淮猪血统,并且在苏淮猪长期的人工选育过程中会对背膘、日增重进行选择,因此会间接选择包括滴水损失在内的肉质性状。但是由于苏淮猪基因组包含来自大白猪的基因片段和淮猪的基因片段,这造成了苏淮猪群体内滴水损失性状可能会存在较大变异。本研究发现,苏淮猪滴水损失变异很大,也印证了这一判断。因此,本研究使用苏淮猪作为试验动物,来鉴别影响猪滴水损失变异的候选基因。本研究基于苏淮猪高密度芯片数据,采用Fst和iHS两种选择信号方法合并分析的方式对苏淮猪进行全基因组检测,鉴别影响猪滴水损失的关键变异位点和基因。
由于肌肉滴水损失表型受众多且复杂的因素影响,如运输方式、屠宰前的击晕方式、环境温度等都会影响滴水损失表型的测定,这会对试验带来一些不可避免的误差,以至于影响性状表型的准确性和育种效率,因此本研究控制运输方式和屠宰前的击晕方式一致。而且随着数量遗传学理论的发展,育种学家借助一定的统计方法将性状的表型值进行剖分,并从中估计出可以真实遗传的部分,即育种值。因此,相较于猪早期育种时根据性状表型值的高低来选择试验群体的方法,基于育种值高低选择试验群体的方法可以更好地提高育种的准确性和效率。本研究基于EBV选择高、低滴水损失组各48个个体进行选择信号检测。通过研究发现,高、低EBV组苏淮猪的滴水损失存在极显著差异,进一步说明了本试验样品选择的可靠性。
3.2 影响猪滴水损失性状的受选择基因分析本研究使用两种单一的选择信号方法分别鉴别到了很多受选择的位点,其中单一选择信号方法得到的最显著SNP位点上、下游各50 kb的区域没有发现与已报道的影响猪滴水损失的QTL区域重合,并且附近区域通过基因富集也没有发现功能上可能与滴水损失有关的基因。本研究为了避免单个选择信号检测出现假阳性,选择对两种选择信号的结果进行合并分析的方法,以此来增加结果的可靠性。合并分析后,共发现了175个达到显著水平的SNPs,这些显著SNPs共涉及到73个基因,通过功能富集分析发现,许多基因与肌动蛋白、氧化应激、蛋白质磷酸化以及脂质合成有关。同时在175个显著SNPs中有27个SNPs位于15个与猪滴水损失相关的QTL上,并且涉及到了EZR、MRTFA、VKORC1L1基因,这些基因在功能上与肌动蛋白结合和细胞氧化应激相关。对上述3个分别位于EZR、MRTFA、VKORC1L1基因内含子上的rs80930355、rs340037952和rs320624660位点在苏淮猪全群内进行基因分型后,并与滴水损失进行关联分析。结果发现,rs340037952和rs320624660位点与苏淮猪群体的滴水损失存在显著关联,因此可以做为苏淮猪滴水损失选育提升的潜在分子标记。另外,发现有2个基因(MAP3K4、NFATC3)富集到了MAPK信号通路上,MAPK信号通路是已经报道过的在骨骼肌中产生氧化应激的通路之一。然而结果显示,富集到的与细胞氧化应激相关的条目以及MAPK信号通路并不显著,这可能是由于基因数目太少造成的。
动物屠宰后的一系列生理生化反应都会导致肌肉的滴水损失增加。目前,研究表明,肌肉静电荷、肌肉收缩(肌动蛋白和肌球蛋白相互作用)、蛋白质降解以及钙蛋白主要原酶系统以及氧化反应等均会引起宰后滴水损失的改变[30-32]。
邓婕等[33]和赵海洲等[34]的研究显示,PACRG基因编码的蛋白是一个E3泛素连接酶,它通过泛素-蛋白酶体系统来降解异常折叠蛋白,参与氧化应激及线粒体功能调控等多种途径,发挥保护功能; Neisch和Fehon[35]以及Maniti等[36]的研究表明,Ezrin(EZR) 为细胞膜-细胞骨架连接蛋白,EZR即能参与细胞的生长和信号转导,又可接受胞外信号,使细胞骨架重排; Lundquist等[37]认为,MRTFA编码的蛋白是与血清反应因子(SRF)转录因子相关联的转录辅激活子,在发育、形态发生和细胞迁移过程中控制调节细胞骨架基因的表达,SRF-MRTFA复合物活性对Rho-GTPase诱导的细胞球状肌动蛋白(G-actin)浓度变化作出反应,从而将细胞骨架基因表达与细胞骨架动力学耦合,MRTFA通过RPEL重复序列与G-肌动蛋白结合,调节MRTFA-SRF复合物的活性; Janji等[38]认为,L-质体蛋白是是肌动蛋白丝交联剂大家族的成员之一,L-质体蛋白的磷酸化将蛋白质从低活性状态转换为高活性状态,磷酸化L-质体蛋白可能作为一个整合的信号控制组装肌动蛋白细胞骨架和细胞运动的三维空间。因此,这些与肌动蛋白相关的基因可以作为影响滴水损失的候选基因。Westhofen等[39]认为,VKORC1L1与细胞抵抗自由基有关,VKORC1L1基因敲除后导致对氧化应激更高的敏感性和细胞活力降低; 该基因编码的蛋白功能都与细胞氧化应激相关,而屠宰后肌肉的氧化反应是造成肌肉滴水损失增加的主要因素,因此也可以作为影响苏淮猪滴水损失的候选基因。
4 结论综上所述,本研究使用Fst与iHS两种互补的检测方法在高、低滴水损失苏淮猪群体中找到175个受选择的SNPs,并依此鉴定到影响苏淮猪滴水损失的73个受选择的基因,通过功能注释发现,有5个基因是滴水损失的功能候选基因。其中4个候选基因(PACRG、EZR、MRTFA、LCP1)与肌肉中的肌动蛋白有关; 而VKORC1L1基因与氧化应激有关。同时,还分别在MRTFA和VKORC1L1基因上鉴定到与苏淮猪群体的滴水损失存在显著关联的位点:rs340037952和rs320624660。上述基因和SNPs的检测为未来解析猪滴水损失的遗传机制以及苏淮猪肉质的持续选育提供了参考依据。
| [1] |
王波, 罗海玲. 氧化反应对肌肉滴水损失的影响及抗氧化剂对其调控机制的研究进展[J]. 中国畜牧杂志, 2019, 55(6): 1-5. WANG B, LUO H L. Research progress on the effect of oxidation reaction on drip loss of muscle and the regulation mechanism of antioxidants[J]. Chinese Journal of Animal Science, 2019, 55(6): 1-5. (in Chinese) |
| [2] |
ZHAO X Y, WANG C, WANG Y P, et al. Comparative gene expression profiling of muscle reveals potential candidate genes affecting drip loss in pork[J]. BMC Genet, 2019, 20(1): 89. DOI:10.1186/s12863-019-0794-0 |
| [3] |
ZHAO X, WANG C, WANG Y, et al. Weighted gene co-expression network analysis reveals potential candidate genes affecting drip loss in pork[J]. Anim Genet, 2020, 51(6): 855-865. DOI:10.1111/age.13006 |
| [4] |
LI B J, LIU K Q, WENG Q N, et al. RNA-seq analysis reveals new candidate genes for drip loss in a Pietrain×Duroc×Landrace×Yorkshire population[J]. Anim Genet, 2016, 47(2): 192-199. DOI:10.1111/age.12401 |
| [5] |
VAN WIJK H J, ARTS D J G, MATTHEWS J O, et al. Genetic parameters for carcass composition and pork quality estimated in a commercial production chain[J]. J Anim Sci, 2005, 83(2): 324-333. DOI:10.2527/2005.832324x |
| [6] |
SUZUKI K, IRIE M, KADOWAKI H, et al. Genetic parameter estimates of meat quality traits in Duroc pigs selected for average daily gain, longissimus muscle area, backfat thickness, and intramuscular fat content[J]. J Anim Sci, 2005, 83(9): 2058-2065. DOI:10.2527/2005.8392058x |
| [7] |
MITCHELL G, HEFFRON J J. Porcine stress syndromes[J]. Adv Food Res, 1982, 28: 167-230. |
| [8] |
HAMILTON D N, ELLIS M, MILLER K D, et al. The effect of the halothane and rendement napole genes on carcass and meat quality characteristics of pigs[J]. J Anim Sci, 2000, 78(11): 2862-2867. DOI:10.2527/2000.78112862x |
| [9] |
MA J W, YANG J, ZHOU L S, et al. A splice mutation in the PHKG1 gene causes high glycogen content and low meat quality in pig skeletal muscle[J]. PLoS Genet, 2014, 10(10): e1004710. DOI:10.1371/journal.pgen.1004710 |
| [10] |
WRIGHT S. The genetical structure of populations[J]. Ann Eugen, 1951, 15(4): 323-354. |
| [11] |
VOIGHT B F, KUDARAVALLI S, WEN X Q, et al. A map of recent positive selection in the human genome[J]. PLoS Biol, 2006, 4(3): e72. DOI:10.1371/journal.pbio.0040072 |
| [12] |
TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. DOI:10.1093/genetics/123.3.585 |
| [13] |
NIELSEN R, WILLIAMSON S, KIM Y, et al. Genomic scans for selective sweeps using SNP data[J]. Genome Res, 2005, 15(11): 1566-1575. DOI:10.1101/gr.4252305 |
| [14] |
SUN H, WANG Z, ZHANG Z, et al. Genomic signatures reveal selection of characteristics within and between Meishan pig populations[J]. Anim Genet, 2018, 49(2): 119-126. DOI:10.1111/age.12642 |
| [15] |
张威. 基于重测序揭示猪基因组中的选择信号[D]. 合肥: 安徽农业大学, 2019. ZHANG W. Re-sequencing analysis reveals the selection signatures of the pig genome[D]. Hefei: Anhui Agricultural University, 2019. (in Chinese) |
| [16] |
吴林慧, 孙琦, 王荔茹, 等. 恩施黑猪基因组群体遗传学参数的估计与选择信号研究[J]. 畜牧兽医学报, 2019, 50(3): 485-494. WU L H, SUN Q, WANG L R, et al. A study of the population genetics parameters and selection signatures in Enshi black pig[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(3): 485-494. (in Chinese) |
| [17] |
LUKIĆ B, FERENČAKOVIĆ M, ŠALAMON D, et al. Conservation genomic analysis of the Croatian indigenous black Slavonian and Turopolje pig breeds[J]. Front Genet, 2020, 11: 261. DOI:10.3389/fgene.2020.00261 |
| [18] |
WANG B B, LI P H, ZHOU W D, et al. Association of twelve candidate gene polymorphisms with the intramuscular fat content and average backfat thickness of Chinese Suhuai pigs[J]. Animals (Basel), 2019, 9(11): 858. |
| [19] |
高琛, 韩萍萍, 王彬彬, 等. 苏淮猪肉滴水损失相关候选基因突变位点的鉴别[J]. 南京农业大学学报, 2021, 44(2): 338-345. GAO C, HAN P P, WANG B B, et al. Identification of candidate genes and variants related to muscle drip loss in Suhuai pig[J]. Journal of Nanjing Agricultural University, 2021, 44(2): 338-345. (in Chinese) |
| [20] |
MADSEN P, JENSEN J, LABOURIAU R, et al. DMU-a package for analyzing multivariate mixed models in quantitative genetics and genomics[C]//10th World Congress on Genetics Applied to Livestock Production. Vancouver: WCGALP, 2014: 18-22.
|
| [21] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [22] |
BROWNING B L, BROWNING S R. Genotype imputation with millions of reference samples[J]. Am J Hum Genet, 2016, 98(1): 116-126. DOI:10.1016/j.ajhg.2015.11.020 |
| [23] |
WEIR B S, COCKERHAM C C. Estimating F-statistics for the analysis of population structure[J]. Evolution, 1984, 38(6): 1358-1370. |
| [24] |
DANECEK P, AUTON A, ABECASIS G, et al. The variant call format and VCFtools[J]. Bioinformatics, 2011, 27(15): 2156-2158. DOI:10.1093/bioinformatics/btr330 |
| [25] |
TURNER S D. Qqman: an R package for visualizing GWAS results using Q-Q and manhattan plots[J]. J Open Source Softw, 2018, 3(25): 731. DOI:10.21105/joss.00731 |
| [26] |
SZPIECH Z A, HERNANDEZ R D. Selscan: an efficient multithreaded program to perform EHH-based scans for positive selection[J]. Mol Biol Evol, 2014, 31(10): 2824-2827. DOI:10.1093/molbev/msu211 |
| [27] |
WRIGHT S. Evolution and the genetics of populations, volume 4:variability within and among natural populations[M]. Chicago: The University of Chicago Press, 1984.
|
| [28] |
李开军, 侯黎明, 蒲广, 等. 基于全基因组Fst和nSL分析鉴别苏淮猪中性洗涤纤维表观消化率相关候选基因位点[J]. 畜牧兽医学报, 2021, 52(7): 1809-1819. LI K J, HOU L M, PU G, et al. Identification of candidate gene loci related to apparent NDF digestibility of Suhuai pigs based on Genome-wide Fst and nSL analyses[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(7): 1809-1819. (in Chinese) |
| [29] |
BU D C, LUO H T, HUO P P, et al. KOBAS-i: intelligent prioritization and exploratory visualization of biological functions for gene enrichment analysis[J]. Nucleic Acids Res, 2021, 49(W1): W317-W325. DOI:10.1093/nar/gkab447 |
| [30] |
张玉伟, 罗海玲, 贾慧娜, 等. 肌肉系水力的影响因素及其可能机制[J]. 动物营养学报, 2012, 24(8): 1389-1396. ZHANG Y W, LUO H L, JIA H N, et al. Effect factors of water holding capacity of meats and its potential mechanism[J]. Chinese Journal of Animal Nutrition, 2012, 24(8): 1389-1396. DOI:10.3969/j.issn.1006-267x.2012.08.001 (in Chinese) |
| [31] |
徐晨晨. 钙蛋白酶介导的日粮抗氧化剂降低羊肉滴水损失的机制[D]. 北京: 中国农业大学, 2018. XU C C. The mechanism of calpain-mediated dietary antioxidant reduce drip loss in lamb meat[D]. Beijing: China Agricultural University, 2018. (in Chinese) |
| [32] |
曾珍. 钙调蛋白酶2、20S蛋白酶体和组织蛋白酶B+L对宰后猪肉保水性的影响[D]. 雅安: 四川农业大学, 2018. ZENG Z. The effect of calpain 2, 20S proteasome and cathepsin B+L on postmortem pork water-holding capacity[D]. Ya'an: Sichuan Agricultural University, 2018. (in Chinese) |
| [33] |
邓婕, 李怡芳, 栗原博, 等. 线粒体损伤与修复在帕金森病中的作用[J]. 中国药理学通报, 2015, 31(7): 906-909, 910. DENG J, LI Y F, KURIHARA H, et al. Role of mitochondrial damage and repair in Parkinson's disease[J]. Chinese Pharmacological Bulletin, 2015, 31(7): 906-909, 910. DOI:10.3969/j.issn.1001-1978.2015.07.004 (in Chinese) |
| [34] |
赵海洲, 李军, 马延红, 等. Parkin共调基因调控功能与疾病研究进展[J]. 中国药理学通报, 2018, 34(10): 1333-1336. ZHAO H Z, LI J, MA Y H, et al. Research progress of Parkin co-regulated gene[J]. Chinese Pharmacological Bulletin, 2018, 34(10): 1333-1336. DOI:10.3969/j.issn.1001-1978.2018.10.001 (in Chinese) |
| [35] |
NEISCH A L, FEHON R G. Ezrin, Radixin and Moesin: key regulators of membrane-cortex interactions and signaling[J]. Curr Opin Cell Biol, 2011, 23(4): 377-382. DOI:10.1016/j.ceb.2011.04.011 |
| [36] |
MANITI O, CARVALHO K, PICART C. Model membranes to shed light on the biochemical and physical properties of Ezrin/Radixin/Moesin[J]. Biochimie, 2013, 95(1): 3-11. DOI:10.1016/j.biochi.2012.09.033 |
| [37] |
LUNDQUIST M R, STORASKA A J, LIU T C, et al. Redox modification of nuclear actin by MICAL-2 regulates SRF signaling[J]. Cell, 2014, 156(3): 563-576. DOI:10.1016/j.cell.2013.12.035 |
| [38] |
JANJI B, GIGANTI A, DE CORTE V, et al. Phosphorylation on Ser5 increases the F-actin-binding activity of L-plastin and promotes its targeting to sites of actin assembly in cells[J]. J Cell Sci, 2006, 119(9): 1947-1960. DOI:10.1242/jcs.02874 |
| [39] |
WESTHOFEN P, WATZKA M, MARINOVA M, et al. New insight into the function of VKORC1L1[J]. Hämostaseologie, 2010, 30(S1): S183. |
(编辑 郭云雁)



