肥胖已成为当今世界范围内的健康问题,在发达和发展中国家,肥胖的发生率都在急剧增加[1]。世界卫生组织(2016年)报告称,18岁及以上的人群中约有39%的个体超重[2]。肥胖是产生其他代谢性疾病的重要危险因素之一,如胰岛素抵抗、2型糖尿病、脂肪肝、动脉粥样硬化、高血压和中风等,这引起了公众的广泛关注[3-5]。肥胖的发生是一个复杂的过程,涉及遗传和环境因素(如饮食、食物成分和/或生活方式),普遍被认为是由能量摄入与消耗之间长期的失衡以及体内脂肪过度增加而引起的[6-7]。目前,越来越多的证据表明,肠道微生物及其细菌基因组(微生物组)会影响营养获取、能量调节和脂肪存储[8]。这些发现证明,肠道微生物在调节宿主能量代谢中发挥重要作用,并与肥胖症和相关代谢疾病的发生有关。
肠道微生物是一个复杂且动态的生态系统,与其宿主共同进化[9]。目前,肠道中的微生物群落被认为是一个“器官”,具有许多影响人类健康的代谢、免疫和内分泌样作用[10]。肠道微生物群被认为是影响肥胖的重要因素。肠道微生物对宿主肥胖作用的第一个证据来自对无菌(GF)动物的研究,即无细菌且在无菌隔离器中繁殖的动物。1983年,Wostmann等[11]观察到无菌啮齿动物需要比传统的啮齿动物(拥有自身的微生物群)多30%的热量来维持体重。肥胖个体和较瘦个体间远端肠道微生物组的差异提示,肥胖与两个优势细菌门,即拟杆菌门和厚壁菌门的相对比例有关[12]。然而,这些研究只是简单地比较了肥胖和较瘦个体之间肠道微生物的差异,忽略了肠道微生物的动态变化。本研究以广西巴马小型猪为动物模型,对其进行高脂高糖饮食干预,采用16S rRNA高通量测序方法,以探究在高脂高糖饮食过程中肠道微生物的动态变化,为研究小型猪生长发育过程中肠道微生物的动态变化提供理论依据。
1 材料与方法 1.1 试验动物及样品收集本研究选择7头体重和日龄相近的雌性广西巴马小型猪近交系个体,随机分为普通饮食组(CN组)3头和高脂高糖饮食组(HFD组)4头,每头个体在统一断奶和保育后转入育肥舍分别单笼饲养。试验前,所有个体的饲养环境及饮食条件一致,并且未使用过抗生素。适应饲养1周后,对照组饲喂基础饲粮,基础饲粮成分见表 1;高脂高糖饲粮配方为:53%基础饲粮+37%蔗糖+10%油脂,饮食干预为期30 d。在此期间,猪可自由采食和饮水。分别在第0、7、15和30天时收集个体新鲜粪便于1.5 mL无菌冻存管中,立刻放入液氮速冻,后置于-80 ℃冰箱保存,待测。在收集新鲜粪便的同时,分别称量CN组和HFD组个体体重。在试验结束时测量CN组和HFD组个体的体长和腹围数据。
|
|
表 1 CN组基础饲粮配方及营养成分 Table 1 Basic diet formula and nutritional components of CN group |
根据E.Z.N.A.® soil DNA kit(Omega Bio-tek, Norcross, GA, U.S.)说明书进行粪便样品微生物群落总DNA抽提,使用1%的琼脂糖凝胶电泳检测DNA的质量; 使用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)对16S rRNA基因V3-V4可变区进行PCR扩增,扩增程序如下:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),然后72 ℃稳定延伸10 min,最后在4 ℃进行保存(PCR仪:ABI GeneAmp® 9700型)。PCR反应体系为:5×TransStart FastPfu缓冲液4 μL,2.5 mmol·L-1 dNTPs 2 μL,上游引物(5 μmol·L-1)0.8 μL,下游引物(5 μmol·L-1)0.8 μL,TransStart FastPfu DNA聚合酶0.4 μL,模板DNA 10 ng,ddH2O补足至20 μL。每个样本3个重复。扩增产物经凝胶电泳检测后,送上海美吉生物医药科技有限责任公司使用Illumina公司的Miseq PE300平台进行测序。
1.3 生物信息分析使用fastp软件[13]原始测序序列进行质控,使用FLASH软件[14]进行拼接; 基于默认参数,使用QIIME2流程中的DADA2插件[15]对质控拼接之后的优化序列进行降噪处理。DADA2降噪处理之后的序列通常被称为ASV(即扩增子序列变体)。基于Sliva 16S rRNA数据库(v 138),使用QIIME2中的Naive bayes分类器对ASVs进行物种分类学分析。
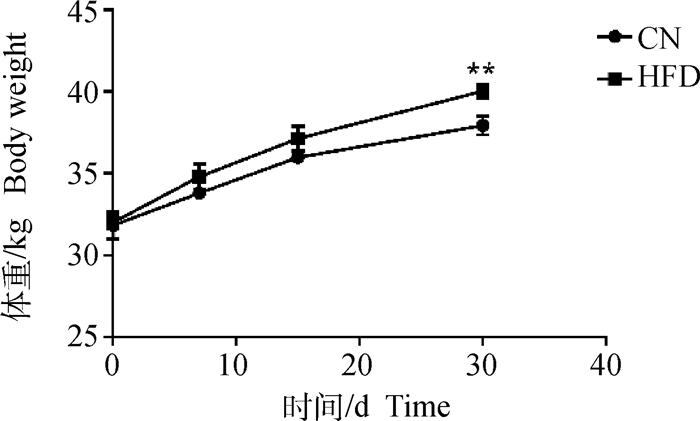
2 结果 2.1 高脂高糖饮食对小型猪体重及体尺的影响在试验0、7、15和30 d分别称量CN组和HFD组个体体重,发现,在试验开始的0、7和15 d时,CN组与HFD组之间个体体重没有显著性差异(P>0.05);在30 d时,CN组的体重为(37.93±0.58) kg,HFD组的体重为(40.03±0.43) kg,两组间存在显著性差异(P≤0.05, 图 1),而动物的体长、腹围等指标差异不显著(P>0.05, 表 2)。
|
**.P < 0.05 图 1 CN组和HFD组猪的体重变化情况 Fig. 1 Body weight change of pigs in CN group and HFD group |
|
|
表 2 CN组和HFD组猪的30 d体重与体尺情况 Table 2 Body weight and body size of pigs in CN and HFD groups at 30 d |
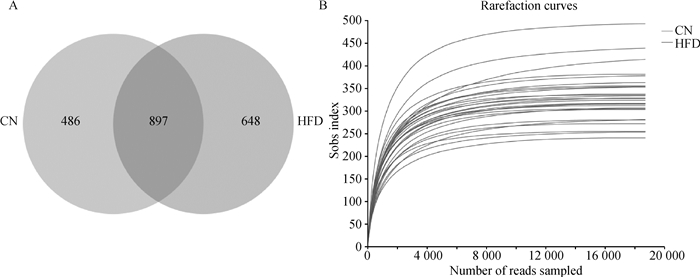
对CN组和HFD组小型猪肠道微生物16S rRNA基因的V3-V4区进行高通量测序,共获得524 524条有效序列。使用QIIME2流程中的DADA2插件对质控拼接之后的优化序列进行降噪处理,共获得2 031个ASVs,其中CN组特有486个ASVs,HFD组特有648个ASVs,两组共有897个ASVs(图 2A); 分为14个门、22个纲、47个目、78个科、191个属和350个种。基于CN组和HFD组样品的Sobs指数得到所有样品的稀释性曲线(图 2B)。结果显示,随着测序量的增加,每个样品的稀释性曲线逐渐趋于平缓,说明样本的测序深度和覆盖范围足够且物种丰富,样品适合下一步的分析。
|
图 2 CN组和HFD组ASV水平韦恩图(A)和Sobs指数曲线(B) Fig. 2 Venn diagram (A) and Sobs index curves (B) of ASV level in CN group and HFD group |
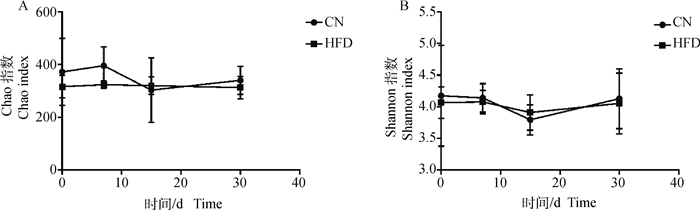
以细菌物种的多样性指数(Shannon)和丰富度指数(Chao)作为猪肠道微生物Alpha多样性的参数(图 3)。发现CN组和HFD组在0、7、15和30 d 4个不同时期的Shannon指数和Chao指数整体变化趋势相似,但是HFD组的多样性指数和丰富度指数在不同时期普遍低于CN组(第15天时除外)。
|
图 3 CN组和HFD组不同时期猪肠道微生物的Alpha多样性 Fig. 3 Alpha diversity of intestinal microbiota of pigs in CN group and HFD group at different periods |
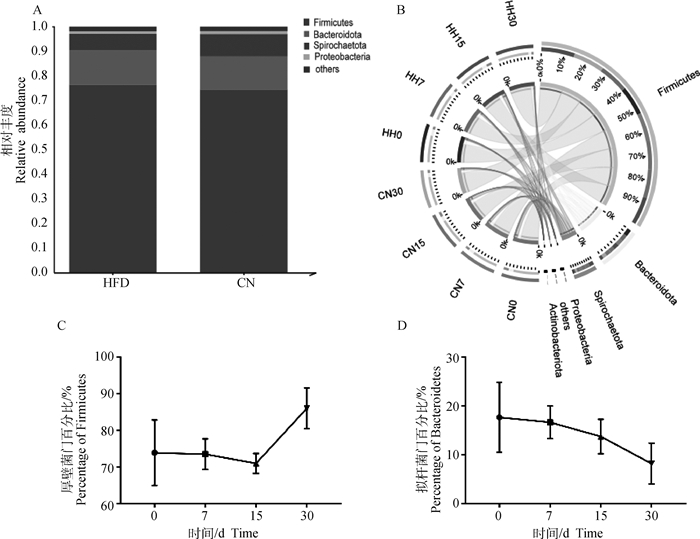
在门水平上,检测到CN组和HFD组的优势门为Firmicutes、Bacteroidetes和Spirochaetota,在两组间的比例略有差异,且均占据了各组总量的95%以上(图 4A)。随着试验的进展,CN组和HFD组在不同时期优势门的比例发生改变(图 4B),这可能是由于高脂高糖饮食所导致的。随即,本研究分析了Firmicutes和Bacteroidetes在高脂高糖饮食饲喂不同时期的变化情况,发现,Firmicutes丰度在HFD组中整体呈现上升趋势并在30 d时达到最高值(图 4C),而Bacteroidetes丰度在HFD组中整体呈现下降趋势并在30 d时达到最低值(图 4D)。
|
A. 物种组成条形图; B. 物种组成和变化Circos图。CN0、CN7、CN15和CN30分别表示CN组第0、7、15和30天; HH0、HH7、HH15和HH30分表示HFD组第0、7、15天和30天,下同; C. HFD组Firmicutes变化折线图; D. HFD组Bacteroidetes变化折线图 A. Species composition bar chart; B. Circos diagram of species composition and change. CN0, CN7, CN15 and CN30 mean the 0, 7, 15 and 30 day of CN group, respectively; HH0, HH7, HH15 and HH30 mean the 0, 7, 15 and 30 day of HFD group, respectively. The same as below; C. Firmicutes change line chart; D. Bacteroidetes change line chart 图 4 CN组和HFD组猪肠道菌群门水平物种组成和变化趋势 Fig. 4 Species composition and variation trends at phylum level of intestinal microbiota of pigs in CN group and HFD group |
在属水平上,共鉴定到180个属水平菌(图 5A),其中CN组和HFD组共有的属有152个,CN组有17个特有的属,HFD组有11个特有的属。CN组和HFD组的优势菌属为Clostridium_sensu_stricto_1和Trepponma(图 5B)。HFD组在试验的不同时期属水平的微生物组成表现出了动态变化,比如Lactobacillus在HFD组丰度逐渐减少而Ruminococcus丰度则不断升高(图 5C、D)。

|
A. 属水平韦恩图; B. 物种组成条形图; C. HFD组Lactobacillus变化折线图; D. HFD组Ruminococcus变化折线图 A. At the genus level form Venn diagram; B. Species composition bar chart; C. Lactobacillus change line chart of HFD group; D.Ruminococcus change line chart of HFD group 图 5 CN组和HFD组猪肠道菌群属水平物种组成和变化趋势 Fig. 5 Species composition and variation trends at genus level of intestinal microbiota of pigs in CN group and HFD group |
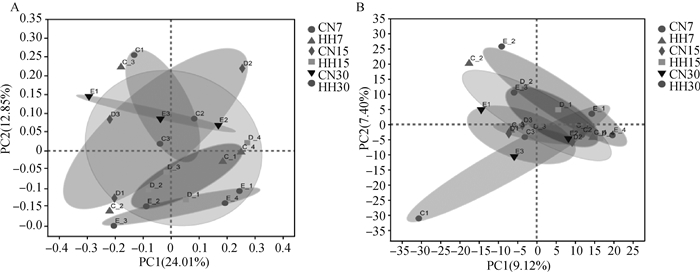
通过PCoA主坐标分析和PCA主成分分析比较CN组和HFD组在7、15和30 d粪便样品微生物的结构相似性(图 6)。结果发现,CN组和HFD组在7 d时有一部分样品交叉聚集到一起,随着饮食干预时间的进展,各组样品按照饮食和干预时间的区分来越明显,在30 d时CN组和HFD组能够明显区分。以上结果说明,CN组和HFD组不同时期样本中的微生物组成存在差异。
|
图 6 饮食干预不同时期ASV水平的PCoA分析(A)和PCA分析(B) Fig. 6 PCoA analysis (A) and PCA analysis (B) of ASV levels in different periods of dietary intervention |
采用LEfSe分析CN组和HFD组之间微生物组成差异性。分支图显示了CN组和HFD组在门、纲、目、科、属分类水平上最丰富的微生物群体(图 7A)。在两组间共发现20个差异项,其中,12个在CN组中富集,8个在HFD组中富集。LDA分析结果(图 7B)显示,12个在CN组中富集的菌群分别为g__Lactobacillus、f__Lactobacillaceae、o__Lactobacillales、f__p_251_o5、g__norank_f__p_251_o5、g__norank_f__norank_o__Oscillospirales、f__norank_o__Oscillospirales、g__Lachnospiraceae_UCG_007、f__unclassified_c__Bacilli、g__unclassified_c__Bacilli、o__unclassified_c__Bacilli和p__Verrucomicrobiota,8个在HFD组中富集的菌群分别为f__Lachnospiraceae、o__Lachnospirales、g__Peptococcus、f__Coriobacteriaceae、g__Collinsella、g__Senegalimassilia、g__Lachnospiraceae_AC2044_group和g__Subdoligranulum。

|
A. LEfSe分析分支图; B. LDA判别图 A. LEfSe analysis branch diagram; B. Linear discriminant analysis (LDA) diagram 图 7 CN组和HFD组小型猪肠道组微生物组成差异 Fig. 7 Differences in microbial composition of mini-pig intestine between CN group and HFD group |
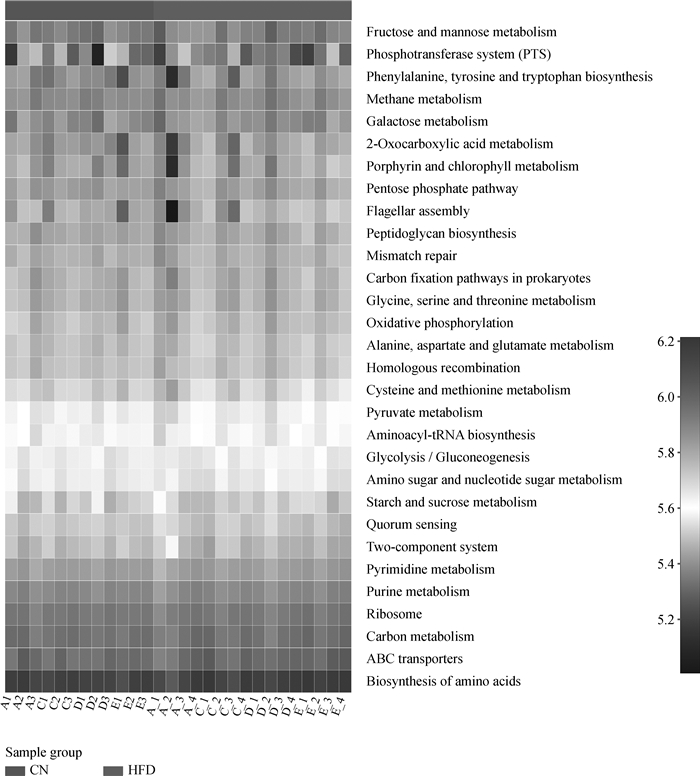
使用PICRUSt2将ASVs序列与其内部的参考序列比对,确定每个样本基因家族的丰度; 将基因家族信息与KEGG数据库(Kyoto Encyclopedia of Genes and Genomes,http://www.genome.jp/kegg/)进行比对,获得KEGG的功能信息和丰度信息(图 8)。结果发现,CN组和HFD组之间肠道微生物基因功能无显著性差异,均主要富集在氨基酸的生物合成、淀粉与蔗糖的代谢、氨基糖和核苷酸糖代谢以及糖酵解和糖异生等方面,预测可能富含能够将相关蛋白分解、转运和纤维素降解酶的相关基因。
|
图 8 CN组和HFD组KEGG三级代谢通路热图 Fig. 8 Heat map of KEGG tertiary metabolic pathway in CN and HFD groups |
广西巴马小型猪是本研究团队三代科研人员经过30余年的品系培育获得的小型猪实验动物近交品系,具有体型矮小、性成熟早、遗传相似性和稳定性高、便于实验操作等优点[16]。此外,猪与人的生理结构、器官大小和饮食结构较为接近,加之广西巴马小型猪的单拷贝同源基因比小鼠更加接近于人(除猕猴等灵长类外),使其更适合作为医学模型用于人类疾病研究[17]。肠道微生物已经成为近年来的研究热点,作为一个动态变化的生态系统,受多种因素的影响,其中,饮食被认为是塑造肠道菌群结构的主要驱动力之一[18]。本研究中,巴马小型猪连续饲喂高糖高脂饲粮1个月,与CN组相比,HFD组动物体重显著增加,而体长、腹围等指标差异不显著,说明高糖高脂饮食导致能量过剩,诱发小型猪朝肥胖方向发展,与相磊等[19]对高糖高脂饲料诱导的小型猪肥胖的研究结果一致。所以,本研究以广西巴马小型猪为实验动物,研究饲喂高脂高糖饮食过程中肠道微生物的动态变化是合理的,突破了以往研究只单纯针对某一时间点的局限。
在本研究中,采用16S rRNA基因高通量测序方法分析了由高脂高糖饮食组(HFD组)和普通饮食组(CN组)的粪便微生物组成的动态变化与组间差异。本研究采用目前最新的QIIME2平台进行分析,使用DADA2插件对质控拼接之后的优化序列进行降噪处理,共获得了2 031个ASVs。DADA2与传统的基于OTUs的分析方法不同,其不再以97%序列相似度进行聚类而只是进行去重(相当于以100%相似度聚类),从而得到“扩增序列变体”ASVs[15]。这大大提高了数据精确度和物种分辨率,比传统OTUs聚类得到的数据更加理想和准确。
基于微生物16S rRNA基因测序结果,对不同干预时期HFD组和CN组进行多样性分析。Beta多样性分析发现,饮食干预7 d时导致微生物组成发生了较小程度的变化,但在第30天时,HFD组相比CN组的变化却很显著,这表明高脂高糖饮食可能导致肠道菌群的组成逐步累积改变。而Alpha多样性分析发现,HFD组肠道微生物的多样性普遍低于CN组,但两组间并没有达到显著性的差异,表明饮食诱导的肠道微生物群组成的变化可能是由较低水平细菌种类的丰度变化引起的。
随着高脂高糖饮食干预时间的增加,观察到了肠道微生物组的总体变化趋势。在门水平,高脂高糖饮食组个体肠道微生物中,Firmicutes丰度增加而Bacteroidetes丰度逐渐下降。先前的研究已发现,肥胖与厚壁菌门丰度增加和拟杆菌门丰度降低有关[12, 20]。在人体相关研究中也得到了类似的结果,Ley等[21]对12名肥胖者进行热量限制1年,发现在开始减肥饮食之前,肥胖者肠道微生物中的拟杆菌门比例(3%)明显少于瘦对照组(25%),厚壁菌门比例相应高于正常体重者,而肥胖者经过减肥后其肠道微生物中拟杆菌门的比例在1年内从3%增加到18%,这是对限制能量摄入的反应。总之,这些在小鼠和人身上得到的结果表明,肥胖改变了肠道微生物的结构,而通过调节肠道微生物的组成可能有助于改善肥胖,这为治疗肥胖等疾病提供了新策略。然而,有几项研究报道称,肥胖与较瘦受试者的肠道中厚壁菌门(Firmicutes)与拟杆菌门(Bacteroidetes)丰度比例与以上结果存在差异,甚至发现二者存在相反的关系[22-23]。因此有人提出,这些肠道微生物的组成结构在门水平的变化目前仍不能被认为是肥胖发生的标志物,应该在更低的微生物分类学水平上寻找与肥胖相关的细菌属甚至特定的细菌种类。
在属水平上,本研究发现Lactobacillus的丰度在HFD组随高能饮食诱导逐渐减少而Ruminococcus丰度则不断升高。乳杆菌属(Lactobacillus)和双歧杆菌属(Bifidobacterium)是研究最多的益生菌,在实验动物中进行的多数研究都清楚地表明,服用益生菌可以有效预防和治疗肥胖症。有研究发现,向小鼠单独或联合饲喂弯曲乳杆菌HY7601和植物乳杆菌KY1032可限制脂质在脂肪组织和肝中的积累,血浆和肝中的胆固醇含量也显著降低[24]。在使用副干酪乳杆菌CNCM I-4270和鼠李糖乳杆菌CNCM I-3690饲喂时,获得了相似的结果[25]。但本研究仍必须谨慎评估从这些研究得出的所有结论,因为在评估益生菌对动物肥胖程度影响的试验中,经常使用不同类型的日粮喂养不同年龄的动物,而且益生菌的剂量和添加时间也不统一。种种迹象表明,在属水平上,肠道微生物的组成可能会影响动物从饮食中摄取能量的效率,进而影响肥胖的发生与发展。Ruminococcus是1948年由Sijpesteijn[26]从牛瘤胃中首次分离出来的,是核心肠道微生物组中的主要成员之一。在草食性反刍动物中,Ruminococcus属微生物可以降解和发酵膳食纤维素,并将其转化为短链脂肪酸(SCFA),对动物的能量代谢至关重要[27-28]。短链脂肪酸不仅可以促进能量吸收及肝中多余的脂肪生成,同时还可以减少炎症发生,促进肠上皮的屏障完整性和增强动物的饱腹感[29]。Den等[30]报道了短链脂肪酸的有益代谢作用,其可以防止高脂饮食(HFD)引起的肥胖并能够改善胰岛素的敏感性。然而,本研究发现,产SCFA的细菌随着高脂高糖饮食的干预在HFD组逐渐富集。有研究表明,肥胖个体的肠道微生物群富集了参与SCFA生成的代谢途径,超重和肥胖个体的粪便中短链脂肪酸的浓度高于较瘦个体[31]。这些存在差异的结果说明,肠道中SCFA的含量以及肠道微生物的组成结构在肥胖发生过程中的作用方式可能十分复杂。
利用LEfSe分析,也发现了CN组和HFD组之间显著差异的肠道微生物。其中,g__Lactobacillus、f__p_251_o5和g__unclassified_c__Bacilli等13个菌群在CN组中富集; f__Lachnospiraceae、g__Collinsella、g__Peptococcus、g__Subdoligranulum和g__Senegalimassilia等7个菌群在HFD组中富集。柯林斯菌属(Collinsella)被认为是2型糖尿病患者中富含的潜在促进炎症的肠道微生物成分,可导致肠道中的SCFA迅速下降[32]。Liu等[33]认为,Collinsella可作为动脉粥样硬化患者的生物标记物,与成年蒙古族人心脑血管疾病的高死亡率相关,说明高脂高糖饮食导致肠道中有害微生物的增加。Mach等[34]研究发现,消化球菌属(Peptococcus)在肥胖猪中含量更高。Kiros等[35]发现,饲喂酵母的断奶猪生长性能更好,且消化球菌属丰度更高,表明消化球菌属对猪的增重有积极作用,这与本研究结果一致。这些微生物可能作为与肥胖发展相关的潜在微生物标志,但还需要进一步的研究来确定潜在微生物标志在肥胖发展中发挥的作用,如利用粪菌移植等方法验证候选微生物是否能调节无菌动物的体重水平。
总而言之,本研究结果表明,由高脂高糖饮食组(HFD组)和普通饮食组(CN组)的肠道微生物组成与结构在不同干预时期呈现动态的变化和差异,表明特定的细菌物种或物种组合可能直接或间接影响肥胖的发生。这些结果增加了对肠道微生物菌群与宿主的相互作用的认识,并将促进通过综合因素调控肠道微生物来治疗肥胖及其并发症方法的研究与应用。
4 结论本研究采用微生物16S rRNA基因高通量测序方法,对广西巴马小型猪高脂高糖饮食组(HFD组)和普通饮食对照组(CN组)的肠道微生物组成的动态变化进行分析。结果发现,在门水平,高脂饮食的干预导致HFD组个体肠道微生物中Firmicutes丰度增加而Bacteroidetes丰度逐渐下降; 在属水平,HFD组中Lactobacillus丰度逐渐减少而Ruminococcus丰度则不断升高,并筛选了g__Peptococcus、g__Collinsella和g__Senegalimassilia等可能与高能饮食密切相关。本研究为利用小型猪动物模型研究人类高能量饮食对肠道微生物的影响提供一定的理论依据。
| [1] |
NG M, FLEMING T, ROBINSON M, et al. Global, regional, and national prevalence of overweight and obesity in children and adults during 1980-2013: a systematic analysis for the Global Burden of Disease Study 2013[J]. Lancet, 2014, 384(9945): 766-781. DOI:10.1016/S0140-6736(14)60460-8 |
| [2] |
WHO. Obesity and overweight[EB/OL]. (2020-04-01)[2021-05-20]. https://www.who.int/zh/news-room/fact-sheets/detail/obesity-and-overweight.
|
| [3] |
FLEGAL K M, GRAUBARD B I, WILLIAMSON D F, et al. Cause-Specific Excess Deaths Associated With Underweight, Overweight, and Obesity[J]. JAMA, 2007, 298(17): 2028-2037. DOI:10.1001/jama.298.17.2028 |
| [4] |
HOTAMISLIGIL G S. Inflammation and metabolic disorders[J]. Nature, 2006, 444(7121): 860-867. DOI:10.1038/nature05485 |
| [5] |
NAHUM M S, VANIA C R, OSCAR R P, et al. New Aspects of Lipotoxicity in Nonalcoholic Steatohepatitis[J]. Int J Mol Sci,, 2018, 19(7): 2034. DOI:10.3390/ijms19072034 |
| [6] |
MCPHEE J B, SCHERTZER J D. Immunometabolism of obesity and diabetes: microbiota link compartmentalized immunity in the gut to metabolic tissue inflammation[J]. Clin Sci (Lond), 2015, 129(12): 1083-1096. DOI:10.1042/CS20150431 |
| [7] |
HILL J O. Understanding and addressing the epidemic of obesity: an energy balance perspective[J]. Endocr Rev, 2006, 27(7): 750-761. DOI:10.1210/er.2006-0032 |
| [8] |
ROSENBAUM M, KNIGHT R, LEIBEL R. The gut microbiota in human energy homeostasis and obesity[J]. Trends Endocrinol Metab, 2015, 26(9): 493-501. DOI:10.1016/j.tem.2015.07.002 |
| [9] |
LEY R, HAMADY M, LOZUPONE C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883): 1647-1651. DOI:10.1126/science.1155725 |
| [10] |
O'HARA A M, SHANAHAN F. The gut flora as a forgotten organ[J]. EMBO Rep, 2006, 7(7): 688-693. DOI:10.1038/sj.embor.7400731 |
| [11] |
WOSTMANN B S, LARKIN C, MORIARTY A, et al. Dietary intake, energy metabolism, and excretory losses of adult male germfree Wistar rats[J]. Lab Anim Sci, 1983, 33(1): 46-50. |
| [12] |
LEY R E, BÄCKHED F, TURNBAUGH P, et al. Obesity alters gut microbial ecology[J]. Proc Natl Acad Sci U S A, 2005, 102(31): 11070-11075. DOI:10.1073/pnas.0504978102 |
| [13] |
CHEN S F, ZHOU Y Q, CHEN Y R, et al. fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34(17): i884-i890. DOI:10.1093/bioinformatics/bty560 |
| [14] |
MAGOČ T, SALZBERG S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [15] |
CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nat Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [16] |
王爱德, 兰干球, 郭亚芬. 广西巴马小型猪的培育[J]. 实验动物科学, 2010, 27(1): 60-63. WANG A D, LAN G Q, GUO Y F. Genetic Breeding of Guangxi Bama mini-pig[J]. Laboratory Animal Science, 2010, 27(1): 60-63. (in Chinese) |
| [17] |
张笠. 广西巴马小型猪全基因组测序及分析研究[D]. 南宁: 广西大学, 2019. ZHANG L. Genome sequencing and research of Bama mini-pig[D]. Nanning: Guangxi University, 2019. (in Chinese) |
| [18] |
DONALDSON G, LEE S, MAZMANIAN S. Gut biogeography of the bacterial microbiota[J]. Nat Rev Microbiol, 2016, 14(1): 20-32. DOI:10.1038/nrmicro3552 |
| [19] |
相磊, 陈华, 赵德明. 高糖高脂饲料诱导的小型猪肥胖模型[J]. 实验动物科学, 2019, 36(4): 1-5, 9. XIANG L, CHEN H, ZHAO D M. High fat and high sugar diet induced the fat model of Bama miniature pigs[J]. Laboratory Animal Science, 2019, 36(4): 1-5, 9. DOI:10.3969/j.issn.1006-6179.2019.04.001 (in Chinese) |
| [20] |
HOOPER L V, WONG M H, THELIN A, et al. Molecular analysis of commensal host-microbial relationships in the intestine[J]. Science, 2001, 291(5505): 881-884. DOI:10.1126/science.291.5505.881 |
| [21] |
LEY R E, TURNBAUGH P J, KLEIN S, et al. Microbial ecology: human gut microbes associated with obesity[J]. Nature, 2006, 444(7122): 1022-1023. DOI:10.1038/4441022a |
| [22] |
JUMPERTZ R, LE D S, TURNBAUGH P J, et al. Energy-balance studies reveal associations between gut microbes, caloric load, and nutrient absorption in humans[J]. Am J Clin Nutr, 2011, 94(1): 58-65. DOI:10.3945/ajcn.110.010132 |
| [23] |
DUNCAN S H, LOBLEY G E, HOLTROP G, et al. Human colonic microbiota associated with diet, obesity and weight loss[J]. Int J Obes (Lond), 2008, 32(11): 1720-1724. DOI:10.1038/ijo.2008.155 |
| [24] |
YOO S R, KIM Y J, PARK D Y, et al. Probiotics L. plantarum and L. curvatus in combination alter hepatic lipid metabolism and suppress diet-induced obesity[J]. Obesity (Silver Spring, Md), 2013, 21(12): 2571-2578. DOI:10.1002/oby.20428 |
| [25] |
WANG J J, TANG H, ZHANG C H, et al. Modulation of gut microbiota during probiotic-mediated attenuation of metabolic syndrome in high fat diet-fed mice[J]. ISME J, 2015, 9(1): 1-15. DOI:10.1038/ismej.2014.99 |
| [26] |
SIJPESTEIJN A K. Cellulose-decomposing bacteria from the rumen of cattle[J]. Antonie van Leeuwenhoek, 1949, 15(1): 49-52. DOI:10.1007/BF02062631 |
| [27] |
QIN J J, LI R Q, RAES J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [28] |
FLINT H J, BAYER E A, RINCON M T, et al. Polysaccharide utilization by gut bacteria: potential for new insights from genomic analysis[J]. Nat Rev Microbiol, 2008, 6(2): 121-131. DOI:10.1038/nrmicro1817 |
| [29] |
MORRISON D J, PRESTON T. Formation of short chain fatty acids by the gut microbiota and their impact on human metabolism[J]. Gut Microbes, 2016, 7(3): 189-200. DOI:10.1080/19490976.2015.1134082 |
| [30] |
DEN BESTEN G, BLEEKER A, GERDING A, et al. Short-chain fatty acids protect against high-fat diet-induced obesity via a PPARγ-Dependent Switch From Lipogenesis to Fat Oxidation[J]. Diabetes, 2015, 64(7): 2398-2408. DOI:10.2337/db14-1213 |
| [31] |
TREMAROLI V, BÄCKHED F. Functional interactions between the gut microbiota and host metabolism[J]. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [32] |
CANDELA M, BIAGI E, SOVERNI M, et al. Modulation of gut microbiota dysbioses in type 2 diabetic patients by macrobiotic Ma-Pi 2 diet[J]. Br J Nutr, 2016, 116(1): 80-93. DOI:10.1017/S0007114516001045 |
| [33] |
LIU W J, ZHANG J C, WU C Y, et al. Unique Features of Ethnic Mongolian Gut Microbiome revealed by metagenomic analysis[J]. Sci Rep, 2016, 6(1): 34826. DOI:10.1038/srep34826 |
| [34] |
MACH N, BERRI M, ESTELL É J, et al. Early-life establishment of the swine gut microbiome and impact on host phenotypes[J]. Environ Microbiol Rep, 2015, 7(3): 554-569. DOI:10.1111/1758-2229.12285 |
| [35] |
KIROS T G, DERAKHSHANI H, PINLOCHE E, et al. Effect of live yeast Saccharomyces cerevisiae (Actisaf Sc 47) supplementation on the performance and hindgut microbiota composition of weanling pigs[J]. Sci Rep, 2018, 8(1): 5315. DOI:10.1038/s41598-018-23373-8 |
(编辑 范子娟)



