2. 信阳师范学院生命科学学院,信阳 464000;
3. 广西大学动物科学技术学院,南宁 530000
2. College of Life Sciences, Xinyang Normal University, Xinyang 464000, China;
3. College of Animal Science and Technology, Guangxi University, Nanning 530000, China
中国拥有丰富的水牛资源。据FAO统计,2019年中国水牛存栏量位于世界第三,仅次于印度和巴基斯坦。随着农业机械化程度的提高,水牛由过去的役用生产逐渐向乳肉兼用的方向发展[1],因此通过提高水牛肉品质,对其品种进行改良十分重要。肌内脂肪(intramuscular fat,IMF)含量决定牛肉的口感、风味及多汁性,是影响肉类产量和品质的重要因素之一。
脂肪沉积是极其复杂的生理过程,受以PPARG和C/EBPα为核心的复杂网络调控[2]。除了编码RNA,目前已有研究表明非编码RNA也参与脂肪沉积过程[3-5]。而环状RNA(circular RNAs,circRNAs)与传统的线性RNA不同,是一类不具有5′末端帽子和3′末端poly(A)尾巴以共价键形成环形结构的非编码RNA。随着RNA测序和生物信息技术的快速发展,circRNA在许多物种中得到鉴定[6-8],且发现参与包括脂肪生成[9-11]在内的多种生物学过程[12-14]。近年来,已有研究对人[15]、小鼠[16]、猪[9]和水牛[17]等脂肪组织或者脂肪相关细胞进行circRNA转录组测序,获得circRNA的表达谱,从而筛选潜在影响脂肪沉积的功能性circRNA。
前期对水牛脂肪组织进行转录组测序分析发现,N-肉豆蔻酰基转移酶1(N-myristoyltransferase 1,NMT1)产生的circRNA19:45387150|45389986可能参与脂质代谢[17]。为了探究该circRNA在脂肪沉积中所发挥功能,本研究通过确定circNMT1真实性,对其进行核质定位,明确时空表达模式,进一步探究其对3T3-L1细胞和水牛脂肪细胞脂质生成的影响。该研究为提高水牛肉品质提供分子理论基础。
1 材料与方法 1.1 试验材料本研究分别采集3头中国沼泽水牛的心、肝、脾、肺、肾和背最长肌、背部皮下脂肪组织样本。水牛集中饲养于信阳水牛养殖场(中国,河南,信阳),以相同日粮和管理标准进行饲养,水牛在6月龄时断奶,30月龄时屠宰。屠宰后立即对各组织进行取样,并在液氮中冷冻带回实验室,于-80 ℃冰箱中保存备用。试验中的3T3-L1细胞购于上海ATCC细胞库。对新鲜背部皮下脂肪组织取样,将其保存在含有1%链霉素和青霉素的PBS中,并带回实验室进行脂肪细胞的分离和培养。
1.2 主要试剂RNAiso Plus、PrimeScriptTM RT Master Mix、TB GreenTM Premix Ex TaqTM Ⅱ均购于TaKaRa公司;Lucigen RNR07250 Ribonuclease R kit(RNase R)购自Lucigen-Simplifying Genomics公司;PARIS kit购自Life Technologies公司;Lipofectamine 3000购自Invitrogen公司;2×Power Raq PCR Master购自Bioteke公司;质粒小提试剂盒和胶回收试剂盒购于天根生物科技有限公司;DMEM高糖培养基、青霉素/链霉素抗体、胰酶均购于Hyclone公司;罗格列酮、地塞米松、IBMX、胰岛素和油红O均购自Sigma公司。
1.3 引物的设计与合成根据转录组测序分析获得的circRNA序列信息与Ensembl对比,获得对应的序列。对向引物用以鉴定circRNA接口序列的存在,以及作为定量引物进行定量检测。使用NCBI(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)和Primer5.0进行引物设计(表 1)。
|
|
表 1 qRT-PCR的引物信息 Table 1 Primer information for qRT-PCR |
简单来说,将水牛背部脂肪组织采集后,用含1%链霉素和青霉素的PBS进行洗涤,剥去脂肪组织外表皮,同时剔除组织样品中肉眼可见的结缔组织及血管,将脂肪组织分离成黄豆大小(直径约1~3 cm)的组织块,尽量均匀种植到90 mm细胞培养皿中,将培养皿倒置放入37 ℃、5% CO2的细胞培养箱培养,6 h后,加入基础培养基(20% FBS血清+ 1%链霉素和青霉素+DMEM高糖培养基)正置培养7~15 d。待游离出细胞后,剔除组织块进行消化离心,将细胞分离出单独进行培养。当细胞密度达到80%~90%,将基础培养基更换为诱导培养基(基础培养基+1 μmol·L-1地塞米松+0.5 mmol·L-1 IBMX+0.01 mg·mL-1胰岛素+1 μmol·L-1罗格列酮),对其进行诱导分化。诱导2 d后,将诱导培养基更换为维持培养基(基础培养基+0.01 mg·mL-1胰岛素+1 μmol·L-1罗格列酮),每2 d更换1次维持培养基,直至诱导分化为成熟的脂肪细胞(诱导6~10 d左右)。详细过程见文献[18]。
1.5 RNA的提取、cDNA的合成、半定量PCR和实时荧光定量PCR(qRT-PCR)扩增组织和细胞的总RNA过程与检测方法参考文献[18],水牛脂肪细胞的细胞质、细胞核RNA的提取与分离参考文献[19]。cDNA的合成主要是用PrimeScriptTM RT Master Mix试剂盒进行反转录,反转录体系(20.0 μL):5×PrimerScript RT Master Mix I 4.0 μL,RNA 1 000 ng, RNase Free ddH2O添加至20 μL;反应程序:37 ℃ 15 min;85 ℃ 5 s;4 ℃保存。半定量PCR的过程参考文献[20]。qRT-PCR以GAPDH和β-actin作为内参基因。此外,β-actin也被用作细胞定位中细胞质的标记物。qRT-PCR反应体系(10 μL):TB Green Premix Ex Taq Ⅱ 5 μL,PCR Forward Primer(10 μmol·L-1) 0.5 μL,PCR Reverse Primer(10 μmol·L-1)0.5 μL,cDNA 1 μL,RNase Free dH2O添加至10 μL;反应程序:95 ℃ 120 s;95 ℃ 5 s,60 ℃ 35 s,40个循环;95 ℃ 10 s;60 ℃ 60 s,97 ℃ 1 s。使用2-ΔΔCt方法计算基因的相对表达水平。每个样品进行3次重复,qRT-PCR试验进行3次重复。所用引物序列见表 1。
1.6 RNase R核糖核酸外切酶消化线性试验使用Lucigen RNR07250 Ribonuclease R(RNase R)试剂盒进行RNA线性消化试验。将提取的原始RNA样品等分为两份,一份做RNase R处理,另外一份不处理,此时两组RNA等量。RNase R消化反应体系:RNA < 5 μg:10×Reaction 2 μL,RNase R(20 U·μL-1)3~4 U·μg-1 RNA,RNase Free dH2O添加至20 μL;RNA>5 μg:10×Reaction 5 μL,RNase R(20 U·μL-1)3~4 U·μg-1 RNA,RNase Free dH2O添加至50 μL;反应程序:37 ℃反应25 min。
1.7 载体构建和腺病毒包装mouse-pCD2.1-circNMT1重组载体由通用生物系统(安徽)有限公司构建完成。将小鼠circRNA序列克隆到pCD2.1-ciR载体的Kpn I和BamH I限制性位点。腺病毒包装在汉恒生物技术有限公司(中国上海)进行。将水牛circRNA全长序列连接到AdMax系统以获得Ad-circRNA。其中,EGFP绿色荧光标签用作转导效率的指标,Ad-EGFP为阴性对照。
1.8 质粒转染与腺病毒转导对于3T3-L1细胞,当细胞密度达到80%时,使用Lipofectamine 3000试剂按照说明书将mouse-pCD2.1-circRNA重组载体质粒转染细胞。转染后48 h用诱导培养基处理细胞。诱导2 d后用维持培养基处理细胞。以后,每2 d更换1次维持培养基,直到细胞诱导分化第6天。腺病毒转染与质粒转染类似,根据腺病毒说明书,当水牛前体脂肪细胞密度达到80%时,进行腺病毒转染水牛前体脂肪细胞。诱导分化过程同上。诱导分化6 d后进行油红O染色和异丙醇定量。
1.9 油红O染色和定量诱导分化完成的脂肪细胞用PBS洗涤3次,用等量的10%福尔马林对细胞处理5 min和1 h后,用60%异丙醇洗涤细胞并用油红O工作液染色20 min,随后用PBS洗涤细胞3~5次,在显微镜下观察。为了量化细胞中的脂质积累,使用100% 异丙醇洗脱油红O。最后,以100%异丙醇作为空白对照,在波长为510 nm处对油红O的分光光度吸光度进行定量。
1.10 统计分析使用SPSS软件进行数据分析,P < 0.05被认为表明具有统计学意义,用GraphPad Prism7软件进行绘图。
2 结果 2.1 circNMT1序列信息通过NCBI数据库寻找NMT1基因全长序列,并与测序得到的circRNA序列进行比对。结果表明,circRNA19:45387150|45389986是由NMT1基因(ENSBTAT00000007950)产生的,具体信息在先前的研究已有描述[17]。circRNA19:45387150|45389986是通过剪接和环化NMT1基因的第2和3个外显子形成的(图 1)。因此,将该circRNA命名为circNMT1。

|
最下方图为NMT1-201转录本(ENSBTAG00000006051.6)外显子的位置。蓝色和紫色方块表示NMT1基因第2和第3个外显子。测序结果图主要显示为2个外显子的接口处。碱基序列为水牛-circNMT1;最上方的图是circRNA的结构模拟图 The bottom picture shows the position of exons of NMT1-201 transcript (ENSBTAG00000006051.6). Blue and purple squares represent exon 2 and 3 of NMT1 gene. The sequencing map mainly shows the junction of two exons. The base sequence is buffalo-circNMT1. The top diagram is the structural simulation diagram of circRNA 图 1 水牛-circNMT1组成结构的基本信息 Fig. 1 Basic information of buffalo-circNMT1 composition structure |
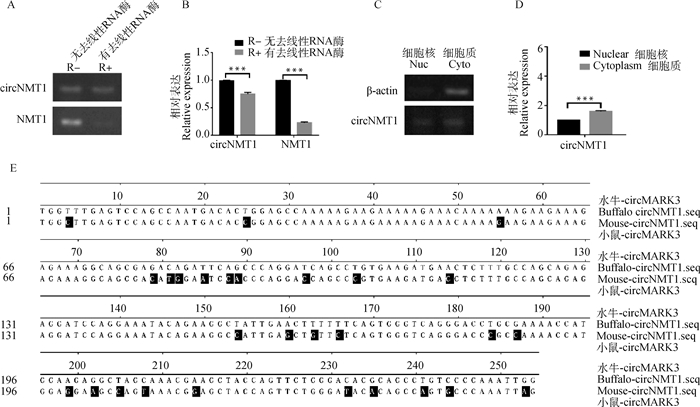
通过核酸外切酶耐受性试验对circNMT1进行鉴定,证实circRNA的真实性,并通过分离水牛前体脂肪细胞的细胞质和细胞核RNA对circNMT1进行定位,推测circRNA可能发挥的功能作用。对线性化RNA处理后,半定量PCR结果表明,circNMT1仍然存在于水牛前体脂肪细胞中(图 2A)。同样的,qRT-PCR的结果表明,尽管经过RNase R处理后,circNMT1的表达量有所减少,但它仍然高度表达(图 2B)。同时,半定量PCR和qRT-PCR结果表明,circNMT1在水牛前体脂肪的细胞核和细胞质中均表达(图 2C,D)。进一步序列比对发现,mouse-circNMT1和buffalo-circNMT1具有一定的保守性(同源率为88.98%,图 2E)。
|
A、B. 半定量PCR和qRT-PCR检测RNase R处理前、后circNMT1的表达量;C、D. 通过半定量和qRT-PCR对circNMT1进行核质定位;E. Mouse-circNMT1和circNMT1(buffalo-circNMT1)序列比对结果。牛-β-actin作为标准化参考基因。数据表示为“平均值± SD”,n = 3,***. P < 0.001 A, B. Semiquantitative PCR and qRT-PCR were used to detect the expression of circNMT1 before and after RNase R treatment; C, D. Semiquantitative PCR and qRT-PCR were used to locate the nuclear and cytoplasmic features of circNMT1; E. The sequence alignment results of mouse-circNMT1 and circNMT1 (buffalo-circNMT1). Bovine-β-actin was used as the reference gene. Data are presented as the "mean ± SD", n = 3, ***. P < 0.001 图 2 Buffalo-circNMT1的表征分析 Fig. 2 Characteristic analysis of buffalo-circNMT1 |
通过对水牛背部脂肪组织进行采集,分离水牛前体脂肪细胞。组织块分离法可以观察到,培养4 d有少数的细胞爬出(图 3A)。对分离得到的细胞培养,并对其进行诱导分化,诱导分化第0天,几乎观察不到脂滴(图 3B)。将基础培养基更换为诱导培养基,对细胞进行诱导分化,2 d后细胞产生少量的脂滴(图 3C)。随后将诱导培养基更换为维持培养基,且以后每2 d更换一次。即诱导分化第4(图 3D)、第6(图 3E)及第10天(图 3F)可以观察到脂滴在不断的增多。表明水牛脂肪细胞的分离、培养及诱导分化体系建立成功。

|
A.组织块法对水牛前体脂肪细胞进行分离培养(4 d),比例尺为100 μm;B. 对前体脂肪细胞诱导分化0 d,比例尺为100 μm;C-F. 对前体脂肪细胞诱导分化2、4、6、10 d,比例尺为50 μm A. The buffalo preadipocytes were isolated and cultured by tissue block method (4 d), scale bar=100 μm. B. Preadipocytes were induced to differentiate for 0 d, scale bar=100 μm. C-F. Preadipocytes were induced to differentiate for 2, 4, 6, 10 d, scale bar=50 μm 图 3 水牛前体脂肪细胞分离、培养与诱导分化 Fig. 3 Isolation, culture and induced differentiation of buffalo preadipocytes |
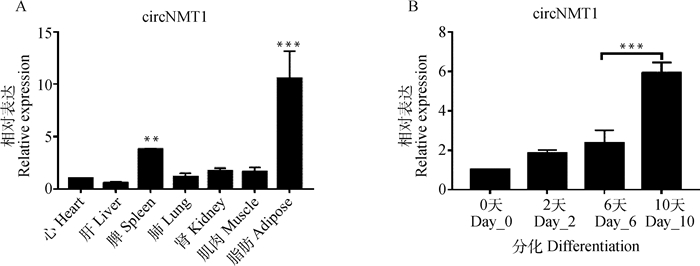
为了研究circNMT1在水牛不同组织(心、肝、脾、肺、肾、肌肉和脂肪组织)中的表达,使用TRIzol从不同组织中收集并提取了总RNA,通过qRT-PCR检测circNMT1实际表达。在脂肪组织中检测到circNMT1具有最高的表达(图 4A,P < 0.001)。进一步检测了circNMT1在水牛脂肪细胞分化不同阶段的表达,发现circNMT1的表达随着细胞分化的程度而增加,并在分化的第10天表达量最高(图 4B,P < 0.001)。
|
A.circNMT1在水牛心、肝、脾、肺、肾、肌肉和脂肪组织中的表达谱(所有数据以心归一化);B. circNMT1在水牛脂肪细胞分化过程中的表达模式(第0、2、6和10天,所有数据以day_0归一化)。GAPDH作为标准化参考基因。数据表示为“平均值± SD”,n = 3,**.P < 0.01,***.P < 0.001,下同 A.The expression patterns of circNMT1 in the heart, liver, spleen, lung, kidney, muscle and adipose tissues of buffalo(all data were normalized according to the expression in heart); B. The expression patterns of circNMT1 during buffalo preadipocytes at different differentiation stages (0, 2, 6 and 10 days, all data were normalized according to the expression on day_0). GAPDH was used as the reference gene. Data are presented as the "mean±SD", n=3, **.P < 0.01, ***.P < 0.001, the same as below 图 4 Buffalo-circNMT1在水牛组织和脂肪细胞中的表达模式 Fig. 4 Expression patterns of buffalo-circNMT1 in tissues and adipocytes of buffalo |
mouse-circNMT1和buffalo-circNMT1序列比对表明具有保守性(图 2E)。为了更好的探究circNMT1在脂肪沉积中发挥的功能,在3T3-L1细胞中进行了mouse-circNMT1的功能获得性试验。正如预期的那样,与pCD2.1-ciR组相比,mouse-pCD2.1-circNMT1组中circNMT1的表达水平显著上调(图 5C,P < 0.001),与此同时,PPARG、C/EBPα和FABP4的表达水平也显著上调(图 5D-F,P < 0.01)。诱导分化后,mouse-pCD2.1-circNMT1组中的脂滴累积显著高于pCD2.1-ciR组(图 5A,B,P < 0.01)。

|
A.将pCD2.1-ciR和mouse-pCD2.1-circNMT1转染到3T3-L1细胞中诱导分化6天后的油红O染色图像,比例尺为200 μm; B.通过分光光度法对油红O染色进行定量分析的柱状图; C-F. 转染48 h后circNMT、PPARG、C/EBPα和FABP4的RNA表达水平,β-actin作为标准化参考基因,control表示pCD2.1-ciR组,circNMT表示mouse-pCD2.1-circNMT1组,NC表示空白对照 A. Images of Oil Red O staining in 3T3-L1 cells transfected with pCD2.1-ciR and mouse-pCD2.1-circNMT1 on day_6 of adipogenic differentiation, scale bar=200 μm; B. Histogram showing the quantitation of Oil Red O staining by spectrophotometry; C-F. The RNA expression of circNMT1, PPARG, C/EBPα and FABP4 on 48 h after transfection, β-actin was used as the reference gene, control means the pCD2.1-ciR group, circNMT1 means the mouse-pCD2.1-circNMT1 group, NC means blank control 图 5 过表达mouse-circNMT1对3T3-L1细胞的影响 Fig. 5 Effects of mouse-circNMT1 overexpression on 3T3-L1 cells |
为了评估circNMT1对水牛脂肪沉积的影响,将circNMT1的全长连接到腺病毒系统中从而实现circRNA的过表达(Ad_circNMT1)。Ad_circNMT1转染细胞2 d后,绿色荧光标签GFP高度表达(图 6A),表明具有较高的转染效率,定量结果也表明在Ad_circNMT1组中circNMT1的表达显著高于Ad_EGFP组(图 6D,P < 0.001)。同时,水牛前体脂肪细胞分化6 d后,相比较Ad_EGFP组,Ad_circNMT1组的脂滴累积显著增多(图 6B,C,P < 0.01)。至于成脂标志基因,PPARG、C/EBPα和FABP4的mRNA表达在转染2 d后显著上调(图 6E-G,P < 0.01)。

|
A.Ad_GFP(对照)和Ad_circNMT1组中含有GFP标签的阳性细胞转染2 d的显微照片,比例尺为200 μm; B.将Ad_GFP和Ad_circNMT1转染到水牛脂肪细胞中诱导分化6 d后的油红O染色图像,比例尺为200 μm; C.分光光度法对油红O染色进行定量分析; D-G. 转染48 h后的circNMT(D)、PPARG(E)、C/EBPα(F)和FABP4(G)的RNA表达水平,GAPDH作为标准化参考基因,NC表示空白对照 A. Micrographs of GFP-positive cells in the Ad_GFP (control) and Ad_circNMT1 groups on day_2 of transfection, scale bar=200 μm; B. Images of Oil Red O staining in buffalo adipocytes transfected with Ad_GFP and Ad_circNMT1 groups on day 6 of adipogenic differentiation, scale bar=200 μm; C. Histogram showing the quantitation of Oil Red O staining by spectrophotometry; D-G. RNA expression of circNMT1(D), PPARG(E), C/EBPα(F) and FABP4(G) on day_2 after transfection, GAPDH was used as the reference gene, NC means blank control 图 6 过表达buffalo-circNMT1对水牛脂肪细胞的影响 Fig. 6 Effects of buffalo-circNMT1 overexpression on buffalo adipocytes |
NMT1是一种关键的细胞酶,通过促进肉蔻酸酯(一种14碳饱和脂肪酸)与几种蛋白质分子的N-末端甘氨酸连接进行脂质修饰并增加蛋白质的亲脂性[21]。编码蛋白基因可通过可变剪切产生circRNA,能被鉴定到的circRNA一般为表达丰度高且具有生物学功能,而且与来源基因之间会存在着一定的相关性[22]。因此,由NMT1基因产生的circNMT1被认为是功能性的circRNA。
环状RNA与线性RNA不同的是,circRNA的环状闭合结构具有核酸外切酶耐受性,可抵抗RNase R酶的消化作用,稳定性较高。circNMT1经核酸外切酶处理后仍稳定表达(图 2A,2B),表明circNMT1在脂肪细胞中以环状结构存在。这与报道的circRNA经去线性处理的研究结果相一致[23-25]。circNMT1在水牛前体脂肪细胞的细胞核和细胞质中均表达(图 2C,2D)。而circRNA因所处于核质的位置不同,发挥的功能具有很大区别。大多数的circRNA主要存在细胞质中,可作为ceRNA与mRNA竞争性结合miRNA,从而调控mRNA的表达[14, 23, 26]。少数通过内含子环化而成的circRNA位于细胞核中,主要发挥对亲本基因转录调控的作用[27]。结果提示,circNMT1发挥的功能可能较为复杂,需要进一步对其参与机制进行研究。circNMT1在水牛和小鼠中具有较高的同源性(同源率为88.98%,图 2E),这与报道的circRNA在不同物种中具有保守性[28-30]相一致。而circNMT1在脂肪组织和成熟的脂肪细胞中高表达(图 4A,4B, P < 0.01),提示该circRNA可能调节脂肪细胞的分化[31]。
动物的脂肪组织从沉积位置分为皮下脂肪、内脏脂肪和IMF。皮下脂肪是指在腹部、背部及腹股沟等动物皮下部位沉积的脂肪组织,内脏脂肪是指在内脏周围沉积的脂肪,而IMF是在肌纤维和肌束间沉积的脂肪组织,含量和分布是衡量牛肉品质的重要指标。由于其位置的特异性,IMF不同于皮下脂肪[32-34]和内脏脂肪[35],但彼此之间相互影响。日本和牛IMF沉积丰富,可作为研究牛IMF沉积的动物模型[36]。有研究发现,日本和牛背最长肌中IMF含量与皮下脂肪百分比[35]、成熟度[37]呈正相关。即IMF与皮下脂肪呈正相关。故而,体外培养的皮下脂肪细胞可作为研究脂肪沉积(IMF)分子调控机制的细胞模型,这在前期研究中也有所提及[18]。3T3-L1小鼠胚胎成纤维细胞(前脂肪)对其进行诱导分化后也是探究脂质累积的常用研究细胞模型[38-40]。
功能获得性试验表明,将circNMT1过表达于3T3-L1细胞和水牛前体脂肪细胞中,会上调脂肪生成标记基因PPARG、C/EBPα和FABP4的表达(图 5D~F;图 6E~G,P < 0.01)。从前体脂肪细胞分化为成熟的脂肪细胞涉及到复杂且高度协调的基因表达。作为脂肪细胞分化的关键调节因子PPARG和C/EBPα,会在启动子区域的多个结合位点上合作,共同调节发育和成熟的脂肪细胞表达各种基因[41-43]。PPARG会激活编码C/EBPα基因的启动子,反之亦然,从而形成一个正反馈回路[2]。此外,PPARG和C/EBPα也会诱导参与胰岛素敏感性、脂肪生成基因以及脂肪分解基因的表达,包括葡萄糖转运蛋白(GLUT4)、脂肪酸结合蛋白(FABP4)、脂蛋白脂肪酶(LPL)、单酰基甘油磷酸酰基转移酶2(AGPAT2)、perilipin、分泌因子脂联素和瘦素[2]。通过众多的脂肪相关基因所参与的复杂调控网络,最终促使前体脂肪细胞分化为含有大量脂滴的成熟脂肪细胞。然而,对过表达circNMT1的前体脂肪细胞进行诱导分化后,相比对照组,脂滴累积极显著增多(图 5A、B;图 6B、C,P < 0.01),表明circNMT会促进脂肪细胞的分化。
IMF是衡量牛肉品质的重要指标[44],牛肉的嫩度、口感、多汁性都会受到其含量的影响[45]。但水牛肉的IMF相对黄牛较低[46]。因此,充分开发水牛肉用性能,找到有效且可用于水牛肉IMF沉积改良的分子标记和关键调控因子至关重要。本研究结果提示,circNMT1可作为影响水牛脂肪沉积(IMF)的潜在调控因子,但参与脂肪生成的调控机制需要进一步探索。
4 结论综上所述,本研究通过对影响脂质代谢的候选circNMT1进行核质定位,时空表达模式分析以及发挥功能进行探究,获得以下结论:1)circNMT1是真实存在且稳定表达的circRNA。2)circNMT1在水牛前体脂肪细胞的细胞核和细胞质中均表达,且在脂肪组织和成熟的脂肪细胞中高表达。3)在3T3-L1细胞和水牛脂肪细胞,过表达circNMT1会上调成脂标志基因PPARG、C/EBPα和FABP4的表达水平。4)在3T3-L1细胞和水牛脂肪细胞,过表达circNMT1会促进脂肪细胞的脂滴累积。结果提示,circNMT1可能是水牛脂肪细胞分化的正调控因子,这为深入探究circNMT1对水牛脂肪沉积的调控机制提供了基础。
| [1] |
PINEDA P S, FLORES E B, HERRERA J R V, et al. Opportunities and challenges for improving the productivity of swamp buffaloes in southeastern Asia[J]. Front Genet, 2021, 12: 629861. DOI:10.3389/fgene.2021.629861 |
| [2] |
LOWE C E, O'RAHILLY S, ROCHFORD J J. Adipogenesis at a glance[J]. J Cell Sci, 2011, 124(16): 2681-2686. DOI:10.1242/jcs.079699 |
| [3] |
岳永起, 华永琳, 熊燕, 等. microRNA调控动物皮下脂肪组织和肌内脂肪沉积的研究进展[J]. 畜牧兽医学报, 2021, 52(10): 2698-2709. YUE Y Q, HUA Y L, XIONG Y, et al. Research progress of microRNA regulation on the fat deposition of subcutaneous adipose tissue and intramuscular fat in animals[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(10): 2698-2709. DOI:10.11843/j.issn.0366-6964.2021.010.002 (in Chinese) |
| [4] |
LEI Z X, WU H G, XIONG Y, et al. ncRNAs regulate bovine adipose tissue deposition[J]. Mol Cell Biochem, 2021, 476(7): 2837-2845. DOI:10.1007/s11010-021-04132-2 |
| [5] |
HUANG J P, ZHENG Q Z, WANG S Z, et al. High-throughput RNA sequencing reveals NDUFC2-AS lncRNA Promotes adipogenic differentiation in Chinese buffalo (Bubalus bubalis L)[J]. Genes, 2019, 10(9): 689. DOI:10.3390/genes10090689 |
| [6] |
ZHANG Y F, GUO X, PEI J, et al. CircRNA expression profile during yak adipocyte differentiation and screen potential circRNAs for adipocyte differentiation[J]. Genes, 2020, 11(4): 414. DOI:10.3390/genes11040414 |
| [7] |
ZHANG M, HAN Y, ZHAI Y H, et al. Integrative analysis of circRNAs, miRNAs, and mRNAs profiles to reveal ceRNAs networks in chicken intramuscular and abdominal adipogenesis[J]. BMC Genomics, 2020, 21(1): 594. DOI:10.1186/s12864-020-07000-3 |
| [8] |
WANG L D, LIANG W S, WANG S S, et al. Circular RNA expression profiling reveals that circ-PLXNA1 functions in duck adipocyte differentiation[J]. PLoS One, 2020, 15(7): e0236069. DOI:10.1371/journal.pone.0236069 |
| [9] |
LI A, HUANG W L, ZHANG X X, et al. Identification and characterization of circRNAs of two pig breeds as a new biomarker in metabolism-related diseases[J]. Cell Physiol Biochem, 2018, 47(6): 2458-2470. DOI:10.1159/000491619 |
| [10] |
LIU X, LIU K Q, SHAN B S, et al. A genome-wide landscape of mRNAs, lncRNAs, and circRNAs during subcutaneous adipogenesis in pigs[J]. J Anim Sci Biotechnol, 2018, 9: 76. DOI:10.1186/s40104-018-0292-7 |
| [11] |
王卫振, 邓占钊, 辛国省, 等. 环状RNA的生物学功能及其在家禽中的研究进展[J]. 畜牧兽医学报, 2021, 52(7): 1778-1788. WANG W Z, DENG Z Z, XIN G S, et al. The biological function of circular RNA and its research progress in poultry[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(7): 1778-1788. (in Chinese) |
| [12] |
LIANG W C, WONG C W, LIANG P P, et al. Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway[J]. Genome Biol, 2019, 20(1): 84. DOI:10.1186/s13059-019-1685-4 |
| [13] |
WEI X F, LI H, YANG J M, et al. Circular RNA profiling reveals an abundant circLMO7 that regulates myoblasts differentiation and survival by sponging miR-378a-3p[J]. Cell Death Dis, 2017, 8(10): e3153. DOI:10.1038/cddis.2017.541 |
| [14] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 |
| [15] |
LIU J Y, ZHANG C H, ZHANG B Y, et al. Comprehensive analysis of the characteristics and differences in adult and newborn brown adipose tissue (BAT): newborn BAT is a more active/dynamic BAT[J]. Cells, 2020, 9(1): 201. DOI:10.3390/cells9010201 |
| [16] |
LONG T, GUO Z Y, HAN L, et al. Differential expression profiles of circular RNAs during osteogenic differentiation of mouse adipose-derived stromal cells[J]. Calcif Tissue Int, 2018, 103(3): 338-352. DOI:10.1007/s00223-018-0426-0 |
| [17] |
HUANG J P, ZHAO J H, ZHENG Q Z, et al. Characterization of circular RNAs in Chinese buffalo (Bubalus bubalis) adipose tissue: a focus on circular RNAs involved in fat deposition[J]. Animals, 2019, 9(7): 403. DOI:10.3390/ani9070403 |
| [18] |
冯雪. 水牛肌内脂肪沉积关键基因的筛选及相关功能研究[D]. 信阳: 信阳师范学院, 2021. FENG X. Screening and functional analysis of important genes for intramuscular fat deposition in buffaloes[D]. Xinyang: Xinyang Normal University, 2021. (in Chinese) |
| [19] |
ZHU R R, FENG X, WEI Y T, et al. lncSAMM50 enhances adipogenic differentiation of buffalo adipocytes with no effect on its host gene[J]. Front Genet, 2021, 12: 626158. DOI:10.3389/fgene.2021.626158 |
| [20] |
冯雪, 洪慧颖, 孙晓菲, 等. 水牛PPARG来源环状RNA的鉴定及表达谱分析[J]. 中国畜牧兽医, 2019, 46(12): 3457-3465. FENG X, HONG H Y, SUN X F, et al. Identification and expression profile analysis of PPARG-derived circular RNA in buffalo[J]. China Animal Husbandry & Veterinary Medicine, 2019, 46(12): 3457-3465. (in Chinese) |
| [21] |
WRIGHT M H, HEAL W P, MANN D J, et al. Protein myristoylation in health and disease[J]. J Chem Biol, 2010, 3(1): 19-35. DOI:10.1007/s12154-009-0032-8 |
| [22] |
XU X L, ZHANG J W, TIAN Y H, et al. CircRNA inhibits DNA damage repair by interacting with host gene[J]. Mol Cancer, 2020, 19(1): 128. DOI:10.1186/s12943-020-01246-x |
| [23] |
DING Z Y, SUN D Y, HAN J, et al. Novel noncoding RNA CircPTK2 regulates lipolysis and adipogenesis in cachexia[J]. Mol Metab, 2021, 53: 101310. DOI:10.1016/j.molmet.2021.101310 |
| [24] |
WANG L, ZHOU J H, ZHANG C, et al. A novel tumour suppressor protein encoded by circMAPK14 inhibits progression and metastasis of colorectal cancer by competitively binding to MKK6[J]. Clin Transl Med, 2021, 11(10): e613. |
| [25] |
CHEN W B, CEN S R, ZHOU X M, et al. Circular RNA CircNOLC1, upregulated by NF-KappaB, promotes the progression of prostate cancer via miR-647/PAQR4 axis[J]. Front Cell Dev Biol, 2021, 8: 624764. DOI:10.3389/fcell.2020.624764 |
| [26] |
HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 |
| [27] |
LI Z Y, HUANG C, BAO C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nat Struct Mol Biol, 2015, 22(3): 256-264. DOI:10.1038/nsmb.2959 |
| [28] |
JECK W R, SORRENTINO J A, WANG K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 |
| [29] |
WANG P L, BAO Y, YEE M C, et al. Circular RNA is expressed across the eukaryotic tree of life[J]. PLoS One, 2014, 9(6): e90859. |
| [30] |
XIAO W, LI J, HU J, et al. Circular RNAs in cell cycle regulation: mechanisms to clinical significance[J]. Cell Prolif, 2021, e13143. DOI:10.1111/cpr.13143 |
| [31] |
WEI X F, HAN S, WANG S Z, et al. ANGPTL8 regulates adipocytes differentiation and adipogenesis in bovine[J]. Gene, 2019, 707: 93-99. DOI:10.1016/j.gene.2019.04.048 |
| [32] |
BOSCH L, TOR M, REIXACH J, et al. Age-related changes in intramuscular and subcutaneous fat content and fatty acid composition in growing pigs using longitudinal data[J]. Meat Sci, 2012, 91(3): 358-363. DOI:10.1016/j.meatsci.2012.02.019 |
| [33] |
TYRA M, ROPKA-MOLIK K, TERMAN A, et al. Association between subcutaneous and intramuscular fat content in porcine ham and loin depending on age, breed and FABP3 and LEPR genes transcript abundance[J]. Mol Biol Rep, 2013, 40(3): 2301-2308. DOI:10.1007/s11033-012-2311-7 |
| [34] |
HUDSON N J, REVERTER A, GRIFFITHS W J, et al. Gene expression identifies metabolic and functional differences between intramuscular and subcutaneous adipocytes in cattle[J]. BMC Genomics, 2020, 21(1): 77. DOI:10.1186/s12864-020-6505-4 |
| [35] |
YAMADA T, KAMIYA M, HIGUCHI M. Fat depot-specific effects of body fat distribution and adipocyte size on intramuscular fat accumulation in Wagyu cattle[J]. Anim Sci J, 2020, 91(1): e13449. |
| [36] |
CAMPOS C F, DUARTE M S, GUIMARAES S E F, et al. Review: animal model and the current understanding of molecule dynamics of adipogenesis[J]. Animal, 2016, 10(6): 927-932. DOI:10.1017/S1751731115002992 |
| [37] |
LIU L, CAO P L, ZHANG L P, et al. Comparisons of adipogenesis- and lipid metabolism-related gene expression levels in muscle, adipose tissue and liver from Wagyu-cross and Holstein steers[J]. PLoS One, 2021, 16(2): e0247559. DOI:10.1371/journal.pone.0247559 |
| [38] |
TUVIA N, PIVOVAROVA-RAMICH O, MURAHOVSCHI V, et al. Insulin directly regulates the circadian clock in adipose tissue[J]. Diabetes, 2021, 70(9): 1985-1999. DOI:10.2337/db20-0910 |
| [39] |
XIAO F, TANG C Y, TANG H N, et al. Long non-coding RNA 332443 inhibits preadipocyte differentiation by targeting Runx1 and p38-MAPK and ERK1/2-MAPK signaling pathways[J]. Front Cell Dev Biol, 2021, 9: 663959. DOI:10.3389/fcell.2021.663959 |
| [40] |
ZHANG H Y, DENG T, GE S H, et al. Exosome circRNA secreted from adipocytes promotes the growth of hepatocellular carcinoma by targeting deubiquitination-related USP7[J]. Oncogene, 2019, 38(15): 2844-2859. |
| [41] |
LEFTEROVA M I, ZHANG Y, STEGER D J, et al. PPARγ and C/EBP factors orchestrate adipocyte biology via adjacent binding on a genome-wide scale[J]. Genes Dev, 2008, 22(21): 2941-2952. DOI:10.1101/gad.1709008 |
| [42] |
NIELSEN R, PEDERSEN T Å, HAGENBEEK D, et al. Genome-wide profiling of PPARγ: RXR and RNA polymerase Ⅱ occupancy reveals temporal activation of distinct metabolic pathways and changes in RXR dimer composition during adipogenesis[J]. Genes Dev, 2008, 22(21): 2953-2967. DOI:10.1101/gad.501108 |
| [43] |
崔胜, 王永, 朱江江, 等. 过表达NR4A1促进山羊皮下脂肪细胞分化[J]. 畜牧兽医学报, 2021, 52(5): 1258-1266. CUI S, WANG Y, ZHU J J, et al. Overexpression of NR4A1 promotes goat subcutaneous adipocytes differentiation[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(5): 1258-1266. (in Chinese) |
| [44] |
JOO S T, HWANG Y H, FRANK D. Characteristics of Hanwoo cattle and health implications of consuming highly marbled Hanwoo beef[J]. Meat Sci, 2017, 132: 45-51. DOI:10.1016/j.meatsci.2017.04.262 |
| [45] |
JUNG E Y, HWANG Y H, JOO S T. The relationship between chemical compositions, meat quality, and palatability of the 10 primal cuts from hanwoo steer[J]. Korean J Food Sci Anim Resour, 2016, 36(2): 145-151. |
| [46] |
HUANG J P, FENG X, ZHU R R, et al. Comparative transcriptome analysis reveals that PCK1 is a potential gene affecting IMF deposition in buffalo[J]. BMC Genomics, 2020, 21(1): 710. |
(编辑 郭云雁)



