2. 广东省兽药研制与安全评价重点实验室,广州 510624;
3. 扬州大学兽医学院,扬州 225009
2. Guangdong Provincial Key Laboratory of Veterinary Pharmaceutics Development and Safety, Guangzhou 510642, China;
3. College of Veterinary Medicine, Yangzhou University, Yangzhou 225009, China
噬菌体被称为细菌病毒,必须寄生于特定的活菌体内。其个体微小,仅由核酸和蛋白质外壳构成。噬菌体在自然界中分布广泛,数量超过1031个[1],分布于水体、土壤、人体及畜禽肠道等各种环境中[2],有细菌的地方就有噬菌体的存在。相对于其他生态环境,水环境中的噬菌体的数量最多[3]。根据其繁殖特点,噬菌体可被分为裂解性噬菌体和溶原性噬菌体两大类。
近年来,细菌耐药问题日益严峻,多重耐药菌株检出率逐年增加,甚至出现同时携带blaNDM-5及mcr-1这样的“超级细菌”[4],严重危害人类和畜禽健康,解决抗生素耐药问题已变得刻不容缓。噬菌体作为一种天然“抗菌药物”,其制剂的毒性低,特异性强和开发周期短等,被视为“后抗生素时代”很好的治疗手段和生态环境净化制剂[5]。用于“噬菌体治疗”的是裂解性噬菌体,“噬菌体治疗”概念的提出最早可以追溯到20世纪20年代,但因为第二次世界大战和抗菌药物的发现,转移了人们对“噬菌体治疗”的关注[6],一定程度上制约了它的发展,直至近10年,噬菌体再次获得了人们的关注[7]。目前, 运用噬菌体治疗多重耐药细菌感染病患已取得了令人振奋的成果[8-9],这些成果不仅证实了噬菌体治疗的可行性,也暗示着噬菌体在兽医生产临床上也具有极大的应用潜力。
广东家禽养殖环境复杂,抗菌药物的不合理使用,使得多重耐药菌株不断检出[10],也给养殖业造成了巨大的损失,严重挑战了兽医临床上对抗大肠杆菌的有效药物的选择。为了解决上述问题,本研究提出利用禽源多重耐药大肠杆菌为宿主菌,以从养殖场池塘水中采集样品为研究对象,试图分离出可裂解携带blaNDM-5及mcr-1耐药基因大肠杆菌的噬菌体,并对其生物学特性进行研究,解析其全基因组序列,旨在为养殖场中blaNDM-5及mcr-1阳性大肠杆菌的防控提供一定的理论依据。
1 材料与方法 1.1 样品来源菌株:宿主菌为华南农业大学兽医药理研究室分离并保存的禽源多重耐药大肠杆菌GZB8C146-1M,菌株详细信息如表 1所示。
|
|
表 1 GZB8C146-1M菌株信息 Table 1 GZB8C146-1M strain information |
池塘水样品来源:广东肇庆某家禽养殖场所采集的池塘水。
1.2 主要试剂麦康凯琼脂和LB肉汤等均购于广州环凯微生物有限公司。
SM缓冲液:称取NaCl 4.68 g,MgSO4 1.58 g,Tris 4.85 g,明胶0.016 g,加入去离子水充分溶解,调节pH至7.5,定容至800 mL,高压灭菌后使用。
噬菌体DNA提取试剂盒购于Omega公司。
1.3 样品的处理将采集回来的鸭场污水离心除去大颗粒杂质,依次使用5.00、1.20、0.45、0.22 μm的微孔滤膜过滤除菌,通过Millipore Pellicon XL超滤膜将滤液浓缩为50 mL的液体,即为噬菌体母液,-4 ℃保存备用。
1.4 噬菌体的分离与纯化分别取100 μL噬菌体母液和100 μL对数期菌悬液加入至2 mL LB肉汤中,37 ℃、160 r·min-1摇床中培养4 h,6 000×g离心5 min,0.22 μm滤膜除菌后再加入100 μL菌悬液,继续培养4 h后,离心过膜除菌,如此重复3次,得到富集液。将富集液用SM缓冲液倍比稀释一定倍数后,加入等体积的菌悬液,静置培养30 min后制作双层平板,37 ℃培养6~8 h,若琼脂平板上显现出噬菌斑,挑取单个透亮圆润的噬菌斑,加入1 mL SM缓冲液中,涡旋混匀后过膜除菌,制作双层平板进行纯化,此步骤约重复3次,直至得到大小均一的噬菌斑。
1.5 噬菌体的透射电镜观察将封口膜平铺在玻璃片上,用镊子把铜网夹至封口膜上,使用移液枪取10 μL噬菌体富集液滴在铜网上,静置10 min后,用滤纸吸去多余液体后,取10 μL 2%磷钨酸滴在铜网上,静置1~3 min后,用滤纸吸去多余液体。室温放置干燥后,放置透射电镜(型号:日本电子JEOL JEM-1200EX)观察,并进行拍照记录。
1.6 噬菌体的最佳感染复数(OMOI)测定大肠杆菌菌液培养至对数期(OD600nm=0.3~0.4),噬菌体液富集3次备用,完成细菌和噬菌体计数后,在2 mL离心管中分别加入100 μL稀释后的菌悬液,按照噬菌体∶细菌(P∶B)=10∶1、1∶1、10-1∶1、10-2∶1、10-3∶1、10-4∶1的数量比例加入倍比稀释后噬菌体(每个比例作3个重复)。在37 ℃、160 r·min-1摇床中培养4 h后,进行噬菌体的效价测试,滴度最高的组对应的MOI,即为最佳感染复数。
1.7 噬菌体的一步生长曲线的测定在2 mL离心管中加入1 mL的细菌菌悬液,按照P∶B=10的比例,加入富集后的噬菌体液,37 ℃静置15 min,10 000 × g离心30 s弃去上清液后,加入1 mL LB液体培养基迅速吹打菌体沉淀,离心弃上清,重复洗涤2次,加入1 mL LB液体培养基并吹打沉淀,转移混合液至LB肉汤试管中(总体积5 mL),放置37 ℃、180 r·min-1摇床中培养,每间隔10 min取200 μL培养液,离心过膜处理后,测定培养液中噬菌体的效价,一共持续取样测定90 min,本试验设置3组平行。根据爆发量计算公式:爆发量=爆发末期噬菌体滴度/感染初期宿主菌浓度,计算试验结果结果,并绘制噬菌体的一步生长曲线图。
1.8 噬菌体的温度稳定性测定将1 mL噬菌体富集液(每组设3个重复)分别置于4 ℃冰浴及37、45、50、55、60、70 ℃的水浴锅中处理1 h后,对样品进行倍比稀释,测定不同温度处理后的噬菌体效价。
1.9 噬菌体的pH稳定性测定配制pH为1~12的LB肉汤,将100 μL噬菌体富集液分别加入到不同pH的1 mL LB肉汤中(每组设3个重复),37 ℃、180 r·min-1摇床下孵育1 h后,测定不同pH条件下处理后的噬菌体效价。
1.10 宿主谱测定挑取本实验室中102株与GZB8C146-1M同一批分离出的blaNDM-5阳性大肠杆菌和大肠杆菌标准菌株ATCC 25922,其中12株大肠杆菌同时携带blaNDM-5和mcr-1耐药基因,采用点样法测定噬菌体的宿主谱。取100 μL对数期菌液加入5 mL半固体中,摇晃均匀后静置放凉即得到试验所需的双层平板,将2 μL噬菌体液点板于双层平板上,37 ℃培养6~8 h后,读取试验结果。
1.11 噬菌体的全基因组测序及生物信息学分析使用Omega公司病毒提取试剂盒进行噬菌体DNA的提取,DNA样品送至北京诺禾致源科技股份有限公司进行二代测序。将测序结果导入CLC Genomics Workbench 10.1,通过De Novo Assembly进行拼接,获得contigs,用于进一步分析。利用RAST(https://rast.nmpdr.org/rast.cg)和NCBI中的BLAST工具(http://blast.ncbi.nlm.nih.gov/)注释和验证ORF;利用MEGAX软件及NCBI中BLAST比对相似的10组噬菌体序列构建系统发育进化树;利用网络数据库CGE(https://cge.cbs.dtu.dk/services/)进行耐药基因和毒力基因的查找。
2 结果 2.1 噬菌体的分离与纯化以同时携带blaNDM-5及mcr-1阳性大肠杆菌GZB8C146-1为宿主菌,从鸭场污水中分离纯化出1株噬菌体,将其命名为vB_EcoM-ZQ1(图 1A)。

|
A. 噬菌体vB_EcoM-ZQ1的噬菌斑;B. 噬菌体vB_EcoM-ZQ1的透射电镜图 A. The plaque of the phage vB_EcoM-ZQ1; B. The transmission electron microscope of the phage vB_EcoM-ZQ1 图 1 噬菌体vB_EcoM-ZQ1的基本形态特征 Fig. 1 Basic morphological characteristics of the phage vB_EcoM-ZQ1 |
透射电镜结果(图 1B)显示,vB_EcoM-ZQ1具有较为典型的二十面体头部和尾部,噬菌体全长大小约为180 nm,其中,头部约为72 nm,尾部约为108 nm。通过电镜观察和全基因组测序分析,该噬菌体属于有尾病毒目,肌尾噬菌体科。
2.3 噬菌体的最佳感染复数(OMOI)根据表 2所示结果可知,当感染复数(MOI)为0.1时,噬菌体vB_EcoM-ZQ1效价最高,为6.7×1011 PFU·mL-1,所以噬菌体vB_EcoM-ZQ1的最佳感染复数为0.1。
|
|
表 2 噬菌体vB_EcoM-ZQ1最佳感染复数的测定 Table 2 Determination of the optimal multiplicity of infection of the phage vB_EcoM-ZQ1 |
由图 2可知,噬菌体vB_EcoM-ZQ1的潜伏期为20 min,在这期间噬菌体的效价虽有增长,但是总体趋势较缓慢。在20~30 min,噬菌体效价急剧增加,持续时间10 min后,噬菌体又趋于稳定增长。根据公式可得噬菌体vB_EcoM-ZQ1的裂解量大约为92 PFU·cell-1。
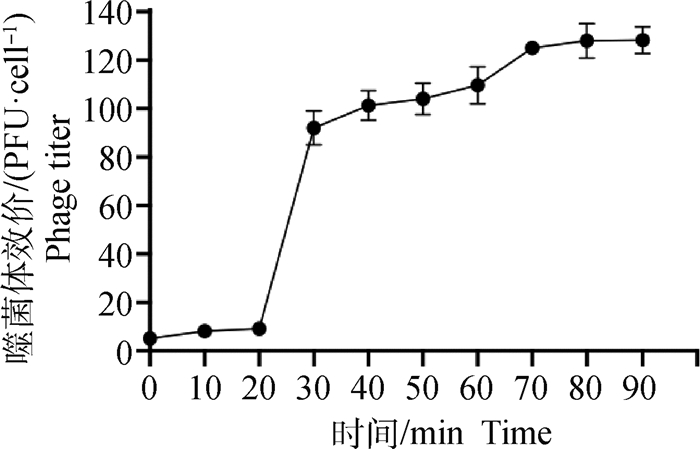
|
图 2 噬菌体vB_EcoM-ZQ1的一步生长曲线 Fig. 2 One-step growth curve of the phage vB_EcoM-ZQ1 |
由图 3A可知,噬菌体vB_EcoM-ZQ1在4~55 ℃,噬菌体的效价变化不大,较为稳定。60 ℃时,噬菌体效价出现明显下降,在70 ℃时,观察不到噬菌体的存活,说明在该温度下,噬菌体vB_EcoM-ZQ1的活力被严重破坏。
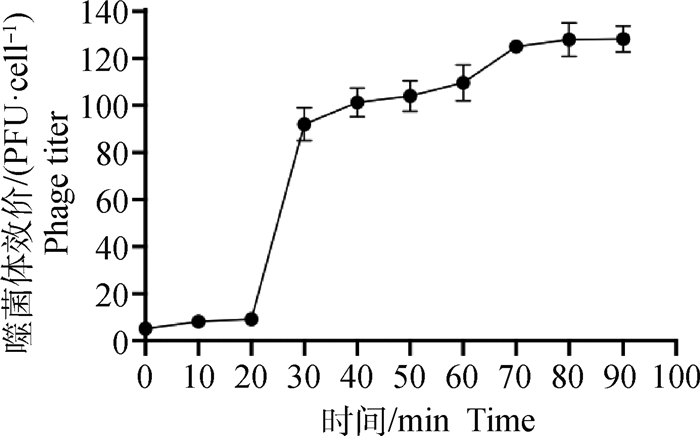
|
A. 噬菌体vB_EcoM-ZQ1温度稳定性试验结果;B. 噬菌体vB_EcoM-ZQ1pH稳定性试验结果 A. Temperature stability test result of the phage vB_EcoM-ZQ1; B. pH stability test results of the phage vB_EcoM-ZQ1 图 3 噬菌体vB_EcoM-ZQ1稳定性试验结果 Fig. 3 Stability test results of the phage vB_EcoM-ZQ1 |
如图 3B所示,噬菌体vB_EcoM-ZQ1在pH耐受范围为4~10,噬菌体对pH有较宽的耐受性,当pH低于3或高于11时,并未发现存活的噬菌体,说明噬菌体vB_EcoM-ZQ1不耐强酸和强碱。
2.7 噬菌体的宿主谱测定噬菌体vB_EcoM-ZQ1能裂解26株大肠杆菌,裂解率为25.5%。26株大肠杆菌中,有24株是携带blaNDM-5耐药基因的大肠杆菌,有2株为同时携带blaNDM-5和mcr-1耐药基因。结果说明噬菌体vB_EcoM-ZQ1对多重耐药细菌有一定的裂解作用,但试验中也发现该噬菌体并不能裂解大肠杆菌标准菌株ATCC 25922,试验所选取菌株信息及裂解情况如表 3所示。
|
|
表 3 宿主谱选用菌株信息及宿主谱测定结果 Table 3 Strain information selected by the host profile and the results of the host profile determination |
基因组装后的噬菌体vB_EcoM-ZQ1基因全长149 112 bp,GC含量为49.1%。通过注释和比对后,该基因组共比对到184个ORF,预测出了2个tRNA,分别为tRNA-Met(CAT)和tRNA-Ser(GCT)。基因组中并未预测出耐药基因和毒力因子,说明了噬菌体ZQ1在临床上应用的安全性。
在184个ORF中,有70个ORF是已知功能的基因,其中约35%的基因与噬菌体的结构与组装有关,如tail spike protein、baseplate wedge subunit、Gp4 head completion protein和Gp13 neck protein等;约61%的基因与噬菌体DNA复制和调控有关,如DNA ligase、putative DNA polymerase和dCMP deaminase等;terminase large subunit、terminase small subunit及Gp20 portal vertex protein of head与噬菌体DNA包装有关,同时有1个基因putative endolysin与宿主裂解相关(图 4)。
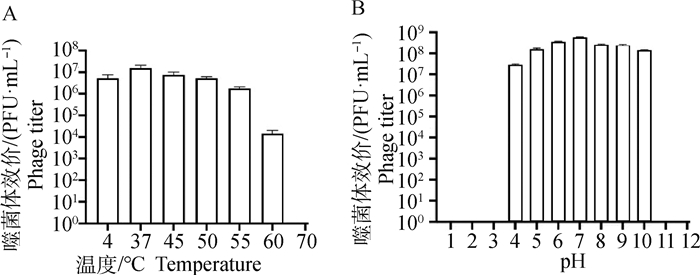
|
图 4 噬菌体vB_EcoM-ZQ1的基因图谱 Fig. 4 Gene map of the phage vB_EcoM-ZQ1 |
由图 5的发育进化树结果显示,噬菌体vB_EcoM-ZQ1与沙门菌噬菌体pSal-SNUABM-02的亲缘关系最近,位于同一分支,其次是沙门菌噬菌体P46FS4,而与肠杆菌噬菌体EspM4VN的亲缘关系最远。
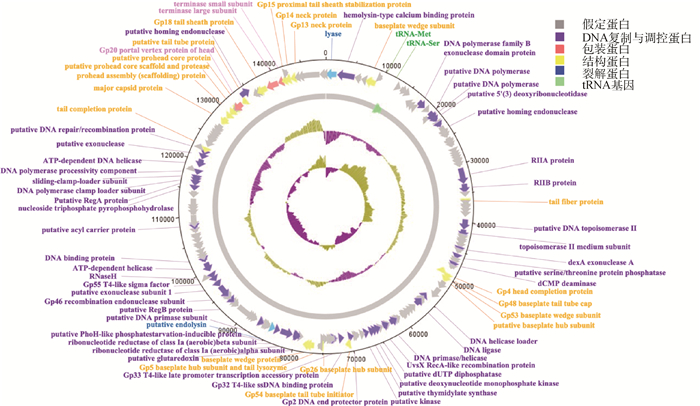
|
图 5 基于噬菌体末端酶大亚基构建的同源进化树 Fig. 5 The homologous evolutionary tree constructed based on the large subunit of the phage terminal enzyme |
大肠杆菌广泛分布于养殖场环境和动物体内,大肠杆菌病传统上采取抗菌药物进行治疗,在这个过程中,由于临床养殖人员缺乏足够的理论知识储备,养殖场细菌耐药现象较为严重[11]。“超级细菌”的出现给抗菌药物的运用带来了极大的压力,也严重威胁到了公共卫生健康,于是近些年“噬菌体治疗”概念被提出,由于噬菌体易获得、低成本和安全性高等优点[12],“噬菌体治疗”也被越来越多人所接受,且逐步形成产业化试用于临床[13]。
本研究从鸭场水环境中成功分离出1株对同时携带blaNDM-5及mcr-1耐药基因的家禽源大肠杆菌有裂解作用的噬菌体vB_EcoM-ZQ1。该噬菌体电镜下呈现出多面体的头部和一个可收缩的尾部,噬菌体全长大小约为180 nm,其中,头部约为72 nm,尾部约为108 nm,属于有尾病毒目,肌尾噬菌体科噬菌体[14-15],和基因组分析比对结果相对应,也和葛展霞等[16]报道的噬菌体形态相似。
噬菌体vB_EcoM-ZQ1在温度4~55 ℃范围内都能较稳定生长,与孙利厂等[17]的报道相比,该噬菌体具有更宽的活性范围;在pH4~10时,噬菌体vB_EcoM-ZQ1的活性都较高,耐受范围比杨慧敏等[18]报道的要宽,但当pH低于3或高于11时,并未发现噬菌体的存活,说明噬菌体不耐强酸和强碱,这一结论与绝大多数报道一致[19]。噬菌体的最佳感染复数为0.1,潜伏期为20 min,裂解期为10 min,裂解量为92 PFU·cell-1,噬菌体的裂解期要短于一般文献报道[20],但与Thanh等[21]报道的一步生长曲线相似,并表示该噬菌体能在短时间内完成裂解过程,运用于临床实践中,能快速杀灭耐药大肠杆菌[22]。宿主谱试验中发现,噬菌体的裂解率为25.5%,低于Zhou等[23]的报道,也不能裂解大肠杆菌标准菌株ATCC 25922,考虑与试验选用菌株也有一定关系,但对于治疗多重耐药大肠杆菌甚至携带blaNDM-5及mcr-1依旧有理论意义。
经过全基因组测序分析后,将噬菌体序列上传至NCBI(NCBI序列号:MW650886),该噬菌体基因全长149 112 bp,GC含量为49.1%,无明显的GC偏向性,共有184个ORF,其中有70个ORF是已知功能的基因,表明该基因组绝大部分基因组序列有待研究。噬菌体vB_EcoM-ZQ1中发现了tRNA的存在,噬菌体依靠吞噬宿主菌获得增殖,利用宿主菌的tRNA完成自身生物合成的过程[24],在很长一段时间,噬菌体被认为不携带tRNA,但随着研究的深入和测序技术的发展,越来越多的噬菌体中发现了tRNA的存在,Bailly-Bechet等[25]指出tRNA可能在一定程度上能提高噬菌体对宿主菌的裂解能力。但在噬菌体中并未发现该噬菌体中含有耐药基因与毒力因子,验证了该噬菌体运用于兽医临床的安全性。噬菌体vB_EcoM-ZQ1和沙门菌噬菌体pSal-SNUABM-02[26]的亲缘关系最近,位于同一分支,与志贺菌phiSboM-AG3[27]有92.37%的相似性,而这些噬菌体的宿主菌分别是志贺菌和肠杆菌,考虑噬菌体vB_EcoM-ZQ1有可能具有裂解其他类型菌的能力,有待进一步探究。
4 结论成功筛选出1株对同时携带blaNDM-5及mcr-1耐药基因的家禽源大肠杆菌有裂解作用的噬菌体vB_EcoM-ZQ1,并对其进行了生物学特性和基因组学研究,明确了噬菌体形态和生物学特征,验证了运用于兽医临床的可行性,从基因组学角度也证明了该噬菌体的安全性,为后期噬菌体防控blaNDM-5及mcr-1阳性大肠杆菌感染奠定了一定基础。
| [1] |
RIPP S. Bacteriophage-based pathogen detection[J]. Adv Biochem Eng Biotechnol, 2010, 118: 65-83. |
| [2] |
DION M B, OECHSLIN F, MOINEAU S. Phage diversity, genomics and phylogeny[J]. Nat Rev Microbiol, 2020, 18(3): 125-138. DOI:10.1038/s41579-019-0311-5 |
| [3] |
张永雨, 黄春晓, 杨军, 等. 海洋微生物与噬菌体间的相互关系[J]. 科学通报, 2011, 56(14): 1071-1079. ZHANG Y Y, HUANG C X, YANG J, et al. Interactions between marine microorganisms and their phages[J]. Chinese Science Bulletin, 2011, 56(17): 1770-1777. (in Chinese) |
| [4] |
KONG L H, LEI C W, MA S Z, et al. Various sequence types of Escherichia coli isolates coharboring blaNDM-5 and mcr-1 genes from a commercial swine farm in China[J]. Antimicrob Agents Chemother, 2017, 61(3): e02167-16. |
| [5] |
KOWALSKA J D, KAZIMIERCZAK J, SOWI AN'U SKA P M, et al. Growing trend of fighting infections in aquaculture environment-opportunities and challenges of phage therapy[J]. Antibiotics (Basel), 2020, 9(6): 301. DOI:10.3390/antibiotics9060301 |
| [6] |
CISEK A A, DABROWSKA I, GREGORCZYK K P, et al. Phage therapy in bacterial infections treatment: one hundred years after the discovery of bacteriophages[J]. Curr Microbiol, 2017, 74(2): 277-283. DOI:10.1007/s00284-016-1166-x |
| [7] |
SALMOND G P C, FINERAN P C. A century of the phage: past, present and future[J]. Nat Rev Microbiol, 2015, 13(12): 777-786. DOI:10.1038/nrmicro3564 |
| [8] |
QIN J H, WU N N, BAO J, et al. Heterogeneous Klebsiella pneumoniae co-infections complicate personalized bacteriophage therapy[J]. Front Cell Infect Microbiol, 2021, 10: 608402. DOI:10.3389/fcimb.2020.608402 |
| [9] |
VILA J, MORENO-MORALES J, BALLESTÉ-DELPIERRE C. Current landscape in the discovery of novel antibacterial agents[J]. Clin Microbiol Infect, 2020, 26(5): 596-603. DOI:10.1016/j.cmi.2019.09.015 |
| [10] |
WANG M, CHEN S Y, ZHANG J X, et al. Variations of antibiotic resistance profiles in chickens during administration of amoxicillin, chlortetracycline and florfenicol[J]. J Appl Microbiol, 2018, 125(6): 1692-1701. DOI:10.1111/jam.14065 |
| [11] |
LEVY S B. Factors impacting on the problem of antibiotic resistance[J]. J Antimicrob Chemother, 2002, 49(1): 25-30. DOI:10.1093/jac/49.1.25 |
| [12] |
WERNICKI A, NOWACZEK A, URBAN-CHMIEL R. Bacteriophage therapy to combat bacterial infections in poultry[J]. Virol J, 2017, 14(1): 179. DOI:10.1186/s12985-017-0849-7 |
| [13] |
LOPONTE R, PAGNINI U, IOVANE G, et al. Phage therapy in veterinary medicine[J]. Antibiotics (Basel), 2021, 10(4): 421. DOI:10.3390/antibiotics10040421 |
| [14] |
ACKERMANN H W. Frequency of morphological phage descriptions in the year 2000[J]. Arch Virol, 2001, 146(5): 843-857. DOI:10.1007/s007050170120 |
| [15] |
LEFKOWITZ E J, DEMPSEY D M, HENDRICKSON R C, et al. Virus taxonomy: the database of the International Committee on Taxonomy of Viruses (ICTV)[J]. Nucleic Acids Res, 2018, 46(D1): D708-D717. DOI:10.1093/nar/gkx932 |
| [16] |
葛展霞, 钟希娜, 朱炳海, 等. NDM-5阳性大肠杆菌裂解性噬菌体的生物学特性[J]. 江苏农业学报, 2018, 34(3): 699-705. GE Z X, ZHONG X N, ZHU B H, et al. The biological characteristics of a bacteriophage against NDM-5 positive Escherichia coli[J]. Jiangsu Journal of Agricultural Sciences, 2018, 34(3): 699-705. DOI:10.3969/j.issn.1000-4440.2018.03.032 (in Chinese) |
| [17] |
孙利厂, 周艳, 张莉莉, 等. 1株宽噬菌谱肠出血性大肠杆菌噬菌体的分离及生物学特性分析[J]. 中国动物传染病学报, 2018, 26(6): 58-63. SUN L C, ZHOU Y, ZHANG L L, et al. Isolation and biological characteristics of lytic enterohemorrhagic Escherichia coli bacteriophage[J]. Chinese Journal of Animal Infectious Diseases, 2018, 26(6): 58-63. (in Chinese) |
| [18] |
杨慧敏, 吴圆圆, 屈勇刚, 等. 一株鸡致病性大肠杆菌裂解性噬菌体的生物学特性及其对肠道菌群影响分析[J]. 中国家禽, 2018, 40(23): 18-22. YANG H M, WU Y Y, QU Y G, et al. Biological characteristics of a chicken pathogenic Escherichia coli lytic phage and its effect on intestinal flora[J]. China Poultry, 2018, 40(23): 18-22. (in Chinese) |
| [19] |
路建彪, 王俊丽, 吴伟胜, 等. 一株鸡大肠杆菌噬菌体的分离鉴定及治疗试验[J]. 中国预防兽医学报, 2019, 41(5): 530-533. LU J B, WANG J L, WU W S, et al. Isolation and identification of a strain of chicken coliphage and its treatment effect on Avian colibacillosis[J]. Chinese Journal of Preventive Veterinary Medicine, 2019, 41(5): 530-533. (in Chinese) |
| [20] |
CHEN L, LIU Q, FAN J Q, et al. Characterization and genomic analysis of ValSw3-3, a new Siphoviridae bacteriophage infecting vibrio alginolyticus[J]. J Virol, 2020, 94(10): e00066-20. |
| [21] |
THANH N C, NAGAYOSHI Y, FUJINO Y, et al. Characterization and genome structure of virulent phage EspM4VN to control Enterobacter sp. M4 isolated from plant soft rot[J]. Front Microbiol, 2020, 11: 885. DOI:10.3389/fmicb.2020.00885 |
| [22] |
HYMAN P, ABEDON S T. Practical methods for determining phage growth parameters[M]//CLOKIE M R, KROPINSKI A M. Bacteriophages. Totowa: Humana Press, 2009: 175-202.
|
| [23] |
ZHOU Y, BAO H D, ZHANG H, et al. Isolation and characterization of lytic phage vB_EcoM_JS09 against clinically isolated antibiotic-resistant avian pathogenic Escherichia coli and Enterotoxigenic Escherichia coli[J]. Intervirology, 2015, 58(4): 218-231. DOI:10.1159/000437426 |
| [24] |
AL-SHAYEB B, SACHDEVA R, CHEN L X, et al. Clades of huge phages from across Earth's ecosystems[J]. Nature, 2020, 578(7795): 425-431. DOI:10.1038/s41586-020-2007-4 |
| [25] |
BAILLY-BECHET M, VERGASSOLA M, ROCHA E. Causes for the intriguing presence of tRNAs in phages[J]. Genome Res, 2007, 17(10): 1486-1495. DOI:10.1101/gr.6649807 |
| [26] |
KWON J, KIM S G, KIM H J, et al. Bacteriophage as an alternative to prevent reptile-associated Salmonella transmission[J]. Zoonoses Public Health, 2021, 68(2): 131-143. DOI:10.1111/zph.12804 |
| [27] |
ANANY H, LINGOHR E J, VILLEGAS A, et al. A Shigella boydii bacteriophage which resembles Salmonella phage ViI[J]. Virol J, 2011, 8: 242. DOI:10.1186/1743-422X-8-242 |
(编辑 白永平)



