藏兔(Lepus tibetanus Waterhouse, 1841)隶属于兔形目(Lagomorpha)兔科(Leporidae)兔属(Lepus)。藏兔身型纤细,身体毛色主要呈沙黄色或浅褐色。分布于阿富汗、印度、吉尔吉斯斯坦、巴基斯坦、塔吉克斯坦、中国以及蒙古[1]。在中国境内主要分布于新疆西南部帕米尔高原地区,与新疆特有兔种塔里木兔(Lepus yarkandensis)的分布区有一定程度的重叠,易产生杂交和基因渗透现象[2]。藏兔主要以荒漠植物为食,但由于其分布区存在大量农田,也会对农作物产生危害,且藏兔皮毛及兔肉具有一定的经济价值,对当地畜牧农业发展有一定影响。
由于藏兔的分类地位一直存在争议,因此曾将藏兔和托氏兔(Lepus tolai)作为非洲草兔(Lepus capensis)的亚种进行研究[3]。但是,罗泽珣[4]和程承等[5]的研究表明,非洲草兔与新疆“草兔”间的形态学指标存在差异。Wang和Yang[6]基于线粒体全基因组水平的研究指出,中国“草兔”与南非草兔亲缘关系较远。Lado等[7]基于核基因和线粒体基因对非洲草兔的进化历史进行了研究,结果表明非洲草兔与中国兔属物种间的亲缘关系较远。单文娟等[8-11]基于线粒体基因及线粒体全序列探讨了新疆“草兔”的亚种分化及新疆兔属物种的分类,支持将新疆帕米尔高原的野兔划分为藏兔帕米尔亚种。以上研究表明,无论是基于形态学的头骨测量数据还是基于分子生物学的系统发育关系,均支持将生活在新疆帕米尔高原地区的野兔归为藏兔。但目前对藏兔的研究主要集中于分类地位的探讨,对该地区藏兔的群体遗传学研究鲜有报道。
帕米尔高原是由印度板块与欧亚板块碰撞形成的一处山结,平均海拔4 500 m,主要山峰均在6 000 m以上,属于显著的大陆性高山气候[12],其中约三分之一坐落于中国新疆西南部,即东帕米尔高原地区,该地区包括塔什库尔干县全部,阿克陶县大部及乌恰县南部3个行政区。东帕米尔高原地区环境的主要特点为:海拔高、气压低、含氧量低、降水少、辐射多、气候寒冷,这种极端生境对当地动物的生存是一种挑战。近年来,高原极端环境对动物遗传多样性与遗传结构的影响成为广受关注的研究热点,如有学者基于线粒体基因以及全基因组SNP等分子遗传工具对藏猪、藏鸡、藏狐以及藏羚羊等高原物种的遗传多样性与遗传结构进行了探讨,其结果表明高原环境是影响群体遗传多样性与遗传结构的一项重要因素[13-16],也是进一步研究极端环境适应性的基础。但高原极端环境会对藏兔的遗传多样性、遗传结构及遗传分化产生何种影响尚未见报道。
线粒体分子标记具有母系遗传、变异性强、成本低等特点,张玉琮等[17]在新疆兔属物种DNA条形码筛选的研究中发现,CO1与ND4基因表现出更强的变异水平和更高的鉴定准确率。因此,本研究选用CO1与ND4基因作为分子标记对采集自东帕米尔高原地区的藏兔进行研究,旨在探讨高原极端环境对藏兔的遗传多样性、遗传结构及遗传分化产生的影响。
1 材料与方法 1.1 样本信息本研究在东帕米尔高原及周边的4个行政区共收集了基于64项头骨指标[18]和线粒体基因[8]初步鉴定为藏兔的37例野兔整只样本,以下简称藏兔。将全部藏兔样本按照其地理位置及海拔高度分为4个地理种群,分别为位于东帕米尔高原南部的塔什库尔干县红其拉甫口岸种群(TXH)共13只样本,当地平均海拔4 700 m,是典型高原地区;阿克陶县及与其接壤的塔什库尔干县大同乡种群(AKT)共8只样本,当地平均海拔3 800 m;位于东帕米尔高原北部乌恰县种群(WQ)共8只样本,当地平均海拔2 100 m;位于东帕米尔高原周边的喀什市疏附县、疏勒县及泽普县种群(KS)共8只样本,当地平均海拔1 300 m,海拔高度相对较低(表 1)。
|
|
表 1 东帕米尔高原地区藏兔样本采集信息表 Table 1 The detailed information of L. tibetanus samples in East Pamirs |
本试验选择藏兔的耳尖组织通过试剂盒法提取全基因组DNA,所用试剂盒为天根生化科技(北京)有限公司生产的血液/细胞/组织基因组DNA提取试剂盒(离心柱型),提取后的全基因组DNA通过0.8%的琼脂糖凝胶进行电泳检测。
1.3 目的基因PCR扩增选择电泳条带清晰明亮的藏兔基因组总DNA样本作为模板,使用表 2中已发表的引物[17]和宝日医生物技术(北京) 有限公司生产的Premix Taq,用25 μL标准PCR反应体系(其中模板DNA 1 μL,终浓度为1.0 μmol·L-1的上、下游引物各1 μL,Premix Taq酶13 μL,去离子水9 μL)扩增CO1与ND4两个基因。反应条件:98 ℃预变性5 min;95 ℃变性10 s,两种引物退火条件相同均为51 ℃ 30 s,72 ℃延伸60 s,进行30个循环;72 ℃终末延伸5 min。扩增产物经1.5%的琼脂糖凝胶电泳检测合格后,送至生工生物工程(上海)股份有限公司进行测序。
|
|
表 2 藏兔线粒体基因PCR扩增反应所用引物 Table 2 Primers for PCR reaction of L. tibetanus mitochondrial genes |
用DNAMAN v6.0.40(Lynnon BioSoft)将测序合格的seq序列转换成fasta格式的文件便于后续分析;用MEGA7[19]提供的ClustalW[20]进行序列比对,比对条件均为默认,比对后将序列裁剪整齐;使用PhyloSuite v1.2.2[21]将CO1与ND4两个基因片段串联用于后续多位点序列分析(Multi-locus Sequence Analysis, MLSA);用MEGA 7与DnaSP6[22]对串联序列进行序列特征分析,并分选单倍型序列;用MEGA 7基于串联序列构建单倍型系统发育ML树,选择家兔(Oryctolagus cuniculus)作为外群,并基于CO1和ND4两个基因序列计算各地理种群间的遗传距离;对于藏兔杂交及线粒体基因渗透的探讨,为了使分析简明且有效,本研究使用部分有代表性的藏兔样本CO1基因与从GenBank中下载的包括塔里木兔在内的其他兔属物种的CO1基因一起构建ML树,仍选择家兔作为外群,表 3为本研究所用线粒体基因GenBank登录号;用Network[23]绘制单倍型网络图;用Arlequin[24]与DnaSP6基于串联序列进行遗传多样性分析、中性检验分析、分子方差分析及FST值的计算,根据公式
|
|
表 3 本研究所用线粒体基因GenBank登录号 Table 3 Mitochondrial gene accession number of Lepus downloaded from GenBank in this study |
以藏兔耳尖组织提取全基因组DNA作为模板,分别加入CO1、ND4特异性引物后进行PCR反应,扩增产物经电泳检测与预期产物大小一致,如图 1所示,选择条带清晰明亮的扩增产物进行测序分析。
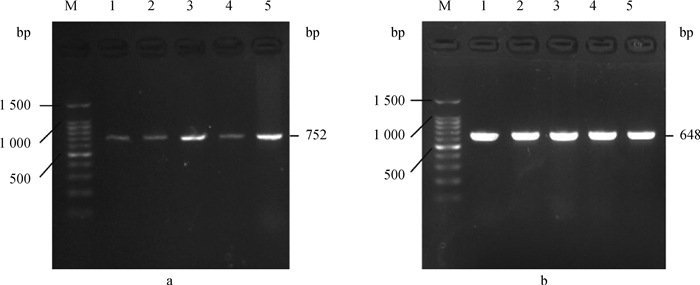
|
M. DNA相对分子质量标准;a. 藏兔CO1基因扩增产物;b. 藏兔ND4基因扩增产物 M. DL1500 marker; a. Amplification fragments of L.tibetanus CO1 gene; b. Amplification fragments of L.tibetanus ND4 gene 图 1 藏兔线粒体基因PCR扩增结果电泳图 Fig. 1 Results of PCR amplification of L. tibetanus mitochondria genes |
将藏兔两个线粒体基因CO1与ND4拼接,得到总长为1 145 bp的序列。碱基平均含量分别为A:27.4%,T:33.4%,C:25.2%,G:14.0%,T+A:60.8%,C+G:39.2%。共发现多态性位点(variable sites)77个,突变位点(mutations sites)78个,简约信息位点(parsimony informative sites)59个,转换(transition)12个、颠换(transversion)1个,转换颠换比(Ti/Tv)12,插入和缺失位点(insertion and deletion)共9个。
2.3 遗传多样性参数基于37条藏兔CO1、ND4基因串联序列共得到17种单倍型,分别按照4个不同地理种群计算各项遗传多样性参数,结果见表 4。总核苷酸多样性(π)为(0.012±0.006),其中WQ种群核苷酸多样性相对最低,为(0.003±0.002);TXH与AKT种群核苷酸多样性相对较高,分别为(0.009±0.005)、(0.011±0.006)。KS种群核苷酸多样性相对最高,为(0.021±0.012)。总单倍型多样性(h)为(0.935±0.021),其中TXH种群的相对较低为(0.833±0.082),AKT、WQ与KS 3个种群的单倍型多样性相对较高,分别为(0.893±0.086)、(0.893±0.111)、(0.893±0.111)。中性检验Tajima`s D值中,TXH、KS种群为正,AKT、WQ种群及总体结果为负;Fu`s Fs值中,除WQ种群为负值以外,其它均为正值,所有结果P值均未达到显著水平。
|
|
表 4 基于藏兔CO1、ND4基因计算的遗传多样性参数 Table 4 Genetic diversity parameters calculated based on L. tibetanus CO1 and ND4 genes |
2.4.1 系统发育分析 以家兔为外群,基于藏兔17种串联序列单倍型,通过ML法构建的系统发育树显示(图 2),全部单倍型聚为明显的两枝,其中一个大枝又以较高的置信度分出两个小枝,共3个小枝,分别用Clade A-C表示。Clade A枝中的单倍型囊括了全部4个采样地的样本,且不同地理种群之间有共享单倍型;Clade B中仅包含来自TXH种群的4个单倍型;Clade C则由两个来自KS地区的单倍型和一个来自AKT地区的单倍型组成。
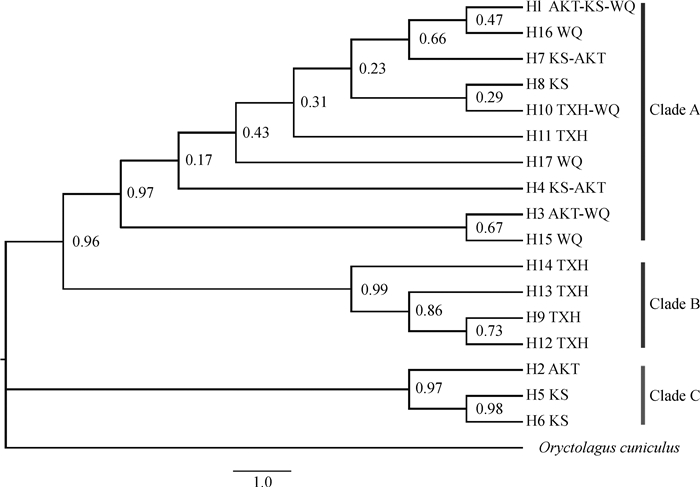
|
图 2 基于藏兔17种串联序列单倍型构建的ML树 Fig. 2 The ML tree based on 17 haplotypes of Lepus tibetanus concatenate sequences |
2.4.2 遗传距离 本研究中藏兔4个地理种群之间的遗传距离见表 5,结果显示CO1基因计算出的遗传距离相对ND4较小,但变化趋势基本一致。KS种群与另外3个地理种群间的遗传距离均相对较大,分别为KS-TXH=1.63%/1.96%,KS-AKT=1.35%/1.67%,KS-WQ=1.21%/1.44%;WQ-AKT群体遗传距离最小为0.57%/0.82%。种群内遗传距离计算结果中,KS种群相对最大为1.93%/2.23%,WQ种群相对最小为0.21%/0.38%。
|
|
表 5 基于藏兔CO1、ND4基因计算的遗传距离 Table 5 Genetic distance calculated based on Lepus tibetanus CO1 and ND4 genes |
2.4.3 单倍型中介网络图 基于藏兔17种串联序列单倍型构建的中介网络图如图 3所示,结果与藏兔系统发育树一致,全部单倍型共聚为左右两枝,左边又可以细分为两小枝,左下角的小枝由来自4个地理种群的混合单倍型组成;左上角的小枝由来自TXH种群的4种单倍型组成;右边小枝由来自KS种群的2种单倍型与来自AKT种群的1种单倍型组成,且与另外两枝间距离较远。

|
图 3 基于藏兔17种串联序列单倍型构建的中介网络图 Fig. 3 The median-joining network based on 17 haplotypes of L. tibetanus concatenate sequences |
2.5.1 兔属物种系统发育树 选取图 2藏兔系统发育树CladeA、B两枝中的部分样本以及Clade C中全部样本的CO1基因,与GenBank中发表的其他兔属物种的CO1基因一起构建了ML树(图 4)。结果显示,Clade C中全部3个样本与塔里木兔聚成一枝,显示出与塔里木兔更近的亲缘关系。CladeA、Clade B的藏兔聚成一枝,加上Clade C以及塔里木兔一起聚为一个的大枝,该枝与雪兔及托氏兔亲缘关系稍近,与非洲草兔亲缘关系最远。
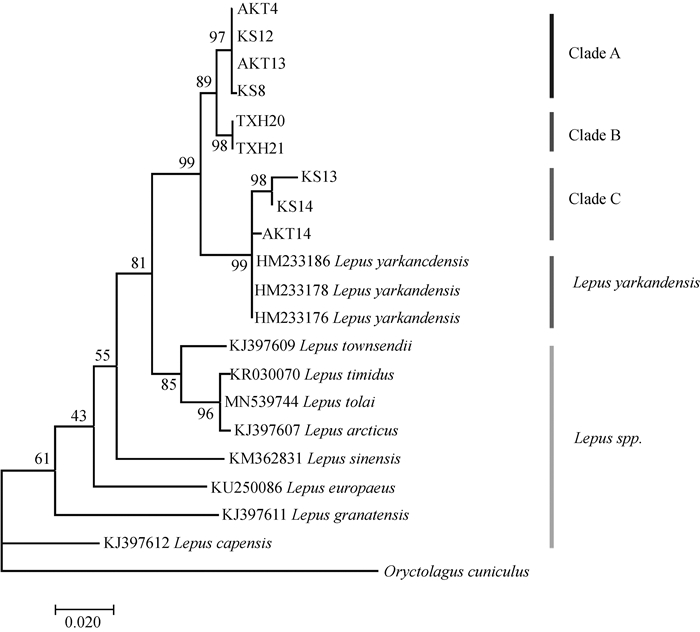
|
图 4 基于兔属物种CO1基因构建的ML树 Fig. 4 ML tree constructed based on CO1 gene of Lepus |
2.5.2 AMOVA及分化指数FST 分子方差分析(Analysis of Molecular Variance, AMOVA)中,将样本分为4个基因池[TXH][AKT][WQ][KS]和2个基因池[TXH][AKT WQ KS],[AKT][TXH WQ KS]、[WQ][TXH AKT KS]、[KS][TXH AKT WQ]分别计算分子变异与FST值,结果如表 6所示。按照不同地理种群分为4个基因池时,种群间变异小于种群内变异,FST值为0.168;将每个地理种群与剩余地理种群分为2个基因池时,种群间变异均小于种群内变异,TXH种群FST值相对最高为0.228(P < 0.01),AKT、WQ和KS种群FST值相对较小,分别为0.002(P < 0.01)、0.063(P < 0.05)、0.097(P < 0.01)。
|
|
表 6 藏兔分子方差分析 Table 6 Analysis of molecular variance of L. tibetanus |
2.5.3 遗传分化指数与基因流 藏兔不同地理种群间的遗传分化指数FST与基因流Nm结果显示(表 7),TXH与WQ种群间FST值最大为0.344,AKT与WQ种群间FST值最小为0.015,各种群间FST的P值均小于0.01。根据FST计算出的基因流中,AKT-WQ组基因流最大为16.495,其次为AKT-KS组为4.500,以及WQ-KS组为2.077,TXH-AKT、TXH-WQ、TXH-KS 3个组的基因流相对较小,均低于1。
|
|
表 7 藏兔遗传分化指数FST与基因流 Table 7 FST and gene flow among L. tibetanus populations |
单倍型多样性(h)与核苷酸多样性(π)是反映遗传多样性的重要指标,遗传多样性高代表物种基因库积累的变异丰富,应对环境变化能力强;反之则说明该物种受到了选择压力的影响或经历了种群收缩。本研究中,藏兔样本总核苷酸多样性及海拔较高的TXH、AKT、WQ 3个种群的核苷酸多样性均小于0.012,低于已经报道的其他兔属物种如塔里木兔(0.045±0.022)、托氏兔(0.033±0.018)、欧兔(0.027±0.003)、雪兔(0.057±0.029)等,仅海拔较低的KS种群核苷酸多样性较高为(0.021±0.012)与其他兔属物种接近。相应的,海拔最高的TXH种群的单倍型多样度也较低为(0.833±0.082),AKT、WQ和KS 3个种群单倍型多样度相对较高为(0.893±0.111),但仍低于塔里木兔(0.979±0.014)和欧兔(0.988±0.004)[25-27]。中性检验结果的两个指标均未检测到近期种群扩张的显著证据。因此认为在线粒体基因水平上,东帕米尔高原地区的藏兔随着海拔的升高,核苷酸多样性与单倍型多样性均有降低,且当地藏兔遗传多样性水平总体偏低,应对环境变化能力弱,可能需要补充制定相关保护措施。
3.2 藏兔的群体遗传结构地理隔离影响物种遗传结构、基因交流与物种分化,帕米尔高原山地中沟壑纵横且分布有大量现代冰川,是形成地理隔离的因素之一[28]。系统发育分析表明(图 2),东帕米尔高原地区的藏兔样本共分成3枝Clade A-C,除Clade C枝中的样本被怀疑受到了杂交的影响外,其余两枝由海拔相异的4个地理种群的藏兔相互混杂在一起,并未呈现出较强的地理分布模式。中介网络图(图 3)也呈现出与此一致的结果。值得注意的是,海拔最高的TXH种群在系统发育树中被分为两部分,一部分与其他地理种群一起聚在Clade A中,另一部分单独聚类在Clade B中,同一个地区的样本出现了相互独立的两个进化枝。王玉涛等[29]在对东帕米尔高原雪鸡的系统发育研究中,也发现了类似的聚类情况。Sarabia等[30]在关于非洲金狼的研究中同样发现了类似的聚类情况,并认为这种现象是由于物种占据了不同的生态型而产生的,虽然为同一个采样地的同一个物种,但其种群内遗传结构仍存在差异,物种在不断扩张和收缩的过程中,不同种群由于食物和栖息地竞争而形成了食性不同或栖息地碎片化的不同生态型,由于高原环境容易形成地理屏障,阻止了不同生态型群体之间的基因交流,进而使同一地理区域分布的同物种产生了一定的分化。
在此基础上,本研究分析了4个藏兔地理种群两两群体间的遗传距离,遗传距离反映不同群体间的差异大小和亲缘关系,遗传距离越近,两个群体相似程度越高,亲缘关系也越近;反之则表示两群体间差异越大,亲缘关系越远[31-32]。从结果可以看出(表 5),WQ-AKT组遗传距离最小。KS种群与其他地理种群间遗传距离最大,且是唯一一个种群内遗传距离大于种群间遗传距离的群体,这可能是受到了Clade C中两个来自KS地区杂交样本的影响。
3.3 藏兔的群体遗传分化为了进一步检测高原环境影响下不同群体间的差异与基因交流情况,本研究进行了AMOVA分子方差分析,并计算了固定系数FST和基因流。固定系数取值范围从1到0,值越高,则说明群体间差异越大,分化程度也越大,一般认为,FST值大于0.25就说明群体间已经产生了明显的分化[33]。本研究分析结果显示(表 6),4个基因池分析时,4个地理种群之间FST值为0.168,2个基因池分析时,TXH种群FST值最大为0.228,P < 0.01,其他种群FST相对较小(均低于0.1,P < 0.05),说明不同种群间分子差异主要来源于TXH种群。各种群间分化指数与基因流的分析结果也显示(表 7),TXH与AKT、WQ、KS种群间的FST较高(均大于0.4),且基因流较小(均小于1),表明TXH种群分化程度最大。从地理上看,由于TXH种群采样地的海拔为所有采样点中最高(4 700 m),当地藏兔需面临更为严苛的高原环境的挑战,且Naldi等[34]在其研究中指出,高原陡坡会影响欧兔的迁移和分布,红其拉甫口岸的极端高海拔环境形成的地理屏障加大了当地兔属物种与周边地区兔属物种进行基因交流的难度,使遗传分化指数升高。
而AKT-WQ组的FST值相对最小,基因流最大(FST=0.015,Nm=16.495),表明AKT与WQ种群间分化程度不明显,且保持着密切的基因交流,这与表 5中计算的AKT-WQ组遗传距离结果一致。AKT-KS、WQ-KS两组的基因流也相对较高,分别为4.500和2.007。且两地之间多有绿洲、耕地相连。AKT种群采样地阿克陶县虽然海拔相对较高(平均海拔3 800 m),但北部与乌恰县(平均海拔2 100 m)相连,南部有叶儿羌河将其与东部喀什地区的泽普县(平均海拔1 300 m)相连。说明AKT、WQ和KS 3个种群的藏兔虽然海拔高度有差异,但依托周围地理条件,仍保持着一定的基因交流。
3.4 藏兔线粒体基因渗透现象虽然本研究中的全部样本基于形态学及分子生物学数据被初步划分为藏兔,但基于藏兔线粒体基因串联序列单倍型构建的系统发育树(图 2)及中介网络图(图 3)显示,Clade C枝中2个KS地区的藏兔样本与1个AKT地区的藏兔样本均以单系分布,与其他地区藏兔样本形成的另外两小枝组成的一大支呈并系关系。由于全世界其他兔属物种CO1基因数据较为全面,因此选用CO1基因构建世界兔属物种系统发育树(图 4),结果显示Clade C枝中的3个样本与数据库中下载的塔里木兔亲缘关系最近。基于藏兔两个线粒体基因(CO1、ND4)分别计算的遗传距离结果也显示,KS种群与其他种群遗传距离较大,且种群内遗传距离大于种群间遗传距离,说明KS种群内的样本可能发生过杂交。从地理上看,该枝中的3个样本所处的地理位置(喀什市与阿克陶县)位于塔里木盆地边缘,与塔里木兔的分布范围重叠[8],是两个物种的分布接触区。
Alves等[35]早在2003年研究兔属物种遗传结构时就发现该属存在由于杂交导致的线粒体基因渗透(introgression of mitochondria DNA)现象,Ferreira等[36]在其关于欧兔与伊比利亚野兔的研究中指出,在两种野兔分布范围的接触区,由于物种入侵和收缩,导致了反复杂交引起的线粒体基因渗透现象,Wu等[2]及Liu等[37]曾报道,中国兔属不同种之间存在杂交和线粒体基因渗透现象,可以看出,在兔属物种接触区发生杂交及线粒体基因渗透是频繁且常见的现象。综上所述,认为Clade C中的3个藏兔样本可能受到了与塔里木兔杂交而产生的线粒体基因渗透的影响。
4 结论本研究基于东帕米尔高原地区藏兔线粒体CO1及ND4两个基因的分析表明,TXH、AKT、WQ种群的藏兔受高原极端环境影响,遗传多样性水平较低,应对环境变化能力较弱,应加强当地的种质资源保护意识,制定相关保护政策。在遗传结构上不同采样地种群没有呈现出较强的系统地理分布模式。红其拉甫口岸的藏兔种群由于高原地理屏障作用,可能形成了不同生态型且与周边地区藏兔存在一定程度的分化。此外,本研究还发现,藏兔与塔里木兔分布范围接触区可能存在线粒体基因渗透现象。
| [1] |
SMITH A T, JOHNSTON C H, ALVES P C, et al. Lagomorphs: pikas, rabbits, and hares of the world[M]. Baltimore: Johns Hopkins University Press, 2018.
|
| [2] |
WU Y, XIA L, ZHANG Q, et al. Bidirectional introgressive hybridization between Lepus capensis and Lepus yarkandensis[J]. Mol Phylogenet Evol, 2011, 59(3): 545-555. DOI:10.1016/j.ympev.2011.03.027 |
| [3] |
相雨, 杨奇森, 夏霖. 中国兔属动物的分类现状和分布[J]. 四川动物, 2004, 23(4): 391-397. XIANG Y, YANG Q S, XIA L. Distribution and taxonomy of genus Lepus (Hares) in China[J]. Sichuan Journal of Zoology, 2004, 23(4): 391-397. DOI:10.3969/j.issn.1000-7083.2004.04.026 (in Chinese) |
| [4] |
罗泽珣. 中国野兔[M]. 北京: 中国林业出版社, 1988. LUO Z X. The Chinese hare[M]. Beijing: China Forestry Publishing House, 1988. (in Chinese) |
| [5] |
程承, 葛德燕, 夏霖, 等. 中国"草兔"头骨的形态计量学研究[J]. 兽类学报, 2012, 32(4): 275-286. CHENG C, GE D Y, XIA L, et al. Morphometrics study on the so called'Cape hare'(Lagomorpha: Leporidae: Lepus) in China[J]. Acta Theriologica Sinica, 2012, 32(4): 275-286. (in Chinese) |
| [6] |
WANG J L, YANG G. Complete mitogenome of cape hare Lepus capensis (Lagomorpha: Lepo-ridae) and its phylogenetic considerations[J]. Acta Theriol Sin, 2012, 32(1): 1-11. |
| [7] |
LADO S, ALVES P C, ISLAM M Z, et al. The evolutionary history of the Cape hare (Lepus capensis sensu lato): insights for systematics and biogeography[J]. Heredity, 2019, 123(5): 634-646. DOI:10.1038/s41437-019-0229-8 |
| [8] |
单文娟, 代慧英, 张玉琮. 基于线粒体基因的新疆三种野兔分类及遗传多样性[J]. 畜牧兽医学报, 2020, 51(10): 2403-2412. SHAN W J, DAI H Y, ZHANG Y C. Classification and genetic diversity of three hare species in Xinjiang based on mitochondrial DNA[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(10): 2403-2412. DOI:10.11843/j.issn.0366-6964.2020.10.008 (in Chinese) |
| [9] |
SHAN W J, ZHANG Y C, TURSUN M, et al. The complete mitochondrial genome sequence of Lepus tibetanus pamirensis[J]. Mitochondrial DNA B, 2020, 5(2): 1359-1360. DOI:10.1080/23802359.2020.1736959 |
| [10] |
SHAN W J, TURSUN M, ZHOU S Y, et al. The complete mitochondrial genome sequence of Lepus tolai in Xinjiang[J]. Mitochondrial DNA B, 2020, 5(2): 1336-1337. DOI:10.1080/23802359.2020.1735267 |
| [11] |
单文娟, 刘江, 马合木提哈力克. 新疆草兔的种群遗传结构和亚种分化[J]. 动物学研究, 2011, 32(2): 179-187. SHAN W J, LIU J, HALIK M. Genetic structure and subspecies divergence of Lepus capensis in Xinjiang[J]. Zoological Research, 2011, 32(2): 179-187. (in Chinese) |
| [12] |
张芬芬. 帕米尔东北缘乌泊尔地区新生代构造特征[D]. 杭州: 浙江大学, 2010: 1-14. ZHANG F F. Cenozoic structure features of Wupoer area in northeastern margin of pamir[D]. Hangzhou: Zhejiang University, 2010: 1-14. (in Chinese) |
| [13] |
ZHAO C, ZHANG H H, LIU G S, et al. The complete mitochondrial genome of the Tibetan fox (Vulpes ferrilata) and implications for the phylogeny of Canidae[J]. C R Biol, 2016, 339(2): 68-77. DOI:10.1016/j.crvi.2015.11.005 |
| [14] |
GE R L, CAI Q L, SHEN Y Y, et al. Draft genome sequence of the Tibetan antelope[J]. Nat Commun, 2013, 4: 1858. DOI:10.1038/ncomms2860 |
| [15] |
蔡原, 赵生国. 藏猪遗传多样性及系统关系研究[J]. 国外畜牧学-猪与禽, 2011, 31(5): 60-62. CAI Y, ZHAO S G. Study on genetic diversity and phylogenetic relationships of Tibetan pigs[J]. Animal Science Abroad (Pigs and Poultry), 2011, 31(5): 60-62. DOI:10.3969/j.issn.1001-0769.2011.05.027 (in Chinese) |
| [16] |
杜志强, 曲鲁江, 李显耀, 等. 藏鸡群体遗传多样性研究[J]. 遗传, 2004, 26(2): 167-171. DU Z Q, QU L J, LI X Y, et al. Genetic diversity in Tibetan chicken[J]. Hereditas (Beijing), 2004, 26(2): 167-171. (in Chinese) |
| [17] |
张玉琮, 曾婉琴, 许盼, 等. 新疆野兔DNA条形码筛选[J]. 畜牧兽医学报, 2020, 51(2): 270-278. ZHANG Y C, ZENG W Q, XU P, et al. The screening of DNA barcode for hares in Xinjiang[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 270-278. (in Chinese) |
| [18] |
代慧英, 单文娟. 基于头骨形态指标的新疆野兔年龄鉴定[J]. 黑龙江畜牧兽医, 2021(1): 138-141. DAI H Y, SHAN W J. Age identification of Xinjiang hare based on skull morphology indexes[J]. Heilongjiang Animal Science and Veterinary Medicine, 2021(1): 138-141. (in Chinese) |
| [19] |
KUMAR S, STECHER G, TAMURA K. MEGA7:molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [20] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [21] |
ZHANG D, GAO F L, JAKOVLI Ć I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Mol Ecol Resour, 2020, 20(1): 348-355. DOI:10.1111/1755-0998.13096 |
| [22] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [23] |
POLZIN T, DANESHMAND S V. On Steiner trees and minimum spanning trees in hypergraphs[J]. Oper Res Lett, 2003, 31(1): 12-20. DOI:10.1016/S0167-6377(02)00185-2 |
| [24] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Mol Ecol Resour, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [25] |
PIERPAOLI M, RIGA F, TROCCHI V, et al. Hare populations in Europe: intra and interspecific analysis of mtDNA variation[J]. C R Biol, 2003, 326(Suppl 1): 80-84. |
| [26] |
SHAN W J, LIU J, YU L, et al. Genetic consequences of postglacial colonization by the endemic Yarkand hare (Lepus yarkandensis) of the arid Tarim Basin[J]. Chin Sci Bull, 2011, 56(13): 1370-1382. DOI:10.1007/s11434-011-4460-9 |
| [27] |
KUMAR B, CHENG J L, GE D Y, et al. Phylogeography and ecological niche modeling unravel the evolutionary history of the Yarkand hare, Lepus yarkandensis (Mammalia: Leporidae), through the Quaternary[J]. BMC Evol Biol, 2019, 19(1): 113. DOI:10.1186/s12862-019-1426-z |
| [28] |
尚华明, 范煜婷, 张瑞波, 等. 帕米尔高原东部径流量变化及其对气候变化的响应[J]. 气候变化研究进展, 2021, 17(3): 352-360. SHANG H M, FAN Y T, ZHANG R B, et al. Streamflow variation in the eastern Pamirs and its response to climate change[J]. Climate Change Research, 2021, 17(3): 352-360. (in Chinese) |
| [29] |
王玉涛, 潘建飞, 黄翠翠, 等. 东帕米尔高原喜马拉雅雪鸡遗传多样性及系统发育地位[J]. 生态学报, 2018, 38(1): 316-324. WANG Y T, PAN J F, HUANG C C, et al. Genetic diversity and phylogenetic status of Tetraogallus himalayensis in East Pamir, Xinjiang, China[J]. Atca Ecologica Sinica, 2018, 38(1): 316-324. (in Chinese) |
| [30] |
SARABIA C, VONHOLDT B, LARRASOAÑA J C, et al. Pleistocene climate fluctuations drove demographic history of African golden wolves (Canis lupaster)[J]. Mol Ecol, 2020. DOI:10.1111/mec.15784 |
| [31] |
HEBERT P D N, STOECKLE M Y, ZEMLAK T S, et al. Identification of Birds through DNA Barcodes[J]. PLoS Biol, 2004, 2(10): e312. DOI:10.1371/journal.pbio.0020312 |
| [32] |
徐春燕, 刘勇, 马超, 等. 基于COⅠ基因的DNA条形码技术在石首鱼科鱼类系统分类中的应用[J]. 渔业研究, 2019, 41(5): 359-365. XU C Y, LIU Y, MA C, et al. Application of DNA barcoding based on COⅠ gene in phylogenetic classification of Sciaenidae[J]. Journal of Fisheries Research, 2019, 41(5): 359-365. (in Chinese) |
| [33] |
CURNOW R N, WRIGHT S. Evolution and the genetics of populations, Volume 4:variability within and among natural populations[J]. Biometrics, 1979, 35(1): 359. DOI:10.2307/2529965 |
| [34] |
NALDI L, GRECO I, FERRETTI M, et al. Density estimates and habitat preferences of the european hare (Lepus europaeus) on mountainous areas in Italy[J]. Mamm Study, 2020, 45(2): 123-131. DOI:10.3106/ms2019-0057 |
| [35] |
ALVES P C, FERRAND N, SUCHENTRUNK F, et al. Ancient introgression of Lepus timidus mtDNA into L.granatensis and L.europaeus in the Iberian Peninsula[J]. Mol Phylogenet Evol, 2003, 27(1): 70-80. DOI:10.1016/S1055-7903(02)00417-7 |
| [36] |
FERREIRA M S, JONES M R, CALLAHAN C M, et al. The legacy of recurrent introgression during the radiation of hares[J]. Syst Biol, 2021, 70(3): 593-607. DOI:10.1093/sysbio/syaa088 |
| [37] |
LIU J, YU L, ARNOLD M L, et al. Reticulate evolution: frequent introgressive hybridization among Chinese hares (genus Lepus) revealed by analyses of multiple mitochondrial and nuclear DNA loci[J]. BMC Evol Biol, 2011, 11: 223. DOI:10.1186/1471-2148-11-223 |
(编辑 郭云雁)



