2. 西南医科大学科技处 实验动物中心, 泸州 646000;
3. 西南医科大学体育学院, 泸州 646000
2. Experimental Animal Center, Technology Department, Southwest Medical University, Luzhou 646000, China;
3. School of Physical Education, Southwest Medical University, Luzhou 646000, China
副猪嗜血杆菌(Glaesserella parasuis)是一种非运动性、烟酰胺腺嘌呤二核苷酸(NAD) 依赖的细菌[1],可引起仔猪的猪格氏病(Glässer’s disease)[2],给全世界养猪行业带来巨大的经济损失[3-4]。副猪嗜血杆菌主要临床症状为发热、咳嗽、呼吸困难、跛行、共济失调、皮毛粗糙[5-6]。副猪嗜血杆菌常定植于猪上呼吸道,宿主遇到压力时,可穿透黏膜屏障进入血流,导致严重的血管病变和多种综合征[7]。
双组分系统由位于细胞膜上的组氨酸蛋白激酶(HK)和细胞质中的反应调节因子(RR)组成[8],参与调控细菌的生长繁殖、代谢、运动、耐药和毒力因子的表达[9]。双组分系统广泛存在于副猪嗜血杆菌、大肠杆菌、沙门菌、流感嗜血杆菌和胸膜肺炎放线杆菌中[10-15]。
QseBC双组分系统是肠杆菌科细菌和巴氏杆菌中重要的毒力调控因子[16]。QseC是一种跨膜蛋白,作为密度感应的组氨酸激酶受体[17],参与体内细菌运动、细胞侵袭和竞争性定植。在大肠杆菌中,QseC诱导autoinducer-3和/或肾上腺素/去甲肾上腺素信号分子的自磷酸化,并将磷酸基团转移到QseB,调节毒力相关基因的转录[18]。qseC基因缺失时,细胞内细菌群落(IBCs)形成的缺陷削弱了副猪嗜血杆菌感染小鼠的能力[11, 19-20]。QseC在大肠杆菌中诱导肾上腺素并促进生物膜形成。QseB调控伤寒沙门菌的生物膜形成和毒力[21]。
在这项研究中,作者通过自然转化法构建了ΔqseBC双基因缺失株,并对其在血清抗性、生物膜形成、抗生素抗性和胁迫耐受性中的作用进行了研究,以确定QseBC双组分系统在副猪嗜血杆菌致病过程中的作用。
1 材料与方法 1.1 菌株、引物及细菌生长条件本研究中使用的菌株见表 1,引物见表 2。野生型菌株SC1401(血清型11型)由四川农业大学动物医学院猪病研究中心提供。ΔqseC、ΔqseBC基因缺失株和互补菌株(C-ΔqseBC)由本实验室在前期研究中构建并保存[22]。副猪嗜血杆菌在胰蛋白酶大豆肉汤(TSB;Difco,美国)或胰蛋白酶大豆琼脂(TSA;Difco NJ,美国)平板上生长,补充5% 灭活牛血清和0.1% 烟酰胺腺嘌呤二核苷酸(NAD; Sigma-Aldrich, USA)。培养基因缺失株或互补株时,培养基中添加卡那霉素(50 μg·mL-1)或庆大霉素(20 μg·mL-1)。
|
|
表 1 本研究所用的菌株 Table 1 Strains used in this study |
|
|
表 2 本研究所用的引物 Table 2 Primers used in this study |
ΔqseBC基因缺失株和互补株C-ΔqseBC由本实验室在之前的研究中构建和保存[22]。将冻干菌株SC1401、ΔqseBC和C-ΔqseBC从-80 ℃冰箱中取出。用接种环小心挑取细菌,放入装有TSB培养基的试管中,37 ℃培养13 h,吸取菌液进行PCR鉴定。用软件Primer Premier 5.0设计基因缺失及鉴定引物(表 2)。
1.3 抗血清杀菌试验抗血清杀菌试验按照之前描述的方法进行[23]。取健康仔猪血清,用0.22 μm滤膜过滤。部分血清在56 ℃下处理30 min,使补体失活。SC1401、ΔqseBC和C-ΔqseBC在TSB中培养至OD600 nm为0.8。然后,80 μL细菌培养物与20 μL新鲜血清或热处理血清混合。混合物在37 ℃下轻轻摇动孵育1 h,随后将混合物稀释后涂布于TSA板上,37 ℃孵育24 h。通过测定新鲜血清和热处理血清中菌落的比例来计算存活率。每个样本至少进行3次重复。
1.4 压力耐受试验压力耐受试验根据之前描述的方法进行。将50 μL SC1401、ΔqseBC和C-ΔqseBC的过夜培养物以1∶100的稀释度传代培养到5 mL TSB中,培养至OD600 nm为0.8。对于渗透压耐受能力测定,细菌分别在50、100、150和200 mmol·L-1 NaCl的TSA板上培养。对于氧化应激耐受试验,将细菌分别用1、2、4、8、16 mmol·L-1的H2O2处理30 min。对于热休克试验,细菌分别在39、42或45 ℃中孵育30 min。每个菌株未处理菌液作为阴性对照纳入每个试验。培养后,在PBS中连续稀释培养物,并通过平板计数测定CFU。抗应激细胞的百分比计算为[(应激样品CFU·mL-1)/(对照样品CFU·mL-1)]× 100%。每项分析单独进行3次。
1.5 微孔板生物被膜检测试验生物被膜的形成有助于细菌抵御不良环境的侵害,帮助其逃避宿主的免疫系统和抗生素的作用,导致细菌的抵抗能力增强[24]。由于副猪嗜血杆菌野生型菌株CF7066及其突变株ΔclpP可以在48 h形成生物膜[7],并且流感嗜血杆菌也具有在24 h形成生物膜的能力[25]。参考之前报道进行生物被膜形成试验[14],通过96孔微孔板定量检测副猪嗜血杆菌的生物被膜形成[26]。利用生物被膜内物质可与某些染料结合的特点,通过对生物被膜染色的方法进行定性或定量分析。为了确定基因缺失菌株的生物膜形成是否随着时间的推移始终低于野生菌株。将生物膜形成观察时间延长至96 h。将SC1401、ΔqseBC和C-ΔqseBC接种96孔圆底微量滴定板,37 ℃分别培养48、72和96 h。去除细菌悬浮液,流水洗涤松散黏附在微孔板上的细菌,将微孔板上的生物膜用99%的甲醇溶液固定15 min,移除甲醇,自然干燥后用0.1%的Hucker’s结晶紫(CV)溶液染色30 min,再经水洗、干燥,33%的乙酸溶解被结合的结晶紫。最后在595 nm处测量光密度(OD)。副猪嗜血杆菌SC1401、ΔqseBC和C-ΔqseBC均设8个重复孔,试验独立进行3次。采用t检验分析,P < 0.05为差异有统计学意义。
1.6 抗生素敏感性测定抗生素敏感性试验参考文献[27]进行。浮游细菌的最小抑制浓度(MIC-P)通过肉汤微量稀释测定法测定,使用新鲜TSB培养基进行连续2倍稀释。微量滴定板在37 ℃下孵育,并在24 h后对生长或不生长进行目测评分。
生物膜细菌的最小杀菌浓度(MBC-B)按所述方法确定[28]。将96孔微量滴定板中过夜培养的生物膜菌暴露于连续稀释的抗生素至少24 h。然后,用新鲜TSB替换含抗生素的培养基并培养24 h。将菌液涂布于TSA培养观察菌落个数来评估生物膜中细菌的生存能力。所有试验独立进行3次。
1.7 数据分析试验数据使用GraphPad PRISM 5.0软件分析,当两个试验组进行比较时使用t检验分析,当3个或更多试验组进行比较时使用ANOVA分析。
2 结果 2.1 ΔqseBC缺失株和互补株的复苏与鉴定对复苏的细菌进行PCR鉴定(图 1)。只有SC1401和C-ΔqseBC中能扩增出qseB(672 bp)和qseC(1 401 bp)条带,而ΔqseBC中无法扩增出对应条带。当使用引物Kan-F/R检测时,亲本株不产生条带,缺失株和互补株产生条带,大小为935 bp。
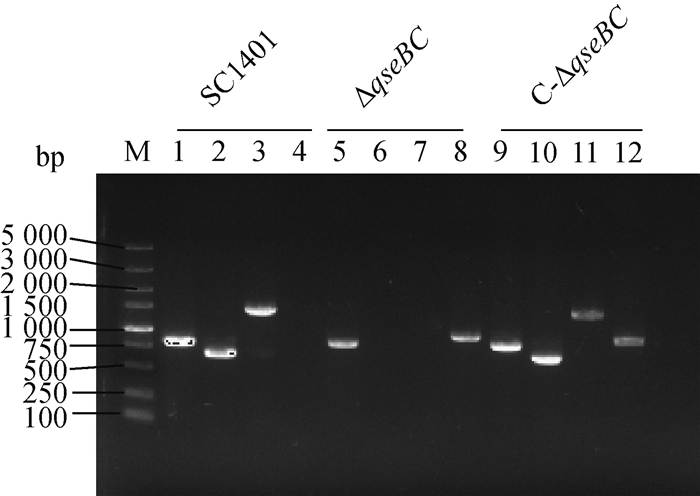
|
M.DL5000 DNA相对分子质量标准;1、5、9. 引物HPS-F/ R鉴定;2、6、10. 引物qseB-F/ R鉴定;3、7、11. 引物qseC-F/ R鉴定;4、8、12. 引物Kan-F/ R鉴定 M.DL5000DNA marker; 1, 5, 9. Identification with primers HPS-F/ R; 2, 6, 10. Identification with primers qseB-F/R; 3, 7, 11. Identification with primers qseC-F/R; 4, 8, 12. Identification with primers Kan-F/ R 图 1 ΔqseBC基因缺失株和互补株的鉴定 Fig. 1 Identification of ΔqseBC mutant and complementary strain C-ΔqseBC |
SC1401、ΔqseBC和C-ΔqseBC均能扩增出822 bp条带,表明三者均为副猪嗜血杆菌。使用引物P5 (qseB-F)/P6 (qseB-R) 从野生株和互补株中扩增出672 bp的qseB基因片段,但未从基因缺失株中扩增出此条带。使用引物P7 (qseC-F)/P8 (qseC-R) 从野生株和互补株中扩增出1 401 bp的qseC基因片段,但未从基因缺失株中扩增出此条带。使用引物P1(Kan-F)/P2(Kan-R)从基因缺失株和互补株中扩增出935 bp的kan片段,但野生型菌株SC1401中没有扩增出此条带,表明ΔqseBC基因缺失株及其互补菌株构建成功。
2.2 亲本株和缺失株抗血清杀菌能力比较结果(图 2)显示,野毒株SC1401具有更高水平的抗血清杀菌能力(P < 0.05)。ΔqseBC、SC1401和C-ΔqseBC的存活率分别为55.12%、83.76%和81.81%。这些结果表明QseBC可能与副猪嗜血杆菌抗血清杀菌能力相关。
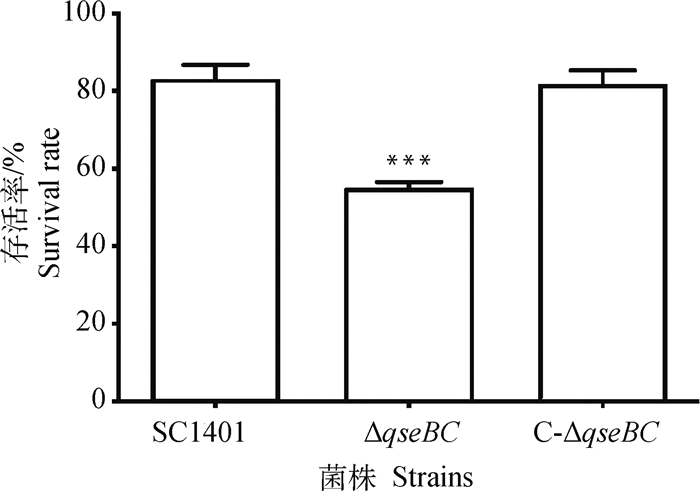
|
试验进行3次重复,每次重复设3个平行组,误差线代表 3次重复试验的标准差, 星号表示使用双向方差分析的统计显著性(***. P < 0.001) Data indicate the means of three independent experiments performed in duplicate, and error bars show standard deviations. Asterisks indicate a statistical significance using a two-way ANOVA (***. P < 0.001) 图 2 SCl401、ΔqseBC和C-ΔqseBC在猪血清中的存活率 Fig. 2 Survival rate of SCl401, ΔqseBC and C-ΔqseBC treated with porcine serum |
本研究测定了SC1401、ΔqseBC和C-ΔqseBC在不同胁迫条件下的存活率。当细胞暴露于50、100和150 mmol·L-1 NaCl,ΔqseBC的存活率分别为84.12%、45.35%和15.75%,远低于SC1401(92.96%、82.85% 和35.5%)(图 3A)。细胞用1、2、4、8和16 mmol·L-1 H2O2处理30 min时,ΔqseBC的存活率为50.71%~68.90%,远低于SC1401(67.14%~94.70%)(图 3B)。在热休克试验中也观察到类似结果,ΔqseBC的存活率在39、42 ℃分别为94.57%和82.12%,与SC1401(94.57%和82.12%)相比显著降低。在45 ℃下孵育时,SC1401、ΔqseBC和C-ΔqseBC菌株无法存活(图 3C)。C-ΔqseBC恢复了对渗透压、氧化应激和热休克的耐受能力。这些结果表明,qseBC双基因的缺失削弱了副猪嗜血杆菌对氧化、高渗和热休克应激的耐受力,证实qseBC是副猪嗜血菌中的胁迫相关基因。
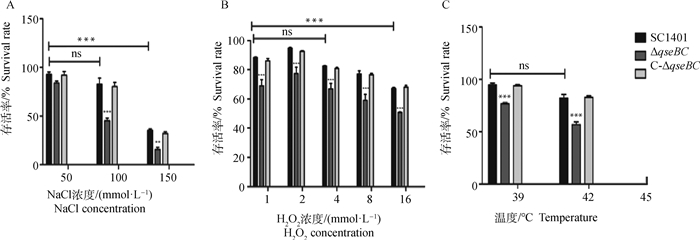
|
A. 渗透压(50、100和150 mmol·L-1 NaCl);B. 氧化应激(1、2、4、8和16 mmol·L-1 H2O2);C. 高温(39和42 ℃水浴)。存活百分比计算为在应激条件下处理的细菌数量与在无应激条件下存活的细菌数量的比值。数据表示3个独立试验的平均值,误差线表示标准偏差。星号表示使用双向方差分析的统计显著性(**. P < 0.01; ***. P < 0.001) A. Osmotic pressure (50, 100 and 150 mmol·L-1 NaCl); B. Oxidative stress (1, 2, 4, 8 and 16 mmol·L-1 H2O2); C. High temperature (Incubated in 39 and 42 ℃ water baths). Percent survival was calculated as the ratio of the number of bacteria that treated with stress conditions to the number that survived without stress conditions. Data indicate the means of three independent experiments performed in duplicate, and error bars show standard deviations. Asterisks indicate a statistical significance using a two-way ANOVA (**. P < 0.01; ***. P < 0.001) 图 3 副猪嗜血杆菌SCl401、ΔqseBC和C-ΔqseBC对渗透压、氧化应激和高温的耐受力 Fig. 3 Analysis of the stress tolerance of Glaesserella parasuis strains SC1401, ΔqseBC and C-ΔqseBC |
本研究采用生物被膜结晶紫染色法对其含量进行定量测定。如图 4所示,缺失株ΔqseBC的生物被膜经过Hucker’s结晶紫(CV)染色后着色较浅,然后用33%的乙酸溶解被结合的结晶紫,发现ΔqseBC的OD595 nm显著低于野毒株SC1401和互补株。结果表明QseBC直接或间接参与了生物膜形成的调控。
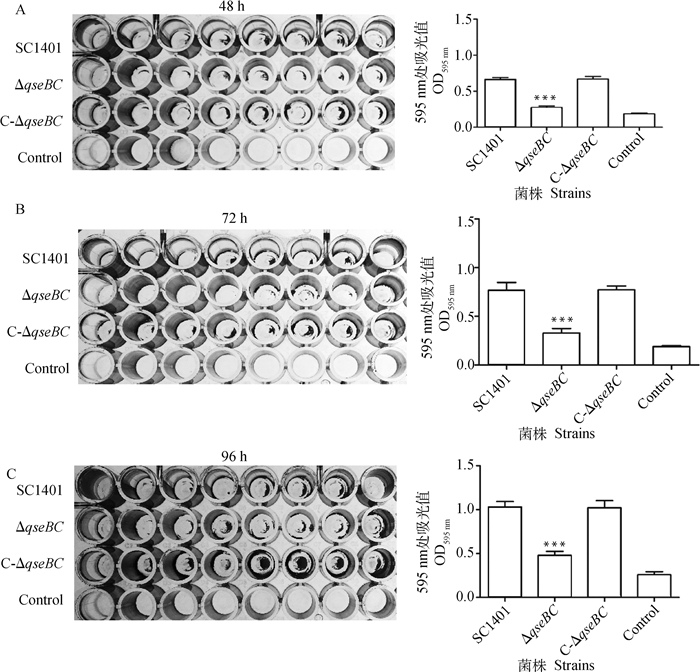
|
细菌分别培养48(A)、72(B)和96 h(C)。通过测量OD595 nm对生物膜生成情况进行定量。左图为生物膜染色结果,右图为采用33%的乙酸溶解结晶紫的脱色结果。试验独立进行3次,误差线代表 3个独立试验的标准偏差。星号表示使用双向方差分析(***. P < 0.001)的统计显著性 Biofilms were cultured for 48 (A), 72 (B) and 96 h (C), respectively. The remaining biofilms were quantitated by OD595nm measurement. The left picture shows the result of biofilm staining, and the right picture shows the decolorization result of crystal violet dissolved with 33% acetic acid. The experiments were performed three times independently in triplicates. Quantification of biofilm production. Error bars represent the standard deviations of three independent experiments. Asterisks indicate statistical significance using a two-way ANOVA (***. P < 0.001) 图 4 副猪嗜血杆菌SC1401、ΔqseBC和C-ΔqseBC在96孔微量滴定板中形成的生物被膜 Fig. 4 Detachment of biofilm colonies of G.parasuis WT, ΔqseBC and C-ΔqseBC grown in 96-well microtiter plates |
如表 3所示,野毒株SC1401对对头孢噻呋、氨苄青霉素、多黏菌素B和强力霉素的MIC分别是△qseBC相应值的16、8、4、4倍,而对恩诺沙星、庆大霉素、青霉素、林可霉素和替米考星的MIC分别是△qseBC相应值的2倍。然后,作者使用MBC-B来检查QseBC是否参与生物膜细胞的抗生素抗性(表 3),缺失株与野毒株表现出相似的抗性水平。QseBC在浮游细胞和生物膜细胞中均对同一类抗生素产生抗性,但氟苯尼考除外,△qseBC的MBC-B较SC1401降低了50%(表 3)。与新霉素的MIC-P结果相似,△qseBC对新霉素的抗性较SC1401增加100%。大多数抗性在互补菌株C-△qseBC中恢复,表明基因缺失株的抗生素敏感性不是由极性效应引起的。综上,这些结果表明QseBC参与了副猪嗜血杆菌的浮游和生物膜细胞对多种抗生素药物的耐药性。
|
|
表 3 副猪嗜血杆菌野生菌株SC1401、△qseBC缺失株和互补株对不同抗菌物质的敏感性 Table 3 Susceptibility of SC1401, △qseBC and C-△qseBC to different antimicrobials |
根据NCBI数据库中已经报道的副猪嗜血杆菌的基因组,主要有4对双组分系统,分别是CpxA/R、UhpA/Uhpb、QseB/C和ArcA/B。先前研究中,qseB (cheY) 或qseC单基因的缺失影响了副猪嗜血杆菌的部分生物学特性[10, 29]。然而,这可能是由于不同双组分系统之间的强串扰所致,并不能直接表明是qseB或qseC基因的作用。为进一步研究QseBC双组分系统与副猪嗜血杆菌致病机制的关系,本研究采用自然转化法探究从副猪嗜血杆菌SC1401基因组中同时敲除qseB和qseC基因获得ΔqseBC双基因缺失株。继而比较了缺失株和野生株的抗血清杀菌能力、压力耐受能力、生物被膜形成能力和对部分抗生素的敏感性。
细菌生物被膜的形成会增加病原菌对抗生素的抵抗力, 生物被膜形成对于引起持续感染至关重要[30-32]。QseBC双组分系统参与了伴放线菌和大肠杆菌的生物被膜形成[3, 19, 33-35],并通过修饰毒力相关的表面结构,如鞭毛、菌毛和分泌系统等,调节迟发性大肠杆菌的细胞内毒力[5]。副猪嗜血杆菌中PotD、galU、galE、ClpP等基因均参与了该菌生物被膜等形成[7, 23, 36],在先前研究中发现,副猪嗜血杆菌的ΔqseB和ΔqseC基因缺失株与野毒株相比,生物被膜形成能力明显减弱[10, 29]。在本研究中,副猪嗜血杆菌qseB和qseC双基因缺失后,形成的生物被膜明显弱于野毒株。
抗血清杀菌能力是病原菌抵抗宿主杀伤作用,引起宿主全身性感染的重要工具。Ding等[37]通过自然转换敲除ArcA/B双组分系统中的arcA基因,发现arcA基因参与副猪嗜血杆菌的血清抗性和毒力。谨瑾等[36]构建了副猪嗜血杆菌potD基因缺失株,发现potD基因的缺失导致该菌的抗血清杀菌能力显著降低。Zou等[23]发现galU和galE基因在副猪嗜血菌SC096菌株的血清抗性中起关键作用。本研究显示,与野毒株SC1401相比,ΔqseBC对猪血清杀菌活性的敏感性明显增加。
细菌侵入宿主面临巨大的生存压力。副猪嗜血杆菌通常定植于猪的上呼吸道和肺部以及其他富含活性氧的部位,对细菌的生存是巨大挑战。QseBC与肠出血性大肠杆菌和鼠伤寒沙门菌适应宿主环境有关,可通过感应肾上腺素、去甲肾上腺素和自诱导剂3来调节毒力相关基因的表达[30, 38]。为了研究QseBC是否影响副猪嗜血杆菌对外界压力的耐受能力,作者在体外使用H2O2、高浓度NaCl和高温对SC1401、ΔqseBC和C-ΔqseBC进行处理,发现ΔqseBC对高温、氧化应激和渗透压的耐受能力明显下降,表明QseBC参与了副猪嗜血杆菌对外界压力的耐受能力。
迄今为止,大多数已发表的关于细菌耐药性的研究主要集中在浮游细菌,而对生物膜细菌知之甚少。Cao等[33]研究发现Cpx双组分系统与副猪嗜血杆菌应激耐受和对大环内酯类药物的耐药性相关。Feng等[39]发现acrB基因缺失后副猪嗜血杆菌对新生霉素、红霉素、克拉霉素和阿奇霉素敏感性增强。众所周知,生物膜中的细菌比浮游细菌对抗生素的耐受力更强。然而,其耐药机制仍不完全清楚,需进一步研究。本研究表明,与亲本菌株相比,ΔqseBC基因缺失株的浮游细菌和生物膜细菌对抗生素药物的敏感性显著增加。作者推测QseBC可能参与了一些耐药相关基因的转录调控,导致ΔqseBC较野生菌株SC1401增加了药物敏感性。而具体的耐药机制有待进一步研究。
4 结论qseB/C双基因的缺失影响副猪嗜血杆菌对渗透压、氧化应激和高温的耐受性,导致该菌生物被膜形成能力、抗血清杀菌能力和对抗生素的耐药性减弱。QseBC双组分系统可能与副猪嗜血杆菌的致病性相关。
| [1] |
LI J, PENG H, XU L G, et al. Draft genome sequence of Haemophilus parasuis gx033, a serotype 4 strain isolated from the swine lower respiratory tract[J]. Genome Announc, 2013, 1(3): e00224-13. |
| [2] |
OLIVEIRA S, PIJOAN C. Haemophilus parasuis: new trends on diagnosis, epidemiology and control[J]. Vet Microbiol, 2004, 99(1): 1-12. DOI:10.1016/j.vetmic.2003.12.001 |
| [3] |
CAI X W, CHEN H C, BLACKALL P J, et al. Serological characterization of Haemophilus parasuis isolates from China[J]. Vet Microbiol, 2005, 111(3-4): 231-236. DOI:10.1016/j.vetmic.2005.07.007 |
| [4] |
ZHANG L H, LI Y, DAI K, et al. Establishment of a successive markerless mutation system in Haemophilus parasuis through natural transformation[J]. PLoS One, 2015, 10(5): e0127393. DOI:10.1371/journal.pone.0127393 |
| [5] |
ESPÍNDOLA J P, BALBINOTT N, GRESSLER L T, et al. Molecular serotyping of clinical strains of Haemophilus (Glaesserella) parasuis brings new insights regarding Glässer's disease outbreaks in Brazil[J]. PeerJ, 2019, 7: e6817. DOI:10.7717/peerj.6817 |
| [6] |
SHEN Y J, ZHOU N N, AN J H, et al. Haemophilus parasuis infection in 3D4/21 cells induces autophagy through the AMPK pathway[J]. Cell Microbiol, 2019, 21(8): e13031. |
| [7] |
HUANG J C, WANG X R, CAO Q, et al. ClpP participates in stress tolerance and negatively regulates biofilm formation in Haemophilus parasuis[J]. Vet Microbiol, 2016, 182: 141-149. DOI:10.1016/j.vetmic.2015.11.020 |
| [8] |
STOCK A M, ROBINSON V L, GOUDREAU P N. Two-component signal transduction[J]. Annu Rev Biochem, 2000, 69: 183-215. DOI:10.1146/annurev.biochem.69.1.183 |
| [9] |
APPLEBY J L, PARKINSON J S, BOURRET R B. Signal transduction via the multi-step phosphorelay: not necessarily a road less traveled[J]. Cell, 1996, 86(6): 845-848. DOI:10.1016/S0092-8674(00)80158-0 |
| [10] |
HE L Q, DAI K, WEN X T, et al. QseC mediates osmotic stress resistance and biofilm formation in Haemophilus parasuis[J]. Front Microbiol, 2018, 9: 212. DOI:10.3389/fmicb.2018.00212 |
| [11] |
KOSTAKIOTI M, HADJIFRANGISKOU M, PINKNER J S, et al. QseC-mediated dephosphorylation of QseB is required for expression of genes associated with virulence in uropathogenic Escherichia coli[J]. Mol Microbiol, 2009, 73(6): 1020-1031. DOI:10.1111/j.1365-2958.2009.06826.x |
| [12] |
LIU J L, HU L L, XU Z F, et al. Actinobacillus pleuropneumoniae two-component system QseB/QseC regulates the transcription of PilM, an important determinant of bacterial adherence and virulence[J]. Vet Microbiol, 2015, 177(1-2): 184-192. DOI:10.1016/j.vetmic.2015.02.033 |
| [13] |
LUO Z, WANG M, DU H, et al. The putative sensor kinase QseC of Salmonella enterica serovar Typhi can promote invasion in the presence of glucose[J]. Food Res Int, 2012, 45(2): 1004-1010. DOI:10.1016/j.foodres.2011.01.019 |
| [14] |
VNAL C M, SINGH B, FLEURY C, et al. QseC controls biofilm formation of non-typeable Haemophilus influenzae in addition to an AI-2-dependent mechanism[J]. Int J Med Microbiol, 2012, 302(6): 261-269. DOI:10.1016/j.ijmm.2012.07.013 |
| [15] |
XU J, FU S L, LIU M L, et al. The two-component system NisK/NisR contributes to the virulence of Streptococcus suis serotype 2[J]. Microbiol Res, 2014, 169(7-8): 541-546. DOI:10.1016/j.micres.2013.11.002 |
| [16] |
WEIGEL W A, DEMUTH D R. QseBC, a two-component bacterial adrenergic receptor and global regulator of virulence in Enterobacteriaceae and Pasteurellaceae[J]. Mol Oral Microbiol, 2016, 31(5): 379-397. DOI:10.1111/omi.12138 |
| [17] |
BEARSON B L, BEARSON S M D. The role of the QseC quorum-sensing sensor kinase in colonization and norepinephrine-enhanced motility of Salmonella enterica serovar Typhimurium[J]. Microb Pathog, 2008, 44(4): 271-278. DOI:10.1016/j.micpath.2007.10.001 |
| [18] |
SPERANDIO V, TORRES A G, KAPER J B. Quorum sensing Escherichia coli regulators B and C (QseBC): a novel two-component regulatory system involved in the regulation of flagella and motility by quorum sensing in E. coli[J]. Mol Microbiol, 2002, 43(3): 809-821. DOI:10.1046/j.1365-2958.2002.02803.x |
| [19] |
CLARKE M B, HUGHES D T, ZHU C R, et al. The QseC sensor kinase: a bacterial adrenergic receptor[J]. Proc Natl Acad Sci U S A, 2006, 103(27): 10420-10425. DOI:10.1073/pnas.0604343103 |
| [20] |
HUGHES D T, CLARKE M B, YAMAMOTO K, et al. The QseC adrenergic signaling cascade in Enterohemorrhagic E. coli (EHEC)[J]. PLoS Pathog, 2009, 5(8): e1000553. DOI:10.1371/journal.ppat.1000553 |
| [21] |
JI Y, LI W L, ZHANG Y, et al. QseB mediates biofilm formation and invasion in Salmonella enterica serovar Typhi[J]. Microb Pathog, 2017, 104: 6-11. DOI:10.1016/j.micpath.2017.01.010 |
| [22] |
何绿琴. 副猪嗜血杆菌双组份系统QseB/QseC调控功能研究[D]. 成都: 四川农业大学, 2018. HE L Q. Study on the regulatory function of two-component system QseB/QseC of Haemophilus parasuis[D]. Chengdu: Sichuan Agricultural University, 2018. (in Chinese) |
| [23] |
ZOU Y, FENG S X, XU C G, et al. The role of galU and galE of Haemophilus parasuis SC096 in serum resistance and biofilm formation[J]. Vet Microbiol, 2013, 162(1): 278-284. DOI:10.1016/j.vetmic.2012.08.006 |
| [24] |
BLANCHETTE-CAIN K, HINOJOSA C A, AKULA SURESH BABU R, et al. Streptococcus pneumoniae biofilm formation is strain dependent, multifactorial, and associated with reduced invasiveness and immunoreactivity during colonization[J]. mBio, 2013, 15;4(4): e00745-13. DOI:10.1128/mBio.00745-13 |
| [25] |
MORIYAMA S, HOTOMI M, SHIMADA J, et al. Formation of biofilm by Haemophilus influenzae isolated from pediatric intractable otitis media[J]. Auris Nasus Larynx, 2009, 36(5): 525-531. DOI:10.1016/j.anl.2008.10.010 |
| [26] |
杨开杰, 冯赛祥, 周琪, 等. 副猪嗜血杆菌qseC基因缺失突变对生物被膜形成的影响[J]. 畜牧兽医学报, 2017, 48(8): 1499-1504. YANG K J, FENG S X, ZHOU Q, et al. Effect of qseC gene mutation of Haemophilus parasuis on biofilm formation[J]. Acta Veterinaria et Zootechnica Sinica, 2017, 48(8): 1499-1504. (in Chinese) |
| [27] |
LI Y, CAO S J, ZHANG L H, et al. A TolC-like protein of Actinobacillus pleuropneumoniae is involved in antibiotic resistance and biofilm formation[J]. Front Microbiol, 2016, 7: 1618. |
| [28] |
ZHANG L, MAH T F. Involvement of a novel efflux system in biofilm-specific resistance to antibiotics[J]. J Bacteriol, 2008, 190(13): 4447-4452. DOI:10.1128/JB.01655-07 |
| [29] |
HE L Q, WEN X T, YAN X F, et al. Effect of cheY deletion on growth and colonization in a Haemophilus parasuis serovar 13 clinical strain EP3[J]. Gene, 2016, 577(1): 96-100. DOI:10.1016/j.gene.2015.11.046 |
| [30] |
DONLAN R M, COSTERTON J W. Biofilms: survival mechanisms of clinically relevant microorganisms[J]. Clin Microbiol Rev, 2002, 15(2): 167-193. DOI:10.1128/CMR.15.2.167-193.2002 |
| [31] |
STEWART P S, COSTERTON J W. Antibiotic resistance of bacteria in biofilms[J]. Lancet, 2001, 358(9276): 135-138. DOI:10.1016/S0140-6736(01)05321-1 |
| [32] |
赵良友, 高雪丽, 刘超男, 等. 副猪嗜血杆菌vacJ基因缺失菌株构建及其生物学特性分析[J]. 华北农学报, 2017, 32(5): 19-24. ZHAO L Y, GAO X L, LIU C N, et al. Construction and biological characterization of vacJ gene deletion strain of Haemophilus parasuis[J]. Acta Agriculturae Boreali-Sinica, 2017, 32(5): 19-24. (in Chinese) |
| [33] |
CAO Q, FENG F F, WANG H, et al. Haemophilus parasuis CpxRA two-component system confers bacterial tolerance to environmental stresses and macrolide resistance[J]. Microbiol Res, 2018, 206: 177-185. DOI:10.1016/j.micres.2017.10.010 |
| [34] |
CERDÀ-CUÉLLAR M, ARAGON V. Serum-resistance in Haemophilus parasuis is associated with systemic disease in swine[J]. Vet J, 2008, 175(3): 384-389. DOI:10.1016/j.tvjl.2007.01.016 |
| [35] |
DAI K, HE L Q, CHANG Y F, et al. Basic characterization of natural transformation in a highly transformable Haemophilus parasuis strain SC1401[J]. Front Cell Infect Microbiol, 2018, 8: 32. DOI:10.3389/fcimb.2018.00032 |
| [36] |
谨瑾, 张禄滑, 文心田, 等. 副猪嗜血杆菌potD基因缺失株的构建及其部分生物学特性[J]. 畜牧兽医学报, 2016, 47(11): 2274-2279. JIN J, ZHANG L H, WEN X T, et al. Construction and characterization of a Haemophilus parasuis potD mutant strain[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(11): 2274-2279. DOI:10.11843/j.issn.0366-6964.2016.11.015 (in Chinese) |
| [37] |
DING L Q, WEN X T, HE L Q, et al. The arcA gene contributes to the serum resistance and virulence of Haemophilus parasuis serovar 13 clinical strain EP3[J]. Vet Microbiol, 2016, 196: 67-71. DOI:10.1016/j.vetmic.2016.10.011 |
| [38] |
DAI K, YANG Z, CHANG Y F, et al. Construction of targeted and integrative promoter-reporter plasmids pDK-K and pDK-G to measure gene expression activity in Haemophilus parasuis[J]. Microb Pathog, 2019, 134: 103565. DOI:10.1016/j.micpath.2019.103565 |
| [39] |
FENG S X, XU L N, XU C G, et al. Role of acrAB in antibiotic resistance of Haemophilus parasuis serovar 4[J]. Vet J, 2014, 202(1): 191-194. DOI:10.1016/j.tvjl.2014.05.045 |
(编辑 白永平)



