2. 江西正邦养殖有限公司, 南昌 330096;
3. 中国农业科学院北京畜牧兽医研究所, 北京 100193
2. Jiangxi Zhengbang Breeding Co. Ltd., Nanchang 330096, China;
3. Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
虽然我国畜禽品种资源非常丰富,但本土种质资源的开发利用率一直偏低。因此,开展遗传资源基因组水平上的开发和利用是现代畜禽遗传改良的重要方向之一。分子标记的相关研究持续发展,最初的研究主要集中于限制性片段长度多态性(restriction fragment length polymorphism,RFLP)和短串联重复序列(short tandem repeat,STR)两种遗传标记。直到1996年,Lander[1]在科学杂志上正式提及到人类基因组中的单核苷酸多态性(single nucleotide polymorphism,SNP),使得基因组学的研究发展到新的水平,SNP也被公认为是第三代遗传标记。SNP分型技术历经了从低通量分型的凝胶电泳到目前应用广泛的高通量分型的发展过程。测序法是获得SNP分型最直接的方法,Sanger测序是最早应用的SNP分型检测技术,也是目前DNA测序的金标准。1991年Affymetrix合成了首张寡核苷酸的基因芯片,基因芯片成为SNP分型检测的主要方法。到目前为止,SNP标记的分型检测已经发展到高通量的第二代测序技术和第三代测序技术。随着基因芯片、高通量测序和组学大数据技术的突飞猛进,SNP标记的分型检测成本已大幅降低,从而带动了畜禽育种由传统的BLUP法向全基因组选择育种发展的浪潮。全基因组SNP分型技术的出现是基因组选择技术从理论研究到实际应用的关键。当前,基因组选择技术为畜禽育种带来了革命性的变化,不仅使育种效率大幅度提高,还能实现育种企业的早期选择,提升企业的降本增效能力。所以该项技术已经成为国际畜禽育种领域研究和竞争的热点[2]。
准确高效的SNP分型是畜禽基因组研究和育种应用的关键,SNP作为第三代分子标记,具有数量多、分布广、遗传稳定等特点,在全基因组关联分析、地方资源遗传背景分析、基因组选择信号等方向广泛应用。全基因组SNP分型技术以SNP芯片技术和基于二代测序的SNP分型技术为主。本文概述了全基因组SNP分型技术的原理、分型技术在全基因组关联分析、选择信号和畜禽遗传资源背景分析等基础研究和畜禽遗传育种中的应用,以期为畜禽基因组的研究和育种应用提供借鉴和参考。
1 全基因组SNP分型技术 1.1 基因芯片技术基因芯片(gene chip), 又称为DNA芯片、生物芯片或DNA微阵列等,是将大量的探针分子(一般是指DNA片段)有规律地排列和固化于固相支持物上,构成一个二维DNA探针阵列。根据研究方向不同,基因芯片可以分为SNP基因芯片、比较基因组杂交基因芯片、表达谱基因芯片、DNA甲基化基因芯片和染色质免疫共沉淀芯片等[3]。在畜禽育种领域,50K以上的中高密度SNP基因芯片应用较为广泛,主要由Illumina公司的Infinium芯片技术平台和Thermo Fisher公司的Axiom芯片技术平台定制生产。这两大芯片平台虽然在分型原理上相同,都是利用红、绿荧光蛋白及激光发光基团来进行SNP的分型,但是在芯片的设计方面有很大的区别。Illumina芯片是光纤微珠技术,其优点是探针长度50 bp,针对单个SNP有15~30次重复设计,微珠能100%质量控制,应用上灵活性较高;Thermo Fisher芯片是原位光刻合成技术,其优点是基因芯片几乎无批次效应,能够兼容20 bp以上的SNP,且设计位点转化率较高。
随着我国企业自主育种意识的加强,近年国内出现了一批以我国育种需求为设计理念的国产基因芯片,大大降低了企业对于国外芯片产品的依赖。基因分型成本的降低也激发了育种企业应用高密度SNP芯片开展基因组育种的热情。
1.2 基于二代测序的SNP分型技术随着测序技术的迅猛发展和测序成本的超摩尔速度降低,基于二代测序的SNP分型技术在畜禽分子育种领域应用与日俱增。从测序原理角度,通过测序不仅可以直接获得待测群体的真实变异数据、不受物种是否具有参考基因组的限制,而且能够获得覆盖低、中、高密度甚至全基因组范围内的所有变异信息,同时能够通过提高测序的深度达到检测稀有变异信息的目的。目前,基于二代测序技术开发的标记检测技术主要包括:全基因组重测序(whole genome resequencing,WGS)、简化基因组测序(reduced-representation genome sequencing,RRGS)、低深度重测序(low-depth resequencing)和靶向捕获测序(target capture sequencing, TCS)等。
1.2.1 全基因组重测序 全基因组重测序(whole genome resequencing,WGS)是通过对基因组序列己知的物种进行不同个体的基因组测序,在个体或群体水平上进行差异化分析的测序方法。其基本流程是:首先通过基因组片段化、末端修复、连接测序接头和扩增富集获得全基因组的测序文库;然后利用基因测序平台进行全基因组文库的扫描测序,经全基因组重测序的序列比对,可以得到大量的单核苷酸多态性位点(single nucleotide polymorphism, SNP)、插入缺失(insertion-deletion, InDel)、拷贝数变异(copy number variations, CNV)和结构变异(structure variation, SV)等变异信息,进一步通过生物信息学分析可以找到与疾病、经济性状及功能相关的遗传标记。随着测序成本的大幅度降低以及测序效率的数量级提升,再加上己知基因组序列的物种增多,全基因组重测序已经成为动植物遗传差异研究、功能基因挖掘等最可靠且常用的方法。Tao等[4]利用WGS对云上黑山羊品种的两个亚群(多胎组和单胎组)进行重测序,通过GWAS分析发现了12个包含具有最小P值的OSBPL8等候选基因。Banos等[5]利用重测序技术对两种不同的埃塞俄比亚本土鸡生态型进行了包括估计遗传参数、GWAS分析、估算基因组育种值(GEBV)、基因组预测等联合分析,用于研究本土鸡与重要健康和生产力特征相关的基因组结构,并探索进行跨生态型基因组选择的可行性。
1.2.2 简化基因组测序 简化基因组测序(reduced-representation genome sequencing, RRGS)是利用限制性内切酶将基因组DNA进行片段化,对筛选到的特定酶切片段进行高通量测序以此获得大量遗传标记的测序策略。酶切位点可以出现在基因组的任意位置,由于同一物种的不同个体或者近缘物种间的酶切位点位置相对保守,共享相同的酶切位点,为简化测序提供了先决条件[6]。目前应用较多的主要是GBS (genotyping by sequencing)、dd-RAD (double digest restriction associated sequencing, ddRAD-seq)[7]和RAD-seq (restriction associated DNA sequencing) [8],其主要区别在于在测序接头连接后是否进行片段大小的筛选和PCR富集后是否进行片段选择。与全基因组重测序相比,简化基因组测序因只对基因组上很少的一部分区域进行测序,极大的简化了基因组,因此,该技术不仅能够降低测序成本,而且能降低测序数据量和缩短生物信息分析的周期。
GBS分型技术利用限制性内切酶对基因组DNA进行片段化,通过筛选一定比例的酶切片段进行高深度测序来获得覆盖全基因组的高密度遗传标记。一方面GBS分型技术因文库构建操作流程简单,具有高效率、低成本等优势;另一方面,该技术所获得的遗传标记大多是待测群体中多态性较高的SNP位点,使得GBS分型技术比较适合在我国地方品种或高度纯化的品系中进行分型并获得大量的遗传标记信息[9]。目前,简化基因组测序分型技术已经广泛应用于牛、猪、羊、鸡、鸭等畜禽物种研究中[10],其中GBS分型技术已经被广东温氏公司应用于杜洛克猪群体的基因组选择育种研究,并取得了较为显著的遗传进展[11]。
1.2.3 低深度重测序 全基因组低深度重测序是继简化基因组测序技术之后的新一代低成本标记检测的方法,该技术首先对群体中所有的个体进行全基因组低深度重测序和变异检测,然后根据变异位点间的连锁不平衡对缺失基因型进行填充,最终获得大规模样本的全基因组水平的高密度遗传标记[12]。由于测序深度与基因组变异信息的覆盖度高度相关,提高测序深度不仅能够降低假阳性的比例,而且在稀有变异检测上也有着显著的优势。但是在畜禽育种领域,高深度的重测序需要较高的测序成本,限制了该技术在企业育种上的应用。为了控制检测成本,可以通过基因组文库构建方法的改进来降低成本,如基于酶切法打断建库(包括非特异性外切酶和Tn5转座酶等),还可以尝试降低测序深度,开发低深度重测序方法,结合基因型填充流程实现全基因组水平的变异位点检测。Nicod等[13]对1 887只远缘杂交系的小鼠个体进行了0.15×的超低深度重测序后,通过GWAS定位到156个与92个性状相关的独立的遗传标记,该项报道为畜禽基因组研究中实施大规模群体的低深度SNP分型技术提供了新的思路。Yang等[12]用低深度重测序(平均测序深度0.73×)方法,对2 885头杜洛克公猪进行4个经济性状全基因组关联分析,关联到2个QTLs可能与乳头数量和背部脂肪厚度特征有关。
1.2.4 靶向捕获测序 靶向捕获测序是利用二代测序技术对基因组上目标区域进行捕获后测序,目前主要有两种技术体系,一种是基于多重PCR技术,一种是基于探针杂交。这两种技术体系均可以实现对基因组上的非高度重复区域进行定向捕获,可以同时检测各种变异类型,如SSR、SNP和InDel等。基于多重PCR的捕获测序流程是:第一轮PCR对目标位点进行扩增,第二轮PCR过程中引入测序接头和Barcode,获得可以上机测序的文库,然后通过高通量测序仪进行测序。与常规PCR不同在于,多重PCR捕获技术能够在一管反应中实现上千位点的扩增。基于探针杂交的捕获测序原理是基于目标区域序列与液相探针的互补结合,对目标位点捕获并测序,其主要流程为:首先构建全基因组文库,然后利用生物素修饰的探针进行捕获,利用链霉亲和素的磁珠对探针结合的分子进行吸附,最后对捕获序列进行扩增富集得到测序文库(图 1)。与全基因组测序相比,靶向捕获测序缩小了测序的区域,降低了样品的测序成本。这项检测方法中,探针合成的费用是主要成本,检测步骤较多,数据稳定性依赖于测序深度以及对目标区域的捕获效率等因素。考虑成本和数据质量等因素,靶向捕获测序技术目前主要用在需求量不大、没有商业化SNP基因芯片的物种上,集中于中低密度Panel的定制开发。Lippold等[14]利用多重液相捕获方法研究了来自44个品种的59匹家养马和1匹普氏野马(Equus przewalski)的整个线粒体基因组,并发现家马中有473个可变位置,提供了一个很好解析的系统发育树。Newman等[15]使用全外显子组捕获测序,研究了安格斯牛EPAS1氧降解域中EPAS1 (HIF2a) 双变体与高海拔肺动脉高压(HAPH)的高度关联。
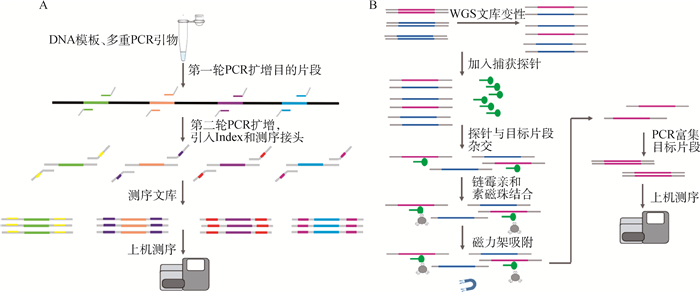
|
A.基于多重PCR的靶向捕获测序技术原理;B.基于探针杂交的靶向捕获测序技术原理 A. Principle of the multiplex PCR-based target capture sequencing technology; B. Principle of the probe hybridization-based target capture sequencing technology 图 1 基于多重PCR和探针杂交的靶向捕获测序技术原理图 Fig. 1 Schematic diagram of target capture sequencing technology based on multiplex PCR and probe hybridization |
全基因组关联分析(genome wide association analysis, GWAS)是基于全基因组上大量遗传标记的多态性,对每个多态性标记(即基因型)与表型性状进行关联分析,根据统计学阈值或P值筛选与目标性状关联的分子标记的分析策略,也是在畜禽研究中用来挖掘与经济性状相关的分子标记的最常用的方法[16]。目前,GWAS已成为畜禽重要性状基因定位的常用方法,据Animal genome网站统计,截至2022年4月,QTLdb上已发布并公开了192 925个牛性状相关的QTLs、16 637个鸡性状相关的QTLs、35 384个猪性状相关的QTLs、4 207个绵羊性状相关的QTLs。
基因芯片作为第一个用于全基因组关联分析研究的高通量技术一直被广泛应用至今[17]。由于过低的SNP密度会影响性状关联标记的挖掘效率,所以目前进行畜禽重要性状关键基因定位的基因芯片密度都在50K以上。在猪的体重性状研究方面,Ji等[18]利用Illumina Procine SNP60K芯片在611个和79个与白杜洛克和二花脸猪杂交二代群体中筛选出与210 d体尺(体高、体长、胸围、胸深、胸宽、管围、腹围和臀围)和体重关联的SNPs标记,并鉴定出7个新的QTLs和5个候选基因。在猪的骨性状研究方面,邱恒清[19]利用Illumina Procine SNP60K芯片对300日龄巴马香猪检出的CNV进行GWAS,挖掘到18个位于2、5和7号染色体上并显著影响骨骼长度的拷贝数变异区域。在鸡体重研究方面,Lien等[20]使用60K Illumina SNPChip对844个个体的0、4、8、12和16周龄的体重、8周龄的小腿长度、16周龄的梳区大小及11周龄(初次免疫后第7和14天)的红细胞水平进行GWAS分析,鉴定到与178个SNPs显著关联的47个QTLs和714个效应位点。在牛的胸高和臀高研究方面,Zhang等[21]使用Bovine SNP50 v2 BeadChip对中国荷斯坦牛的4个生长阶段(6、12、18、24月龄)的胸围和臀高进行GWAS分析发现了66个候选基因在16个信号通路和互作网络中发挥重要的生物学功能。在山羊体型研究方面,Rahmatalla等[22]利用Goat SNP52K BeadChip对苏丹4个山羊品种的14个体型性状进行GWAS分析发现,位于2号染色体上的CNTNAP5基因与胸宽显著关联,位于3号染色体上的SNP位点56482-scaffold89-467312与体长显著关联。另外,Seroussi等[23]使用Illumina Ovine SNP50 BeadChip进行GWAS分析挖掘到影响绵羊绒细度的候选基因AKT1和ALX4。
全基因组重测序技术的发展、各物种基因组序列的测序完成,为从基因组水平上研究目标性状提供了便利。张易[24]采用case-control设计,对F2群体308只鸭子羽色性状进行全基因组关联分析,结果显示共关联显著水平相关的SNP位点8 423个,通过参考基因组序列比对和GenBank功能注释,结合KEGG富集、GO分析和CNV关联,预测MITF基因是鸭白羽性状显著相关基因。简化基因组测序相比于全基因组重测序,能降低测序成本和缩短数据分析所需的时间。谈成[25]利用GBS测序对3 757头杜洛克公猪进行测序分型和GWAS分析,鉴定到大量与一种或多种性状相关联的显著性SNPs。靶向捕获测序也是利用二代测序技术进行分析的最有效方法之一,相比全基因组测序法,该方法大幅缩小了目标区域的捕获范围,在保证检测到目标区段所有变异的前提下,能够降低样品测序成本。乔贤[26]使用绒山羊66K靶向捕获panel,对432个个体羊绒进行捕获测序获得目标SNP位点分型,对羊绒细度性状进行了关联分析。
基因组学技术的快速发展推动了新一代测序技术的迅猛发展,测序成本也随之呈现超摩尔速度降低,这为进一步开发和应用动物高密度芯片的研究奠定了基础。目前基于50K以上标记密度为主的基因芯片在牛、猪、绵羊、山羊、鸡等物种上得到了广泛应用,推动了全基因组关联分析在畜禽遗传育种上的快速发展[27]。随着各物种基因组测序的完成和测序成本的降低,全基因组重测序在挖掘和经济性状相关的分子标记上的应用也越来越多,相对于基因芯片还可以挖掘到新的或者稀有的SNP标记。因重测序一般要求测序深度大于10×,对于较大基因组的物种来说测序费用较基因芯片高,所以如果有商业化的基因芯片建议直接应用;如果是较大的群体检测,可以选取部分有代表性的个体进行重测序,利用重测序的数据进行高密度基因芯片的开发,从而实现大群体的检测和标记的挖掘。
2.2 选择信号分析选择信号(selection signature)是指在生物群体进化过程中,由于人工选择或自然选择的作用使生物群体的表型特征发生变化并且在基因组上留下大量的痕迹,一般表现为基因型纯合或者某些位点或DNA片段的多态性降低,这些信号通常与动物的选育方向以及驯化适应机制紧密相关。因此,对选择信号进行检测有助于挖掘与动物经济性状相关的基因,了解性状形成的潜在的遗传学基础,对于畜禽遗传改良具有重要意义[28]。
早期对于畜禽选择信号的研究都是通过基因芯片实现的,如绵羊和山羊的基因组芯片分别于Kijas等[29]和Tosser-klopp[30]等的研究中问世。刘恩民[31]应用Illumina Goat SNP 50K Beadchip对我国16个山羊品种(其中2个野生山羊群体)进行了遗传多样性、群体结构和选择信号分析,发现在1、8和14号染色体上的强受选择区域存在一些未知功能的基因,包括PLGRKT、LOC106502473、LOC1021856、XXYLT1等。Jin等[32]利用Illumina Caprine 50K BeadChip对53只内蒙古绒山羊、辽宁绒山羊和黄淮山羊进行基因分型,确定了一些积极选择的SNPs。Edea等[33]利用Porcine SNP70K BeadChip对488头杜洛克猪和155头杜洛克×韩国本土猪(DKNP) 进行基因分型鉴定到了与生长/身高、胴体和肉质相关的定向选择基因。李景[34]使用高密度SNP基因芯片对来自5个藏区和28个低原地区群体的共593头猪进行基因分型,同时整合了来自Dryad网站的6个中西方猪种共85头猪的60K基因芯片分型数据,发现5个藏猪群体在进化和地理位置上有明显的分化。Zhang等[35]使用鸡60K SNP芯片在两个经过了11代腹部脂肪含量选择的品系中进行多种选择信号分析,确定了多个受选择的基因区域。
全基因组重测序技术由于其高通量的输出基因组序列的特点,速度和准确性均较高,已经广泛应用到选择信号的研究中。陈建兴等[36]对4个驴品种60个样本进行全基因组重测序和群体遗传分化系数(Fst)、核苷酸多样性比值(πratio)分析找到了39个落入选择信号区域的候选基因,主要在免疫、生殖、细胞作用等通路中发挥重要的作用,说明山东小毛驴在免疫力和生殖能力等性状上经历了人工选择。Li等[37]对5个地方猪种(包括白眉猪、金华猪、荣昌猪、梅山猪、藏猪) 与西方5个家猪的测序数据进行选择信号分析,发现中西方人工选择方向的不同导致地方猪与西方猪存在较大的遗传差异,中国地方猪的遗传变异较高。金川牦牛作为独立于其他牦牛的一个分支,其驯化程度和选择强度均大于其他牦牛群体,Lan等[38]基于重测序对四川省金川县牦牛进行全基因组分析发现,与其他牦牛品种相比,金川牦牛有339个基因(包括与节律、神经系统、突触发育等相关基因)受到显著的正向选择。Guo等[39]分析了不同表型山羊的选择特征,基因扫描发现藏山羊有4个起源,品种间遗传分化较高,毛色上的选择区域受体较多。Zhang等[40]对藏鸡和低海拔鸡研究发现,藏鸡中与适应高海拔生活相关的差异表达基因参与了心肺系统发育、炎症和免疫反应以及辐射的反应。
简化基因组测序,由于被测序的是基因组的一部分,在相同的测序通量和测序深度的情况下能够测得更多的个体,这也为群体遗传学中大量样本的基因分型提供了可能[41]。Liu等[42]使用RAD-seq从6个中国本土兔品种和2个进口兔品种中获得了1 006 496个SNPs标记,对具有对比毛色的两个群体进行选择信号分析,发现了以四川白兔和新西兰兔为参考群体,闽西南黑兔和万仔兔为目标群体的受选择特征基因。马士龙等[43]对麦洼牦牛3个保种群粉嘴群、全黑群和弗洛群进行GBS简化基因组测序检测到了126 122个SNPs标记,利用Fst和π法对3个保种群进行选择信号分析,发现有104个受选择基因广泛参与生殖机能、免疫系统、胚胎发育等条目以及生殖激素、内/外分泌、信号传递等通路,其中部分基因提示麦洼牦牛的繁殖、肉质、毛色性状以及应激反应得到了人工选择。夏树立等[44]利用简化基因组测序技术分析和比较天津猴鸡群体与其他鸡种群体的遗传差异,并对进化过程中受到选择的基因进行基因功能注释。结果显示,在天津猴鸡群体中检测出265 869个SNPs标记,受选择基因主要参与氨基酸生物合成、细胞内信号转导以及神经系统发育等通路或生物学过程;检测到6个与种质特性相关的受选择基因,它们与神经系统发育、激素调节和抗热应激等生理功能紧密相关,研究结果揭示了天津猴鸡的裸颈性状形成机制和抗热应激特性,为天津猴鸡的保护和种质特性评价提供重要理论依据。
早期对畜禽选择信号的研究是基于基因芯片,标记密度一般在5万个以上。随着测序技术的发展、测序成本逐步降低,基于基因组重测序、简化基因组测序等技术的选择信号分析同样可以快速准确地筛选出受选择区域和定位经济性状相关的候选基因。
2.3 畜禽遗传资源背景分析遗传背景分析可以揭示生物群体的遗传结构与进化的历史动态。群体遗传多样性是指群体内所有个体的遗传变异信息的总和,自然选择和群体间的基因交流均能影响群体遗传的多样性。近年来,全基因组基因分型技术迅猛发展,基因检测效率大幅提高,为探讨生物群体的遗传结构、追踪不同群体经历的选择提供了可能。
中高密度的基因芯片能够从全基因组范围内对地方畜禽遗传资源进行遗传结构和长纯合片段检测等分析,为地方遗传资源保护和开发提供参考。胡亮等[45]利用600K基因芯片对20个藏系绵羊品种(西藏、青海、甘肃、云南和四川地区)进行藏系绵羊间的遗传关系和遗传多样性分析发现,西藏、云南地区藏系绵羊的近交系数明显高于青海、甘肃和四川地区的藏系绵羊。戴丽荷等[46]利用Illumina CAUPorcine 50K SNP芯片检测54头淳安花猪的单核苷酸多态性(SNP),分析群体遗传多样性和群体结构,将群体划分为6个家系,为保种和选配提供了依据。屠云洁等[47]为揭示广西麻鸡2个群体里当鸡和灵山香鸡之间的遗传距离和亲缘关系,利用“京芯一号”芯片对2个群体的亲缘关系进行遗传分析,结果显示广西麻鸡2个群体个体间近交程度较低,个体间亲缘关系相对较远,为培育市场需求的优质麻鸡配套系提供参考和帮助。
基于测序的分型技术在畜禽遗传资源背景分析中的应用也越来越多。在家鸡的遗传资源鉴定中,陈彬龙[48]利用WGS技术对来自不同地理位置的78只家鸡进行群体结构分析发现,6个具有地理代表性的藏鸡群体至少存在3个明显的分支,表明藏鸡可能与其他家鸡一样是多起源的。Gebreselase[49]利用WGS对埃塞俄比亚和中国山羊群体进行群体结构和系统发育分析,将山羊品种按照其起源地分成了4个分支。张俸伟[50]对21头隆林牛、18头南丹牛和17头涠洲牛进行全基因组重测序、群体结构分析、核苷酸多样性分析、LD衰减分析、ROH计算、线粒体基因组分析,得到广西的这3个黄牛品种全基因组遗传变异非常丰富,主要为中国瘤牛起源,具有抵抗疱疹病毒基因、分子黏附相关基因(NCR3、FAT4)和6个参与补体激活的基因,具有独特的瘤牛I1a亚单倍型组;对Y染色体雄性特异区SNP分析发现中国瘤牛Y3a亚单倍型组占绝对优势。相比于全基因组重测序,简化基因组测序只对很小一部分的基因组进行测序,能降低测序成本和数据分析所需的时间。盛中华等[51]利用简化基因组测序技术构建了上海白猪(上系)分子保种数据库;通过基因组结构特性分析,将上海白猪(上系)、西方猪种和太湖猪种分成3类,证实了上海白猪(上系)经过长期的选育,已形成独特的遗传结构,具有独特的基因组结构和群体结构。兰蓉等[52]采用简化基因组测序(GBS)技术对来自云南省种羊推广中心的37只黄色波尔山羊公羊进行测序,分析了群体的遗传结构并将群体划分为11个家系,为波尔山羊黄色群体在云南黄山羊新品种培育中的合理利用提供了科学依据,也为评估山羊个体近交水平、防止近交衰退、优化选种选配方案提供了有力的技术手段。
3 全基因组SNP分型在畜禽育种中的应用 3.1 基因组选择育种概述基因组选择(genomic selection, GS)是畜禽经济性状选育改良的重要方法,利用覆盖全基因组高密度遗传标记信息计算个体基因组估计育种值(genomic estimated breeding value, GEBV)[51]。与常规基于系谱及表型信息评估育种值(estimated breeding value, EBV)方法相比,GEBV通常能获得更高的估计准确性[53-55]。由于计算个体的GEBV可以不依赖系谱和表型信息,为实现早期选育提供了可能。基因组选择的方法不仅可以提高遗传进展、缩短世代间隔、降低育种成本[56],而且对于低遗传力性状和难以测量的性状也具有明显的优势。
随着GS技术的应用与发展,已经有了丰富的算法和模型可以进行育种值估算。根据统计模型的不同,现在常用的算法和模型分为两种[57]:第一类是间接法,是基于估计等位基因效应来计算GEBV[58]常见的方法,如最小二乘法[59]、随机回归-最佳线性无偏预测[60]、贝叶斯方法[61]等;第二类是直接法,直接法模型的原理和经典的BLUP相同,只是采用SNP标记或者SNP结合系谱构建关系矩阵, 然后利用混合线性模型估计GEBV,常见的方法有GBLUP[62]、SSGBLUP[63-64]等。
进行全基因组选择时,首先需要组建参考群,参考群必须有准确的表型记录和基因型分型数据。特别需要说明的是,在SSGBLUP模型中有准确系谱和表型的个体也被视为参考群[65];因此,SSGBLUP方法是利用参考群的表型数据、系谱数据及基因分型数据计算群体的方差组分,并优化运算模型因子构成,完成参考群运算模型构建;然后通过候选群的基因分型数据和系谱数据对有基因型和无基因型数据的个体进行GEBV估计,按照综合指数权重计算综合育种值并根据排名进行选留,如图 2所示。
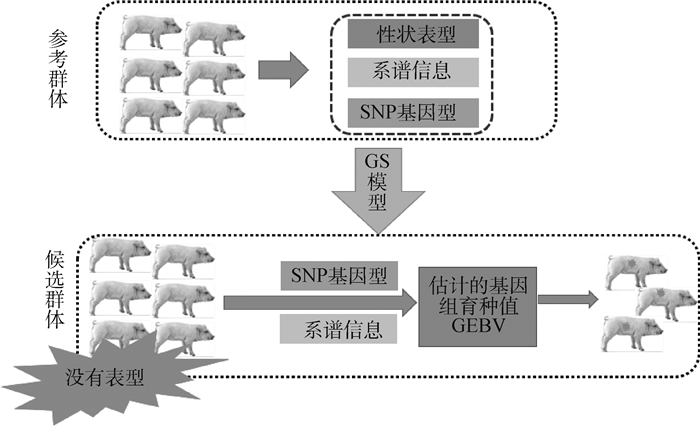
|
图 2 基因组选择流程图 Fig. 2 Flowchart of genome selection |
从2001年基因组选择的概念首次提出至今,各个国家在畜禽育种方面已经陆续使用。美国和加拿大在2009年率先向全球发布奶牛基因组选择的成果。2009—2015年基因组选择使美国奶牛育种的世代间隔大幅度缩短,公牛父亲世代间隔从原来的7年左右下降到2.5年,而公牛母亲从4年降到了2.5年。某些中高遗传力性状(乳蛋白、产奶量、乳脂量等)的年遗传进展提升50%~100%,而低遗传力性状(体细胞评分等)提升的更加显著,年遗传进展提升了3~4倍[66]。截至2017年,美国对奶牛利用基因组芯片的检测量达到200万头。英国猪育种PIC公司从2010年起每年检测量已达10万头。目前,全球主要的发达国家都在奶牛、肉牛、猪、羊、鸡等物种上全面开展了基因组选择育种,使选育进展大幅度提升,选育成本进一步降低[67]。在我国也已经初步建立了猪、鸡、鸭、肉牛、奶牛等物种的基因组选择育种技术体系,相继成立猪基因组选择北京联盟、肉鸡基因组选择育种联盟、北京联育肉牛育种科技有限公司等组织。奶牛方面,我国于2008年开始启动奶牛基因组育种(GS)研究,2012年正式将GS技术应用于荷斯坦奶牛的遗传评估中,主要评估了产奶量、体细胞计数、体型评分等14个性状,其中产奶性状基因组预测准确性较常规BLUP方法提升了0.13~0.30,基因组预测准确性为0.59~0.76。荷斯坦奶牛基因组选择技术体系的建立,实现了大规模的产业化应用,同时使我国奶牛育种技术跻身于国际先进行列。
3.2 畜禽选育产品开发及育种应用畜禽育种产品的开发需要大量遗传变异信息。全基因组重测序技术可以检测出个体或群体全基因组范围的变异信息,从而可以用于全基因组选择育种、目标性状的全基因组关联分析、品种间差异分析等分子标记和产品的开发。
Liu等[68]对8个中国地方鸡品种128个个体进行全基因组重测序和SNP位点挖掘,结合GWAS分析,设计出一款包含21.41 K全新位点的55K鸡育种基因芯片。以江西农业大学研究成果为基础集成了国内十余所高校及科研单位研究成果开发的“中芯一号”基因芯片,囊括了多肋、肉色、肉质和疾病等重要性状因果基因位点,该产品不仅适用于我国地方猪种,同时适用于商业化猪种的基因组检测。Qi等[69]对427个太平洋牡蛎样本(采集地点包括中国、日本、韩国、加拿大)进行全基因组重测序,设计了一款200K的牡蛎基因组育种芯片,从而方便对牡蛎(动物物种中基因组DNA变异水平最高物种之一)进行现有全基因组关联分析、精细连锁图谱和群体遗传学研究。Yáñez等[70]对来自3个群体的326个罗非鱼样本进行全基因组重测序,对分析得到的SNP位点进行筛选(包括基因型和位点质量、孟德尔错误率、非特异位点)设计了一款50K高质量SNP芯片,这些入选的SNPs位点在3个群体中表现出良好的多样性,94%~99%的位点符合哈迪-温博格平衡,76%~90%的SNPs位点MAF(minor allele frequency)大于0.05,这款芯片有助于分析罗非鱼经济相关性状、基因组选择加强育种计划以及罗非鱼养殖群体的遗传研究。乔贤[26]对我国著名地方品种内蒙古绒山羊和辽宁绒山羊73个样本进行全基因组重测序,结合国内外其他山羊品种基因组数据库,采用叠瓦式探针设计方案,设计了一款66K位点的山羊捕获测序产品,用于全基因组关联分析研究和绒毛品质的选育。
在品种鉴定产品开发方面,刘继强[71]利用全基因组重测序技术开发了滩羊基因身份证检测产品,该项目挑选性状差异显著的滩羊、乌珠穆沁羊、呼伦贝尔羊、蒙古羊、宁夏蒙古羊,进行全基因组重测序分析,构建二者单碱基多态性SNP和结构变异SV等遗传变异库,采用机器学习的方法,通过对训练集的学习,得到优化的位点组合模型,通过盲测及模型优化得到最终检测位点集。范欢欢等[72]对249只梅花鹿、206只马鹿、一代杂交鹿(F1)23只、二代杂交鹿(F2)20只和三代杂交鹿(F3)20只共518个个体进行全基因组重测序,以染色体级别梅花鹿基因组为参考序列,对所有个体进行变异检测,结合检测个体的表型信息将梅花鹿和马鹿参考进行群体划分,计算两个参考群体SNP的遗传分化指数(genetic differentiation index, Fst),根据定制算法和严格的位点筛选原则,最终选取1 000个梅花鹿特异性SNPs位点用于1K梅花鹿基因芯片的开发(鹿芯壹号),该芯片可以准确对待测样本(即梅花鹿纯度)进行鉴别(表 1)。
|
|
表 1 畜禽分子育种产品开发案例 Table 1 Development cases of livestock and poultry molecular breeding products |
在猪和鸡的GS应用过程中,系谱的准确性往往对结果有较大的影响。但是根据现有的研究及实际应用的经验,国内大多数企业的系谱均有10%~20%的错误[73]。这些错误一般是由引种时系谱混乱、现场种畜耳标脱落、人工授精操作不规范、人为记录失误等原因造成的。近年来随着一些企业GS的开展,企业在进行基因分型样本的采集时,个体ID的对应关系及记录也出现了一定概率的错误,而且这种错误极难通过育种企业内部筛查进行修正。因此,基于SNP数据的系谱纠偏对于育种企业来说非常有意义。基于孟德尔遗传定律,即每个等位基因位点均以孟德尔遗传方式由亲本传递给后代[74]。在疑似亲本和后代个体所构成的待检测亲子对间,对每一个双等位基因的遗传位点进行孟德尔错误判定,理论上少量合适的标记就能得到准确率为99.99%以上的判断[75]。郭立平[76]从SNP标记芯片(BovineSNP50 Genotyping Bead Chip)中筛选了50个多态性高的SNPs标记位点作为西门塔尔牛亲子鉴定的SNP标记组合,用SNP标记组合对938头西门塔尔牛进行亲子推断,80%的置信度水平385头找到最似父亲,其中268头置信度超过95%,117头置信度介于95%~80%。张哲等[77]提出了一种基于全基因组高密度SNP标记的亲子鉴定新方法,命名为EasyPC,并利用191头杜洛克猪的全基因组SNP数据和2 180头中国荷斯坦牛的全基因组SNP芯片数据分别使用EasyPC和Cervus软件进行了对比,结果显示猪的系谱错误率为6%,牛的系谱错误率为20%,但EasyPC运行效率较Cervus更高。因此该方法可以快速、准确地判别系谱的正确性,同时还能够矫正错误的系谱。
4 SNP分型技术的选择基因组检测技术不断发展,全基因组范围内的SNP分型技术也日渐丰富。不同的分型技术适用于不同的应用场景,科研和育种工作者可以根据不同检测技术的优势及自身的研究需求灵活选择。
基因芯片具有检测周期短、数据稳定和分析流程容易等特点,在畜禽基因组研究和育种上广泛应用。根据应用方向的不同,基因芯片可以划分为以科学研究为主的基因芯片和以产业应用为主的基因芯片。科学研究用基因芯片的遗传标记数量占物种全基因组的比例较大,主要用于如QTL、全基因组关联分析、基因定位和群体进化分析等基础研究中。产业应用的基因芯片遗传标记数量占物种全基因组的比例较小,主要用于育种公司大量样本的遗传育种值和重要性状分布的评估[78]。全基因组重测序技术能够捕捉到个体全基因组上所有的变异信息,但对于基因组较大的物种基因分型成本高,目前主要应用于科学研究中,在产业化育种上应用较少(表 2)。低深度重测序技术能够通过降低测序深度来降低个体的检测成本,但需要大量样本的检测和高计算量为基础适用于大型育种群体研究。靶向捕获测序技术针对特定的目标区域设计探针,进行目标区域捕获和测序,能够提升目标区域的测序深度,相对于重测序具有较高的测序效率,未来在畜禽基因组研究和基因组育种中具有一定的潜力。
|
|
表 2 不同全基因组SNP分型技术在畜禽基因组研究中的比较 Table 2 Comparison of different genome-wide SNP typing techniques in livestock and poultry genome research |
在SNP分型技术选择上,如果研究的对象有相应的芯片产品,由于芯片检测简单快速、数据更易存储和处理,建议优先使用基因芯片产品。如果没有相应的芯片产品,则可以选择基于测序技术的相关分型策略,但测序后数据处理、存储以及对分析人员的要求相对较高。当然,若待检测的群体很大,为了兼顾两种技术的优势,也可以选取群体中有代表性的少量个体进行测序分型,根据测序的结果获得具有一定代表性的标记集合,进一步定制基因芯片或者靶向捕获测序芯片完成剩余个体的检测和基因分型。总之,在实际育种应用中,育种工作者应该根据具体的情况综合考虑,包括检测周期、数据情况和成本投入等因素,选择最适合的全基因组SNP分型检测技术。
5 展望随着基因芯片和重测序成本的不断降低,畜禽基因组研究中SNP分型技术的选择更加多元化。一方面利用基因芯片进行SNP分型具有分型准确性高、分析周期快的优势,在科学研究和育种中仍具有较大的应用空间;另一方面以测序为基础的SNP分析技术能够在全基因组范围捕获到更高密度的变异信息,为基础研究提供了强大数据支持。但是基于二代测序的SNP分型技术在产业育种应用上仍然存在分型准确性和时效性问题,针对分型准确性问题可以通过优化测序策略和建立严格的数据质控参数提高;针对时效性问题,可以借助于优化文库构建流程、采用测序效率更高的测序仪器和建立标准化、智能化的数据分析流程来解决。随着芯片和测序技术的发展,更低成本、更高准确性的SNP分型策略势必能够加快畜禽基因组研究和遗传育种的发展,为我国的畜禽种业振兴提供技术支撑。
| [1] |
LANDER E S. The new genomics: global views of biology[J]. Science, 1996, 274(5287): 536-539. DOI:10.1126/science.274.5287.536 |
| [2] |
林敏. 农业生物育种技术的发展历程及产业化对策[J]. 生物技术进展, 2021, 11(4): 405-417. LIN M. The development course and industrialization countermeasure of agricultural biological breeding technology[J]. Current Biotechnology, 2021, 11(4): 405-417. (in Chinese) |
| [3] |
孙兵, 闫彩霞, 张廷婷, 等. 基因芯片技术在植物基因克隆中的应用研究进展[J]. 基因组学与应用生物学, 2009, 28(1): 153-158. SUN B, YAN C X, ZHANG T T, et al. Research progress of gene chip technology in plant gene cloning[J]. Genomics and Applied Biology, 2009, 28(1): 153-158. (in Chinese) |
| [4] |
TAO L, HE X Y, JIANG Y T, et al. Combined approaches to reveal genes associated with litter size in Yunshang black goats[J]. Anim Genet, 2020, 51(6): 924-934. DOI:10.1111/age.12999 |
| [5] |
BANOS G, LINDSAY V, DESTA T T, et al. Integrating genetic and genomic analyses of combined health data across ecotypes to improve disease resistance in indigenous African chickens[J]. Front Genet, 2020, 11: 543890. DOI:10.3389/fgene.2020.543890 |
| [6] |
王宇哲. 利用简化基因组测序精细定位鸡生长性状功能基因[D]. 北京: 中国农业大学, 2017. WANG Y Z. Fine mapping the QTL for growth traits in chicken by improved genotyping by sequencing[D]. Beijing: China Agricultural University, 2017. (in Chinese) |
| [7] |
BAIRD N A, ETTER P D, ATWOOD T S, et al. Rapid SNP discovery and genetic mapping using sequenced RAD markers[J]. PLoS One, 2008, 3(10): e3376. DOI:10.1371/journal.pone.0003376 |
| [8] |
WANG S, MEYER E, MCKAY J K, et al. 2b-RAD: a simple and flexible method for genome-wide genotyping[J]. Nat Methods, 2012, 9(8): 808-810. DOI:10.1038/nmeth.2023 |
| [9] |
谈成, 边成, 杨达, 等. 基因组选择技术在农业动物育种中的应用[J]. 遗传, 2017, 39(11): 1033-1045. TAN C, BIAN C, YANG D, et al. Application of genomic selection in farm animal breeding[J]. Hereditas (Beijing), 2017, 39(11): 1033-1045. (in Chinese) |
| [10] |
ANDREWS K R, GOOD J M, MILLER M R, et al. Harnessing the power of RADseq for ecological and evolutionary genomics[J]. Nat Rev Genet, 2016, 17(2): 81-92. DOI:10.1038/nrg.2015.28 |
| [11] |
TAN C, WU Z F, REN J L, et al. Genome-wide association study and accuracy of genomic prediction for teat number in Duroc pigs using genotyping-by-sequencing[J]. Genet Sel Evol, 2017, 49(1): 35. DOI:10.1186/s12711-017-0311-8 |
| [12] |
YANG R F, GUO X L, ZHU D, et al. Accelerated deciphering of the genetic architecture of agricultural economic traits in pigs using a low-coverage whole-genome sequencing strategy[J]. GigaScience, 2021, 10(7): giab048. DOI:10.1093/gigascience/giab048 |
| [13] |
NICOD J, DAVIES R W, CAI N, et al. Genome-wide association of multiple complex traits in outbred mice by ultra-low-coverage sequencing[J]. Nat Genet, 2016, 48(8): 912-918. DOI:10.1038/ng.3595 |
| [14] |
LIPPOLD S, MATZKE N J, REISSMANN M, et al. Whole mitochondrial genome sequencing of domestic horses reveals incorporation of extensive wild horse diversity during domestication[J]. BMC Evol Biol, 2011, 11: 328. DOI:10.1186/1471-2148-11-328 |
| [15] |
NEWMAN J H, HOLT T N, COGAN J D, et al. Increased prevalence of EPAS1 variant in cattle with high-altitude pulmonary hypertension[J]. Nat Commun, 2015, 6: 6863. DOI:10.1038/ncomms7863 |
| [16] |
SCUTERI A, SANNA S, CHEN W M, et al. Genome-wide association scan shows genetic variants in the FTO gene are associated with obesity-related traits[J]. PLoS Genet, 2007, 3(7): e115. DOI:10.1371/journal.pgen.0030115 |
| [17] |
任航行, 李杰, 王高富, 等. 畜禽生产性状主效基因定位技术与方法研究进展[J]. 基因组学与应用生物学, 2020, 39(3): 1094-1100. REN H X, LI J, WANG G F, et al. Progress in gene mapping methods and technologies for the economical traits in farm animals[J]. Genomics and Applied Biology, 2020, 39(3): 1094-1100. (in Chinese) |
| [18] |
JI J X, ZHOU L S, GUO Y M, et al. Genome-wide association study identifies 22 new loci for body dimension and body weight traits in a White Duroc×Erhualian F2 intercross population[J]. Asian-Australas J Anim Sci, 2017, 30(8): 1066-1073. DOI:10.5713/ajas.16.0679 |
| [19] |
邱恒清. 巴马香猪全基因组拷贝数变异的检测及其与五块四肢骨骼长度的关联分析[D]. 南昌: 江西农业大学, 2017. QIU H Q. Genome-wide copy number variation detection and their association with the lengths of five limb bones in Bama Pigs[D]. Nanchang: Jiangxi Agricultural University, 2017. (in Chinese) |
| [20] |
LIEN C Y, TIXIER-BOICHARD M, WU S W, et al. Detection of QTL for traits related to adaptation to sub-optimal climatic conditions in chickens[J]. Genet Sel Evol, 2017, 49(1): 39. DOI:10.1186/s12711-017-0314-5 |
| [21] |
ZHANG X, CHU Q, GUO G, et al. Genome-wide association studies identified multiple genetic loci for body size at four growth stages in Chinese Holstein cattle[J]. PLoS One, 2017, 12(4): e0175971. DOI:10.1371/journal.pone.0175971 |
| [22] |
RAHMATALLA S A, ARENDS D, REISSMANN M, et al. Genome-wide association study of body morphological traits in Sudanese goats[J]. Anim Genet, 2018, 49(5): 478-482. DOI:10.1111/age.12686 |
| [23] |
SEROUSSI E, ROSOV A, SHIRAK A, et al. Unveiling genomic regions that underlie differences between Afec-Assaf sheep and its parental Awassi breed[J]. Genet Sel Evol, 2017, 49(1): 19. DOI:10.1186/s12711-017-0296-3 |
| [24] |
张易. 鸭羽色性状拷贝数变异和全基因组关联分析[D]. 扬州: 扬州大学, 2021. ZHANG Y. Copy number variation and genome-wide association analysis of color traits of duck feather[D]. Yangzhou: Yangzhou University, 2021. (in Chinese) |
| [25] |
谈成. 基于二代测序技术的猪基因组选择及全基因组关联分析研究[D]. 北京: 中国农业大学, 2017. TAN C. Genomic selection and genome-wide association studies in swine using the next generation sequencing technology[D]. Beijing: China Agricultural University, 2017. (in Chinese) |
| [26] |
乔贤. 绒山羊SNP芯片设计及重要经济性状全基因组关联分析研究[D]. 呼和浩特: 内蒙古农业大学, 2020. QIAO X. Study on SNP chip design and genome association analysis of important economic traits in cashmere goats[D]. Hohhot: Inner Mongolia Agricultural University, 2020. (in Chinese) |
| [27] |
汤香, 何俊, 蒋隽. 全基因组关联分析在畜禽中的研究进展[J]. 畜牧与兽医, 2020, 52(5): 147-152. TANG X, HE J, JIANG J. Progress in research on whole genome association studies in livestock and poultry[J]. Animal Husbandry & Veterinary Medicine, 2020, 52(5): 147-152. (in Chinese) |
| [28] |
马云龙, 张勤, 丁向东. 利用高密度SNP检测不同猪品种间X染色体选择信号[J]. 遗传, 2012, 34(10): 1251-1260. MA Y L, ZHANG Q, DING X D. Detecting selection signatures on X chromosome in pig through high density SNPs[J]. Hereditas (Beijing), 2012, 34(10): 1251-1260. (in Chinese) |
| [29] |
KIJAS J W, TOWNLEY D, DALRYMPLE B P, et al. A genome wide survey of SNP variation reveals the genetic structure of sheep breeds[J]. PLoS One, 2009, 4(3): e4668. DOI:10.1371/journal.pone.0004668 |
| [30] |
TOSSER-KLOPP G, BARDOU P, BOUCHEZ O, et al. Correction: design and characterization of a 52K SNP chip for goats[J]. PLoS One, 2016, 11(3): e0152632. DOI:10.1371/journal.pone.0152632 |
| [31] |
刘恩民. 中国地方山羊品种群体遗传结构和选择信号分析[D]. 兰州: 兰州大学, 2018. LIU E M. Analysis of population genetic structure and select signal in Chinese indigenous goat breeds[D]. Lanzhou: Lanzhou University, 2018. (in Chinese) |
| [32] |
JIN M L, LU J, FEI X J, et al. Genetic signatures of selection for cashmere traits in Chinese goats[J]. Animals (Basel), 2020, 10(10): 1905. |
| [33] |
EDEA Z, HONG J K, JUNG J H, et al. Detecting selection signatures between Duroc and Duroc synthetic pig populations using high-density SNP chip[J]. Anim Genet, 2017, 48(4): 473-477. DOI:10.1111/age.12559 |
| [34] |
李景. 基于猪60K芯片数据鉴别藏猪的高原适应性选择信号[D]. 南昌: 江西农业大学, 2014. LI J. Genome-wide detection of selective signals for high-altitude adaptation in Tibetan pigs by porcine60k chip data[D]. Nanchang: Jiangxi Agricultural University, 2014. (in Chinese) |
| [35] |
ZHANG H, HU X X, WANG Z P, et al. Selection signature analysis implicates the PC1/PCSK1 region for chicken abdominal fat content[J]. PLoS One, 2012, 7(7): e40736. DOI:10.1371/journal.pone.0040736 |
| [36] |
陈建兴, 童家兴, 张孝忠, 等. 山东小毛驴全基因组选择信号检测[J]. 河南农业科学, 2021, 50(2): 145-150. CHEN J X, TONG J X, ZHANG X Z, et al. Detection of selection signatures of population-specific whole-genomic regions selected in Shandong little donkey[J]. Journal of Henan Agricultural Sciences, 2021, 50(2): 145-150. (in Chinese) |
| [37] |
LI M Z, CHEN L, TIAN S L, et al. Comprehensive variation discovery and recovery of missing sequence in the pig genome using multiple de novo assemblies[J]. Genome Res, 2017, 27(5): 865-874. DOI:10.1101/gr.207456.116 |
| [38] |
LAN D L, XIONG X R, MIPAM T D. Genetic diversity, molecular phylogeny, and selection evidence of Jinchuan Yak revealed by whole-genome resequencing[J]. G3 (Bethesda), 2018, 8(3): 945-952. DOI:10.1534/g3.118.300572 |
| [39] |
GUO J Z, TAO H X, LI P F, et al. Whole-genome sequencing reveals selection signatures associated with important traits in six goat breeds[J]. Sci Rep, 2018, 8(1): 10405. DOI:10.1038/s41598-018-28719-w |
| [40] |
ZHANG Q, GOU W Y, WANG X T, et al. Genome resequencing identifies unique adaptations of Tibetan chickens to hypoxia and high-dose ultraviolet radiation in high-altitude environments[J]. Genome Biol Evol, 2016, 8(3): 765-776. DOI:10.1093/gbe/evw032 |
| [41] |
王琦, 朱迪, 王宇哲, 等. 全基因组SNP分型策略及基因组预测方法的研究进展[J]. 畜牧兽医学报, 2020, 51(2): 205-216. WANG Q, ZHU D, WANG Y Z, et al. Research progress of genomic-wide SNP genotyping and genomic prediction methods[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(2): 205-216. (in Chinese) |
| [42] |
LIU C M, WANG S H, DONG X G, et al. Exploring the genomic resources and analysing the genetic diversity and population structure of Chinese indigenous rabbit breeds by RAD-seq[J]. BMC Genomics, 2021, 22(1): 573. DOI:10.1186/s12864-021-07833-6 |
| [43] |
马士龙, 李小伟, 李响, 等. 基于GBS简化基因组测序评估3个麦洼牦牛保种群的遗传结构研究[J]. 草业学报, 2022, 31(9): 183-194. MA S L, LI X W, LI X, et al. Assessment of genetic structure of 3 Maiwa yak preserved populations based on genotyping-by-sequencing technology[J]. Acta Prataculturae Sinica, 2022, 31(9): 183-194. (in Chinese) |
| [44] |
夏树立, 周成浩, 赵向华, 等. 基于简化基因组测序的天津猴鸡遗传进化研究[J]. 中国家禽, 2022, 44(7): 1-5. XIA S L, ZHOU C H, ZHAO X H, et al. Genetic evolution mechanism of Tianjin-monkey chicken based on RAD-Seq[J]. China Poultry, 2022, 44(7): 1-5. (in Chinese) |
| [45] |
胡亮, 孙伟, 马月辉. 藏系绵羊群体遗传多样性及遗传结构分析[J]. 畜牧兽医学报, 2019, 50(6): 1145-1153. HU L, SUN W, MA Y H. Study on genetic diversity and genetic structure of Tibetan sheep populations[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(6): 1145-1153. (in Chinese) |
| [46] |
戴丽荷, 褚晓红, 陈晓宇, 等. 利用SNP标记分析淳安花猪群体遗传多样性和群体结构[J]. 养猪, 2021(6): 59-64. DAI L H, CHU X H, CHEN X Y, et al. Analysis of genetic diversity and genetic structure in Chun'an spotted pigs conserved population based on SNP chip[J]. Swine Production, 2021(6): 59-64. DOI:10.3969/j.issn.1002-1957.2021.06.019 (in Chinese) |
| [47] |
屠云洁, 唐燕飞, 蒋华莲, 等. 利用基因芯片开展广西麻鸡2个群体亲缘关系的遗传分析[J]. 中国畜牧杂志, 2021, 57(8): 143-146. TU Y J, TANG Y F, JIANG H L, et al. Genetic analysis of two populations of Guangxi Ma Chickens using gene chips[J]. Chinese Journal of Animal Science, 2021, 57(8): 143-146. (in Chinese) |
| [48] |
陈彬龙. 基因组重测序揭示鸡的遗传多样性和进化选择模式[D]. 雅安: 四川农业大学, 2018. CHEN B L. Genomic resequencing reveals genetic diversity and evolutionary selection patterns in chickens[D]. Ya'an: Sichuan Agricultural University, 2018. (in Chinese) |
| [49] |
GEBRESELASE H B. 全基因组重测序和SNP芯片对埃塞俄比亚和中国山羊群体的全基因组种质特性研究[D]. 北京: 中国农业科学院, 2019. GEBRESELASE H B. Genome-wide characterization of the Ethiopian and Chinese indigenous goat populations by whole genome re-sequencing and SNP array[D]. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese) |
| [50] |
张俸伟. 广西3个黄牛品种全基因组遗传多样性分析[D]. 杨凌: 西北农林科技大学, 2020. ZHANG F W. The whole-genome genetic diversity of three cattle breeds in Guangxi[D]. Yangling: Northwest A&F University, 2020. (in Chinese) |
| [51] |
盛中华, 陈国宏, 潘玉春, 等. 基于基因组遗传标记的上海白猪(上系)杂交优势预测分析[J]. 上海农业学报, 2018, 34(6): 60-64. SHENG Z H, CHEN G H, PAN Y C, et al. Prediction and analysis of heterosis in Shanghai white pigs(Shang) based on genome-wide genetic markers[J]. Acta Agriculturae Shanghai, 2018, 34(6): 60-64. (in Chinese) |
| [52] |
兰蓉, 朱兰, 江炎庭, 等. 基于简化基因组测序的黄色波尔山羊公羊亲缘关系及近交系数分析[J]. 中国畜牧兽医, 2021, 48(8): 2878-2888. LAN R, ZHU L, JIANG Y T, et al. Analysis on genetic relationship and inbreeding coefficient of yellow Boer rams by genotyping-by-sequencing[J]. China Animal Husbandry & Veterinary Medicine, 2021, 48(8): 2878-2888. (in Chinese) |
| [53] |
PSZCZOLA M, CALUS M P L. Updating the reference population to achieve constant genomic prediction reliability across generations[J]. Animal, 2016, 10(6): 1018-1024. |
| [54] |
OLSON K M, VANRADEN P M, TOOKER M E, et al. Differences among methods to validate genomic evaluations for dairy cattle[J]. J Dairy Sci, 2011, 94(5): 2613-2620. |
| [55] |
GODDARD M E, HAYES B J. Mapping genes for complex traits in domestic animals and their use in breeding programmes[J]. Nat Rev Genet, 2009, 10(6): 381-391. |
| [56] |
VANRADEN P M, VAN TASSELL C P, WIGGANS G R, et al. Invited review: reliability of genomic predictions for North American Holstein bulls[J]. J Dairy Sci, 2009, 92(1): 16-24. |
| [57] |
尹立林, 马云龙, 项韬, 等. 全基因组选择模型研究进展及展望[J]. 畜牧兽医学报, 2019, 50(2): 233-242. YIN L L, MA Y L, XIANG T, et al. The progress and prospect of genomic selection models[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 233-242. (in Chinese) |
| [58] |
李宏伟, 王瑞军, 王志英, 等. 家畜基因组选择研究进展[J]. 遗传, 2017, 39(5): 377-387. LI H W, WANG R J, WANG Z Y, et al. The research progress of genomic selection in livestock[J]. Hereditas (Beijing), 2017, 39(5): 377-387. (in Chinese) |
| [59] |
王景霖. 肉用绵羊基因组选择方法的模拟研究[D]. 太原: 山西农业大学, 2014. WANG J L. Study on the methods of genomic selection for meat sheep by simulation[D]. Taiyuan: Shanxi Agricultural University, 2014. (in Chinese) |
| [60] |
WHITTAKER C J, THOMPSON R, DENHAM M C. Marker-assisted selection using ridge regression[J]. Genet Res, 2000, 75(2): 249-252. |
| [61] |
GIANOLA D, DE LOS CAMPOS G, HILL W G, et al. Additive genetic variability and the Bayesian alphabet[J]. Genetics, 2009, 183(1): 347-363. |
| [62] |
VANRADEN P M. Efficient methods to compute genomic predictions[J]. J Dairy Sci, 2008, 91(11): 4414-4423. |
| [63] |
CHRISTENSEN O F, LUND M S. Genomic prediction when some animals are not genotyped[J]. Genet Sel Evol, 2010, 42: 2. |
| [64] |
MISZTAL I, LEGARRA A, AGUILAR I. Computing procedures for genetic evaluation including phenotypic, full pedigree, and genomic information[J]. J Dairy Sci, 2009, 92(9): 4648-4655. |
| [65] |
CHRISTENSEN O F, MADSEN P, NIELSEN B, et al. Single-step methods for genomic evaluation in pigs[J]. Animal, 2012, 6(10): 1565-1571. |
| [66] |
GARCÍA-RUIZ A, COLE J B, VANRADEN P M, et al. Changes in genetic selection differentials and generation intervals in US Holstein dairy cattle as a result of genomic selection[J]. Proc Natl Acad Sci U S A, 2016, 113(28): E3995-E4004. |
| [67] |
林敏. 农业生物育种技术的发展历程及产业化对策[J]. 生物技术进展, 2021, 11(4): 405-417. LIN M. The development course and industrialization countermeasure of agricultural biological breeding technology[J]. Current Biotechnology, 2021, 11(4): 405-417. (in Chinese) |
| [68] |
LIU R R, XING S Y, WANG J, et al. A new chicken 55K SNP genotyping array[J]. BMC Genomics, 2019, 20(1): 410. |
| [69] |
QI H G, SONG K, LI C Y, et al. Construction and evaluation of a high-density SNP array for the Pacific oyster (Crassostrea gigas)[J]. PLoS One, 2017, 12(3): e0174007. |
| [70] |
YÁÑEZ J M, YOSHIDA G, BARRIA A, et al. High-throughput single nucleotide polymorphism (SNP) discovery and validation through whole-genome resequencing in Nile Tilapia (Oreochromis niloticus)[J]. Mar Biotechnol (NY), 2020, 22(1): 109-117. |
| [71] |
刘继强. 一种鉴定滩羊与非滩羊的8种SNP位点及其应用: 中国, CN109182544B[P]. 2021-09-28. LIU J Q. A kind of eight SNP loci for identification of Tan sheep and non-Tan sheep and its application: CN, CN109182544B[P]. 2021-09-28. (in Chinese) |
| [72] |
范欢欢, 王天骄, 张然然, 等. 1K梅花鹿基因芯片的应用[J]. 特产研究, 2022, 44(3): 68-72. FAN H H, WANG T J, ZHANG R R, et al. Application of 1K Sika deer (Cervus nippon) gene chip[J]. Special Wild Economic Animal and Plant Research, 2022, 44(3): 68-72. (in Chinese) |
| [73] |
罗元宇, 吴鹏, 贺金龙, 等. 畜禽群体中基于SNP标记的亲子鉴定及亲本推断方法[J]. 华中农业大学学报, 2016, 35(5): 68-74. LUO Y Y, WU P, HE J L, et al. Parentage identification and paternity inference based on SNP markers in livestock population[J]. Journal of Huazhong Agricultural University, 2016, 35(5): 68-74. (in Chinese) |
| [74] |
HEATON M P, LEYMASTER K A, KALBFLEISCH T S, et al. SNPs for parentage testing and traceability in globally diverse breeds of sheep[J]. PLoS One, 2014, 9(4): e94851. |
| [75] |
BOERNER V. On marker-based parentage verification via non-linear optimization[J]. Genet Sel Evol, 2017, 49(1): 50. |
| [76] |
郭立平. 利用微卫星和SNP标记对西门塔尔牛进行亲子推断的研究[D]. 北京: 中国农业科学院, 2013. GUO L P. Parentage testing with microsatellite and SNP markers in Simmental[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. (in Chinese) |
| [77] |
张哲, 罗元宇, 李晴晴, 等. 一种基于高密度遗传标记的亲子鉴定方法及其应用[J]. 遗传, 2014, 36(8): 835-841. ZHANG Z, LUO Y Y, LI Q Q, et al. Developing and applying of a parentage identification approach based on high density genetic markers[J]. Hereditas (Beijing), 2014, 36(8): 835-841. (in Chinese) |
| [78] |
马丽娜, 刘永进, 王锦, 等. 芯片技术在畜禽育种中的应用研究进展[J]. 中国畜牧兽医, 2020, 47(1): 98-106. MA L N, LIU Y J, WANG J, et al. Research advance on application of chip technology in livestock and poultry breeding[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(1): 98-106. (in Chinese) |
(编辑 郭云雁)



