2. 奶牛遗传育种与繁殖北京市重点实验室,北京 100192;
3. 农业农村部奶牛遗传育种与繁殖重点实验室,北京 100192;
4. 北京市畜牧总站,北京 100107;
5. 中国农业大学动物科技学院,北京 100193
2. Beijing Key Laboratory of Dairy Cattle Genetic, Breeding and Reproduction, Beijing 100192, China;
3. Key Laboratory of Dairy Cattle Genetic, Breeding and Reproduction, Ministry of Agriculture and Rural Affairs, Beijing 100192, China;
4. Beijing General Station of Animal Husbandry, Beijing 100107, China;
5. College of Animal Science and Technology, China Agricultural University, Beijing 100193, China
产奶性状是奶牛最重要的经济性状,包括产奶量、乳脂量、乳蛋白量、乳脂率和乳蛋白率[1],属于受大量基因和诸多环境因素共同影响的数量性状。在过去的几十年里,数量性状位点(QTL)、候选基因分析和全基因组关联研究(GWAS)被广泛用于鉴定奶牛的产奶性状基因,例如DGAT1、GHR和ABCG2等基因已被证实与产奶性状显著相关[2-7]。
RNA测序(RNA-seq)通过高通量技术进行测序分析,已成为分析基因表达模式的一种全面、准确的工具[8]。通过对具有极端高、低乳蛋白率和乳脂率的4头中国荷斯坦牛乳腺组织进行RNA-seq转录组测序,结果共鉴定出31个差异表达基因[9],有14个基因既位于奶牛乳蛋白、乳脂性状QTL区段又临近显著SNP位点,其中包括RPL23A等基因[10]。牛核糖体蛋白L23A(ribosomal protein L23a, RPL23A)基因位于19号染色体上,全长2 871 bp,由5个外显子和4个内含子组成,mRNA全长614 bp,共编码156个氨基酸。RPL23A是一种核糖体蛋白,是60S亚单位的一个组成部分,在细胞分化、分裂和凋亡等功能蛋白合成过程中起重要作用[11]。所以,RPL23A基因可以作为影响产奶性状的候选基因进行遗传效应分析。
乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACC)是催化脂肪酸合成代谢第一步反应的限速酶,将乙酰辅酶A羧化生成丙二酰单酰辅酶A,属于生物素依赖性酶。ACC广泛存在于多种生物中,在人类和其它哺乳动物中该酶属于组织特异性酶,存在两种变构体ACC1(或称ACCα)和ACC2(或称ACCβa),分别由ACACA和ACACB基因编码[12-13]。乙酰辅酶A羧化酶B(ACACB)是一种促进脂肪合成的限速酶,在糖脂代谢中起重要作用。之前的研究表明,ACACB基因位于已知对产奶量和乳成分性状有很大遗传效应的区域[14-15]。牛的ACACB基因位于第17号染色体上,全长116.36 kb,包含58个外显子和57个内含子。其mRNA全长7 007 bp,共编码2 331个氨基酸,mRNA序列与人的同源性为86%,氨基酸序列与人的同源性为88%。ACACB基因被认为是影响牛奶成分的一个潜在重要候选基因。课题组前期RNA-seq研究鉴定了一批候选功能基因的遗传效应,确定RPL23A和ACACB基因与中国荷斯坦牛产奶量和乳成分性状显著关联;因此本研究选择RPL23A和ACACB基因的3个SNPs位点在荷斯坦群体中进行验证。
本研究采用检测成本低、准确性高的微流控芯片SNP分型技术,对前期研究获得的与产奶性状潜在关联的RPL23A和ACACB基因的3个SNPs位点进行大样本验证及关联性分析,并进行群体多态性分析,为奶牛产奶性状功能基因的挖掘以及高乳成分性状群体的选育提供可靠的候选基因标记。
1 材料与方法 1.1 样品采集本研究以北京地区8个牛场的1 596头中国荷斯坦母牛为试验群体,所有个体均为3胎以内的泌乳牛,每头牛所有测定日的产奶量、乳脂率、乳蛋白率等数据来源于中心奶牛生产性能测定实验室。试验个体通过EDTA抗凝真空管采集尾根血样4 mL,-20 ℃保存备用。
1.2 主要试剂血液DNA提取试剂盒以及RNA酶购自天根生化科技(北京)有限公司,其他无水乙醇、异丙醇等均为国产分析纯。
1.3 方法1.3.1 DNA提取 采用天根DNA提取试剂盒,根据试剂盒说明书提取血样DNA,TB缓冲液洗脱溶解, 1.0%琼脂糖凝胶电泳检测DNA质量,同时采用NanoDrop 2000核酸蛋白测定仪检测DNA浓度和质量。将OD260 nm /OD280 nm值在1.7 ~2.0之间的样本调整浓度约50 ng·μL-1,置于96孔板中,-20 ℃保存备用。
1.3.2 位点的开发与设计 检索NCBI数据库发布的RPL23A(GenBank登录号为NC_037346.1)、ACACB基因序列信息(GenBank登录号为NC_037344.1),根据3个SNPs位点(g.20146771G>A、g.63962768C>T、g.63878254T>C)上、下游各150 bp序列信息,利用Primer Express3.0软件进行引物设计。两个等位基因特异的正向引物(分别为FAM标记、HEX标记),一个用于野生型等位基因,另一个用于突变型等位基因,以及一个通用的反向引物(下游引物R),被用于3个SNPs的分型检测,具体引物信息见表 1。
|
|
表 1 高乳成分位点扩增引物序列 Table 1 Primer sequences for high milk composition SNPs |
1.3.3 SNP分型检测 本研究中SNP基因型检测由北京博奥生物技术有限公司完成,SNP分型采用基于微流控芯片的SNP检测系统,其原理是在微流控芯片上完成基于等位基因特异扩增原理的基因分型。SNP分型检测流程如下:1)配置PCR扩增反应体系,用移液器将配置的反应体系从芯片入口注入到芯片里,并密封进出口。2)将芯片放入到离心机中4 000 r·min-1离心1 min;放入芯片热封仪中进行热封1 s。3) 放入平板PCR仪上进行扩增反应,PCR扩增体系约1 μL: DNA模板20 ng, Mix(2×) 0.5 μL,0.14 μL引物混合物(包括两种等位基因特异性上游引物各12 μmol·L-1,通用下游引物30 μmol·L-1),以及0.36 μL ddH2O。4)PCR扩增程序: 95 ℃预变性15 min; 95 ℃变性20 s, 61~55 ℃ 60 s,共10个降落扩增循环(每循环降0.6 ℃),之后26个扩增循环,95 ℃变性20 s, 55 ℃ 60 s; 37 ℃ 60 s完成扩增。5)PCR扩增程序运行结束后,通过LuxScan-10K/D扫描仪进行扫描,生成tif文件,通过软件转换成数据信号值,然后将原始数据导入SNPTyper软件中,可输出每个位点SNP分型结果,例如基因型、信号值、检出率等。
1.4 数据分析1.4.1 表型数据及基因型数据的质量控制 牛只具有完整的系谱信息和规范的DHI测定记录,包括第一泌乳期记录989条和第二泌乳期记录666条。表型数据包括305 d产奶量、乳脂率、乳脂量、乳蛋白率和乳蛋白量5个性状。
1.4.2 关联分析 本研究采用SAS 9.4软件中的MIXED过程对试验群体的中国荷斯坦母牛产奶量、乳脂量、乳脂率、乳蛋白量和乳蛋白率5个产奶性状和SNP位点基因型进行关联分析,采用动物模型如下:
| $ Y=\mu+h y s+b \times M+G+a+e $ |
其中,y为个体产奶性状(产奶量、乳脂量、乳脂率、乳蛋白量和乳蛋白率)表型值,μ为总体均值,hys为场年季效应,b为协变量M的回归系数,M为产犊月龄效应,G为基因型,a为个体随机加性效应,e为随机残差效应。多重比较采用Bonferroni法进行矫正,本模型中场年季水平合计47个。
1.4.3 遗传参数计算 利用GenAlE x6.5软件计算3个SNPs位点在荷斯坦牛群中的等位基因频率及基因型频率等遗传参数。
2 结果 2.1 DNA提取结果对血液样本进行了DNA提取,经琼脂糖凝胶电泳检测,条带清晰单一,DNA的OD260 nm /OD280 nm值、浓度、体积均达到位点分型检测要求,部分样本电泳检测结果见图 1。
|
M.DL2000 marker; 1.170062; 2.170063; 3.170064; 4.170065; 5.170066; 6.070067; 7.180001; 8.180003 图 1 8个样本DNA电泳检测结果图 Fig. 1 Plot of DNA electrophoresis results from 8 samples |
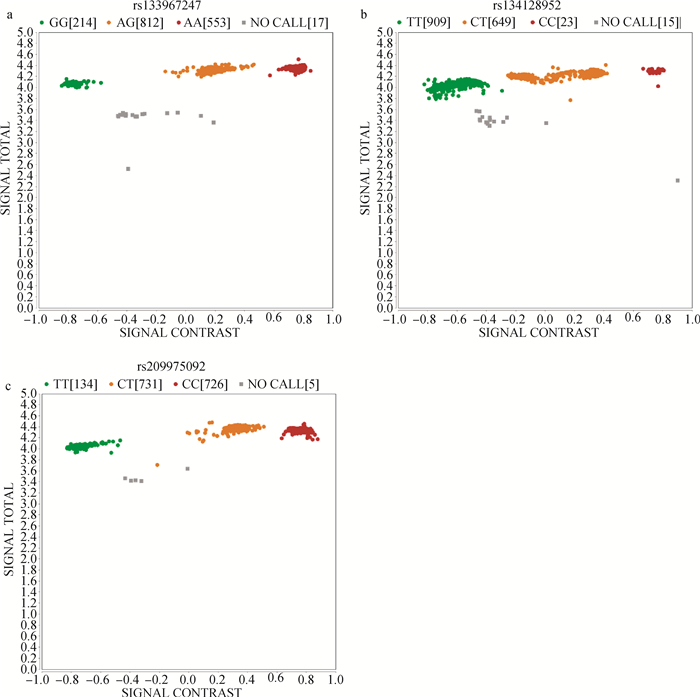
采用微流控平台进行PCR扩增,根据荧光信号的散点图,确定测定个体基因型,每个位点既有位点纯合子,也有杂合子。通过对高乳成分位点的检测,3个高乳成分位点的检出率均在98.9%以上,位点分型结果见图 2,X轴代表两种荧光(FAM和HEX)的比值,Y轴代表荧光的强度。
|
a. rs133967247分型结果图;b. rs134128952分型结果图;c. rs209975092分型结果图。X轴代表两种荧光的比值,Y轴代表荧光的强度 a. rs133967247 typing result diagram; b. rs134128952 typing result diagram; c. rs209975092 typing result diagram. The X-axis represents the ratio of two kinds of fluorescence, and the Y-axis represents the intensity of fluorescence 图 2 3个SNPs位点分型结果图 Fig. 2 Genotyping results of 3 SNPs |
利用高通量技术对北京地区荷斯坦牛群体中的高乳成分位点进行检测,3个SNPs位点经微流控芯片技术分型,结果显示3个位点基因型检出率均大于90%,位点基因型检出率及等位基因频率见表 2,分型数据可用于后续关联分析。群体中RPL23A基因优势位点rs134128952等位基因G的频率为68.62%,A等位基因的频率为31.38%;ACACB基因优势位点rs209975092等位基因C的频率为30.22%,T等位基因的频率为69.78%;ACACB基因优势位点rs133967247等位基因T的频率为60.74%,C等位基因的频率为39.26%。通过精准的选种选配可进一步提高优势等位基因的频率。
|
|
表 2 优势位点等位基因频率结果统计 Table 2 Statistics of allele frequency results of dominant loci |
RPL23A基因的SNP位点g.20146771G>A与5个产奶性状的关联分析结果见表 3。在第一泌乳期,g.20146771G>A与产奶量、乳脂量、乳脂率和乳蛋白量均达到极显著关联(P<0.01),与乳蛋白率关联不显著(P>0.05);第二泌乳期,g.20146771G>A与5个产奶性状均呈极显著关联(P<0.01)。
|
|
表 3 RPL23A基因SNP位点与5个产奶性状的关联分析(最小二乘均值±标准误) Table 3 Association analysis between SNP locus of RPL23A gene and 5 milk production traits(least squares mean±standard error) |
ACACB基因的2个SNPs位点与5个产奶性状的关联分析结果见表 4。SNP位点g.63962768C> T在第一泌乳期,与产奶量、乳脂量、乳脂率和乳蛋白量均呈极显著关联(P<0.01),与乳蛋白率呈不显著关联(P>0.05);在第二泌乳期,与产奶量、乳脂量、乳脂率和乳蛋白率呈极显著关联(P<0.01),与乳蛋白量关联不显著(P>0.05)。SNP位点g.63878254T>C在第一泌乳期中,仅与乳脂量呈极显著关联(P<0.01),与其余4个产奶性状均无显著关联(P>0.05),在第二泌乳期中,除乳蛋白量无显著关联外(P>0.05),其余4个产奶性状均有极显著关联(P<0.01)。
|
|
表 4 ACACB基因的2个SNPs位点与5个产奶性状的关联分析(最小二乘均值±标准误) Table 4 Association analysis between two SNPs of ACACB gene and 5 milk production traits (least squares mean±standard error) |
RPL23A基因是真核生物核糖体不可或缺的组成部分,在蛋白质合成、折叠及排列中具有重要作用。它的主要功能之一是在核糖体的合成和功能中发挥关键作用,可提高rRNA合成蛋白质的催化能力[16]。核糖体蛋白可以调节基因转录、mRNA翻译、细胞凋亡和DNA修复[17]。根据之前的研究,RPL23A可能是一种正调控乳蛋白量的基因[18]。本研究结果提示,RPL23A基因是一个影响奶牛乳脂率、乳蛋白率性状的重要关键基因,RPL23A基因位点rs134128952与第一、第二泌乳期的乳脂率、乳蛋白率均达到显著关联水平(P<0.01)。Li等[11]首次在RPL23A基因中发现了1个单倍型块与产奶性状关联显著,同时发现1个SNP位点(g.20702122 C>G)可能通过改变转录因子结合位点而显著影响产奶性状。
ACACB是调节脂肪代谢的限速酶,定位于线粒体外膜肉碱棕榈酰转移酶附近[18],它可能对脂代谢起调节作用。脂肪酸氧化研究发现,人类ACACB的等位基因变体可能与代谢综合征相关[19],据报道ACACB的等位基因变异与产奶特性有关,如在山羊和牛中[20-21]。本研究结果表明,ACACB基因的2个SNPs位点与第一、二泌乳期的乳脂率显著相关,此外ACACB是脂肪酸氧化的关键调节因子[22],因此认为ACACB及其SNPs可能影响牛奶中的乳脂成分。Han等[13]研究发现,ACACB基因2个SNPs位点,rs135874354 (g.66218726T>C)和rs210928430 (g.66218117G>A)与产奶量、脂肪含量显著相关,转录因子结合位点分析表明,rs135874354被认为是一个潜在的功能性突变。
奶牛全基因组评估自2008年开始,随着基因分型成本的降低,进行基因分型的奶牛数量持续增加,这些记录将成为用于基因组评估的基本数据[23-27]。在选择目标中,产奶性状是奶牛的主要生产性状,挖掘奶牛的产奶性状功能基因位点和重要遗传变异一直是我国奶牛分子育种的关键。此外,产奶性状是一种数量性状,由微效多基因决定,影响奶牛产奶性状的候选基因如EEF1D、AGPAT3、SAA2、OPN等已被文献证实[28-32],可以通过挖掘影响奶牛产奶性状的关键基因,进一步改良奶牛产奶性状,为从分子水平提高奶牛产奶性能提供依据。
研究表明,AGPAT3 SNP g.12264 C>T的主要等位基因C与试验日产奶量、蛋白质百分比和305日产奶量呈正相关,进一步分析表明,该位点的C/T转换产生两个新的转录因子结合位点(TFB),E2F1和Nkx3-2[33]。SAA2基因SNP g.14061A>G和g.14819C>T和组合基因型Hap1/5的关联分析表明,中国荷斯坦奶牛的产奶性状显著受这些基因位点的影响(P<0.05)[34]。功能SNP位点与产奶性状之间的关联可用于标记辅助选择,用于产奶性状的遗传改良,并可能与其他性状一起用于选择,以提高奶牛的适应性[35-37]。
根据对荷斯坦奶牛泌乳相关基因研究进展进行综合分析发现,目前大部分泌乳相关基因未进行相关性分析和功能验证。功能SNP位点验证可通过与产奶性状关联分析、在牛乳腺上皮细胞中开展RNAi功能分析或信号通路分析等[38-40]。在后期的研究中,需进一步扩大泌乳基因研究范围,并积极开展功能验证,以便于更准确地应用于奶牛群体遗传改良。
4 结论通过奶牛功能性SNP与产奶性状之间的关联分析,揭示了单核苷酸多态性对产奶量和乳成分的影响。本研究结果表明,RPL23A、ACACB基因3个SNPs是影响奶牛产奶量和乳成分的潜在功能性突变,RPL23A、ACACB基因可以作为影响中国荷斯坦奶牛产奶性状的候选基因用于标记辅助选择,以上基因位点可能通过直接或间接的途径影响奶牛的乳脂或乳蛋白性状,对产奶性状起到重要调控作用。本研究为荷斯坦奶牛后续的标记辅助选择奠定了良好的基础。
| [1] |
SPELMAN R J, COPPIETERS W, KARIM L, et al. Quantitative trait loci analysis for five milk production traits on chromosome six in the Dutch Holstein-Friesian population[J]. Genetics, 1996, 144(4): 1799-1808. DOI:10.1093/genetics/144.4.1799 |
| [2] |
PAUSCH H, EMMERLING R, GREDLER-GRANDL B, et al. Meta-analysis of sequence-based association studies across three cattle breeds reveals 25 QTL for fat and protein percentages in milk at nucleotide resolution[J]. BMC Genomics, 2017, 18(1): 853. DOI:10.1186/s12864-017-4263-8 |
| [3] |
LI F Y, CAI C C, QU K X, et al. DGAT1 K232A polymorphism is associated with milk production traits in Chinese cattle[J]. Anim Biotechnol, 2021, 32(4): 427-431. DOI:10.1080/10495398.2020.1711769 |
| [4] |
GRISART B, FARNIR F, KARIM L, et al. Genetic and functional confirmation of the causality of the DGAT1 K232A quantitative trait nucleotide in affecting milk yield and composition[J]. Proc Natl Acad Sci U S A, 2004, 101(8): 2398-2403. DOI:10.1073/pnas.0308518100 |
| [5] |
BLOTT S, KIM J J, MOISIO S, et al. Molecular dissection of a quantitative trait locus: a phenylalanine-to-tyrosine substitution in the transmembrane domain of the bovine growth hormone receptor is associated with a major effect on milk yield and composition[J]. Genetics, 2003, 163(1): 253-266. DOI:10.1093/genetics/163.1.253 |
| [6] |
OLSEN H G, NILSEN H, HAYES B, et al. Genetic support for a quantitative trait nucleotide in the ABCG2 gene affecting milk composition of dairy cattle[J]. BMC Genet, 2007(8): 32-41. |
| [7] |
BHATTARAI D, DAD R, WORKU T, et al. The functions and mechanisms of sequence differences of DGAT1 gene on milk fat synthesis between dairy cow and buffalo[J]. J Dairy Res, 2020, 87(2): 170-174. DOI:10.1017/S0022029920000126 |
| [8] |
COHEN-ZINDER M, SEROUSSI E, LARKIN D M, et al. Identification of a missense mutation in the bovine ABCG2 gene with a major effect on the QTL on chromosome 6 affecting milk yield and composition in Holstein cattle[J]. Genome Res, 2005, 15(7): 936-944. DOI:10.1101/gr.3806705 |
| [9] |
LI C, CAI W, ZHOU C, et al. RNA-Seq reveals 10 novel promising candidate genes affecting milk protein concentration in the Chinese Holstein population[J]. Scientific Reports, 2016, 6: 26813. DOI:10.1038/srep26813 |
| [10] |
CUI X G, HOU Y L, YANG S H, et al. Transcriptional profiling of mammary gland in Holstein cows with extremely different milk protein and fat percentage using RNA sequencing[J]. BMC Genomics, 2014, 15: 226. DOI:10.1186/1471-2164-15-226 |
| [11] |
LI Y, HAN B, LIU L, et al. Genetic association of DDIT3, RPL23A, SESN2 and NR4A1 genes with milk yield and composition in dairy cattle[J]. Anim Genet, 2019, 50(2): 123-135. DOI:10.1111/age.12750 |
| [12] |
BENNEWITZ J, REINSCH N, GROHS C, et al. Combined analysis of data from two granddaughter designs: a simple strategy for QTL confirmation and increasing experimental power in dairy cattle[J]. Genet Sel Evol, 2003, 35(3): 319-338. DOI:10.1186/1297-9686-35-3-319 |
| [13] |
HAN B, LIANG W, LIU L, et al. Genetic association of the ACACB gene with milk yield and composition traits in dairy cattle[J]. Anim Genet, 2018, 49(3): 169-177. DOI:10.1111/age.12651 |
| [14] |
JIANG J P, GAO Y H, HOU Y L, et al. Whole-genome resequencing of Holstein bulls for indel discovery and identification of genes associated with milk composition traits in dairy cattle[J]. PLoS One, 2016, 11(12): e0168946. DOI:10.1371/journal.pone.0168946 |
| [15] |
COLE J B, WIGGANS G R, MA L, et al. Genome-wide association analysis of thirty one production, health, reproduction and body conformation traits in contemporary U.S. Holstein cows[J]. BMC Genomics, 2011, 12: 408. DOI:10.1186/1471-2164-12-408 |
| [16] |
DAS S, SHAH P, BAHARIA R K, et al. Over-expression of 60 s ribosomal L23a is associated with cellular proliferation in SAG resistant clinical isolates of Leishmania donovani[J]. PLoS Negl Trop Dis, 2013, 7(12): e2527. DOI:10.1371/journal.pntd.0002527 |
| [17] |
WOOL I G. Extraribosomal functions of ribosomal proteins[J]. Trends Biochem Sci, 1996, 21(5): 164-165. DOI:10.1016/S0968-0004(96)20011-8 |
| [18] |
CHANG K C, WEN J D, YANG L W. Functional importance of mobile ribosomal proteins[J]. Biomed Res Int, 2015, 2015: 539238. |
| [19] |
ABU-ELHEIGA L, ALMARZA-ORTEGA D B, BALDINI A, et al. Human acetyl-CoA carboxylase 2:molecular cloning, characterization, chromosomal mapping, and evidence for two isoforms[J]. J Biol Chem, 1997, 272(16): 10669-10677. DOI:10.1074/jbc.272.16.10669 |
| [20] |
CECCHINATO A, RIBECA C, CHESSA S, et al. Candidate gene association analysis for milk yield, composition, urea nitrogen and somatic cell scores in Brown Swiss cows[J]. Animal, 2014, 8(7): 1062-1670. DOI:10.1017/S1751731114001098 |
| [21] |
FEDERICA S, FRANCESCO N, GIOVANNA D M, et al. Identification of novel single nucleotide polymorphisms in promoter Ⅲ of the acetyl-CoA carboxylase-α gene in goats affecting milk production traits[J]. J Hered, 2009, 100(3): 386-389. DOI:10.1093/jhered/esn098 |
| [22] |
WIGGANS G R, COLE J B, HUBBARD S M, et al. Genomic selection in dairy cattle: the USDA experience[J]. Annu Rev Anim Biosci, 2017, 5: 309-327. DOI:10.1146/annurev-animal-021815-111422 |
| [23] |
OLIVEIRA H R, CANT J P, BRITO L F, et al. Genome-wide association for milk production traits and somatic cell score in different lactation stages of Ayrshire, Holstein, and Jersey dairy cattle[J]. J Dairy Sci, 2019, 102(9): 8159-8174. DOI:10.3168/jds.2019-16451 |
| [24] |
STERI R, MOIOLI B, CATILLO G, et al. Genome-wide association study for longevity in the Holstein cattle population[J]. Animal, 2019, 13(7): 1350-1357. DOI:10.1017/S1751731118003191 |
| [25] |
ATASHI H, SALAVATI M, DE KOSTER J, et al. Genome-wide association for milk production and lactation curve parameters in Holstein dairy cows[J]. J Anim Breed Genet, 2020, 137(3): 292-304. DOI:10.1111/jbg.12442 |
| [26] |
YANG Z, LIAN Z, LIU G, et al. Identification of genetic markers associated with milk production traits in Chinese Holstein cattle based on post genome-wide association studies[J]. Anim Biotechnol, 2021, 32(1): 67-76. DOI:10.1080/10495398.2019.1653901 |
| [27] |
IUNG LHS, PETRINI J, RAMÍREZ-DÍAZ J, et al. Genome-wide association study for milk production traits in a Brazilian Holstein population[J]. J Dairy Sci, 2019, 102(6): 5305-5314. DOI:10.3168/jds.2018-14811 |
| [28] |
WANG Y H, ZHAI J Y, YANG C H, et al. Complement component 3 haplotypes influence serum complement activity and milk production traits in Chinese Holstein cattle[J]. PLoS One, 2022, 17(6): e0268959. DOI:10.1371/journal.pone.0268959 |
| [29] |
HAGIYA K. Development of genetic evaluation for milk production traits of Holsteins in Japan[J]. Anim Sci J, 2019, 90(4): 457-461. DOI:10.1111/asj.13190 |
| [30] |
RAZA S H A, KHAN R, PANT S D, et al. Genetic variation in the OPN gene affects milk composition in Chinese Holstein cows[J]. Anim Biotechnol, 2021, 15: 1-7. |
| [31] |
JIANG J C, MA L, PRAKAPENKA D, et al. A large-scale genome-wide association study in U.S. Holstein cattle[J]. Front Genet, 2019, 10: 412. DOI:10.3389/fgene.2019.00412 |
| [32] |
ABDOLLAHI-ARPANAHI R, CARVALHO M R, RIBEIRO E S, et al. Association of lipid-related genes implicated in conceptus elongation with female fertility traits in dairy cattle[J]. J Dairy Sci, 2019, 102(11): 10020-10029. DOI:10.3168/jds.2019-17068 |
| [33] |
SUN X M, LIANG Y, GAO Q S, et al. AGPAT3 Gene polymorphisms are associated with milk production traits in Chinese Holstein cows[J]. J Dairy Res, 2021, 88(3): 247-252. DOI:10.1017/S0022029921000546 |
| [34] |
RAZA S H A, LIANG C C, ALSHAMMARI A M, et al. Genetic variants in the serum amyloid A2 (SAA2) gene as a potential marker for milk production traits in Chinese Holstein cows[J]. Vet Med Sci, 2022, 8(4): 1835-1840. DOI:10.1002/vms3.796 |
| [35] |
CAI Z X, GULDBRANDTSEN B, LUND M S, et al. Prioritizing candidate genes post-GWAS using multiple sources of data for mastitis resistance in dairy cattle[J]. BMC Genom, 2018, 19(1): 656. DOI:10.1186/s12864-018-5050-x |
| [36] |
GALLIOU J M, KISER J N, OLIVER K F, et al. Identification of loci and pathways associated with heifer conception rate in U.S. Holsteins[J]. Genes, 2020, 11(7): 767. DOI:10.3390/genes11070767 |
| [37] |
LU H, WANG Y, BOVENHUIS H. Genome-wide association study for genotype by lactation stage interaction of milk production traits in dairy cattle[J]. J Dairy Sci, 2020, 103(6): 5234-5245. DOI:10.3168/jds.2019-17257 |
| [38] |
CHEN S Y, OLIVEIRA H R, SCHENKEL F S, et al. Using imputed whole-genome sequence variants to uncover candidate mutations and genes affecting milking speed and temperament in Holstein cattle[J]. J Dairy Sci, 2020, 103(11): 10383-10398. DOI:10.3168/jds.2020-18897 |
| [39] |
杨洋, 周子薇, 张京一, 等. 奶牛SP1基因结构及对乳脂合成功能的初步分析[J]. 畜牧兽医学报, 2022, 53(9): 2970-2981. YANG Y, ZHOU Z W, ZHANG J Y, et al. Analysis on SP1 gene structure and its function on milk fat synthesis in holstein dairy cows[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(9): 2970-2981. (in Chinese) |
| [40] |
HOU Y L, XIE Y, YANG S H, et al. EEF1D facilitates milk lipid synthesis by regulation of PI3K-Akt signaling in mammals[J]. FASEB J, 2021, 35(5): e21455. |
(编辑 郭云雁)



