猪圆环病毒2型属于圆环病毒科、圆环病毒属的单链环状DNA病毒,是引起猪圆环病毒相关性疾病(porcine circovirus-associated diseases,PCVAD)的主要病原[1]。PCV2侵害免疫组织,造成淋巴细胞衰竭及免疫抑制,常与猪繁殖与呼吸综合征病毒、猪流行性腹泻病毒、猪细小病毒等混合感染[2]。近年来,变异的PCV2毒株不断出现,猪群中PCV2疫苗免疫失败的情况也时有发生,给全世界的养猪业带来巨大的经济损失[3]。PCV2基因组主要包含两个方向相反的开放阅读框(open reading frame,ORF),ORF1编码病毒复制相关蛋白Rep及Rep′,ORF2编码具有免疫原性的衣壳蛋白Cap[4]。两个开放阅读框的5′端之间是PCV2基因组的复制起始位点,Ori区域的特征是包含一段保守九聚核苷酸的茎环结构以及位于茎环结构下游的4个六聚体重复序列(5′-CGGCAG-3′)[5]。PCV2基因组复制需要Rep复合体(Rep-Rep′)和Ori的相互作用[6]。为了启动基因组合成,Rep复合体结合茎环右臂的H1/H2六聚体重复序列并引起构象改变,茎环结构暴露,Rep/Rep′蛋白特异切割茎环的九聚核苷酸并共价结合在5′端起始PCV2基因组的复制[7-8]。
聚(ADP-核糖)聚合酶1[poly (ADP-ribose) polymerase 1, PARP1]蛋白是PARP家族的成员,调节多种DNA损伤修复通路,在发生基因损伤后,PARP1被招募到DNA断裂处帮助DNA损伤修复蛋白对DNA进行修复[9]。研究表明,PARP1在DNA病毒复制中也发挥重要作用。PARP1与乙型肝炎病毒(hepatitis B, HBV)核心启动子结合,增强病毒基因转录和HBV DNA整合[10]。单纯疱疹病毒1型(herpes simplex virus 1, HSV-1)通过激活PARP1蛋白,促进病毒复制[11]。
PCV2是自然界最小的动物病毒之一,基因组资源十分有限,病毒复制的多个过程都需要蛋白的参与,然而哪些蛋白参与了病毒复制复合体的组装,报道十分有限。Ori是PCV2复制的起始位点及病毒复制复合体的组装部位,本研究用DNA-protein pull down的方法筛选与PCV2 Ori互作的蛋白,发现了多种可与病毒Ori互作的蛋白,并对PARP1蛋白与PCV2 Ori的互作进行了验证,这对病毒的复制机制以及抗病毒靶标的研究具有重要意义。
1 材料与方法 1.1 材料人胚胎肾细胞系(HEK293T)、猪肾细胞(PK-15)、真核质粒p3×FLAG-CMV-14及pCAGGS-HA均由本实验室保存。PCV2-WH株由华中农业大学农业微生物国家重点实验室何启盖教授馈赠。限制性内切酶、T4 DNA连接酶购自大连宝生物公司。链霉亲和素磁珠、Protein A/G磁珠、Flag标签抗体磁珠及HA标签抗体磁珠均购自MCE公司。Flag、HA、β-actin、PARP1标签抗体及鼠源IgG购自武汉三鹰生物技术有限公司。银染试剂盒购自Sigma-Aldrich公司。荧光定量PCR试剂盒购自ABclonal公司。病毒DNA提取试剂盒与2×F8 FastLong PCR MasterMix购自北京艾德莱生物科技有限公司。引物由武汉擎科生物技术有限公司合成。一步法高效RT-PCR试剂盒购自南京诺唯赞生物科技股份有限公司。
1.2 DNA-protein pull down蛋白结合探针的制备以PCV2基因组DNA为模板进行Ori片段(139 bp)的扩增,引物见表 1,上、下游引物的5′端均用生物素标记。使用序列相同的普通引物扩增。反应体系:25 μL 2 × F8 Mix,2 μL F1,2 μL R1,模板2 μL,19 μL ddH2O。PCR反应条件:95 ℃预变性5 min;循环参数为94 ℃ 30 s,54 ℃ 30 s,72 ℃ 10 s,共35个循环;72 ℃延伸10 min,20 g·L-1琼脂糖凝胶电泳检测。用DNA纯化试剂盒纯化PCR产物、回收扩增片段,并测序鉴定。
|
|
表 1 PCR引物序列设计及用途 Table 1 Design and application of PCR primers |
收集培养的PK-15细胞,用RIPA裂解液裂解细胞,提取细胞总蛋白。M280链霉亲和素磁珠与上述生物素标记的DNA探针室温结合30 min,以无生物素标记的DNA探针-磁珠为阴性对照组。向磁珠-DNA混合物中加入细胞裂解液(约含蛋白1 mg),4 ℃孵育过夜。洗涤磁珠-DNA探针-蛋白复合物,最后洗脱蛋白复合物,使用SDS-PAGE胶电泳分离蛋白条带并银染。从银染后的蛋白胶中小心切下胶条,充分洗涤后送深圳华大基因股份有限公司进行质谱鉴定。
1.4 LC-MS/MS蛋白质谱鉴定与数据处理蛋白胶条经酶解后抽提出肽段,将抽提好的肽段通过Thermo公司UltiMate 3000 UHPLC进行分离。使用串联质谱仪Q-Exactive HF X(Thermo Fisher Scientific,San Jose,CA)进行DDA(Data Dependent Acquisition)模式检测,随后使用软件Mascot v2.3.02进行数据库搜索。搜索引擎产生的结果使用Percolator进行预处理并重新打分,然后对输出的结果以谱图水平FDR ≤1%进行过滤,从而获得显著性鉴定的谱图和肽段列表。最后基于蛋白鉴定列表,选择特异性肽段≥2的蛋白,使用DAVID在线基因功能注释数据库(DAVID, v6.7, https://david.ncifcrf.gov/summary.jsp)对蛋白进行GO(Gene ontology)分析及KEGG(Kyoto encyclopedia of genes and genomes)通路富集分析,Dr.Tom在线分析系统(https://biosys.bgi.com/)进行数据可视化绘图,利用STRING(https://string-db.org/cgi/input.pl)在线数据库对蛋白与蛋白之间相互作用(protein protein interaction,PPI)进行分析,将PPI数据导入Cytoscape软件绘图。
1.5 真核质粒的构建以PK-15总RNA为模板,使用一步法RT-PCR试剂盒扩增细胞蛋白PARP1基因,引物见表 1;以PCV2 DNA为模板,使用表 1中的特异性引物扩增病毒复制蛋白Rep基因。将p3×FLAG-CMV-14、pCAGGS-HA空载体分别与纯化后的宿主蛋白PARP1及病毒蛋白Rep基因分别双酶切(所用酶见表 1),将酶切后经纯化回收的目的片段分别与载体连接,构建p3×FLAG-CMV-PARP1及pCAGGS-HA-Rep真核表达质粒。将重组真核质粒进行双酶切鉴定及测序验证,保存对应质粒和菌液备用。
1.6 DNA免疫共沉淀(DNA-Immunoprecipitation, DNA IP)将带有Flag标签的PARP1蛋白真核表达质粒转染PK-15细胞24 h后,按1 MOI的接毒剂量感染PCV2 WH株。37 ℃培养2 h后,PBS洗3遍,换含2%血清的培养基继续培养36 h后收细胞。用RIPA弱裂解液将细胞进行裂解,收集细胞总蛋白,将细胞总蛋白分成试验组与对照组两份,试验组加入鼠源Flag标签抗体磁珠,对照组加入提前结合鼠源IgG的Protein A/G磁珠,4 ℃摇转孵育过夜。磁珠-DNA-蛋白复合物经PBS洗涤3遍后,70 ℃洗脱30 min,DNase Ⅰ 37 ℃处理30 min,使用病毒DNA提取试剂盒提取复合物中剩余DNA,使用表 1中引物Ori-IP通过qPCR方法对PCV2基因组Ori片段进行定量[12]。
1.7 蛋白质免疫共沉淀(Co-Immunoprecipitation, Co-IP)293T细胞汇合度至80%,同时转染两种带不同标签的真核质粒,36 h后,用RIPA裂解液裂解细胞,提取细胞总蛋白,分为两份,分别加入带有Flag或HA蛋白标签抗体的磁珠。4 ℃孵育4 h后,洗涤磁珠-蛋白复合物,最后将磁珠上结合的蛋白洗脱下来,使用SDS-PAGE胶电泳分离蛋白条带,并用与磁珠上标记不同的另一种蛋白标签抗体进行Western blot。
1.8 过表达或沉默PARP1蛋白对PCV2复制的影响PK-15细胞转染PARP1蛋白真核质粒,或通过转染siRNA沉默PARP1蛋白表达,36 h后感染PCV2病毒1 MOI,使用SYBR real-time PCR试剂盒将细胞提取的RNA反转为cDNA进行qPCR,qCap引物检测病毒DNA的拷贝数,PCR体系:5 μL 2×SYBR Green PCR Master Mix,0.5 μL上下游引物,3 μL cDNA,1.5 μL ddH2O。PCR程序:95 ℃ 15 s,60 ℃ 1 min,共40个循环。
1.9 统计学分析应用统计学分析软件计算各组数据的平均值(x)和标准差(s),各组数据以“x±sx”表示,并用t检验分析各组数据的均数差异显著性。*. P < 0.05表示差异显著;**. P < 0.01表示差异极显著;***. P < 0.001表示差异非常显著。
2 结果 2.1 DNA-protein pull down筛选与Ori互作的宿主蛋白使用生物素标记的引物对Ori序列进行扩增,20 g·L-1琼脂糖凝胶电泳分离片段(图 1),结果显示,扩增到与目的片段大小一致的序列(139 bp),测序结果显示,与PCV2-WH株Ori序列一致(GenBank: AF027217)。随后使用纯化后的片段进行DNA-protein pull down试验以及银染(图 2)。结果显示,生物素标记双链Ori组(Biotin-dsOri)以及生物素标记单链Ori组(Biotin-ssOri)与无生物素标记Ori组(Ori)相比,存在肉眼可见的7条差异条带。
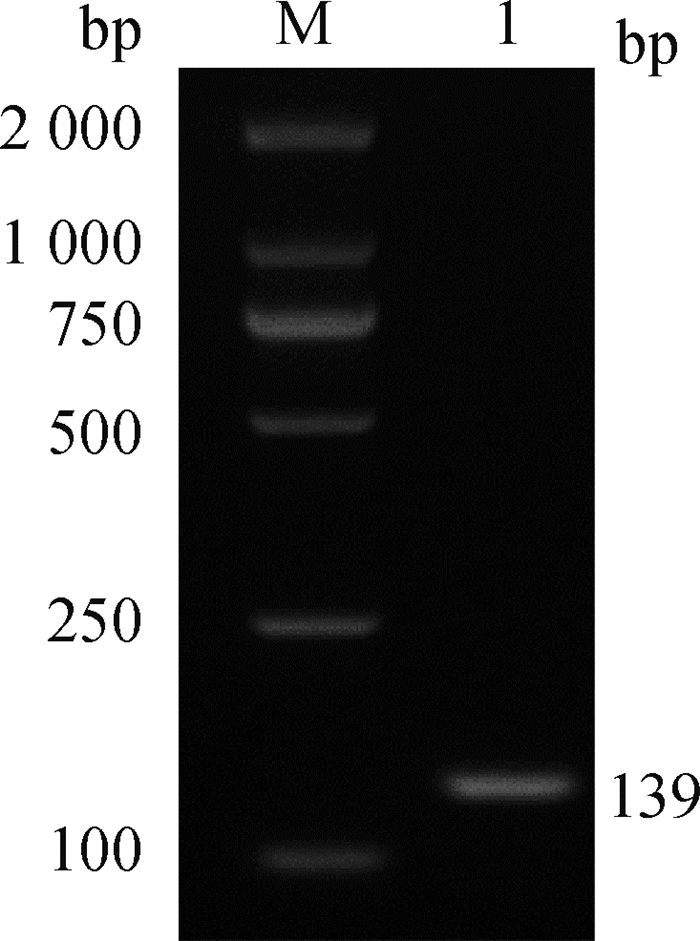
|
M. DNA相对分子质量标准;1. 生物素标记的双链Ori DNA M. DNA marker; 1. Biotin labeled double strand Ori DNA 图 1 PCV2基因组Ori片段的扩增 Fig. 1 Amplification of PCV2 Ori DNA by PCR |
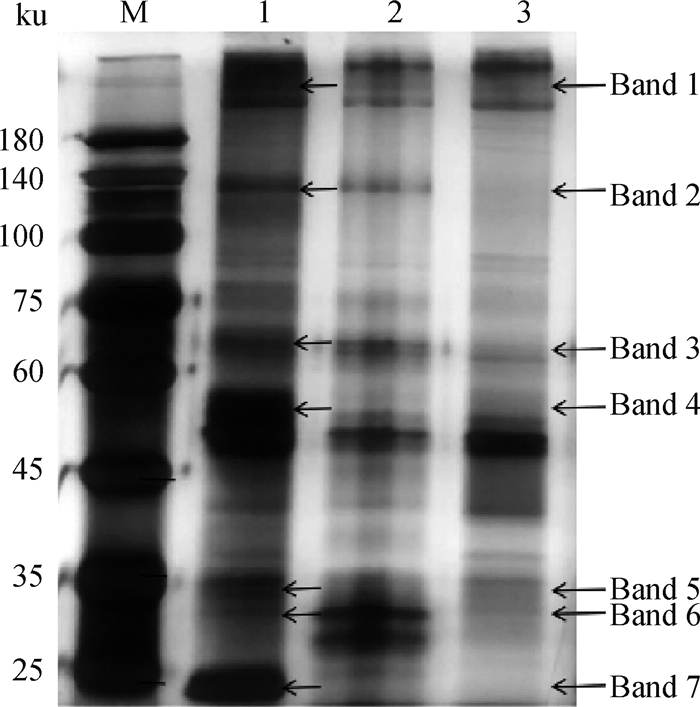
|
M. 蛋白质相对分子质量标准;1. 生物素标记双链Ori;2. 生物素标记单链Ori;3. 无标记Ori M. Protein marker; 1. Biotin-dsOri; 2. Biotin-ssOri; 3. Ori 图 2 DNA-protein pull down筛选Ori结合蛋白 Fig. 2 Screening of Ori binding proteins by DNA-protein pull down assay |
复制起始,PCV2基因组从单链变为双链,因此本研究重点关注生物素标记双链Ori组互作的宿主蛋白。将图 2中的1、3泳道分别切下,加入超纯水,进行质谱鉴定。根据质谱结果,生物素标记双链Ori组共得到谱图 42 607张,通过搜索引擎完成鉴定后,匹配到的谱图有3 501张,共鉴定到621个蛋白,2 552个肽段。无标记Ori组得到谱图 42 579张,通过搜索引擎完成鉴定后,匹配到的谱图共3 638张,鉴定到864个蛋白,3 006个肽段。去除各组中鉴定到的蛋白中特异性肽段数≤2的蛋白后,使用Dr. Tom在线分析软件绘制两组鉴定蛋白的韦恩图(图 3),生物素标记双链Ori鉴定到互作蛋白242个,除去与无标记双链Ori有交集的112个蛋白后,共鉴定到130个互作蛋白。
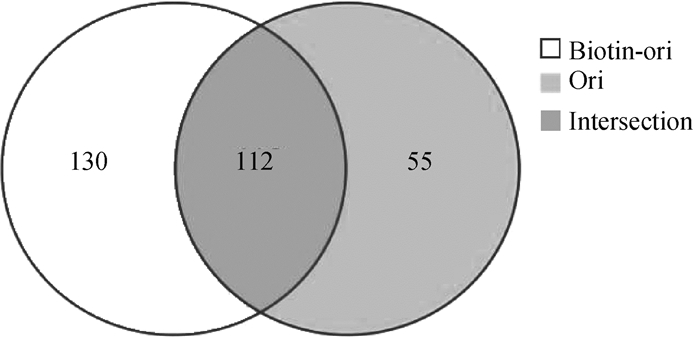
|
图 3 LC-MS/MS胶条蛋白韦恩图 Fig. 3 Identification of protein Venn diagram by LC-MS/MS |
GO分析显示,筛选到的蛋白主要位于细胞核内,参与DNA复制及损伤修复等细胞过程(图 4),其中,105种具有蛋白结合功能,45种具有DNA结合功能。随后进行KEGG通路富集分析,将Q值小于0.05的功能分类列出(表 2),互作蛋白主要涉及错配修复、DNA复制、RNA聚合酶等通路;随后,对鉴定到的互作蛋白根据互作评分绘制蛋白互作网络图(图 5),越靠近圆心的蛋白在整个互作网络中有更高的互作关系。在该图中,共有511条蛋白相互作用,最内圈的7个蛋白,每个蛋白与其他蛋白之间的相互作用数目都超过20个。
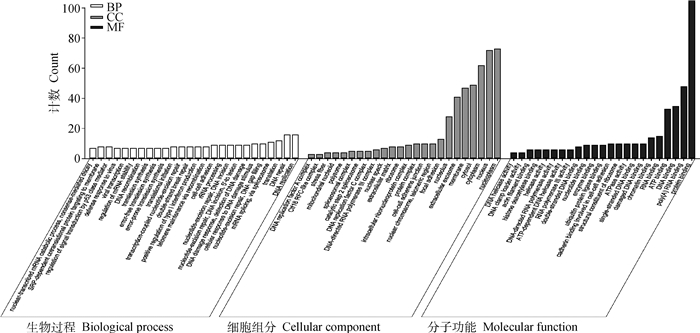
|
图 4 蛋白质的GO功能分类 Fig. 4 Functional classification of proteins based on gene ontolog |
|
|
表 2 PCV2 Ori互作蛋白的通路分析 Table 2 Pathway analysis of PCV2 Ori interacting proteins |

|
圆形代表蛋白质,黑色的线条表示蛋白之间存在互作关系,颜色由浅到深的渐变表示蛋白互作数递增(彩图中为黄到红,扫描文章首页OSID码可查看彩图) The ellipses represent proteins, the black lines represent interactions between proteins, and the gradient from light to dark indicates increasing interactions (In the color picture, the gradient was from yellow to red. The color picture can be found by scanning the OSID code on the front page of the article) 图 5 蛋白-蛋白互作网络(PPI) Fig. 5 Protein-protein interaction network |
选择质谱蛋白评分最高的PARP1蛋白进行进一步研究,为了研究PARP1蛋白与Ori的互作关系及是否参与病毒复制过程。分别构建PARP1蛋白及病毒Rep蛋白的真核质粒。对构建好的重组真核表达质粒进行双酶切鉴定,结果显示,除载体条带外,分别切出与PARP1(3 057 bp)、Rep(945 bp)基因大小一致的目的片段(图 6)。质粒经测序,与目标序列一致,说明p3×FLAG-CMV-PARP1、pCAGGS-HA-Rep真核表达质粒构建成功。
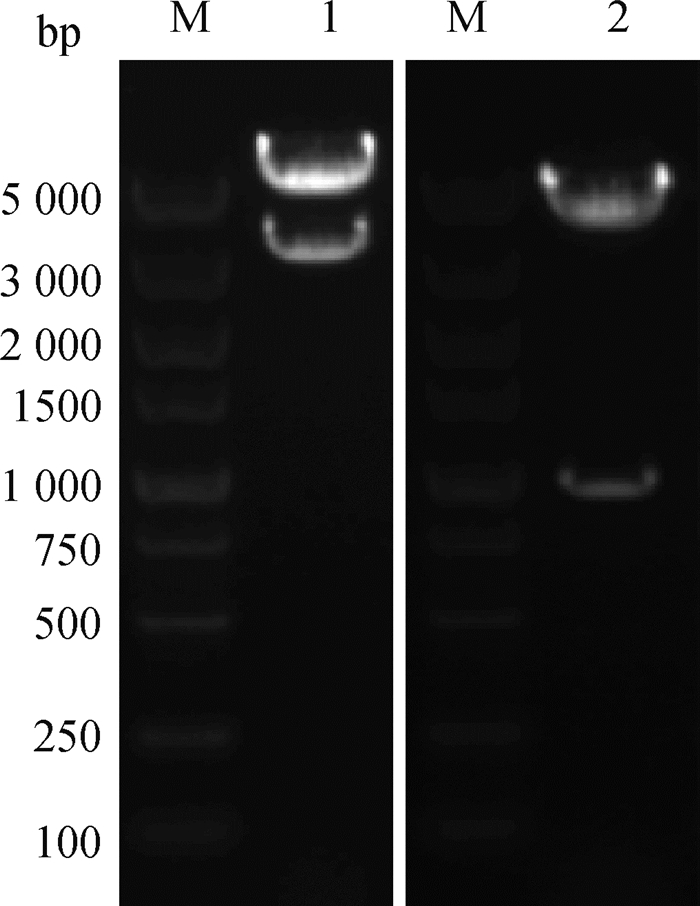
|
M. DNA相对分子质量标准;1. 重组载体p3×FLAG-CMV-PARP1经HindⅢ和XbaⅠ双酶切;2. 重组载体pCAGGS-HA-Rep经EcoRⅠ和XhoⅠ双酶切 M. DNA marker; 1. Recombinant plasmid p3×FLAG-CMV-PARP1 digested by HindⅢ and XbaⅠ; Recombinant plasmid pCAGGS-HA-Rep digested by EcoRⅠ and XhoⅠ 图 6 重组表达载体p3×FLAG-CMV-PARP1、pCAGGS-HA-Rep的双酶切鉴定 Fig. 6 Identification of the recombinant plasmid of p3×FLAG-CMV-PARP1, pCAGGS-HA-Rep by enzymes digestion |
为进一步验证PARP1蛋白与PCV2 Ori的互作,用DNA-protein pull down方法钓取与PCV2 Ori DNA互作的细胞蛋白,用PARP1蛋白抗体进行Western blot,结果显示,生物素标记Ori组有PARP1蛋白条带,而无标记组没有PARP1蛋白条带(图 7),证明PCV2 Ori在细胞外能够与PARP1发生相互作用。
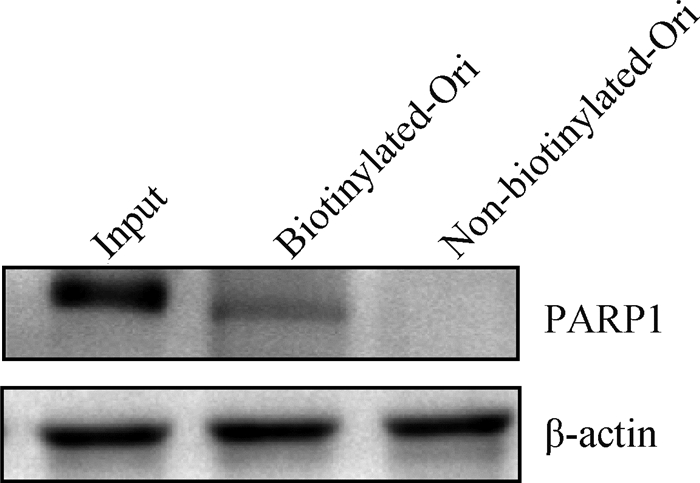
|
图 7 DNA pull down验证PARP1与Ori互作 Fig. 7 DNA pull down assay confirmed the interaction between PARP1 and Ori |
同时,在细胞中过表达PARP1蛋白后感染PCV2,使用带有蛋白标签抗体的磁珠与上述细胞的裂解液进行孵育,经过洗脱过程以及DNase Ⅰ处理后,检测磁珠-蛋白复合物中结合的Ori的量,结果显示,与同种属IgG抗体磁珠组相比,Flag标签抗体磁珠组有更强的Ori序列结合能力,差异非常显著(P < 0.001)(图 8)。上述试验结果表明,PARP1蛋白在细胞内与PCV2基因组Ori相互作用。
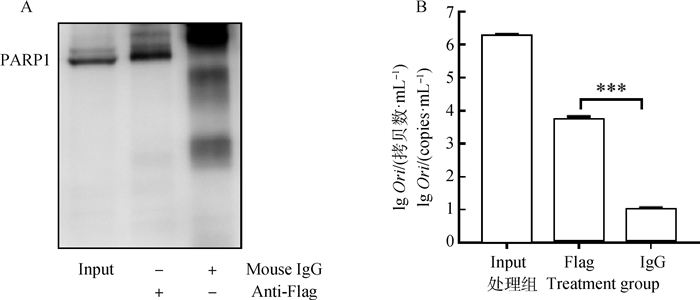
|
A. WB检测样品中PARP1蛋白;B. qPCR检测Ori DNA片段。***.P < 0.001 A. PARP1 protein in samples was detected by WB; B. qPCR was used to detect Ori DNA fragments. ***.P < 0.001 图 8 DNA IP试验验证PARP1与Ori互作 Fig. 8 DNA IP assay confirmed the interaction between PARP1 and Ori |
为阐明PARP1蛋白与病毒复制相关蛋白Rep的相互作用情况,在HEK293T细胞中分别共转染Rep-HA和PARP1-Flag真核表达质粒,转染后分别使用两种标签抗体的磁珠进行Co-IP试验。如图 9所示,当使用HA标签抗体进行IP时,Rep蛋白因为相互作用可将PARP1蛋白从裂解液中拉出;当使用Flag标签抗体进行IP时,PARP1蛋白因为相互作用可将Rep蛋白从裂解液中结合出来,结果显示,PCV2 Rep蛋白与PARP1蛋白存在相互作用。
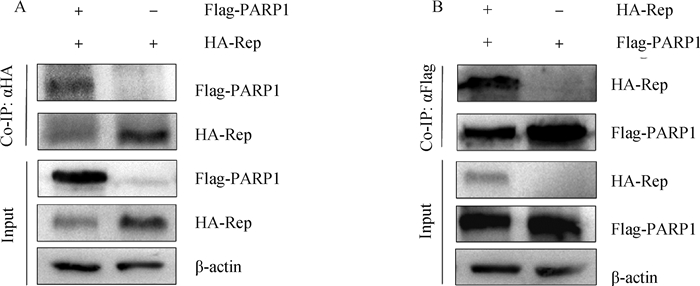
|
A. HA标签抗体Co-IP;B. Flag标签抗体Co-IP A. Co-IP with HA-labeled antibody; B. Co-IP with Flag-labeled antibody 图 9 Co-IP试验验证PARP1与PCV2 Rep蛋白互作 Fig. 9 Co-IP assay verified the interaction between PARP1 and PCV2 Rep protein |
将针对PARP1蛋白的siRNA转染细胞后,发现该siRNA可成功抑制PARP1蛋白的表达(图 10),PCV2感染后,用qPCR方法检测细胞内PCV2基因组拷贝数的变化,结果显示,当PARP1蛋白表达下降,PCV2在细胞内的基因组拷贝数也下降,差异非常显著(P < 0.001)(图 10)。过表达PARP1蛋白后,细胞内PCV2基因组拷贝数增高,差异非常显著(P < 0.001)(图 10)。上述结果说明,PARP1蛋白可促进PCV2在细胞内的复制。
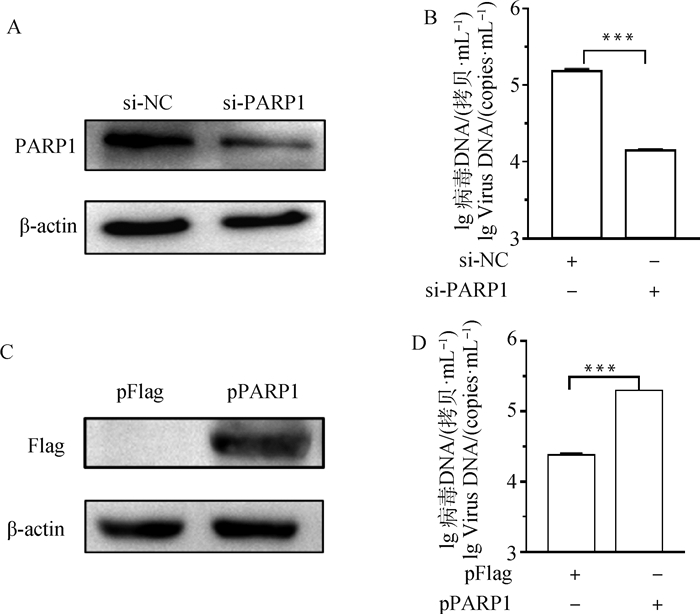
|
A、B. PARP1沉默抑制病毒复制;C、D. PARP1过表达促进病毒复制。***.P < 0.001 A, B. PARP1 silencing inhibits virus replication; C, D. Overexpression of PARP1 promotes viral replication. ***.P < 0.001 图 10 PARP1蛋白过表达或沉默对PCV2复制的影响 Fig. 10 Effects of overexpression or knockdown of PARP1 protein on PCV2 replication |
作为已知能够感染哺乳动物的最小的DNA病毒,PCV2基因组小且结构紧凑,编码能力非常有限。由于自身缺乏酶类,PCV2的生命周期在很大程度上依赖宿主细胞[13-14]。在过去几年中,基于酵母双杂交技术,许多蛋白被鉴定与PCV2基因组DNA、Rep/Rep′、Cap和ORF3蛋白存在相互作用[5, 15-16]。目前,关于PCV2感染引起的蛋白组学、转录组学变化研究较多[17-18]。研究发现,氧化应激、未折叠蛋白反应/内质网(ER)应激和PCV2诱导的自噬可促进病毒复制[19-21]。
PCV2基因组复制起始区Ori序列高度保守,其包括一段茎环结构区域,茎环部分九聚核苷酸中的AT之间可被病毒Rep蛋白识别并切割,从而起始病毒的复制[3, 14]。张辉等[22]基于酵母单杂交技术,鉴定到Elf4、AP2α等蛋白可结合到PCV2基因组茎环结构上。Sun等[12]发现PK-15细胞中具有DNA修复功能的HMGB1蛋白可以与PCV2基因组Ori进行结合,显著抑制病毒的复制。本研究旨在从宿主细胞与Ori在分子水平上的相互作用出发,对病毒复制起始过程中可能参与的宿主因素进行筛选,为进一步开展该病毒复制的研究奠定基础。本研究使用DNA-protein pull down结合LC-MS/MS的试验方法,共鉴定到130个可能与PCV2 Ori互作的宿主蛋白,大多与DNA的复制与修复相关。在对这些蛋白进行GO功能分类时,发现大多数蛋白定位于细胞核,这与PCV2基因组复制的位点一致。KEGG通路富集分析发现,宿主蛋白主要参与遗传信息处理过程,这与作者的预期相符,因为在该分类下的蛋白质大多具有核酸结合能力,其中,参与复制与修复功能、翻译及转录功能的蛋白数量是蛋白功能分类中最多的。但是也有部分蛋白没有DNA结合功能,推测这类蛋白是因为与DNA结合蛋白有相互作用而被结合下来。研究表明,DNA修复通路与先天免疫反应的调动以及抗病毒反应有密切关系[23]。在甲型流感病毒感染过程中,DNA错配修复通路的上调强烈诱导细胞内抗病毒基因的表达,从而清除病原体感染[24]。
根据与Ori互作的宿主蛋白绘制的互作网络图发现,FEN1、WRN、XRCC6、RPL8、RFC4、PARP1、PCNA与其他蛋白的互作数目均超过20个,除RPL8蛋白外,其余蛋白均具有遗传物质损伤修复功能,提示遗传物质损伤修复通路可能参与PCV2基因组的复制起始。PCNA与RFC作为DNA复制与损伤修复的关键因子,也是DNA病毒猿猴病毒(VP40)在人源细胞中复制的关键因子[25];多种病毒聚合酶发挥功能依赖于PARP1的活性,研究表明,沉默PARP1蛋白后,细胞培养释放的甲型流感病毒滴度降低超过50%[26-27]。
DNA-protein pull down及DNA IP试验均验证了PARP1具有结合PCV2 Ori的能力。病毒基因组中携带PARP1结合基序“ACTTCAAA”,可被PARP1识别并激活转录[28]。人类T细胞白血病病毒(HTLV)[29]和卡波西肉瘤相关病毒(KSHV)复制过程中也存在病毒DNA与PARP1蛋白的相互作用[30-31]。Sun等[12]研究发现,HMGB1蛋白与PCV2 Ori紧密结合抑制了PCV2的复制。本研究结果显示,PARP1蛋白可结合PCV2 Ori并促进PCV2基因组的复制。两种蛋白因功能不同,在PCV2复制起始的过程中的作用也截然不同。为研究PARP1蛋白与病毒复制复合体Rep-Rep′在Ori上的结合有无关联,使用Co-IP试验进行了PARP1蛋白与Rep蛋白的互作研究,结果表明,PARP1蛋白与PCV2 Rep蛋白有相互作用。PARP1蛋白可以对多种蛋白进行翻译后修饰,在DNA损伤修复过程中PARP1蛋白可以作为平台招募DNA损伤修复蛋白到DNA损伤位点[32-33],推测在PCV2 Ori位点,PARP1蛋白可招募Rep蛋白,并增强其对Ori的识别、切割,从而促进PCV2基因组的复制。关于PARP1蛋白如何促进Rep对PCV2 Ori的识别与切割需进一步研究。
4 结论本研究鉴定到130个可能与PCV2 Ori互作的宿主蛋白,主要涉及宿主DNA损伤修复及DNA复制功能。成功构建了宿主蛋白PARP1及病毒蛋白Rep真核表达质粒,验证了PARP1蛋白与PCV2基因组Ori区域互作,并证明PARP1蛋白促进PCV2的复制,为进一步研究PCV2在细胞中的复制起始过程奠定了基础。
| [1] |
OUYANG T, ZHANG X W, LIU X H, et al. Co-Infection of swine with porcine circovirus type 2 and other swine viruses[J]. Viruses, 2019, 11(2): 185. DOI:10.3390/v11020185 |
| [2] |
KARUPPANNAN A K, OPRIESSNIG T. Porcine circovirus type 2 (PCV2) vaccines in the context of current molecular epidemiology[J]. Viruses, 2017, 9(5): 99. DOI:10.3390/v9050099 |
| [3] |
TURLEWICZ-PODBIELSKA H, AUGUSTYNIAK A, POMORSKA-MÓL M. Novel porcine circoviruses in view of lessons learned from porcine circovirus type 2-epidemiology and threat to pigs and other species[J]. Viruses, 2022, 14(2): 261. DOI:10.3390/v14020261 |
| [4] |
LV Q Z, GUO K K, ZHANG Y M. Current understanding of genomic DNA of porcine circovirus type 2[J]. Virus Genes, 2014, 49(1): 1-10. DOI:10.1007/s11262-014-1099-z |
| [5] |
TIMMUSK S, FOSSUM C, BERG M. Porcine circovirus type 2 replicase binds the capsid protein and an intermediate filament-like protein[J]. J Gen Virol, 2006, 87(Pt 11): 3215-3223. |
| [6] |
MEEHAN B M, CREELAN J L, MCNULTY M S, et al. Sequence of porcine circovirus DNA: affinities with plant circoviruses[J]. J Gen Virol, 1997, 78(Pt 1): 221-227. |
| [7] |
MANKERTZ A, PERSSON F, MANKERTZ J, et al. Mapping and characterization of the origin of DNA replication of porcine circovirus[J]. J Virol, 1997, 71(3): 2562-2566. DOI:10.1128/jvi.71.3.2562-2566.1997 |
| [8] |
STEINFELDT T, FINSTERBUSCH T, MANKERTZ A. Demonstration of nicking/joining activity at the origin of DNA replication associated with the rep and rep' proteins of porcine circovirus type 1[J]. J Virol, 2006, 80(13): 6225-6234. DOI:10.1128/JVI.02506-05 |
| [9] |
KIM C, WANG X D, YU Y H. PARP1 inhibitors trigger innate immunity via PARP1 trapping-induced DNA damage response[J]. Elife, 2020, 9: e60637. DOI:10.7554/eLife.60637 |
| [10] |
KO H L, REN E C. Novel poly (ADP-ribose) polymerase 1 binding motif in hepatitis B virus core promoter impairs DNA damage repair[J]. Hepatology, 2011, 54(4): 1190-1198. DOI:10.1002/hep.24502 |
| [11] |
NA T Y, KA N L, RHEE H, et al. Interaction of hepatitis B virus X protein with PARP1 results in inhibition of DNA repair in hepatocellular carcinoma[J]. Oncogene, 2016, 35(41): 5435-5445. DOI:10.1038/onc.2016.82 |
| [12] |
SUN R J, SUN S Q, ZHANG Y K, et al. PCV2 induces reactive oxygen species to promote nucleocytoplasmic translocation of the viral DNA binding protein HMGB1 to enhance its replication[J]. J Virol, 2020, 94(13): e00238-20. |
| [13] |
CHEUNG A K. Transcriptional analysis of porcine circovirus type 2[J]. Virology, 2003, 305(1): 168-180. DOI:10.1006/viro.2002.1733 |
| [14] |
CHEUNG A K. Porcine circovirus: transcription and DNA replication[J]. Virus Res, 2012, 164(1-2): 46-53. DOI:10.1016/j.virusres.2011.10.012 |
| [15] |
FINSTERBUSCH T, STEINFELDT T, DOBERSTEIN K, et al. Interaction of the replication proteins and the capsid protein of porcine circovirus type 1 and 2 with host proteins[J]. Virology, 2009, 386(1): 122-131. DOI:10.1016/j.virol.2008.12.039 |
| [16] |
MANKERTZ A. Molecular interactions of porcine circoviruses type 1 and type 2 with its host[J]. Virus Res, 2012, 164(1-2): 54-60. DOI:10.1016/j.virusres.2011.11.001 |
| [17] |
ZHANG X, ZHOU J Y, WU Y P, et al. Differential proteome analysis of host cells infected with porcine circovirus type 2[J]. J Proteome Res, 2009, 8(11): 5111-5119. DOI:10.1021/pr900488q |
| [18] |
HE J, LENG C L, PAN J Z, et al. Identification of lncRNAs involved in PCV2 infection of PK-15 cells[J]. Pathogens, 2020, 9(6): 479. DOI:10.3390/pathogens9060479 |
| [19] |
CHEN X X, REN F, HESKETH J, et al. Interaction of porcine circovirus type 2 replication with intracellular redox status in vitro[J]. Redox Rep, 2013, 18(5): 186-192. DOI:10.1179/1351000213Y.0000000058 |
| [20] |
ZHU B L, ZHOU Y S, XU F, et al. Porcine circovirus type 2 induces autophagy via the AMPK/ERK/TSC2/mTOR signaling pathway in PK-15 cells[J]. J Virol, 2012, 86(22): 12003-12012. DOI:10.1128/JVI.01434-12 |
| [21] |
ZHOU Y S, QI B Z, GU Y X, et al. Porcine circovirus 2 deploys PERK pathway and GRP78 for its enhanced replication in PK-15 cells[J]. Viruses, 2016, 8(2): 56. DOI:10.3390/v8020056 |
| [22] |
张辉, 唐青海, 危艳武, 等. 与猪圆环病毒2型基因组茎环结构相互作用的宿主细胞蛋白的筛选[J]. 中国兽医科学, 2013, 43(12): 1211-1216. ZHANG H, TANG Q H, WEI Y W, et al. Screening of host proteins interacting with porcine circovirus type 2 genome stem-loop structure[J]. Chinese Veterinary Science, 2013, 43(12): 1211-1216. (in Chinese) |
| [23] |
ZHU H F, ZHENG C F. When PARPs meet antiviral innate immunity[J]. Trends Microbiol, 2021, 29(9): 776-778. DOI:10.1016/j.tim.2021.01.002 |
| [24] |
CHAMBERS B S, HEATON B E, RAUSCH K, et al. DNA mismatch repair is required for the host innate response and controls cellular fate after influenza virus infection[J]. Nat Microbiol, 2019, 4(11): 1964-1977. DOI:10.1038/s41564-019-0509-3 |
| [25] |
OHASHI E, TSURIMOTO T. Functions of multiple clamp and clamp-loader complexes in eukaryotic DNA replication[M]//MASAI H, FOIANI M. DNA Replication. Singapore: Springer, 2017: 135-162.
|
| [26] |
MAYER D, MOLAWI K, MARTÍNEZ-SOBRIDO L, et al. Identification of cellular interaction partners of the influenza virus ribonucleoprotein complex and polymerase complex using proteomic-based approaches[J]. J Proteome Res, 2007, 6(2): 672-682. DOI:10.1021/pr060432u |
| [27] |
BORTZ E, WESTERA L, MAAMARY J, et al. Host- and strain-specific regulation of influenza virus polymerase activity by interacting cellular proteins[J]. Mbio, 2011, 2(4): e00151-11. |
| [28] |
HAGEN T M, HUANG S, CURNUTTE J, et al. Extensive oxidative DNA damage in hepatocytes of transgenic mice with chronic active hepatitis destined to develop hepatocellular carcinoma[J]. Proc Natl Acad Sci U S A, 1994, 91(26): 12808-12812. DOI:10.1073/pnas.91.26.12808 |
| [29] |
ZHANG Z, HILDEBRANDT E F, SIMBULAN-ROSENTHAL C M, et al. Sequence-specific binding of poly (ADP-ribose) polymerase-1 to the human T cell leukemia virus type-Ⅰ tax responsive element[J]. Virology, 2002, 296(1): 107-116. DOI:10.1006/viro.2002.1385 |
| [30] |
OHSAKI E, UEDA K, SAKAKIBARA S, et al. Poly (ADP-ribose) polymerase 1 binds to kaposi's sarcoma-associated herpesvirus (KSHV) terminal repeat sequence and modulates KSHV replication in latency[J]. J Virol, 2004, 78(18): 9936-9946. DOI:10.1128/JVI.78.18.9936-9946.2004 |
| [31] |
WANG Y, LI H, TANG Q Y, et al. Kaposi's sarcoma-associated herpesvirus ori-Lyt-dependent DNA replication: involvement of host cellular factors[J]. J Virol, 2008, 82(6): 2867-2882. DOI:10.1128/JVI.01319-07 |
| [32] |
KIM M Y, ZHANG T, KRAUS W L. Poly (ADP-ribosyl) ation by PARP-1:'PAR-laying' NAD+ into a nuclear signal[J]. Genes Dev, 2005, 19(17): 1951-1967. DOI:10.1101/gad.1331805 |
| [33] |
KRIETSCH J, ROULEAU M, PIC É, et al. Reprogramming cellular events by poly (ADP-ribose)-binding proteins[J]. Mol Aspects Med, 2013, 34(6): 1066-1087. DOI:10.1016/j.mam.2012.12.005 |
(编辑 孟培)



