羧肽酶B2(carboxypeptidase B2,CPB2),是凝血酶激活的纤溶抑制物(thrombin activatable fibrinolysis inhibitor,TAFI)的编码基因[1],故CPB2基因又称之为TAFI基因。TAFI前体酶原主要在肝中合成,含有423个氨基酸,在去除22个残基的信号肽后,以56 ku分子量的酶原(TAFI)分泌到血液循环中[2]。TAFI包含92个残基的(N末端)活化肽(Phe1-Arg92)和309个残基的催化结构域(Ala93-Val401)[3]。TAFI具有较低的羧肽酶酶原活性,其真正发挥羧肽酶活性需要利用胰蛋白酶样蛋白酶对Arg92-Ala93肽键切割去除激活肽后形成活化的TAFI(activated TAFI,TAFIa)[2, 4-5]。TAFIa是一种锌依赖性外肽酶,可切割羧基末端(赖氨酸)肽键[6-7]。目前,已经在蛋白质数据库(PDB)中在线发表了17个包含人、牛和猪的TAFI或TAFIa的结构[3]。
TAFIa主要被认为具有抗纤溶的作用,可参与凝血与纤溶、伤口愈合等过程;除上述作用外,其还具有一定的抗炎症作用,能够灭活炎症蛋白如过敏毒素C3a和C5a等[8-10]。TAFI异常与心血管疾病和血栓性疾病有关,例如有研究中Tafi敲除鼠未表现出可观察的明显表型,但在FeCl3诱导腔静脉血栓研究中,Tafi敲除鼠中形成的血栓显著减少,显示Tafi在血栓形成过程中存在重要作用[11-12]。也有研究探讨了TAFI编码基因CPB2的多态性与2型糖尿病的关系[13-14]。可见,TAFI紊乱与疾病密切相关,可被用作潜在的治疗靶点。
目前,关于人和鼠的CPB2基因报道较多,通过在NCBI数据库中检索CPB2基因信息,显示人类CPB2基因存在两种已验证转录本(NM_001278541.2和NM_001872.5)[3],一种预测转录本(XM_0170203932),对转录本之间进行比较发现存在外显子跳跃[15-16]和5′端可变剪接事件。目前,对于猪的CPB2基因及可变剪接的研究较少。前期本实验室巴马小型猪肝转录组测序显示,猪CPB2基因可能存在多种可变剪接体。本研究通过RT-PCR技术及基因平末端克隆技术扩增猪CPB2基因的CDS区及部分非编码区,利用生物信息学工具预测CPB2编码蛋白的基本生物学特征,进一步通过RT-PCR技术检测CPB2基因在肝、心、肾、皮下脂肪、肠系膜脂肪、股二头肌和背最长肌中的表达特征;以实验室制备的代谢性疾病易感猪(转PNPLA3I148M、GIPRdn和hIAPP基因猪[17])为试验动物,利用qRT-PCR技术检测在富营养饮食效应下的肝中CPB2基因的表达特征。本研究结果将为CPB2基因功能及转录调控提供理论参考。
1 材料与方法 1.1 试验材料本试验选用健康状态和体重相同的3头12月龄巴马小型猪,安乐死后收集肝、心、肾、皮下脂肪、肠系膜脂肪、股二头肌和背最长肌等组织样本;选用健康状态和体重相同的4头9月龄代谢性疾病易感猪(转PNPLA3I148M、GIPRdn和hIAPP基因猪)饲喂高脂高能量饲料(53%基础日粮、37%蔗糖和10%猪油)作为富营养饮食试验组(TG-HSHFD),4头9月龄代谢性疾病易感猪饲喂基础日粮作为对照组(TG-CD),试验期3个月,安乐死后收集肝等组织样本;采集的所有组织样品全部置于液氮内备用;所有巴马小型猪试验均经中国农业科学院北京畜牧兽医研究所实验动物伦理委员会许可(许可证号:IAS2019-12),单栏饲养,日饲2次,自由饮水,温度16~28 ℃,相对湿度40%~70%。
1.2 试剂TRIzol试剂(Invitrogen,美国);高保真酶2×Phanta Flash Master Mix(Vazyme,中国);50×TAE(Solarbio,中国);琼脂糖(Biowest,西班牙);DNA Mark试剂(Generay,中国);琼脂糖凝胶DNA试剂盒(Tianmobio,中国);5 min TA/Blunt-Zero cloning Kit载体克隆试剂盒(Vazyme,中国);DH5ɑ感受态(Vazyme,中国);PrimeScriptTM RT reagent Kit with gDNA Eraser反转录试剂盒和TB Green® Premix Ex TaqTM荧光定量试剂盒(TaKaRa,日本)等。
1.3 方法1.3.1 引物设计与合成 经NCBI数据库获取CPB2序列(登录号:XM_001929146.4)及基因组序列,利用NCBI Primer BLAST设计CPB2-1、CPB2-2及CPB2-3的全长扩增引物(上游引物Fq和下游引物Rq),根据基因克隆序列设计CPB2-1、CPB2-2及CPB2-3定量引物(上游引物Fd和下游引物Rd),内参基因为18S。引物序列(表 1)由北京擎科生物公司合成。
|
|
表 1 引物基本信息 Table 1 Basic information of primers |
1.3.2 组织总RNA提取及cDNA反转录 传统TRIzol法提取样品组织总RNA,利用1%浓度琼脂糖凝胶和Nano-100检测仪检测RNA完整性和RNA浓度;利用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒合成cDNA(-20 ℃储存)。
1.3.3 RT-PCR试验及基因克隆 以巴马小型猪肝组织cDNA作为模板,扩增CPB2基因序列,RT-PCR反应总体系为50 μL:2×Phanta Mix 25 μL,cDNA 2 μL,上游引物F 2 μL,下游引物R 2 μL,ddH2O补至50 μL。PCR反应程序:95 ℃预变性5 min;95 ℃变性15 s,60 ℃退火15 s,72 ℃延伸60 s,34个循环;72 ℃延伸5 min。反应产物用1.5%琼脂糖凝胶电泳检测,凝胶成像仪显影琼脂糖凝胶目的条带,编辑并保存图片。
利用琼脂糖凝胶DNA回收试剂盒对目的条带进行DNA纯化回收。利用5 min TA/Blunt-Zero cloning Kit试剂盒克隆目的片段,反应总体系为5 μL:5×TA/Blunt-Zero Cloning Mix 1 μL,PCR纯化产物2 μL,ddH2O 2 μL。反应程序:37 ℃连接5 min。采用热应激法将连接产物转化至DH5α感受态细胞,用Amp抗性的固体培养基37 ℃培养12 h,挑取单菌落转入Amp抗性的液体培养基37 ℃培养2 h,菌液PCR检测阳性菌送至北京擎科生物公司双向测序。
1.3.4 qRT-PCR试验 利用TB Green® Premix Ex TaqTM荧光定量试剂盒检测CPB2基因表达量,qRT-PCR反应总体系为20 μL:SYBR Green 10 μL,ddH2O 6.8 μL,上游引物F 0.4 μL,下游引物R 0.4 μL,ROX dye II 0.4 μL,cDNA 2.0 μL。qRT-PCR反应程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃ 34 s,40个循环。每个样品重复2次,18S为内参基因,分析数据使用2-△△Ct法计算各目的基因的mRNA相对表达量。
1.3.5 生物信息学分析 利用Snapgene、DNAMAN 8软件比对基因序列和蛋白序列;利用Swiss-PdbViewer软件编辑蛋白质三维结构;利用Cytoscape软件绘制相关性网络图;其他在线工具如表 2。
|
|
表 2 在线工具基本信息 Table 2 Basic information of online tools |
试验数据经过SAS 9.1软件和GraphPad Prism 8软件进行t-test统计学分析,P < 0.05时认为具有统计学意义。
2 结果 2.1 猪CPB2基因可变剪接体的克隆前期肝转录组测序结果显示,猪CPB2基因可能存在3个不同的转录本,以其特异性区域设计包含CDS区的全长扩增引物(图 1A)。利用RT-PCR方法在肝组织中获得猪CPB2基因的3条不同转录本条带(图 1B)。通过载体pCE2 TA/Blunt-Zero与切胶回收片段连接克隆,筛选阳性菌测序,3种转录本序列长度(包括CDS区及部分非编码区)分别为1 445、1 344和1 268 bp(图 1C)。与数据库序列比对后,显示长片段转录本与NCBI中预测序列XM_001929146.4相同,命名为CPB2-1;中片段和短片段转录本,分别命名为CPB2-2和CPB2-3(已将扩增到的转录本CPB2-1、CPB2-2和CPB2-3序列上传至NCBI GenBank,登录号分别为ON088314、ON088315和ON088316)。
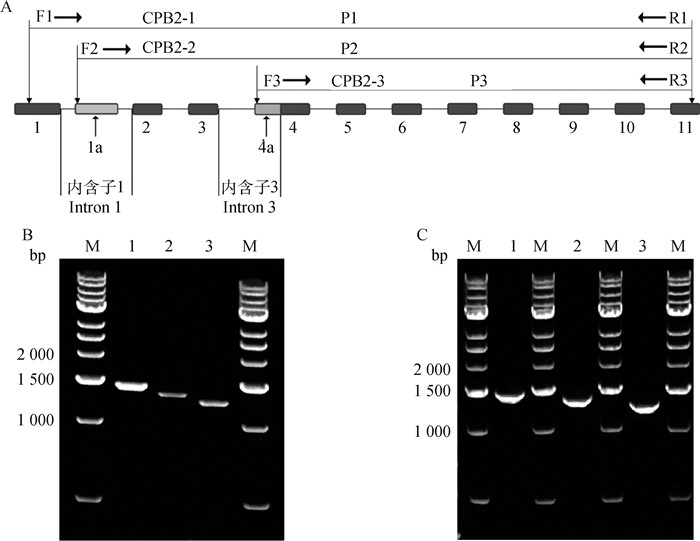
|
A.引物设计(F. 上游引物;R. 下游引物;1~11、1a、4a 为外显子序列号);B. RT-PCR 扩增结果(M. DNA 相对分子质量标准;1. 转录本 CPB2-1;2. 转录本 CPB2-2;3. 转录本 CPB2-3);C. 阳性菌鉴定结果(M. DNA 相对分子质量标准;1. 转录本 CPB2-1;2. 转录本 CPB2-2;3. 转录本 CPB2-3) A. Primer design drawing (F. Forward primer; R. Reverse primer; 1-11, 1a, 4a are exon numbers); B. Gel map amplified by RT-PCR (M. DNA Marker; 1. Transcript CPB2-1; 2. Transcript CPB2-2; 3. Transcript CPB2-3);C. Positive bacteria identification map (M. DNA Marker; 1. Transcript CPB2-1; 2. Transcript CPB2-2; 3. Transcript CPB2-3) 图 1 CPB2-1、CPB2-2和CPB2-3的克隆 Fig. 1 Cloning of CPB2-1, CPB2-2 and CPB2-3 |
将CPB2的3个转录本序列与CPB2基因组进行比对,结果显示,CPB2基因共有13个外显子,其中外显子5~11是3种转录本共有的;与转录本CPB2-1相比,转录本CPB2-2和CPB2-3的第一外显子组成不同,分别由外显子1a和外显子4a+4构成,即均发生A5SS事件(图 2A)。组成3种转录本的第一外显子与第二外显子交界碱基都为5′….AG/TG….3′(图 2B),其中,转录本与基因组比对显示CPB2-1外显子1后端剪接位点与外显子2前端剪接位点分别为G|G和G|T;CPB2-2外显子1a后端剪接位点和外显子2前端剪接位点G|G和G|T;CPB2-3外显子4a与外显子4之间的剪接位点为G|T。

|
A.转录本模式图(1~11、1a、4a 为外显子序列号);B. 转录本部分核苷酸序列比对图 A.Transcription pattern diagram (1-11, 1a, 4a are exon numbers); B. Nucleotide sequence alignment diagram of the transcript portion 图 2 CPB2-1、CPB2-2和CPB2-3的可变剪接分析 Fig. 2 Alternative splicing analysis of CPB2-1, CPB2-2 and CPB2-3 |
2.3.1 蛋白序列比对 经NCBI ORF预测,结果显示,CPB2-1的CDS区1 272 bp,其起始密码子为ATG,位于133 bp处,终止密码子为TAA,位于1 404 bp处,编码包含423个氨基酸的蛋白;CPB2-2的CDS区846 bp,其起始密码子为ATG,位于460 bp处,终止密码子为TAA,位于1 305 bp,编码包含281个氨基酸的蛋白;CPB2-3的CDS区均为846 bp,其起始密码子为ATG,位于383 bp处,终止密码子为TAA,位于1 228 bp处,编码包含281个氨基酸的蛋白。氨基酸序列比对发现,CPB2-2和CPB2-3编码同种蛋白;与CPB2-1蛋白相比,CPB2-2和CPB2-3蛋白缺失了前段的142个氨基酸(图 3)。

|
图 3 CPB2 蛋白序列比对 Fig. 3 Comparison of CPB2 protein sequences |
2.3.2 蛋白理化性质分析 ProtParam软件预测理化性质,结果显示,CPB2-1分子式为C2201H3350N584O635S14,相对分子质量为48 601.18,原子数6 784,理论等电点(PI)为6.83(< 7)属于酸性蛋白,带负电荷残基数(Asp+Glu)为48,带正电荷残基数(Arg+Lys)为46,消光系数为89 770, 预测其半衰期为30 h,不稳定系数为49.79(< 40)属不稳定蛋白,脂肪族氨基酸指数为82.51;而CPB2-2和CPB2-3分子式为C1478H2228N398O411S12,相对分子质量为32 533.09,理论等电点(PI)为8.91(>7)属于碱性蛋白,带负电荷残基数(Asp + Glu)为30,带正电荷残基数(Arg + Lys)为37,消光系数为69 705,预测其半衰期为30 h,不稳定系数为50.36,小于40属不稳定性蛋白,脂肪族氨基酸指数为72.92。ProtScale软件疏水性分析结果显示,CPB2-1蛋白疏水性最强的氨基酸是位于第12位的Ala(正值3.344),亲水性最强的氨基酸是第328位的Lys(负值3.056),总氨基酸中亲水性氨基酸占比达61.23%,氨基酸平均值-0.308 95,推测为亲水性蛋白(图 4C);CPB2-2和CPB2-3蛋白疏水性最强的氨基酸是第50位的Leu(正值1.700),亲水性最强的氨基酸是第186位的Lys(负值3.056),总氨基酸中亲水性氨基酸占比64.77%,氨基酸平均值-0.430 71,推测为亲水性蛋白(图 4D)。
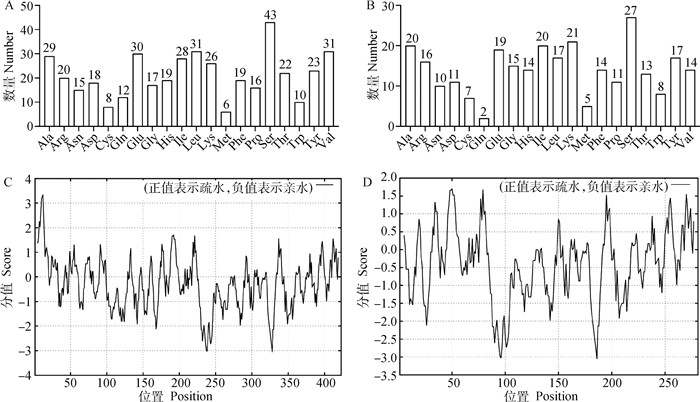
|
A.CPB2-1 蛋白氨基酸分布;B. CPB2-2 和 CPB2-3 蛋白氨基酸分布;C. CPB2-1 蛋白疏水性与亲水性预测;D. CPB2-2 和 CPB2-3 蛋白疏水性与亲水性预测 A. Amino acid distribution of CPB2-1 protein; B. Amino acid distribution of CPB2-2 and CPB2-3 proteins; C. Hydrophobicity and hydrophilicity prediction of CPB2-1 protein; D. Hydrophobicity and hydrophilicity prediction of CPB2-2 and CPB2-3 protein 图 4 CPB2 蛋白理化性质 Fig. 4 Physicochemical properties analysis of CPB2 protein |
2.3.3 蛋白信号肽预测 信号肽是新合成多肽链中用于指导蛋白质跨膜转运的短肽序列。通过在线工具SignalP 5.0预测信号肽,结果表明,CPB2-1蛋白存在一个22个氨基酸的信号肽(图 5A),可能属于分泌蛋白,可信度0.870 1;CPB2-2和CPB2-3蛋白不存在信号肽(图 5B),可能属于不分泌蛋白。
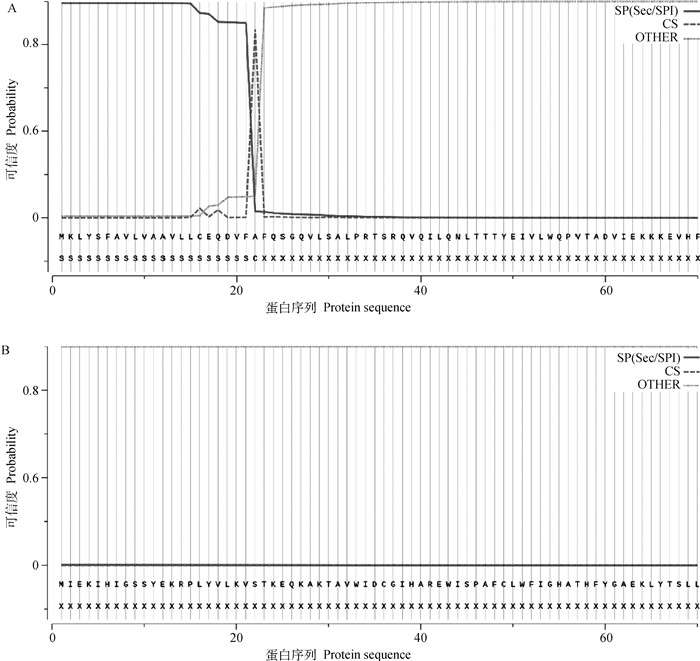
|
A.CPB2-1 蛋白信号肽预测;B. CPB2-2 和 CPB2-3 蛋白信号肽预测 A. Prediction of CPB2-1 protein signal peptide; B. Prediction of CPB2-2 and CPB2-3 protein signal peptide 图 5 CPB2 蛋白信号肽分析 Fig. 5 CPB2 protein signal peptide analysis |
2.3.4 蛋白保守结构域预测 NCBI保守结构域在线预测工具预测CPB2蛋白保守结构域,结果表明,CPB2-1的N端含有1个Propep_M14羧肽酶激活肽结构域;C端含有1个M14_CPB2羧肽酶活性功能域,由1个锌结合位点和活性位点组成(图 6A)。与CPB2-1相比,截断蛋白CPB2-2与CPB2-3缺少了Propep_M14羧肽酶激活肽结构域,但具有相同的M14_CPB2羧肽酶活性功能域,推测其羧肽酶活性功能正常,但是功能激活方式可能存在一定差异(图 6B)。

|
A.CPB2-1 蛋白保守结构域预测;B. CPB2-2 和 CPB2-3 蛋白保守结构域预测 A. Prediction of CPB2-1 protein conserved domain; B. Prediction of CPB2-2 and CPB2-3 proteins conserved domain 图 6 CPB2 蛋白保守结构域 Fig. 6 Conserved domain analysis of CPB2 protein |
2.3.5 蛋白二级结构与三级结构预测 利用SOPMA软件对CPB2蛋白进行二级结构预测,结果显示,CPB2-1、CPB2-2和CPB2-3的二级结构均主要由ɑ-螺旋(h)、延伸(e)、β-折叠(t)和无规则卷曲(c)组成,这4种二级结构在CPB2-1蛋白中占比分别为36.41%、19.15%、4.26%和40.19%,在CPB2-2和CPB2-3蛋白中占比分别为29.18%、22.78%、6.41%和41.64%(图 7A、7C)。SWISS-MODEL软件预测CPB2基因编码蛋白的三级结构,结果显示,CPB2-1蛋白主要由上、下两部分组成,其中,红色部分为信号肽和Propep_M14羧肽酶激活肽结构域,彩色部分为M14_CPB2羧肽酶活性功能域(图 7B);CPB2-2蛋白缺少红色部分结构,但是彩色部分与CPB2-1蛋白对应位置的三维结构相同(图 7D)。

|
A.CPB2-1 蛋白二级结构预测;B. CPB2-1 蛋白三级结构预测;C. CPB2-2 和 CPB2-3蛋白二级结构预测;D. CPB2-2 和 CPB2-3 蛋白三级结构预测 A. Prediction of secondary structure of CPB2-1 protein; B. Prediction of tertiary structure of CPB2-1 protein; C. Prediction of secondary structure of CPB2-2 and CPB2-3 proteins; D. Prediction of tertiary structure of CPB2-2 and CPB2-3 proteins 图 7 CPB2 蛋白二级与三级结构 Fig. 7 Secondary and tertiary structure analysis of CPB2 protein |
2.3.6 蛋白互作网络分析 利用String网站,选择物种猪(Sus scrofa),选择可信度0.7,对猪CPB2蛋白互作网络进行预测。结果显示,与CPB2具有互作关系的蛋白有SERPINC1、AHSG、F2、PROC、PLG、AGT、C8B、F7、CPE和DBT等(图 8)。对CPB2及其互作基因进行富集分析,GO、KEGG和REAC结果显示,其可能通过肽酶水解及肽酶活性调节,参与纤溶、补体与凝血级联和创伤反应等过程;HP结果显示其与凝血异常、静脉血栓和静脉炎等疾病存在一定关系(表 3)。
|
|
表 3 CPB2 互作相关基因富集分析 Table 3 Enrichment analysis of CPB2 interaction related genes |
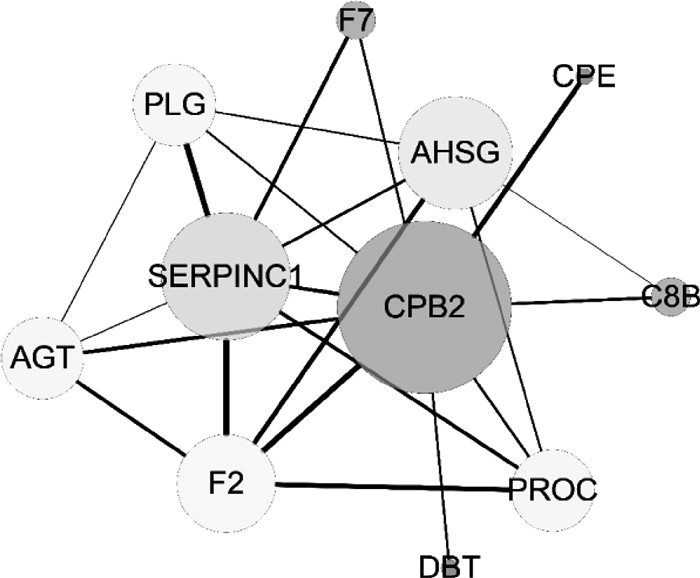
|
图 8 CPB2 蛋白互作网络 Fig. 8 Protein interaction network analysis of CPB2 |
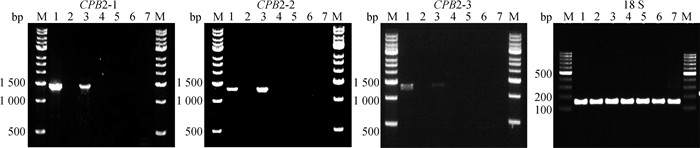
为研究CPB2的3个转录本在巴马小型猪不同组织内表达情况,利用RT-PCR检测其全长转录本表达分布。结果显示,CPB2-1、CPB2-2和CPB2-3只在肝和肾中表达,与CPB2-1和CPB2-2相比,CPB2-3表达量相对较低,可以看出CPB2基因的3种转录本存在明显的组织特异性,这与它的功能特点密切相关(图 9)。
|
M. DNA 相对分子质量标准;1. 肝;2. 心;3. 肾;4. 皮下脂肪;5. 肠系膜脂肪;6. 股二头肌;7. 背最长肌 M. DNA marker; 1. Liver; 2. Heart; 3. Kidney; 4. Subcutaneous fat; 5. Mesenteric fat; 6. Biceps femoris; 7. Longissimus dorsi 图 9 CPB2-1、CPB2-2和CPB2-3 全长转录本的组织表达谱 Fig. 9 Tissue expression profiles of the full-length transcripts of CPB2-1, CPB2-2 and CPB2-3 |
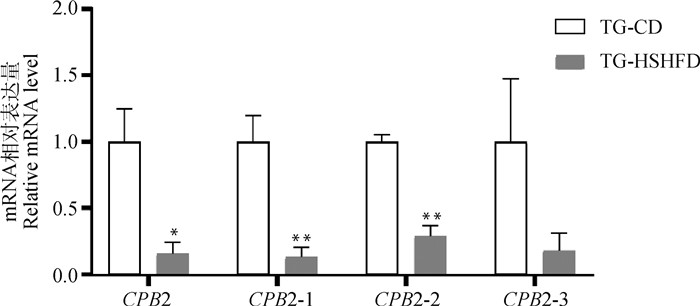
为研究富营养饮食下代谢性疾病易感猪肝中CPB2基因的表达模式,在猪CPB2基因的3个转录本共有区域设计引物检测CPB2基因总表达量,在3个转录本特异区域设计定量引物检测CPB2-1、CPB2-2和CPB2-3的表达量。结果显示,CPB2基因总表达量显著降低(P < 0.05);CPB2-1和CPB2-2两种转录本极显著降低(P < 0.01);转录本CPB2-3表达量降低,但未达到显著水平(P=0.14,图 10)。
|
*. P < 0.05;**. P < 0.01 图 10 CPB2 基因及3种转录本的 qRT-PCR 验证 Fig. 10 qRT-PCR validation of CPB2 gene and 3 transcripts |
可变剪接(alternative splicing,AS)现象的描述最早出现在20世纪70年代[18-19],是指一个基因的pre-mRNA通过不同的内含子及外显子拼接方式形成多个成熟mRNA的过程。动植物体内发生的可变剪接类型可分为8种,但是常见的可变剪接类型主要分为5种:外显子跳跃(skipped exon,SE)、外显子互斥(mutually exclusive exon,MXE)、内含子保留(retained intron,RI)、5′端可变剪接(alternative 5′splice site,A5SS)和3′端可变剪接(alternative 3′splice site,A3SS)[20-21]。CPB2是凝血酶激活的纤溶抑制物(TAFI)的编码基因,主要具有抗纤溶的作用,可参与凝血与纤溶、伤口愈合、抗炎等过程,也有研究表明其与代谢性疾病(如2型糖尿病、心血管疾病)等存在一定关系[11-14]。在本实验室前期富营养饮食对代谢性疾病易感猪肝组织可变剪接影响的研究中,筛选到差异剪接基因CPB2,富集分析显示,CPB2基因显著富集到与代谢相关的通路中,由代谢相关差异剪接基因的蛋白互作网络分析筛选出的枢纽基因也包括CPB2基因,显示出CPB2基因可能在代谢性疾病中的重要性。本研究通过转录组测序和RT-PCR技术发现了猪CPB2的3个可变剪接体CPB2-1、CPB2-2和CPB2-3,三者后7个外显子为3种转录本共有;与CPB2-1相比,CPB2-2和CPB2-3发生了A5SS事件,在NCBI数据库中人CPB2基因中也观察到可能存在A5SS事件,说明人与猪之间可能存在一定保守的剪接模式,这与之前猪C1QTNF3基因在物种间存在相同剪接模式的研究结果一致[22]。
大约75%的可变剪接事件出现在可变剪接体的编码蛋白区,其对应蛋白一级结构、二级结构和三级结构可能发生改变,进而可影响其结合特性、酶活性、细胞内定位、蛋白稳定性和表观修饰等多个方面[23-25]。经NCBI工具预测,CPB2-1的CDS区为1 272 bp,编码423个氨基酸的蛋白质;由于5′端可变剪接事件的存在,CPB2-2和CPB2-3的翻译起始位点发生改变,CDS区变为846 bp,编码281个氨基酸的蛋白质,两者相差142个氨基酸。经理化性质预测显示,CPB2-1、CPB2-2和CPB2-3半衰期相同且均为不稳定性蛋白,这与之前研究报道蛋白质的半衰期和其稳定性有密切关系的结果一致[26],也与报道中人CPB2蛋白存在自身热不稳定性的结果一致[3, 27-28],这种不稳定性被认为是在抗纤溶活性的自动调节中起作用。
有研究表明,人CPB2蛋白在肝中通过信号肽去除进入血液,在血中去除激活肽后,才能正常发挥羧肽酶功能[3]。根据信号肽和保守结构域预测显示,CPB2-1蛋白因包含信号肽、激活肽和羧肽酶活性功能域,推测其可以行使正常生理功能。CPB2-2蛋白和CPB2-3蛋白因天然缺少信号肽和激活肽,但具有羧肽酶活性功能域,推测其被保留在肝中发挥羧肽酶功能,这两种截断蛋白在肝中发挥羧肽酶活性的高低以及是否行使与完整蛋白相同的生理学功能还需进一步研究。蛋白质二级结构和三级结构预测显示,CPB2-1蛋白结构完整,截断蛋白CPB2-2和CPB2-3羧肽酶活性功能域没有因为缺失信号肽和激活肽结构而发生构像改变,也证明其蛋白可能具有正常羧肽酶活性。猪CPB2蛋白互作网络分析及其富集分析显示,CPB2在猪体内可能通过肽酶水解及肽酶活性调节,参与蛋白质代谢反应、纤溶、补体与凝血级联和创伤反应等过程,功能失调可能导致凝血异常、静脉血栓和静脉炎等疾病。
有研究表明,人CPB2基因与一些凝血因子具有相似的特殊转录启动序列,使其在肝中特异性表达[29],在鼠中也只在肝中检测到Cpb2基因的表达[30]。本研究通过RT-PCR检测CPB2-1、CPB2-2和CPB2-3全长序列在7种组织(肝、心、肾、皮下脂肪、肠系膜脂肪、股二头肌和背最长肌)中的表达情况,发现猪CPB2基因的3种转录本同样具有明显的组织特异性,除在肝中特异性表达外,在肾中也有表达。前期本实验室关于代谢性疾病易感猪的研究,已发现其肝内靶向转入代谢性疾病风险基因PNPLA3I148M,肝表现出炎症病灶等特征,与人类非酒精性脂肪性肝炎(NASH)等慢性肝病存在一定的相似性[17]。本研究表明,在短期富营养饮食诱导的代谢性疾病易感猪肝中,CPB2总体表达量、转录本CPB2-1和转录本CPB2-2显著降低,这与已有研究中CPB2表达与慢性肝病存在负相关关系[31-32]的结果相似,也与已有报道中NASH患者具有较低水平的CPB2编码蛋白TAFI值的结果相似[33];也可以看出CPB2-1和CPB2-2表达趋势与CPB2总体表达量相同,这两个转录本可能在猪体内起主要作用。
4 结论本研究成功在肝中克隆了猪CPB2基因的3种可变剪接体,推测CPB2-1是行使纤溶与凝血等生理功能的正常转录本;新发现转录本CPB2-2和CPB2-3被留在肝和肾中可能具有羧肽酶活性和重要生理学功能;CPB2基因与代谢相关的慢性肝病存在一定联系。本研究结果将为CPB2基因功能及转录调控提供理论基础。
| [1] |
REDLITZ A, TAN A K, EATON D L, et al. Plasma carboxypeptidases as regulators of the plasminogen system[J]. J Clin Invest, 1995, 96(5): 2534-2538. DOI:10.1172/JCI118315 |
| [2] |
EATON D L, MALLOY B E, TSAI S P, et al. Isolation, molecular cloning, and partial characterization of a novel carboxypeptidase B from human plasma[J]. J Biol Chem, 1991, 266(32): 21833-21838. DOI:10.1016/S0021-9258(18)54713-X |
| [3] |
SILLEN M, DECLERCK P J. Thrombin activatable fibrinolysis inhibitor (TAFI): An updated narrative review[J]. Int J Mol Sci, 2021, 22(7): 3670. DOI:10.3390/ijms22073670 |
| [4] |
WILLEMSE J L, POLLA M, HENDRIKS D F. The intrinsic enzymatic activity of plasma procarboxypeptidase U (TAFI) can interfere with plasma carboxypeptidase N assays[J]. Anal Biochem, 2006, 356(1): 157-159. DOI:10.1016/j.ab.2006.05.020 |
| [5] |
FOLEY J H, KIM P, NESHEIM M E. Thrombin-activable fibrinolysis inhibitor zymogen does not play a significant role in the attenuation of fibrinolysis[J]. J Biol Chem, 2008, 283(14): 8863-8867. DOI:10.1074/jbc.M800127200 |
| [6] |
DECLERCK P J. Thrombin activatable fibrinolysis inhibitor[J]. Hamostaseologie, 2011, 31(3): 165-173. DOI:10.5482/ha-1155 |
| [7] |
VERCAUTEREN E, GILS A, DECLERCK P J. Thrombin activatable fibrinolysis inhibitor: A putative target to enhance fibrinolysis[J]. Semin Thromb Hemost, 2013, 39(4): 365-372. DOI:10.1055/s-0033-1334488 |
| [8] |
LEUNG L L K, MYLES T, NISHIMURA T, et al. Regulation of tissue inflammation by thrombin-activatable carboxypeptidase B (or TAFI)[J]. Mol Immunol, 2008, 45(16): 4080-4083. DOI:10.1016/j.molimm.2008.07.010 |
| [9] |
SWAISGOOD C M, SCHMITT D, EATON D, et al. In vivo regulation of plasminogen function by plasma carboxypeptidase B[J]. J Clin Invest, 2002, 110(9): 1275-1282. DOI:10.1172/JCI0215082 |
| [10] |
TE VELDE E A, WAGENAAR G T M, REIJERKERK A, et al. Impaired healing of cutaneous wounds and colonic anastomoses in mice lacking thrombin-activatable fibrinolysis inhibitor[J]. J Thromb Haemost, 2003, 1(10): 2087-2096. DOI:10.1046/j.1538-7836.2003.00404.x |
| [11] |
NAGASHIMA M, YIN Z F, ZHAO L, et al. Thrombin-activatable fibrinolysis inhibitor (TAFI) deficiency is compatible with murine life[J]. J Clin Invest, 2002, 109(1): 101-110. DOI:10.1172/JCI0212119 |
| [12] |
WANG X K, SMITH P L, HSU M Y, et al. Deficiency in thrombin-activatable fibrinolysis inhibitor (TAFI) protected mice from ferric chloride-induced vena cava thrombosis[J]. J Thromb Thrombolysis, 2007, 23(1): 41-49. DOI:10.1007/s11239-006-9009-4 |
| [13] |
吴晓本. 凝血酶激活的纤溶抑制物编码区基因CPB2多态性与2型糖尿病关系的研究[D]. 济南: 山东大学, 2009. WU X B. Study on the association of genetic variations in thrombin activatable fibrinolysis inhibitor (TAFI) encoding region and the risk of type 2 diabetes Mellitus[D]. Ji'nan: Shandong University, 2009. (in Chinese) |
| [14] |
李敏, 孙玉倩, 范博, 等. 血浆凝血酶激活的纤溶抑制物、视黄醇结合蛋白与2型糖尿病血管并发症的关系[J]. 牡丹江医学院学报, 2011, 32(4): 44-46. LI M, SUN Y Q, FAN B, et al. Relationship between plasma thrombin activated fibrinolytic inhibitors, retinol binding protein and vascular complications in type 2 diabetes mellitus[J]. Journal of Mudanjiang Medical University, 2011, 32(4): 44-46. (in Chinese) |
| [15] |
CAGLIANI R, FUMAGALLI M, RIVA S, et al. Polymorphisms in the CPB2 gene are maintained by balancing selection and result in haplotype-preferential splicing of exon 7[J]. Mol Biol Evol, 2010, 27(8): 1945-1954. DOI:10.1093/molbev/msq082 |
| [16] |
LIN J H H, NOVAKOVIC D, RIZZO C M, et al. The mRNA encoding TAFI is alternatively spliced in different cell types and produces intracellular forms of the protein lacking TAFIa activity[J]. Thromb Haemost, 2013, 109(6): 1033-1044. DOI:10.1160/TH12-09-0668 |
| [17] |
ZHANG K Y, TAO C, XU J P, et al. CD8+ T cells involved in metabolic inflammation in visceral adipose tissue and liver of transgenic pigs[J]. Front Immunol, 2021, 12: 690069. DOI:10.3389/fimmu.2021.690069 |
| [18] |
BERGET S M, MOORE C, SHARP P A. Spliced segments at the 5' terminus of adenovirus 2 late mRNA[J]. Proc Natl Acad Sci U S A, 1977, 74(8): 3171-3175. DOI:10.1073/pnas.74.8.3171 |
| [19] |
CHOW L T, GELINAS R E, BROKER T R, et al. An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA[J]. Cell, 1977, 12(1): 1-8. DOI:10.1016/0092-8674(77)90180-5 |
| [20] |
GRAVELEY B R. Alternative splicing: Increasing diversity in the proteomic world[J]. Trends Genet, 2001, 17(2): 100-107. DOI:10.1016/S0168-9525(00)02176-4 |
| [21] |
WANG E T, SANDBERG R, LUO S J, et al. Alternative isoform regulation in human tissue transcriptomes[J]. Nature, 2008, 456(7221): 470-476. DOI:10.1038/nature07509 |
| [22] |
杨阳, 张雪莲, 张万锋, 等. 猪C1QTNF3基因可变剪接体的鉴定及介导miR-101调控成脂分化的研究[J]. 畜牧兽医学报, 2021, 52(11): 3042-3052. YANG Y, ZHANG X L, ZHANG W F, et al. Study of alternative splicing of porcine C1QTNF3 gene and its regulation on adipogenesis by mediating miR-101[J]. Acta Veterinaria et Zootechnica Sinica, 2021, 52(11): 3042-3052. (in Chinese) |
| [23] |
STAMM S, BEN-ARI S, RAFALSKA I, et al. Function of alternative splicing[J]. Gene, 2005, 344: 1-20. DOI:10.1016/j.gene.2004.10.022 |
| [24] |
KELEMEN O, CONVERTINI P, ZHANG Z Y, et al. Function of alternative splicing[J]. Gene, 2013, 514(1): 1-30. DOI:10.1016/j.gene.2012.07.083 |
| [25] |
ULE J, BLENCOWE B J. Alternative splicing regulatory networks: functions, mechanisms, and evolution[J]. Mol Cell, 2019, 76(2): 329-345. DOI:10.1016/j.molcel.2019.09.017 |
| [26] |
贾浩, 张小白, 宋晓峰. 人类胞内蛋白半衰期与其亚细胞定位的相关性研究[J]. 计算机与应用化学, 2011, 28(4): 411-414. JIA H, ZHANG X B, SONG X F. Relationship between intracellular protein half-life and subcellular localization in human cells[J]. Computers and Applied Chemistry, 2011, 28(4): 411-414. (in Chinese) |
| [27] |
BAJZAR L, MORSER J, NESHEIM M. TAFI, or plasma procarboxypeptidase B, couples the coagulation and fibrinolytic cascades through the thrombin-thrombomodulin complex[J]. J Biol Chem, 1996, 271(28): 16603-16608. DOI:10.1074/jbc.271.28.16603 |
| [28] |
BOFFA M B, BELL R, STEVENS W K, et al. Roles of thermal instability and proteolytic cleavage in regulation of activated thrombin-activable fibrinolysis inhibitor[J]. J Biol Chem, 2000, 275(17): 12868-12878. DOI:10.1074/jbc.275.17.12868 |
| [29] |
BOFFA M B, REID T S, JOO E, et al. Characterization of the gene encoding human TAFI (thrombin-activable fibrinolysis inhibitor; plasma procarboxypeptidase B)[J]. Biochemistry, 1999, 38(20): 6547-6558. DOI:10.1021/bi990229v |
| [30] |
张京华. 凝血酶激活的纤溶抑制物(TAFI)、超敏C反应蛋白(hsCRP)和2型糖尿病微量白蛋白尿症的关系[D]. 沈阳: 中国医科大学, 2020. ZHANG J H. The relationship between plasminogen inhibitor activated by hemomimetic enzyme and high sensitivity C-reactive protein and microalbuminuria in type2 diabetes mellitus[D]. Shenyang: China Medical University, 2020. (in Chinese) |
| [31] |
王胜江, 徐成伟. 慢性肝病患者血浆中凝血酶激活的纤溶抑制物(TAFI)水平与慢性肝损伤的相关性[J]. 山东大学学报: 医学版, 2012, 50(4): 83-86. WANG S J, XU C W. TAFI levels in patients with chronic liver diseases: Correlation with chronic hepatic damage[J]. Journal of Shandong University: Health Sciences, 2012, 50(4): 83-86. (in Chinese) |
| [32] |
SEJIMA H, SATOH S, DANSAKO H, et al. Molecular mechanism underlying the suppression of CPB2 expression caused by persistent hepatitis C virus RNA replication[J]. Acta Med Okayama, 2016, 70(2): 75-88. |
| [33] |
YENER S, AKARSU M, DEMIR T, et al. Plasminogen activator inhibitor-1 and thrombin activatable fibrinolysis inhibitor levels in non-alcoholic steatohepatitis[J]. J Endocrinol Invest, 2007, 30(10): 810-819. |
(编辑 孟培)



