2. 西藏农牧学院动物科学学院, 林芝 860000
2. College of Animal Science, Tibet Agricultural and Animal Husbandry University, Linzhi 860000, China
仔猪腹泻可导致仔猪发病率、死亡率提高,生长速度减缓等,给我国养猪业带来了巨大损失。细菌感染,尤其是致病性大肠杆菌(Escherichia coli)感染,是引起仔猪腹泻的主要原因之一[1]。我国已于2020年7月正式实施饲料禁抗,细菌性的腹泻成为禁抗后最核心的问题,开发新型高效绿色的“替抗产品”成为畜牧业关注焦点。噬菌体作为一种可杀死细菌却对真核细胞无害的病毒,被认为是具有较好前景的抗生素替代品[2-4]。根据噬菌体对宿主菌的裂解效应不同,将噬菌体分为烈性噬菌体和溶原性噬菌体[5]。烈性噬菌体可直接裂解宿主菌导致其死亡,而溶原性噬菌体大多数情况下会将其基因组整合到宿主菌的染色体上, 并随着宿主基因组的复制而复制,通常情况下并不裂解宿主菌[6],因此临床上仅用烈性噬菌体治疗细菌性感染。噬菌体的宿主谱是决定其治疗效果的重要因素[7],然而1株噬菌体在感染不同细菌时感染能力可能存在差异[8-9],仅测定宿主谱并不能全面说明噬菌体的应用潜力,需明确噬菌体在各株菌上的感染特性,才能更好地将噬菌体应用于治疗。本研究测定了此前从断奶前仔猪粪样中分离得到的1株大肠杆菌噬菌体C6在致病性大肠杆菌上的宿主谱,并比较了该噬菌体在不同致病菌上的感染特性,为进一步开发噬菌体作为新型抗菌剂提供研究基础。
1 材料与方法 1.1 噬菌体及菌株来源试验所用噬菌体为本实验室此前在1头健康仔猪断奶前期的粪样中分离得到的1株大肠杆菌噬菌体C6。
用于噬菌体宿主谱测定和感染特性分析的大肠杆菌菌株均为本实验室此前从猪粪中分离得到的90株大肠杆菌(限于篇幅,请到OSID开放科学数据与内容查看详细信息),其中包括1株非致病性大肠杆菌(E. coli 22),6株产肠毒素大肠杆菌(ETEC 101、ETEC 102、ETEC 103、ETEC 104、ETEC 105和ETEC 106),1株大肠杆菌O157(E. coli W1),和2株血清型为O157:H7的产志贺毒素大肠杆菌(STEC W3、STEC W5),2株肠道致病性大肠杆菌(EPEC 143、EPEC 162)。菌株具体信息见表 1。噬菌体C6是以E. coli 22为宿主菌分离得到。
|
|
表 1 大肠杆菌噬菌体C6的宿主谱 Table 1 Host bacteria of phage C6 |
双层平板法扩增噬菌体后用10 mL SM液将平板上的噬菌体洗脱下,用0.22 μm滤器过滤除菌后,加入100 ku超滤管中, 4 ℃,4 800 g离心20 min,浓缩噬菌体液至500 μL,滴度为1010~1011 pfu·mL-1。将噬菌体浓缩液滴在铜网上,作用15 min,从侧面用滤纸吸干多余液体,加1滴2%磷钨酸(PTA,pH7.2)于铜网,然后置于干燥滤纸上,自然干燥后,进行透射电镜观察。
1.3 基因组提取、全基因组测序和分析首先按“1.2”介绍方法浓缩噬菌体至500 μL,加入DNase I和RNaseA(购自TaKaRa,大连宝生物)至终浓度为1 μg·mL-1,37 ℃作用1 h。接着加入EDTA至终浓度为20 mol·L-1,80 ℃作用8 min。然后参照《分子克隆实验指南》提取噬菌体基因组[10],最后将基因组送至上海凌恩生物科技有限公司进行全基因组测序,测序结果上传至GenBank。
1.4 噬菌体热稳定性、pH稳定性测定取1 mL的效价为107 pfu·mL-1的噬菌体,分别放置在30~70 ℃的水浴锅中恒温作用1 h,样品水浴完成后迅速取出放置在冰块中冷却,使用双层平板法测定不同温度处理后的样品中噬菌体效价。
取100 μL效价为108pfu·mL-1的噬菌体分别与900 μL的pH为2~13的SM缓冲液均匀混合,后置于37 ℃中恒温水浴2 h,使用双层平板法测定不同pH处理后的样品中噬菌体效价。
1.5 噬菌体宿主谱测定结合点滴法和双层平板法测定噬菌体C6在90株大肠杆菌上的宿主谱。点滴法:用无菌棉签沾取处在对数生长期的细菌后均匀涂布于LB平板上,待干后,取5 μL噬菌体液滴在平板上,37 ℃孵育6 h后,若菌苔上出现透亮的空斑则判定噬菌体可裂解该菌。双层平板法:以点滴检测结果为阳性的菌作为宿主菌,用双层平板法观察成斑情况,出现噬菌斑的菌即视为可被裂解感染的细菌。同时观察噬菌体C6在不同株菌上噬菌斑的形态。
1.6 噬菌体最佳感染复数测定将噬菌体以感染复数(multiplicity of infection, MOI)102∶1、10∶1、1、1∶10、1∶102、1∶103、1∶104、1∶105分别接种至处于对数期的各株大肠杆菌中,37 ℃培养4.5 h,得到噬菌体效价最高的MOI即为最佳MOI。
1.7 噬菌体成斑率测定参照文献[11]介绍的方法,以相同量噬菌体分别与各株大肠杆菌铺双层平板后统计噬菌斑数量,以在E. coli 22上出现斑的数量为1,计算噬菌体在各株致病性大肠杆菌上的成斑率(efficiency of plating, EOP)。
1.8 噬菌体吸附率测定将噬菌体分别与各株大肠杆菌以MOI=10-1接种于液体LB中,37 ℃孵育10 min后,4 000 g离心5 min,弃上清,用液体LB重悬菌体后,测噬菌体数量。重悬后测得的噬菌体数量/初始接进去的噬菌体数量即为噬菌体吸附率。
1.9 噬菌体生长曲线测定将噬菌体分别与各株大肠杆菌以MOI=1或MOI=10-2接种于液体LB中,其中菌的接种量均为107cfu·mL-1,噬菌体的接种量为107或105pfu·mL-1。37 ℃孵育15 min后,4 000 g离心5 min,弃上清,用液体LB重悬菌体后置于37 ℃震荡培养5 h,每小时取样检测噬菌体滴度,以感染时间为横坐标,以噬菌体滴度的对数为纵坐标,绘制噬菌体的一步生长曲线。
1.10 噬菌体抑菌曲线测定将噬菌体分别与各株致病性大肠杆菌以MOI=10-4~102接种于液体LB后置于37 ℃震荡培养6 h,每小时检测共孵育液在600 nm处的吸光值(OD600 nm)。同时设立阳性对照组,仅接种致病菌菌,每小时检测OD600 nm。以时间为横坐标,OD600 nm为纵坐标,绘制噬菌体在各株致病性大肠杆菌上的抑菌曲线。
2 结果 2.1 噬菌体C6分类鉴定电镜结果(图 1A)显示噬菌体C6为肌尾噬菌体,由头部、颈部和尾部3部分组成,其头部呈正二十面体形,长约95 nm,具有T4样噬菌体形态特征。测序结果显示噬菌体C6基因组全长169 529 bp,测序结果上传至NCBI,登录号为MW679410。比对结果显示,与C6同源性高的噬菌体均为T4样噬菌体。基因组上无基因比对到细菌基因组上,且其基因组注释显示无整合酶基因,故判定为烈性噬菌体。根据形态观察并结合基因测序结果可推断噬菌体C6属于T4样噬菌体属。

|
A.电镜形态;B.热稳定性;C.酸碱稳定性 A. Electronmicrograph; B. Thermal stability; C. pH stability 图 1 噬菌体C6的电镜形态和稳定性 Fig. 1 Electronmicrograph and stability of phage C6 |
热稳定性结果显示噬菌体C6在温度为30~50 ℃时相对稳定;60 ℃时噬菌体开始出现死亡,70 ℃时噬菌体无法存活(图 1B)。
pH稳定性结果显示噬菌体C6在pH为4~11时相对稳定,在pH为3的酸环境或pH为12的碱性环境开始出现死亡,到pH为2或pH为13的条件时,噬菌体无法存活(图 1C)。
2.3 噬菌体C6宿主谱作者选取实验室此前保存的90株大肠杆菌用于噬菌体C6宿主谱检测。试验结果表明,点滴法得到的宿主谱要广于平板法(表 1)。噬菌体C6可感染1株非致病性大肠杆菌(E. coli 22)和4株致病性大肠杆菌,包括大肠杆菌O157(E. coli W1)、2株产志贺毒素大肠杆菌(STEC W3、STEC W5)和1株肠致病性大肠杆菌(EPEC 143)。
2.4 噬菌体C6感染特性噬菌斑结果(图 2A~E)显示, 噬菌体C6在E. coli 22和E. coli W1上的噬菌斑均透亮,直径为0.5~1.0 mm;在EPEC 143上的噬菌斑也较透亮,但直径≤0.5 mm;在STEC W3和STEC W5上的噬菌斑则较淡,且均为针尖般大小,比较二者,C6在STEC W3上的噬菌斑更清晰一些。

|
A. E. coli 22; B. W1; C. EPEC 143; D. STEC W3; E. STEC W5; a. D图部分区域的放大; b. E图部分区域的放大 A. E. coli 22; B. W1; C. EPEC 143; D. STEC W3; E. STEC W5; a. Enlargement of partial Figure D; b. Enlargement of partial Figure E 图 2 噬菌体C6不同大肠杆菌上的噬菌斑形态 Fig. 2 Plaques of phage C6 on different E. coli strains |
最佳MOI结果显示(表 2),噬菌体C6在E. coli 22上的最佳MOI为10-3,在EPEC143、STEC W3和STEC W5上的最佳MOI均需≥100。
|
|
表 2 大肠杆菌噬菌体C6在各株菌上的感染特性 Table 2 Infecting characteristics of phage C6 |
成斑率EOP结果显示(表 2),噬菌体C6在STEC W3、STEC W5和EPEC 143 3株菌间的成斑率均存在显著差异(P < 0.05),且均显著低于在E. coli 22和E. coli W1上的成斑率(P < 0.05)。C6在STEC W5上的成斑率最低,低至10-7。成斑率结果与噬菌斑大小呈显著正相关关系。
吸附率结果显示(表 2),噬菌体C6在E. coli 22和E. coli W1两株菌间的吸附率存在显著差异,且均显著高于在STEC W3、STEC W5和EPEC 143 3株菌上的吸附率。吸附率结果与最佳MOI呈负相关关系。
根据最佳MOI的结果,作者选择了在MOI=10-2(噬菌体接种量为105pfu·mL-1)时测噬菌体C6在E. coli 22和E. coli W1上的生长曲线(图 3A),以及在MOI=1(噬菌体接种量为107pfu·mL-1) 时测C6在STEC W3、STEC W5和EPEC 143上的生长曲线(图 2B)。比较生长曲线上0时的噬菌体滴度可以发现虽然噬菌体初始接种量在STEC W3、STEC W5和EPEC 143比在E. coli 22和E. coli W1上高了两个数量级,但所测得的滴度仍较低,说明噬菌体吸附到E. coli 22和E. coli W1的能力更强,这与吸附率结果一致。比较5 h后的噬菌体最终滴度,噬菌体在E.coli 22和E.coli W1复制得到的子代滴度可达到109~1011pfu·mL-1,显著高于在STEC W3、STEC W5和EPEC 143上的子代滴度(107~109pfu·mL-1)。进一步比较噬菌体在E. coli 22和E. coli W1上的生长曲线,发现虽然噬菌体在E. coli W1上初始吸附的噬菌体少,但是5 h后,噬菌体在E. coli W1复制得到的子代噬菌体数量更多。比较噬菌体在STEC W3、STEC W5和EPEC 143上的生长曲线,发现C6在STEC W3产生最终子代噬菌体的滴度最高,在STEC W5上的生长速度最慢。

|
所标字母相异代表各处理组差异显著(P < 0.05),字母相同代表各处理组差异不显著(P≥0.05) Different letters denotes significant difference between the treatments(P < 0.05), same letter denotes not significant difference between treatments(P≥0.05) 图 3 噬菌体C6在各株菌上的生长曲线 Fig. 3 One-step growth curve of phage C6 |
根据以上各项感染特性指标的结果,选择MOI=10-1~10-5测定了噬菌体C6在E.coli W1上的抑菌效力,选择MOI=1~102测定了噬菌体C6在STEC W3、STEC W5和EPEC 143上的抑菌效力。
结果显示(图 4),在各株菌上均是MOI越大抑菌效果越好,在E. coli W1上,MOI越小,噬菌体C6需要越长的时间发挥抑菌效力。即使在MOI=10-5时,C6在5 h后也能发挥显著的抑制E. coli W1的生长。比较噬菌体C6在STEC W3、STEC W5和EPEC 143上的抑菌效力,C6在STEC W3上的抑菌效果最好。在STEC W5上,噬菌体C6在3 h后出现抑菌效果减弱的现象,细菌的数量开始逐渐增加。
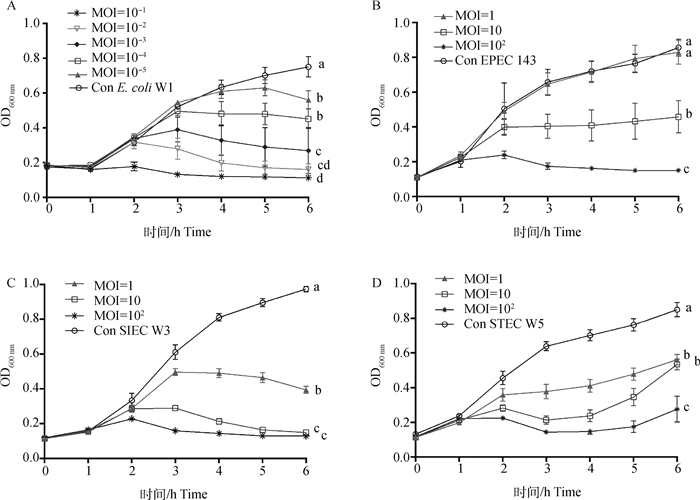
|
A.W1;B.143;C.W3;D.W5。所标字母相异代表各处理组差异显著(P < 0.05),字母相同代表各处理组差异不显著(P≥0.05) A.W1;B.143;C.W3;D.W5.Different letters denotes significant difference between the treatments(P < 0.05), same letter denotes not significant difference between treatments(P≥0.05) 图 4 噬菌体C6在各株致病菌上的抑菌曲线 Fig. 4 Bacteriostatic curves of phage C6 |
本研究测定了实验室此前分离得到的1株大肠杆菌噬菌体C6的生物学特性,发现噬菌体C6有较广的致病菌宿主谱。试验用了点滴法和双层平板法检测了噬菌体C6的宿主谱,发现点滴法得到的宿主谱广于双层平板法。为了排除是由于扩增噬菌体的细菌所分泌的大肠杆菌素导致被测细菌裂解而形成空斑,作者将用于扩增噬菌体的菌株单独培养后,取菌液上清重复点滴试验,并未观测到空斑,说明点滴裂解效应确实由噬菌体产生。这与之前Khan Mirzaei和Nilsson[12]的研究结果一致。革兰阴性菌的噬菌体通过点滴法可裂解细菌却在双层平板上无法形成空斑的主要原因有两种:“lysis from without”或者“abortive infection”[11, 13]。噬菌体的宿主谱是决定其治疗效果的重要因素[7],而点滴法和双层平板法是最常见的两种检测噬菌体宿主谱的方法,但是两者结果的差异说明点滴法得到的宿主谱并不能完全表明噬菌体的感染谱。此外,本试验结果表明即使噬菌体能感染裂解某些细菌,其抑菌能力也存在显著差异,所以评价1株噬菌体是否具有应用潜力,不能简单地看它是否具有较广的宿主谱。
本研究测定了噬菌体C6在多株致病菌上的感染特性,综合噬菌斑形态、最佳MOI、成斑率、吸附率、生长曲线和抑菌曲线,发现它们之间并不完全呈正相关关系。噬菌斑大小与成斑率呈显著正相关,噬菌体的抑菌效果与噬菌体在该菌上的生长能力呈正相关,吸附率与最佳MOI呈显著的负相关关系,吸附率与成斑率之间也无相关性,噬菌体的抑菌效果与噬菌斑大小无相关性。噬菌体C6在5株菌上的噬菌斑大小和成斑率大小均为E.coli22、E.coli W1>EPEC 143>STEC W3>STEC W5。
噬菌体C6在STEC W5在噬菌体与细菌共培养后期,细菌出现增殖的情况,推测是产生了耐受的突变菌,导致噬菌体C6抑菌效力减弱。这也是目前噬菌体治疗细菌感染时最常见的问题[14]。细菌耐受噬菌体可以通过阻止细菌吸附[15]、感染流产[16]和CRISPR-Cas系统等多种途径[17], STEC W5是通过哪种途径产生了耐受还需后续进行更深入地研究确认。但是噬菌体通常也会产生新的突变来应对对细菌的耐受,噬菌体与细菌之间一直存在这种此消彼长,互相抵抗的共同进化的过程[18]。本试验由于仅检测了6 h的抑菌效果,未能观察到噬菌体是否能针对耐受菌产生突变进而产生新一轮的抑菌效果。
细菌表面存在的噬菌体受体是影响噬菌体吸附的最关键因素[19],从吸附率的结果可以看出,E. coli 22、E. coli W1以及EPEC 143 3个菌株上的噬菌体受体结构可能有所不同。抑菌试验结果表明,噬菌体C6在5株菌上均是MOI越大时抑菌效果越好,并非在最佳MOI时抑菌效果最好,这与此前其他学者的试验结果一致[20-21]。
噬菌体C6是从健康断奶前仔猪中分离得到,说明在猪肠道中天然存在着可裂解感染多株致病菌的噬菌体,这些噬菌体可能具有保护仔猪肠道抵抗致病菌侵袭的潜在作用,这对预防仔猪腹泻的发生有着极其重要的意义。此前也有研究表明,内源性的噬菌体可为机体提供潜在的保护作用[22-23]。此外,噬菌体C6可以感染多株致病性大肠杆菌,且在各株致病菌上的感染特性存在差异,可作为较好的模型研究噬菌体与细菌间的互作关系,为进一步开发噬菌体作为新型抗菌剂提供研究基础。
4 结论噬菌体C6为T4样噬菌体,可感染多株致病性大肠杆菌,在不同致病菌上的感染特性存在差异,均能显著抑制致病菌的生长。
| [1] |
GRESSE R, CHAUCHEYRAS-DURAND F, FLEURY M A, et al. Gut microbiota dysbiosis in postweaning piglets: understanding the keys to health[J]. Trends Microbiol, 2017, 25(10): 851-873. DOI:10.1016/j.tim.2017.05.004 |
| [2] |
MORRISETTE T, KEBRIAEI R, LEV K L, et al. Bacteriophage therapeutics: a primer for clinicians on phage-antibiotic combinations[J]. Pharmacotherapy, 2020, 40(2): 153-168. DOI:10.1002/phar.2358 |
| [3] |
DALMASSO M, HILL C, ROSS R P. Exploiting gut bacteriophages for human health[J]. Trends Microbiol, 2014, 22(7): 399-405. DOI:10.1016/j.tim.2014.02.010 |
| [4] |
REHMAN S, ALI Z, KHAN M, et al. The dawn of phage therapy[J]. Rev Med Virol, 2019, 29(4): e2041. |
| [5] |
MIRZAEI M K, MAURICE C F. Ménage à trois in the human gut: interactions between host, bacteria and phages[J]. Nat Rev Microbiol, 2017, 15(7): 397-408. DOI:10.1038/nrmicro.2017.30 |
| [6] |
OFIR G, SOREK R. Contemporary phage biology: from classic models to new insights[J]. Cell, 2018, 172(6): 1260-1270. DOI:10.1016/j.cell.2017.10.045 |
| [7] |
UL HAQ I, CHAUDHRY W N, AKHTAR M N, et al. Bacteriophages and their implications on future biotechnology: a review[J]. Virol J, 2012, 9: 9. DOI:10.1186/1743-422X-9-9 |
| [8] |
MAHMOUD M, ASKORA A, BARAKAT A B, et al. Isolation and characterization of polyvalent bacteriophages infecting multi drug resistant Salmonella serovars isolated from broilers in Egypt[J]. Int J Food Microbiol, 2018, 266: 8-13. DOI:10.1016/j.ijfoodmicro.2017.11.009 |
| [9] |
PARK M, LEE J H, SHIN H, et al. Characterization and comparative genomic analysis of a novel bacteriophage, SFP10, simultaneously inhibiting both Salmonella enterica and Escherichia coli O157:H7[J]. Appl Environ Microbiol, 2012, 78(1): 58-69. DOI:10.1128/AEM.06231-11 |
| [10] |
SAMBROOK J, RUSSELL D W. 分子克隆实验指南[M]. 黄培堂, 译. 3版. 北京: 科学出版社, 2002: 185-187, 195-197. SAMBROOK J, RUSSELL D W. Molecular cloning: a laboratory manual[M]. HUANG P T, trans. 3rd ed. Beijing: Science Press, 2002: 185-187, 195-197. (in Chinese) |
| [11] |
KUTTER E. Phage host range and efficiency of plating[J]. Methods Mol Biol, 2009, 501: 141-149. |
| [12] |
KHAN MIRZAEI M, NILSSON A S. Isolation of phages for phage therapy: a comparison of spot tests and efficiency of plating analyses for determination of host range and efficacy[J]. PLoS One, 2015, 10(3): e0118557. DOI:10.1371/journal.pone.0118557 |
| [13] |
ABEDON S T. Lysis from without[J]. Bacteriophage, 2011, 1(1): 46-49. DOI:10.4161/bact.1.1.13980 |
| [14] |
OECHSLIN F. Resistance development to bacteriophages occurring during bacteriophage therapy[J]. Viruses, 2018, 10(7): 351. DOI:10.3390/v10070351 |
| [15] |
CHEN M M, ZHANG L, XIN S P, et al. Inducible prophage mutant of Escherichia coli can lyse new host and the key sites of receptor recognition identification[J]. Front Microbiol, 2017, 8: 147. |
| [16] |
HYMAN P, ABEDON S T. Bacteriophage host range and bacterial resistance[J]. Adv Appl Microbiol, 2010, 70: 217-248. |
| [17] |
ROSTØL J T, MARRAFFINI L. (Ph)ighting phages: how bacteria resist their parasites[J]. Cell Host Microbe, 2019, 25(2): 184-194. DOI:10.1016/j.chom.2019.01.009 |
| [18] |
LOPEZ PASCUA L, HALL A R, BEST A, et al. Higher resources decrease fluctuating selection during host-parasite coevolution[J]. Ecol Lett, 2014, 17(11): 1380-1388. DOI:10.1111/ele.12337 |
| [19] |
MERCANTI D J, ACKERMANN H W, QUIBERONI A. Characterization of two temperate Lactobacillus paracasei bacteriophages: morphology, kinetics and adsorption[J]. Intervirology, 2015, 58(1): 49-56. DOI:10.1159/000369207 |
| [20] |
LUKMAN C, YONATHAN C, MAGDALENA S, et al. Isolation and characterization of pathogenic Escherichia coli bacteriophages from chicken and beef offal[J]. BMC Res Notes, 2020, 13(1): 8. DOI:10.1186/s13104-019-4859-y |
| [21] |
HUANG C X, SHI J C, MA W J, et al. Isolation, characterization, and application of a novel specific Salmonella bacteriophage in different food matrices[J]. Food Res Int, 2018, 111: 631-641. DOI:10.1016/j.foodres.2018.05.071 |
| [22] |
SHARMA M. Lytic bacteriophages: Potential interventions against enteric bacterial pathogens on produce[J]. Bacteriophage, 2013, 3(2): e25518. DOI:10.4161/bact.25518 |
| [23] |
GÓRSKI A, WEBER-DABROWSKA B. The potential role of endogenous bacteriophages in controlling invading pathogens[J]. Cell Mol Life Sci, 2005, 62(5): 511-519. DOI:10.1007/s00018-004-4403-6 |
(编辑 白永平)



