细菌SOS反应又称为细菌DNA损伤诱导反应,是普遍存在的细菌应对DNA损伤的重要保护机制[1-2]。SOS反应的诱导途径主要有两种,即依赖RecA辅蛋白酶活性的途径和不依赖RecA的通用应激反应途径。依赖于RecA的SOS反应途径主要是利用其辅蛋白酶活性,催化包括LexA、噬菌体CI以及UmuC的自裂解,启动诱导SOS反应;在大多数细菌中,LexA蛋白是RecA表达的重要调节蛋白,是细菌SOS反应的主要抑制因子[3-5]。RecA存在于所有自然状态下存活的细菌中,在整个细菌领域中的平均序列保守性为60%~70%[3]。RecA是参与基因重组和DNA修复的多功能蛋白,调节SOS诱导的DNA修复蛋白的合成与活性,参与突变形成,在基因重组、DNA修复以及DNA配对和链交换中发挥关键作用[3-5]。
细菌的SOS反应首先是在对紫外线的响应中被发现[6],但它也与许多物理的(电离辐射,抗生素,酸碱刺激,重金属破坏)和化学的(烷基化,交联,氧化剂等)损害DNA的因素有关[7]。环境因素如UV,包括抗生素在内的药物和热应激都可以引发和增加活性氧ROS水平,随之对DNA、蛋白质和脂质造成损伤,从而诱导SOS应答[8]。SOS反应还可以通过诱导基因重组和促进抗性基因的水平转移使细菌产生抗性[9]。亚抑菌浓度的抗生素诱导的SOS应答和突变已在包括大肠杆菌、霍乱弧菌和链球菌上得到证实[7, 10-11]。
鸭疫里默氏杆菌感染鸭、鹅、鸡、火鸡等家禽和野禽,主要感染1~5周龄的雏鸭,发病鸭的主要剖检病变为纤维素性浆膜炎。鸭疫里默氏杆菌病在世界各地广泛分布,具有较高的发病率和死亡率,是造成养鸭业重大损失的主要细菌病之一。鸭疫里默氏杆菌的recA基因为1 032 bp,编码大小约37.7 ku的蛋白,但目前尚未报道该蛋白的生物学功能。这项工作首次研究了当鸭疫里默氏杆菌受到紫外线辐照损伤时,RecA蛋白调控菌株的SOS反应,获得的信息有助于增进对鸭疫里默氏杆菌RecA蛋白生物学功能的了解。
1 材料与方法 1.1 试验材料和菌株(及其培养)研究中使用的所有细菌菌株、质粒的相关特性和来源见表 1。胰蛋白酶大豆肉汤(TSB)、胰蛋白酶大豆琼脂(TSA)购自美国BD difcoTM公司,LB肉汤和LB琼脂购自北京Solarbio公司,2, 6-二氨基庚二酸(DPA)购自东京化成工业株式会社;抗生素购自北京Solarbio公司,限制性内切酶SacⅠ、SphⅠ、PstⅠ及质粒提取试剂盒、胶回收试剂盒等均购自TaKaRa公司;多片段一步法快速克隆试剂盒和一步法快速克隆试剂盒购自天津诺禾致源生物信息科技有限公司;0.22 μm无菌硝酸纤维素膜和过滤器购自美国Millipore公司。鸭疫里默氏杆菌RA-GD株和LJW-2株在37 ℃、5%CO2和在含5%小牛血清的TSB或TSA中培养。大肠杆菌X7213株在含有50 μg·mL-1 DPA的LB肉汤或LB琼脂上生长。
|
|
表 1 本研究所用的菌株与质粒 Table 1 Strains and plasmids used in this study |
从四川绵樱鸭业有限公司(中国成都)购得1日龄樱桃谷鸭,将动物饲养于清洁环境,室温保持在28~30 ℃,每天通风两次。
1.2 方法1.2.1 ΔrecA缺失株的构建 本研究中所用的引物见表 2。ΔrecA缺失株的构建如文献[13]所述,将突变株中recA基因的位点替换为红霉素(ermF)抗性基因。以鸭疫里默氏杆菌LJW-2株的DNA为模板,引物ErmF-F/ErmF-R进行扩增,获得红霉素(ermF)抗性基因。以RA-GD菌株的DNA作模板,用引物Up-F/Up-R和Dp-F/Dp-R分别扩增recA基因的上游和下游(各600 bp)同源臂序列。最后用SacⅠ和SphⅠ酶切pRE112质粒以获得载体片段。
|
|
表 2 PCR与qRT-PCR引物序列 Table 2 PCR and qRT-PCR primer sequences |
将上游同源臂、ErmF基因、下游同源臂和酶切后的pRE112质粒用多片段一步法快速克隆试剂盒(Novogene,天津)连接,生成重组质粒pRE112:: ErmF-600H,并转化进大肠杆菌X7213。将携带pRE112:: ErmF-600H重组质粒的大肠杆菌X7213和鸭疫里默氏杆菌RA-GD株共培养,用2 mg·L-1的红霉素筛选阳性菌株,并用保守引物OmpA-F/OmpA-R和鉴定引物RecA-F/RecA-R和ErmF-F/ErmF进行扩增,并将得到的PCR产物进行测序验证。得到的缺失株命名为ΔrecA。
1.2.2 c△recA回复株的构建 E. coli- R. anatipestifer穿梭质粒pCPRA在前期工作中构建完成[12]。以亲本株RA-GD为模板,以引物pCP-RecA-F/pCP-RecA-R扩增携带recA基因的片段,并回收。将pCRRA质粒用PstⅠ和SphⅠ双酶切,回收线性载体片段,用一步法快速克隆试剂盒(Novogene,天津)将recA基因目的片段和载体片段连接,转化X7213大肠杆菌细胞,以100 μg·mL-1的Amp抗性进行筛选,得到供体菌X7213-pCPRA: : RecA。与缺失株ΔrecA共培养,以2 mg·L-1的Cfx进行筛选,得到回复株c△recA。
1.2.3 生长曲线的测定 根据不同时间点的OD600 nm值,在非竞争条件下监测RA-GD、△recA和c△recA菌株的生长曲线。分别将菌株接种至5 mL含5%小牛血清的TSB中,37 ℃ 200 r·min-1培养至OD600 nm约1.0,并测定每个菌株的菌落形成单位(colony-forming unit,CFU),调整每个菌株的起始浓度均为109 CFU·mL-1。以1∶100的比例分别接种至10 mL含血清的TSB中,37 ℃ 200 r·min-1培养。每隔1 h取130 μL菌液用紫外分光光度计测量其OD600 nm值,共测定12 h,每个菌株做3组平行试验。根据OD600 nm值绘制每个菌株的生长曲线。
1.2.4 recA基因的转录 将亲本株RA-GD培养至OD600 nm值达到1.0,用60 μW·cm-2的紫外剂量辐照菌株,辐照时间为0、15、30和45 min,提取的样品mRNA,反转录成cDNA,作为模板备用。以RecA-qRT-F/RecA-qRT-R为引物,用qRT-PCR检测recA基因的转录水平;同时用16S-F/16S-R引物扩增rrsA3基因(产物为16S核糖体RNA),作为内参。利用2-△△Ct方法计算recA基因的相对转录水平[14]。
1.2.5 DNA损伤试验 根据细菌基因组的完整性判定双链DNA(dsDNA)的损伤程度,试验方法参照文献[15],该方法的原理:对细菌细胞施加损伤后,将细胞裂解,提取细胞基因组DNA,此时DNA上会出现断裂点。在控制合适的碱性和温度条件下,dsDNA会从断裂点位置解链,而完整的dsDNA不会解链。加入中和液后,再加入荧光染料,而染料只和未解链的dsDNA结合,不会和断裂点解链的单链DNA结合。因此,检测到的荧光信号越强,说明基因组DNA越完整;检测到的荧光信号越弱,说明dsDNA上的断裂点越多,损伤越严重。
把RA-GD、△recA和c△recA分别培养至OD600 nm为1.0,调整每个菌株的起始浓度均为105 CFU·mL-1。将细菌悬液放入24孔细胞培养板中,每孔加入500 μL悬液(5×104 CFU),用60 μW·cm-2的紫外剂量辐照菌株,辐照时间为45 min,提取细菌基因组DNA。取50 μL DNA溶液加入96孔细胞培养板中,然后加入55 μL 0.2 mol·L-1的NaOH溶液,30 ℃保持60 min,使dsDNA解链。然后加入70 μL中和液(14 mmol·L-1 β-巯基乙醇,1 mol·L-1葡萄糖),同时将温度降至22 ℃。最后加入78 μL SybrGreen溶液(1∶8333),混匀后立即以492 nm激发/520 nm发射波长测定荧光信号。
未经过紫外线处理的菌株作为阳性对照,处理时先把70 μL中和液加入细菌DNA溶液中,再加入55 μL 0.2 mol·L-1的NaOH溶液,以保证细菌dsDNA的完整性。不含有任何细菌DNA的样本(50 μL DNA洗脱液)作为阴性对照。每个样本的相对荧光信号依据以下公式计算:荧光信号(%)=(样本-阴性对照)/(样本-阳性对照)×100%。本研究中采用了依据检测到的荧光信号的强弱从而判定基因组DNA完整性的FADU方法[15-17]。
另外,为了检测RA-GD、△recA和c△recA在紫外线辐照下的存活能力,作者测定了经紫外线辐照不同时间后上述菌株的CFU。
1.2.6 统计分析 使用GraphPad Prism 6.0版Windows软件进行统计分析。t检验用于确定组间差异的重要性。*.P < 0.05表示差异显著,而**.P < 0.01和***.P < 0.001均表示差异极显著。
2 结果 2.1 ΔrecA基因缺失株的构建经PCR扩增验证,ΔrecA中recA基因无法扩增,ermF基因可以扩增出来,说明RA-GD中recA基因已被ermF基因置换(图 1);经对PCR产物测序验证,与目标序列的相似性为100%,说明ΔrecA基因缺失株构建成功。

|
M. DL2000相对分子质量标准;1. ompA基因阳性对照;2、3. ompA基因扩增;4、5. recA基因扩增;6、7. ermF基因扩增;8. ermF基因阴性对照 M. DL2000 relative molecular mass standard; 1. Positive control for ompA gene amplification; 2-3. Colonies for ompA gene amplification; 4-5. Colonies for recA gene amplification; 6-7. Colonies for ermF gene amplification; 8. Negative control for ermF gene amplification 图 1 缺失株ΔrecA的鉴定 Fig. 1 Identification of the deletion strain ΔrecA |
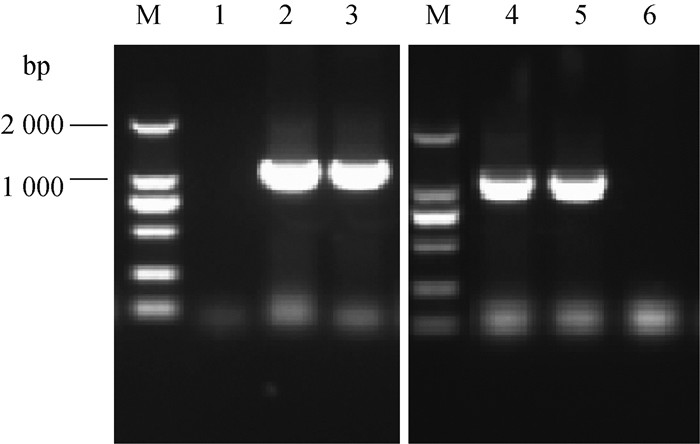
经PCR扩增验证,ΔrecA菌株中转入回复质粒pCPRA: : RecA后,可以扩增到recA基因(图 2);对PCR产物测序验证,与目标序列的相似性为100%,说明回复株cΔrecA构建成功。
|
M. DL2000相对分子质量标准;1. ompA基因阴性对照;2. ompA基因阳性对照;3. ompA基因扩增;4. recA基因阳性对照;5. recA基因扩增;6. recA基因阴性对照 M. DL2000 relative molecular mass standard; 1. Negative control for ompA gene amplification; 2. Positive control for ompA gene amplification; 3. Colonies for ompA gene amplification; 4. Positive control for recA gene amplification; 5. Colonies for recA gene amplification; 6. Negative control for recA gene amplification 图 2 回复株cΔrecA鉴定 Fig. 2 Identification of the complemented strain cΔrecA |
根据12 h内的生长曲线,与亲本株RA-GD相比,缺失株ΔrecA的生长速率没有明显差异(图 3),提示recA基因对于鸭疫里默氏杆菌的生长特性没有明显影响。

|
图 3 亲本株RA-GD和缺失株△recA的生长曲线 Fig. 3 Growth curves of parental strain RA-GD and deletion mutant △recA |
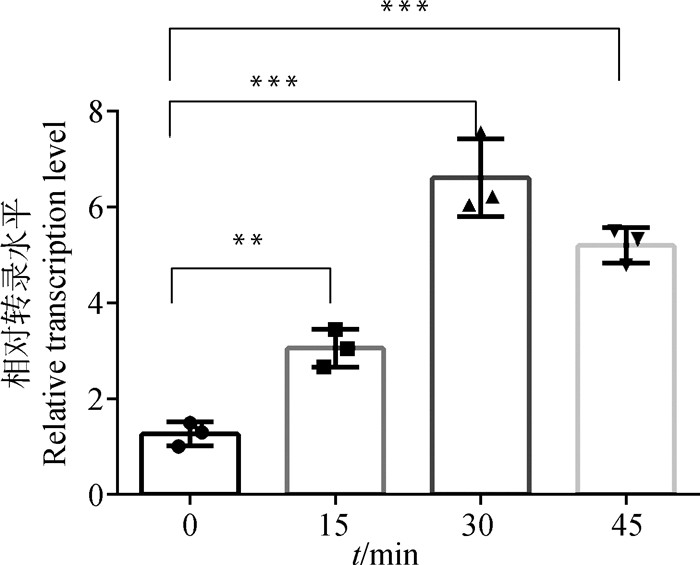
在紫外线处理的条件下,RA-GD株的recA基因的转录水平在0~45 min内随紫外线辐照时间的延长而显著增加(图 4)。
|
图 4 紫外线辐照不同时间的RA-GD菌株recA基因的转录水平 Fig. 4 Transcriptional levels of recA gene of RA-GD strain with UV treatment for different time |
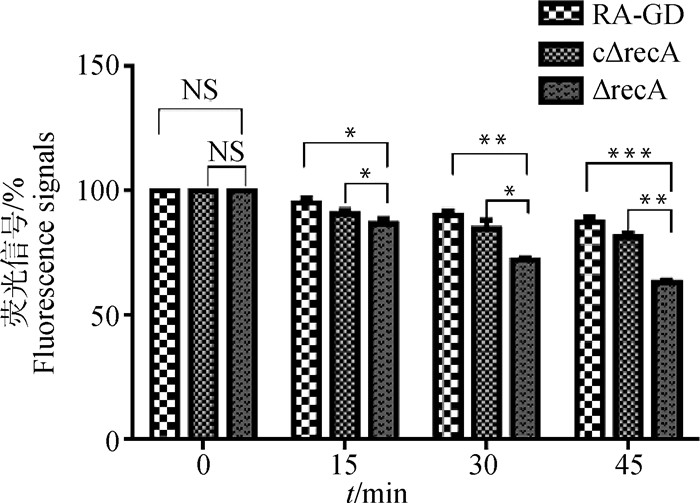
在紫外线辐照的0~45 min内,亲本株RA-GD和回复株cΔrecA dsDNA的荧光信号显著强于缺失株cΔrecA(图 5),说明紫外线可以破坏鸭疫里默氏杆菌dsDNA的完整性,并且recA基因缺失导致dsDNA的完整性明显下降,提示recA基因参与了dsDNA的损伤修复反应(SOS反应)。
|
图 5 紫外线处理不同时间条件下RA-GD、cΔrecA和ΔrecA的荧光信号 Fig. 5 Fluorescence signals of parental strain RA-GD, complemented strain cΔrecA and deletion mutant ΔrecA with UV treatment for different time |
在0~45 min,RA-GD、cΔrecA和ΔrecA的CFU均随着紫外线辐照时长增加而减小,表明紫外线对细菌的杀灭效果随照射时间延长而加强。在相同辐照时长下,RA-GD和cΔrecA的CFU均明显多于ΔrecA;与辐照时间0 min相比,辐照15、30和45 min时,RA-GD/ΔrecA和cΔrecA/ΔrecA的CFU的比值均显著增大,说明recA基因灭活可使RA-GD对UV的抵抗力下降,生存能力降低(表 3)。
|
|
表 3 紫外线处理不同时间条件下RA-GD、cΔrecA和ΔrecA的CFUs Table 3 The CFUs of RA-GD, cΔrecA and ΔrecA with UV treatment for different time |
SOS反应会引发大量的细菌细胞反应,包括DNA修复、细菌细胞伸长、诱导易出错的DNA聚合酶、诱导潜伏噬菌体和抑制细胞分裂等[18-19]。RecA和LexA蛋白在SOS反应中发挥关键调控作用。在正常生长过程中,LexA二聚体通过结合启动子区域的特定操作序列,作为SOS调控基因的转录抑制因子[20-21]。RecA作为一种辅蛋白酶,刺激LexA和其他相关蛋白的自裂解。鸭疫里默氏杆菌RecA蛋白是1个37 ku的蛋白,与大肠杆菌的RecA蛋白具有60.5%的氨基酸序列相似性,但是该蛋白的功能目前还未见报道。
关于鸭疫里默氏杆菌RecA蛋白在其受到外界因素损伤时是否发挥调控SOS反应,目前还未见相关报道。鉴于此,本研究对此问题进行研究。首先构建鸭疫里默氏杆菌recA基因的缺失株和回复株。若recA基因灭活不明显影响菌株的生长能力,可以更好地确定菌株其他性状的改变与recA基因的功能相关。因此,对亲本株和recA基因缺失株的生长能力进行检测。依据菌株在12 h内的生长曲线,发现recA基因缺失没有明显影响菌株的生长能力,说明该基因与鸭疫里默氏杆菌的生长之间没有明显的相关性。此结果与一项关于脆弱拟杆菌的研究结论相符。该研究表明,与正常的野生株相比,脆弱拟杆菌recA突变株在正常条件下的生长时间没有显著延长[22]。
RecA蛋白是参与细菌SOS反应并发挥关键作用的重组酶。当细菌受到外界破坏因素刺激而损害到其基因组DNA时,细菌启动SOS反应以应对生存危机,此时RecA蛋白发挥促进损伤DNA修复的功能,转录和表达水平可能上调。Niccum等[23]通过qRT-PCR法检测了紫外线处理对大肠杆菌recA基因转录水平的影响,结果显示recA基因的转录水平显著上调,表明recA RNA是SOS应答早期能检测到的可靠指标。笔者也检测了鸭疫里默氏杆菌在紫外线辐照下的recA基因的转录水平。结果显示,经紫外线辐照的菌株其recA基因的转录水平显著上调,推测菌株启动了依赖于RecA蛋白的SOS反应。
检测细菌的存活能力及其dsDNA的损伤程度是判定SOS反应的重要指标,需要稳定可靠的检测方法。本试验中,在紫外线损伤下鸭疫里默氏杆菌亲本株RA-GD和回复株cΔrecA的荧光信号显著高于缺失株ΔrecA,说明其dsDNA损伤程度明显小于缺失株,并且其存活能力显著高于缺失株。这些数据说明recA基因的缺失阻碍了DNA的损伤修复反应(SOS应答),提示鸭疫里默氏杆菌RecA蛋白在DNA损伤修复反应中发挥正调控作用。
4 结论鸭疫里默氏杆菌RecA蛋白与其生长能力无明显相关性,参与紫外线损伤后细菌的存活及其基因组dsDNA的修复,调控鸭疫里默氏杆菌的SOS反应。
| [1] |
SHINAGAWA H. SOS response as an adaptive response to DNA damage in prokaryotes[M]//FEIGE U, YAHARA I, MORIMOTO R I, et al. Stress-Inducible Cellular Responses. EXS, Vol 77. Basel: Birkhäuser, 1996: 221-235.
|
| [2] |
COX M M. Alignment of 3 (but not 4) DNA strands within a RecA protein filament[J]. J Biol Chem, 1995, 270(44): 26021-26024. DOI:10.1074/jbc.270.44.26021 |
| [3] |
YAKIMOV A, POBEGALOV G, BAKHLANOVA I, et al. Blocking the RecA activity and SOS-response in bacteria with a short α-helical peptide[J]. Nucleic Acids Res, 2017, 45(16): 9788-9796. DOI:10.1093/nar/gkx687 |
| [4] |
ARANDA J, BARDINA C, BECEIRO A, et al. Acinetobacter baumannii RecA protein in repair of DNA damage, antimicrobial resistance, general stress response, and virulence[J]. J Bacteriol, 2011, 193(15): 3740-3747. DOI:10.1128/JB.00389-11 |
| [5] |
LE S M, CHEN H, ZHANG X H, et al. Mechanical force antagonizes the inhibitory effects of RecX on RecA filament formation in Mycobacterium tuberculosis[J]. Nucleic Acids Res, 2014, 42(19): 11992-11999. DOI:10.1093/nar/gku899 |
| [6] |
ERILL I, CAMPOY S, BARBÉ J. Aeons of distress: an evolutionary perspective on the bacterial SOS response[J]. FEMS Microbiol Rev, 2007, 31(6): 637-656. DOI:10.1111/j.1574-6976.2007.00082.x |
| [7] |
BAHAROGLU Z, MAZEL D. Vibrio cholerae triggers SOS and mutagenesis in response to a wide range of antibiotics: a route towards multiresistance[J]. Antimicrob Agents Chemother, 2011, 55(5): 2438-2441. DOI:10.1128/AAC.01549-10 |
| [8] |
DRLICA K, ZHAO X L. Bacterial death from treatment with fluoroquinolones and other lethal stressors[J/OL]. Expert Revi Anti Infect Ther, 2020, doi: 10.1080/14787210.2021.1840353.
|
| [9] |
HAN N, SHENG D H, XU H. Role of Escherichia coli strain subgroups, integrons, and integron-associated gene cassettes in dissemination of antimicrobial resistance in aquatic environments of Jinan, China[J]. Water Sci Technol, 2012, 66(11): 2385-2392. DOI:10.2166/wst.2012.473 |
| [10] |
ANDERSSON D I, HUGHES D. Microbiological effects of sublethal levels of antibiotics[J]. Nat Rev Microbiol, 2014, 12(7): 465-478. DOI:10.1038/nrmicro3270 |
| [11] |
THI T D, LOPEZ E, RODRIGUEZ-ROJAS A, et al. Effect of recA inactivation on mutagenesis of Escherichia coli exposed to sublethal concentrations of antimicrobials[J]. J Antimicrob Chemother, 2011, 66(3): 531-538. DOI:10.1093/jac/dkq496 |
| [12] |
WANG Y P, LI S D, GONG X W, et al. Characterization of RaeE-RaeF-RopN, a putative RND efflux pump system in Riemerella anatipestifer[J]. Vet Microbiol, 2020, 251: 108852. DOI:10.1016/j.vetmic.2020.108852 |
| [13] |
LI S D, CHEN Q W, GONG X W, et al. RanB, a putative ABC-type multidrug efflux transporter contributes to aminoglycosides resistance and organic solvents tolerance in Riemerella anatipestifer[J]. Vet Microbiol, 2020, 243: 108641. DOI:10.1016/j.vetmic.2020.108641 |
| [14] |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method[J]. Nat Protoc, 2008, 3(6): 1101-1108. DOI:10.1038/nprot.2008.73 |
| [15] |
MORENO-VILLANUEVA M, PFEIFFER R, SINDLINGER T, et al. A modified and automated version of the 'Fluorimetric detection of alkaline DNA unwinding' method to quantify formation and repair of DNA strand breaks[J]. BMC Biotechnol, 2009, 9: 39. DOI:10.1186/1472-6750-9-39 |
| [16] |
BAUMSTARK-KHAN C, HENTSCHEL U, NIKANDROVA Y, et al. Fluorometric analysis of DNA unwinding (FADU) as a method for detecting repair-induced DNA strand breaks in UV-irradiated mammalian cells[J]. Photochem Photobiol, 2000, 72(4): 477-484. DOI:10.1562/0031-8655(2000)072<0477:FAODUF>2.0.CO;2 |
| [17] |
BIRNBOIM H C, JEVCAK J J. Fluorometric method for rapid detection of DNA strand breaks in human white blood cells produced by low doses of radiation[J]. Cancer Res, 1981, 41(5): 1889-1892. |
| [18] |
SINGH R, LEDESMA K R, CHANG K T, et al. Impact of recA on levofloxacin exposure-related resistance development[J]. Antimicrob Agents Chemother, 2010, 54(10): 4262-4268. DOI:10.1128/AAC.00168-10 |
| [19] |
MILLER C, THOMSEN L E, GAGGERO C, et al. SOS response induction by β-lactams and bacterial defense against antibiotic lethality[J]. Science, 2004, 305(5690): 1629-1631. DOI:10.1126/science.1101630 |
| [20] |
ZHANG A P P, PIGLI Y Z, RICE P A. Structure of the LexA-DNA complex and implications for SOS box measurement[J]. Nature, 2010, 466(7308): 883-886. DOI:10.1038/nature09200 |
| [21] |
MEMAR M Y, YEKANI M, CELENZA G, et al. The central role of the SOS DNA repair system in antibiotics resistance: A new target for a new infectious treatment strategy[J]. Life Sci, 2020, 262: 118562. DOI:10.1016/j.lfs.2020.118562 |
| [22] |
STEFFENS L S, NICHOLSON S, PAUL L V, et al. Bacteroides fragilis RecA protein overexpression causes resistance to metronidazole[J]. Res Microbiol, 2010, 161(5): 346-354. DOI:10.1016/j.resmic.2010.04.003 |
| [23] |
NICCUM B A, COPLEN C P, LEE H, et al. New complexities of SOS-induced "untargeted" mutagenesis in Escherichia coli as revealed by mutation accumulation and whole-genome sequencing[J]. DNA Repair, 2020, 90: 102852. DOI:10.1016/j.dnarep.2020.102852 |
(编辑 白永平)



