畜禽遗传资源是生物多样性的重要组成部分,在农业、经济和科学研究中发挥着重要作用。我国幅员辽阔,不同地理和气候条件孕育了丰富的畜禽遗传资源,约占世界畜禽遗传资源总量的六分之一。据2012年版《中国畜禽遗传资源志》统计,我国有777种动物遗传资源正式命名,包括556个地方品种,其中鸡品种116个,北京油鸡是其中之一。
北京油鸡起源于大约300年前的清代早期,以洼里和大屯两个地区最为集中。典型的北京油鸡具有凤冠、胫羽和五趾等外观特征,兼具地方鸡种肉、蛋品质优良,耐粗饲、抗病、抗逆性强等优良特性[1],但生产性能相对于国外品种较差。70年代末,为满足市场需求,企业主推高产品种,北京油鸡数量骤降[2]。为保护北京油鸡这一地方鸡种,中国农业科学院北京畜牧兽医研究所等单位开始承担北京油鸡的品种保护工作。2001年北京油鸡被农业农村部列为国家级畜禽品种资源重点保护品种。
亲缘关系较近个体交配的过程称为近交。在畜禽育种中,一般使用近交系数评估个体之间的近交情况。近交系数是指当个体由于近交而杂合基因减少时,纯合基因或纯合子的百分比[3]。近交可以增加种群的纯合性,也会增加纯合致病基因的概率,导致后代繁殖力下降和表型衰退等情况的发生。群体数量是影响保种情况的重要因素,群体数量较小,更容易导致近交发生。
随着二代全基因组重测序技术的发展,测序成本不断降低,重测序技术越来越多的应用到畜禽保种工作中[4-6]。基于SNP标记计算得到基因组近交系数(genomic inbreeding coefficient based on SNPs,FSNP),成为保种评价的新方法。使用SNP位点信息计算近交系数,可以有效避免系谱信息不完整、误差较大等特点[7]。连续性纯合片段(runs of homozygosity, ROH)[8]是基因组中的一段长纯合片段,长度为数百kb到数Mb不等。自Ferencakovic等[9]首次利用ROH进行近交评估以来,ROH已广泛应用于畜禽近交系数的计算。另有研究发现,FROH的准确性相较于FHOM、FGRM、FUNI计算的结果准确性更高[10]。
本研究以国家级北京油鸡保种场随机交配保种群体为研究对象,基于外貌特征和基因组信息对保种群体的外貌特征、近交系数和有效群体大小进行分析,研究北京油鸡的保种情况。
1 材料与方法 1.1 试验材料与样品采集本试验随机选取国家级北京油鸡保种场2019年随机交配保种群体40只个体,其中公鸡17只,母鸡23只,采用完全随机交配。同时,对北京油鸡凤冠、五趾、胫羽等外貌特征进行整理,对近年外貌特征的变化趋势进行分析。其中,1979年和1991—1993年的外貌特征记录来自优质黄羽肉鸡品系选育和配套研究论文集(内部资料),2001年、2005年外貌特征记录来源于现有群体的早期数据[11]。
翅下静脉采血,于EDTA抗凝管中,-20 ℃保存,用于提取DNA。
1.2 鸡基因组DNA提取采用酚-仿传统法提取DNA,使用两种方法对血液DNA提取效果进行检测:1) 琼脂糖凝胶电泳分析DNA降解的程度以及是否存在RNA污染;2) Nanodrop检测DNA的纯度,OD260 nm/OD280 nm合格范围为1.8~2.0。
合格DNA样品送往北京康普森生物技术有限公司进行二代全基因组重测序,基于Illumina NovaSeq技术测序平台,利用双末端测序(Paired-End)的方法,测序策略为Illumina PE150,测序深度为10×。
1.3 基因组变异检测使用基因组比对软件BWA(v0.7.17)[12]中BWA-MEM算法,将过滤后的Clean Reads比对参考基因组(ftp://ftp.ensembl.org/pub/release-101/fasta/gallus_gallus/dna/),并去掉未比对上、低质量(MQ < 4,MQ为mapping的质量值)、重复的Reads。剩余的高质量Reads用于后续分析。
在比对到参考基因组序列的基础上,通过突变分析软件GATK(v4.1.7.0)[13]检测全基因组中所有SNPs位点,过滤得到高可信度的SNP数据。
1.4 测序数据质控本研究使用PLINK(v1.90)[14]软件对全基因组测序数据生成的文件进行质量控制。质控标准如下:SNP检出率大于0.9,最小等位基因频率大于0.05,哈迪-温伯格平衡P值大于10-6,同时只保留常染色体,避免性别影响。经过质控后,共剩余40只个体的6 252 214个SNPs用于后续分析。使用BEAGLE.18May20.d20[15]软件对缺失基因型进行填充。
1.5 基于基因组信息估计有效群体大小(Ne)本研究使用PLINK软件对质控数据进行连锁不平衡修剪。把r2 < 0.1默认为两个SNPs不连锁。经过连锁不平衡修剪后共剩余43 167个SNPs位点,再使用SNeP(v1.1)[16]软件估计北京油鸡历史世代的有效群体大小,公式[17]:
| $ N_{\mathrm{T}(\mathrm{t})}=\frac{1}{\left(4 f\left(c_{\mathrm{t}}\right)\right)}\left(\frac{1}{E\left[r_{\mathrm{adj}}^{2} \mid c_{\mathrm{t}}\right]}-\alpha\right) $ | (1) |
NT(t)是过去t世代估计的有效群体大小,ct是过去t世代SNP之间特定物理距离的重组率,f(ct)是SNP之间遗传距离的函数,radj2是校正后的连锁不平衡估计,α是对突变发生的校正。对样本偏差进行修正。对SNeP软件计算的结果,使用GraphPad Prism 8绘制图表。
使用NeEstimator(v2.1)[18]软件的基于连锁不平衡方法估计当前世代的有效群体大小[19]。
1.6 基于基因组信息估计近交系数(F)使用PLINK软件对质控后数据计算ROH,设置ROH片段的最小长度为200 kb,组成ROH最少的SNP数量根据Lencz等[20]提出的公式进行计算:
| $ l=\frac{\ln \frac{a}{n_{s} * n_{i}}}{\ln (1-h e t)} $ | (2) |
其中,l为组成ROH的最少SNP数目,a为设定的检测到假阳性ROH的百分率,本研究中设定为0.05,ns为个体的SNP数目,ni为样本数,het为SNP的平均杂合度。本研究中由此公式计算得出组成ROH的最少SNP数目为153个。
根据PLINK软件计算的结果,利用公式计算FROH:
| $ F_{\mathrm{ROH}}=\frac{\sum_{\mathrm{i}} L_{\mathrm{ROHi}}}{L_{\text {auto }}} $ | (3) |
其中∑iLROHi为常染色体上ROH的总长度,Lauto为常染色体的长度,FROH为基于ROH计算的近交系数。
使用PLINK软件对质控后数据计算基于纯合基因型的近交系数(FHOM)和基于联合配子之间相关性计算的近交系数(FUNI),公式如下:
| $ F_{\mathrm{HOM}}=(O-E) /(L-E) $ | (4) |
其中,O为个体观测纯合基因型的数量,E为期望纯合基因型的数量,L为基因型的SNP数目。
| $ F_{\mathrm{UNI}}=\frac{x_{\mathrm{i}}^{2}-\left(1+2 p_{\mathrm{i}}\right) \mathrm{x}_{\mathrm{i}}+2 p_{\mathrm{i}}^{2}}{2 p_{\mathrm{i}}\left(1-p_{\mathrm{i}}\right)} $ | (5) |
其中, pi为第i个SNP位点等位基因A的初始频率,xi是基因型中a的个数,当SNP标记的基因型为AA、Aa或aa时,xi分别为0、1或2。
使用GCTA(v1.93.2beta)软件构建基因组关系G矩阵,再使用R(v3.6.3)软件对构建的G矩阵计算基于基因组关系G矩阵的近交系数(FGRM)[21],公式如下:
| $ F_{\mathrm{GRM}}=\frac{1}{\mathrm{~N}} \sum _{\mathrm{i}=1}^{\mathrm{N}}\left(\frac{\left[x_{\mathrm{i}}-\mathrm{E}\left(x_{\mathrm{i}}\right)\right]^{2}}{2 p_{\mathrm{i}}\left(1-p_{\mathrm{i}}\right)}-1\right) $ | (6) |
其中,pi同上,xi同上,N为SNP的数目。
1.7 FROH、FHOM、FGRM和FUNI不同算法近交系数间的相关性分析使用R的PerformanceAnalytics包对2019年北京油鸡保种群体FROH、FHOM、FGRM和FUNI 4个近交系数进行相关性分析。
1.8 基于各世代实际留种数量估计近交系数根据国家级北京油鸡保种场1979—2019年繁育记录,统计本保种群各世代实际留种公母鸡数,估计2019年北京油鸡保种群有效群体大小(Ne)、近交增量(ΔF)、近交系数(Ft),公式[22]如下:
| $ N e=\frac{4 N_{S} \times N_{D}}{N_{S}+N_{D}} $ | (7) |
| $ \Delta F=\frac{1}{2 N e} $ | (8) |
| $ F_{\mathrm{t}}=1-(1-\Delta F)^{\rm t} $ | (9) |
其中,Ne为有效群体大小,NS为公鸡,ND为母鸡,ΔF为近交增量,t为世代数,Ft为t世代近交系数。
2 结果 2.1 北京油鸡保种群体外貌特征的变化情况对1979年、1991—1993年、2001年、2005年、2017年、2018年和2019年的外貌特征进行统计,结果见图 1。胫羽百分比始终在97%以上,表现稳定。凤冠、五趾的百分比在2005—2017年都有不同程度的下降,凤冠百分比从100%下降到93%,五趾百分比从27%下降到15%。2018年凤冠百分比上升到98%,五趾百分比上升到27%,并保持稳定。

|
图 1 北京油鸡外貌特征统计图 Fig. 1 Statistical graph of appearance characteristics of Beijing You chicken |
根据国家级北京油鸡保种场1979—2019年各世代实际留种公、母鸡数量(共38个世代),估计有效群体大小和近交增量,结果如表 1所示。北京油鸡保种群38个世代平均近交增量为0.001 9,38个世代繁育后,2019年保种群近交系数为0.070 3。
|
|
表 1 北京油鸡保种群有效群体大小、近交系数估计 Table 1 Estimation of effective population size and inbreeding coefficient in Beijing You chicken conservation population |
2.3.1 有效群体大小估计 基于连锁不平衡方法估计2019年北京油鸡保种群体的有效群体大小为193。使用SNeP估计北京油鸡保种群的有效群体大小,结果如图 2所示。发现有效群体大小随着世代的减小逐渐呈现平缓下降的趋势,98世代之前的有效群体大小为595,13世代之前的有效群体大小176。北京油鸡保种群有效群体大小从45世代前开始下降加快,北京油鸡世代间隔大约为1年,70年代刚好是北京油鸡群体数量开始减少的时期,与现实情况相吻合。

|
图 2 北京油鸡历史世代有效群体大小变化图 Fig. 2 Variation of effective population size in Beijing You chicken before each generation |
2.3.2 ROH统计 对40只北京油鸡进行分析,共检测出7 101个ROH。分别统计ROH不同长度所占比例(图 3A)、不同染色体ROH数量所占比例(图 3B)、不同染色体上ROH的平均长度(图 3C)和ROH数量与长度相关统计(图 3D)。较短的ROH(0~0.5 Mb)所占比例最多,约占总数的75%,且ROH数量随ROH长度的增加逐渐减少。常染色体ROH数量各不相同,ROH分布不均匀。每条染色体ROH的数量大体随染色体长度的增加而增加。其中1号染色体ROH数量最多,约占ROH总数的19%,16、25、30、31、32号染色体ROH数量最少,而不同染色体上ROH的平均长度没有显著区别。在不同个体中,ROH的数量与ROH覆盖的长度也有很大的不同。随着ROH数量的增加,ROH的总长度也在增加。在这一群体中,ROH数量最多的个体有232个ROH,总长度约为110 Mb,ROH数量最少的个体有34个ROH,总长度约为8.7 Mb。

|
图 3 ROH长度及分布统计图 Fig. 3 Length and distribution statistics of ROH |
2.3.3 基因组近交系数比较 比较基于ROH、纯合基因型、基因组关系G矩阵、联合配子之间的相关性4种不同算法下的基因组近交系数(FROH、FHOM、FGRM、FUNI),结果如表 2所示。2019年保种群FROH平均值为(0.079 8±0.017 1),范围为0.009 1~ 0.115 5。3种基于SNP位点计算的近交系数FHOM、FGRM、FUNI平均值分别为(0.060 5±0.039 8)、(0.066 2±0.034 7)、(0.063 7±0.035 4),范围分别为-0.021 8~0.176 1、-0.003 2~0.162 3、0.000 6~ 0.164 3,分布范围明显大于FROH结果,且3种基于SNP位点计算的近交系数离散程度较大。
|
|
表 2 基因组信息不同算法计算的近交系数 Table 2 Inbreeding coefficients calculated by different methods of genome information |
2.3.4 不同算法近交系数相关性分析 计算2019年保种群体FROH、FHOM、FGRM和FUNI4种不同算法所得近交系数值间的相关性,结果如图 4所示。从图 4中上三角可以看出,FROH与FGRM显著相关(P < 0.01),相关系数为0.45。其他各组两两之间相关极显著(P < 0.001),相关系数都大于0.5。从图 4中下三角可以看出,FHOM与FGRM,FHOM与FUNI以及FGRM与FUNI之间存在较高线性相关。
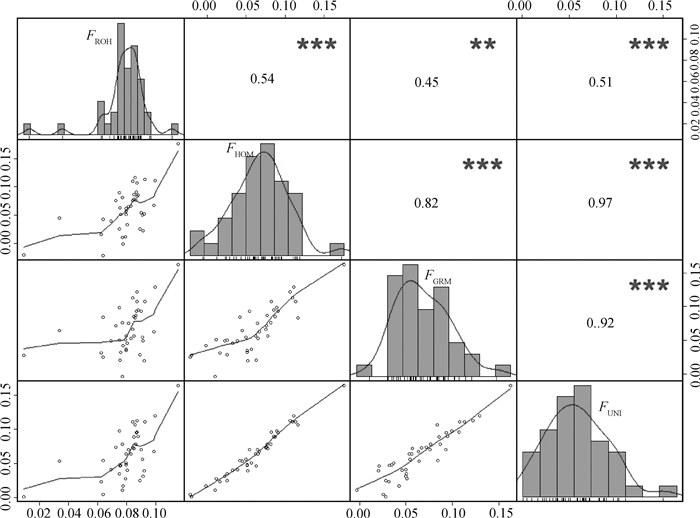
|
对角线:近交系数的频率分布直方图,图中曲线是百分位曲线。对角线上方:不同近交系数之间的Spearman相关系数。*. P < 0.05,**. P < 0.01,***. P < 0.001。对角线以下:不同近交系数之间的散点图,散点位置由上侧和右侧F值共同确定,图中曲线是趋势线 Diagonal: Frequency distribution histogram of inbreeding coefficient, the line is percentile curve. Above the diagonal: Spearman's correlation coefficients among the different inbreeding coefficients. *. P < 0.05, **. P < 0.01, ***. P < 0.001. Below the diagonal: Scatter plots among the different inbreeding coefficients, the scatter position is determined by the upper and right F values, the line is trendline 图 4 2019年保种群不同算法近交系数值间相关性分析 Fig. 4 Correlation analysis of inbreeding coefficient values obtained by different methods |
北京油鸡保种群体胫羽、凤冠、五趾等品种特有外貌特征得到了良好保留。2005—2017年北京油鸡五趾、凤冠百分比有所下降,自2017年起,繁育过程中适当增加具有凤冠、五趾特征的留种比例,至2018年凤冠、五趾特征百分比上升,恢复到2017年前的水平,并保持稳定。对1979—2019年各世代的外貌特征百分比进行统计,这一数据为国家级北京油鸡保种场利用品种特有外貌特征指导保种工作提供了依据。
3.2 基因组信息对保种工作的指导传统计算近交系数的方式是基于系谱记录估计的,但经常遇到系谱信息不完整和错误等问题,且忽略初代个体间近交系数也会影响计算准确性[23]。使用系谱信息是基于亲缘关系的血缘同源概率估计期望值,未考虑减数分裂过程中基因重组是随机事件,使用基因组信息估计的是个体间实际近交情况[4],更能反映保种群体的真实状况。FHOM、FGRM、FUNI是基于状态同源估计的[4, 24],无法区分血缘同源与状态同源,且某些个体近交系数为负值,因此,分别使用上述几种方法估计近交系数都是不够准确的。FHOM、FGRM、FUNI是通过单点计算得到,结果会受等位基因频率估算的影响,而FROH直接反映了自身长纯合片段占参考基因组长度的比例,受DNA测序样品的质量影响更小[25],利用ROH估计近交系数可以解决其他近交系数估计存在的缺陷[26-28]。同时,在人类[29-30]、牛[31-32]、猪[33-35]研究中也发现,基于ROH估计近交系数准确性最高。Peripolli等[36]的研究中,系谱计算的近交系数与FROH相关性最强,提出FROH可以作为代替系谱评估保种群体近交系数的有效方式。
本研究中,FROH的平均值大于3种基于SNP位点计算的近交系数FHOM、FGRM、FUNI,可能是由于FHOM、FGRM、FUNI中某些个体值为负数导致的近交系数偏低,造成的结果不准确,这一结果与Alemu等[37]和Shi等[33]的研究一致。相关性分析发现,FHOM与FGRM,FHOM与FUNI以及FGRM与FUNI具有较高的线性相关,且都是基于单点计算的,与曹愉夏等[38]的研究一致。由于FROH是基于血缘同源的估计,而FHOM、FGRM、FUNI是基于状态同源的估计,FROH与FHOM、FROH与FGRM、FROH与FUNI呈中度相关,在近交系数估计时尽量避免使用FHOM、FGRM、FUNI 3种计算方法。
4 结论北京油鸡1979—2019年40年来外貌特征明显且百分比保持稳定,经过40余年保种工作后FROH为0.079 8,近交系数增长缓慢,说明国家级北京油鸡保种场随机交配保种群体的保种工作是切实可行的。将来对北京油鸡随机交配保种群体每年随机选取一定数量的个体进行二代全基因组重测序检测,有利于以后对保种状况进行动态监控,随时调整保种工作方案。
| [1] |
邹剑敏. 我国肉鸡遗传资源保护、评价与利用的最新进展[J]. 中国家禽, 2019, 41(3): 1-5. ZOU J M. Advances in protection, evaluation and utilization of broiler genetic resources in China[J]. China Poultry, 2019, 41(3): 1-5. (in Chinese) |
| [2] |
李阳, 雷进, 李甘乔, 等. 北京油鸡文化遗产保护与发展分析[J]. 农业展望, 2019, 15(9): 77-80. LI Y, LEI J, LI G Q, et al. Protection and development of Beijing fatty chicken cultural heritage[J]. Agricultural Outlook, 2019, 15(9): 77-80. DOI:10.3969/j.issn.1673-3908.2019.09.013 (in Chinese) |
| [3] |
杨湛澄, 黄河天, 闫青霞, 等. 利用高密度SNP标记分析中国荷斯坦牛基因组近交[J]. 遗传, 2017, 39(1): 41-47. YANG Z C, HUANG H T, YAN Q X, et al. Estimation of genomic inbreeding coefficients based on high-density SNP markers in Chinese Holstein cattle[J]. Hereditas (Beijing), 2017, 39(1): 41-47. (in Chinese) |
| [4] |
PERIPOLLI E, MUNARI D P, SILVA M V G B, et al. Runs of homozygosity: current knowledge and applications in livestock[J]. Anim Genet, 2017, 48(3): 255-271. DOI:10.1111/age.12526 |
| [5] |
MARRAS G, GASPA G, SORBOLINI S, et al. Analysis of runs of homozygosity and their relationship with inbreeding in five cattle breeds farmed in Italy[J]. Anim Genet, 2015, 46(2): 110-121. DOI:10.1111/age.12259 |
| [6] |
VANAVERMAETE D, FOSTIER J, MAENHOUT S, et al. Preservation of genetic variation in a breeding population for long-term genetic gain[J]. G3 (Bethesda), 2020, 10(8): 2753-2762. DOI:10.1534/g3.120.401354 |
| [7] |
KARDOS M, LUIKART G, ALLENDORF F W. Measuring individual inbreeding in the age of genomics: marker-based measures are better than pedigrees[J]. Heredity (Edinb), 2015, 115(1): 63-72. DOI:10.1038/hdy.2015.17 |
| [8] |
GIBSON J, MORTON N E, COLLINS A. Extended tracts of homozygosity in outbred human populations[J]. Hum Mol Genet, 2006, 15(5): 789-795. DOI:10.1093/hmg/ddi493 |
| [9] |
FERENCAKOVIC M, HAMZIC E, GREDLER B, et al. Runs of homozygosity reveal genome-wide autozygosity in the Austrian fleckvieh cattle[J]. Agric Conspec Sci, 2011, 76(4): 325-329. |
| [10] |
MEHRNUSH F, MAHYARI S A, BAES C, et al. Inbreeding and runs of homozygosity before and after genomic selection in North American Holstein cattle[J]. BMC Genomics, 2018, 19(1): 98. DOI:10.1186/s12864-018-4453-z |
| [11] |
赵桂苹, 陈继兰, 文杰, 等. 北京油鸡品种资源特性描述[J]. 中国家禽, 2005, 27(23): 49-50. ZHAO G P, CHEN J L, WEN J, et al. Breed characteristic description of Beijing fatty chicken[J]. China Poultry, 2005, 27(23): 49-50. DOI:10.3969/j.issn.1004-6364.2005.23.020 (in Chinese) |
| [12] |
LI H, DURBIN R. Fast and accurate short read alignment with burrows-wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760. DOI:10.1093/bioinformatics/btp324 |
| [13] |
MCKENNA A, HANNA M, BANKS E, et al. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Res, 2010, 20(9): 1297-1303. DOI:10.1101/gr.107524.110 |
| [14] |
PURCELL S, NEALE B, TODD-BROWN K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [15] |
BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels[J]. Am J Hum Genet, 2018, 103(3): 338-348. DOI:10.1016/j.ajhg.2018.07.015 |
| [16] |
BARBATO M, OROZCO-TERWENGEL P, TAPIO M, et al. SNeP: a tool to estimate trends in recent effective population size trajectories using genome-wide SNP data[J]. Front Genet, 2015, 6: 109. |
| [17] |
CORBIN L J, LIU A Y H, BISHOP S C, et al. Estimation of historical effective population size using linkage disequilibria with marker data[J]. J Anim Breed Genet, 2012, 129(4): 257-270. DOI:10.1111/j.1439-0388.2012.01003.x |
| [18] |
DO C, WAPLES R S, PEEL D, et al. NeEstimator v2:re-implementation of software for the estimation of contemporary effective population size (Ne) from genetic data[J]. Mol Ecol Resour, 2014, 14(1): 209-214. DOI:10.1111/1755-0998.12157 |
| [19] |
WAPLES R S, DO C. Linkage disequilibrium estimates of contemporary Ne using highly variable genetic markers: a largely untapped resource for applied conservation and evolution[J]. Evol Appl, 2010, 3(3): 244-262. DOI:10.1111/j.1752-4571.2009.00104.x |
| [20] |
LENCZ T, LAMBERT C, DEROSSE P, et al. Runs of homozygosity reveal highly penetrant recessive loci in schizophrenia[J]. Proc Natl Acad Sci U S A, 2007, 104(50): 19942-19947. DOI:10.1073/pnas.0710021104 |
| [21] |
VANRADEN P M. Efficient methods to compute genomic predictions[J]. J Dairy Sci, 2008, 91(11): 4414-4423. DOI:10.3168/jds.2007-0980 |
| [22] |
吴淑琴. 群体有效大小和留种方式对保种的影响[J]. 张家口农专学报, 1996, 12(4): 56-58. WU S Q. The effect of effective population size and breeders selection manners on breeds conservation[J]. Journal of Zhangjiakou Agricultural College, 1996, 12(4): 56-58. (in Chinese) |
| [23] |
杨湛澄, 闫青霞, 黄河天, 等. 中国荷斯坦公牛系谱完整性研究[J]. 中国奶牛, 2017(2): 22-25. YANG Z C, YAN Q X, HUANG H T, et al. Evalutation of pedigree completeness in Chinese Holstein bulls[J]. China Dairy Cattle, 2017(2): 22-25. (in Chinese) |
| [24] |
WELLS D A, CANT M A, NICHOLS H J, et al. A high-quality pedigree and genetic markers both reveal inbreeding depression for quality but not survival in a cooperative mammal[J]. Mol Ecol, 2018, 27(9): 2271-2288. DOI:10.1111/mec.14570 |
| [25] |
ZHANG Q Q, CALUS M P L, GULDBRANDTSEN B, et al. Estimation of inbreeding using pedigree, 50k SNP chip genotypes and full sequence data in three cattle breeds[J]. BMC Genet, 2015, 16: 88. |
| [26] |
SAURA M, FERNÁNDEZ A, VARONA L, et al. Detecting inbreeding depression for reproductive traits in Iberian pigs using genome-wide data[J]. Genet Sel Evol, 2015, 47(1): 1. DOI:10.1186/s12711-014-0081-5 |
| [27] |
HOWRIGAN D P, SIMONSON M A, KELLER M C. Detecting autozygosity through runs of homozygosity: a comparison of three autozygosity detection algorithms[J]. BMC Genomics, 2011, 12: 460. DOI:10.1186/1471-2164-12-460 |
| [28] |
PERIPOLLI E, STAFUZZA N B, MUNARI D P, et al. Assessment of runs of homozygosity islands and estimates of genomic inbreeding in Gyr (Bos indicus) dairy cattle[J]. BMC Genomics, 2018, 19(1): 34. DOI:10.1186/s12864-017-4365-3 |
| [29] |
LEUTENEGGER A L, PRUM B, GÉNIN E, et al. Estimation of the inbreeding coefficient through use of genomic data[J]. Am J Hum Genet, 2003, 73(3): 516-523. DOI:10.1086/378207 |
| [30] |
WOODS C G, VALENTE E M, BOND J, et al. A new method for autozygosity mapping using single nucleotide polymorphisms (SNPs) and EXCLUDEAR[J]. J Med Genet, 2004, 41(8): e101. DOI:10.1136/jmg.2003.016873 |
| [31] |
MASTRANGELO S, SARDINA M T, TOLONE M, et al. Genome-wide identification of runs of homozygosity islands and associated genes in local dairy cattle breeds[J]. Animal, 2018, 12(12): 2480-2488. DOI:10.1017/S1751731118000629 |
| [32] |
WILLIAMS J L, HALL S J G, DEL CORVO M, et al. Inbreeding and purging at the genomic Level: the Chillingham cattle reveal extensive, non-random SNP heterozygosity[J]. Anim Genet, 2016, 47(1): 19-27. DOI:10.1111/age.12376 |
| [33] |
SHI L Y, WANG L G, LIU J X, et al. Estimation of inbreeding and identification of regions under heavy selection based on runs of homozygosity in a Large White pig population[J]. J Anim Sci Biotechnol, 2020, 11: 46. DOI:10.1186/s40104-020-00447-0 |
| [34] |
LUKIĆ B, FERENČAKOVIĆ M, ŠALAMON D, et al. Conservation genomic analysis of the Croatian indigenous black slavonian and turopolje pig breeds[J]. Front Genet, 2020, 11: 261. DOI:10.3389/fgene.2020.00261 |
| [35] |
GORSSEN W, MEYERMANS R, BUYS N, et al. SNP genotypes reveal breed substructure, selection signatures and highly inbred regions in Piétrain pigs[J]. Anim Genet, 2020, 51(1): 32-42. DOI:10.1111/age.12888 |
| [36] |
PERIPOLLI E, STAFUZZA N B, MUNARI D P, et al. Assessment of runs of homozygosity islands and estimates of genomic inbreeding in Gyr (Bos indicus) dairy cattle[J]. BMC Genomics, 2018, 19(1): 34. DOI:10.1186/s12864-017-4365-3 |
| [37] |
ALEMU S W, KADRI N K, HARLAND C, et al. An evaluation of inbreeding measures using a whole-genome sequenced cattle pedigree[J]. Heredity (Edinb), 2021, 126(3): 410-423. DOI:10.1038/s41437-020-00383-9 |
| [38] |
曹愉夏, 韩威, 李国辉, 等. 基于RAD-seq简化基因组测序估算狼山鸡分子亲缘系数和近交系数[J]. 中国家禽, 2019, 41(10): 14-19. CAO Y X, HAN W, LI G H, et al. Estimation of genomic kinship coefficients and inbreeding coefficients based on rad-seq simplified genome sequencing data in Langshan chicken[J]. China Poultry, 2019, 41(10): 14-19. (in Chinese) |
(编辑 郭云雁)



