脊椎是哺乳动物整个躯体的框架及支柱,处于背部正中处。脊椎的前体是胚胎发育早期的体节(somites),体节是在胚胎发育时期沿胚前后轴方向形成的大小相同的重复片段[1]。由于遗传活动的振荡波(体节发生波),体节从前体节中胚层(PSM)顺次出现[2]。在哺乳动物绝大多数物种中,体节和脊椎的数量高度保守,猪[3]、羊[4]等哺乳动物的脊椎数存在变异。体节的形成与遗传振荡有关,遗传振荡似乎是PSM细胞固有的,在该区域由于存在一个体节振荡器(一系列相关基因的节律性表达域)使得细胞周期化的增殖分化,从而形成一个个体节[5]。体节作为脊椎的前体这一重要结构,在一定程度上决定了椎骨数量,研究它的形成、分化过程及调控通路尤为重要,体节形成后,根据每个区域中体节细胞的位置,它们将分化成生骨节、生肌节和生皮节。体节产生的细胞将构成脊柱、肋骨、皮肤真皮和骨骼肌以及体壁和四肢。Gomez和Pourquié[6]研究报道了控制体节总数的因素:1) 在体节发育过程中对体轴后方生长的控制,在较长的体轴上可以形成更多相同大小的体节;2) 体节大小,在相同大小的体轴上可以形成更多较小的体节。然而具体的体节分节机制尚未可知,研究表明,时钟波峰模型是被广泛接受的体节形成模型,而且受多种信号通路的调控[7-9]。振荡的遗传时钟控制着体节这一胚胎结构的发育,该结构产生了保护脊髓的椎骨[10]。脊椎是通过上一个体节后部的生骨节与下一个体节前部的生骨节融合形成的[11]。总之,椎骨总数取决于中胚层体节数,通过研究脊椎的胚胎发育过程、体节分节机制及控制体节总数的因素有利于探索脊椎数变异的调控机制。
1 胚胎发育期脊椎及体节的形成 1.1 脊椎的形成脊椎是由脊椎动物胚胎发育早期形成的体节发育而来,体节是在胚胎发育时期沿胚前后轴方向形成的大小相同的重复片段[1]。对于高级哺乳动物来说,正常成熟的椎体由受精卵卵裂成多细胞,经过囊胚期进入原肠胚时期。原肠胚包括内胚层、中胚层和外胚层等3个胚层。胚胎中胚层[12]在骨形成蛋白生长因子(BMP)的浓度控制下分化为背部、轴旁、中间、侧板和头部中胚层。轴旁中胚层形成轴旁中胚层带,在鸟类中称为体节板,在哺乳类中称为不分节中胚层,随着原条退化和神经褶在胚胎中央合拢, 轴旁中胚层分隔成细胞模块,称为体节[13]。在次级神经胚形成时期,轴旁中胚层所形成的体节是脊椎的前体结构。
体节是脊椎动物在胚胎发育的过程中沿身体前后轴形成一定数目的暂时性结构,但是在组建脊椎动物胚胎的分节模式中非常重要。第一对体节在胚胎前端形成,新体节以规则的间隔从吻端轴旁中胚层萌发。由于胚胎发育速度略有差异,因此,体节数目通常是发育进程最佳指标,所形成体节总数具有种的特异性。随着胚胎继续发育形成体节,根据各区体节细胞的位置,体节将分化为生骨节、生皮节和生肌节。生骨节是来自体节的中胚层细胞块,将分化为脊柱的椎骨软骨生骨节,随后发育为脊椎软骨细胞,进而形成脊椎、肋骨、椎间软骨及韧带等结构[14]。脊椎是通过上一个体节后部的生骨节与下一个体节前部的生骨节融合形成的[11]。在两个胸部椎体之间衍生出左右一对肋骨。因此,体节数越多脊椎数和肋骨数也会越多。
1.2 体节的位置标识如果胚胎发育过程中体节发育分化出现紊乱,会导致成熟个体脊椎畸形及肋骨融合缺陷。对鸡胚的研究[14-15]显示,体节是由前体细胞群组成的临时器官,可形成重要的身体结构, 体节成对出现在神经管的两侧和胚胎的脊索上。体节板细胞组织形成轮状,称为体节小体(somitomere)。随着最前端的体节小体变成致密结构,体节小体便转变成体节,它们分化形成相同的细胞类型,由这些细胞形成的结构根据它们在前后轴的位置而发生变化,这点在脊椎的结构中体现最明显。例如,胸椎含有肋骨,而腰椎和荐椎没有。这说明体节沿着前后轴具有唯一的位置标识(在轴旁中胚层形成后不久就建立了)。研究表明,时间共线性导致早期脊椎动物发育期间的空间共线性[16],在早期的鸡和斑马鱼胚胎中[17]通过反BMP信号将动态变化的时间共线Hox序列转换为A-P位置信息的相对稳定的空间共线轴向Hox模式。鸡胃中的时间共线性决定了原始条纹细胞迁移到结点的顺序[18],动态变化的原始条纹细胞群体之间发生了相互作用。Hox基因簇在小鸡体内的时间共线序列中被激活,但是这些基因在小鼠胚胎中激活的确切时间的信息尚不完整[19],在鸡胚化过程中Hox的时间共线性在发育过程中产生了位置信息(例如前肢位置)[20]。
2 调控体节发育的关键信号通路体节的形成及分化涉及多个基因家族和信号通路,其中包括BMP信号通路、Hox基因家族及其信号通路、Wnt信号通路、FGF信号通路及RA信号通路和Notch信号通路等,这些信号通路及基因通过协同合作对体节的形成进行周期性调控,以确保其正常的形成及分化。
2.1 BMP信号通路骨形态发生蛋白(bone morphogenetic protein,BMP)是一大类分泌型生长因子的统称,BMP以其引起骨分化的能力和与细胞表面受体相互作用的分泌蛋白而得名[21]。BMPR1A是BMP通路的关键受体,BMPR1A介导的信号通路对成骨细胞的数量有一定影响。非特异性敲除BMPR1A基因小鼠在胚胎期因中胚层发育受阻引起死亡[22]。Yu等[23]研究发现,骨形态发生蛋白Ⅰ型受体的抑制会减少异位骨化,使用不同的启动子于成骨细胞中特异性敲除BMPR1A后可导致骨量增加,而Mishina等[24]研究表明,在小鼠出生后破坏BMPR1A的特异性,会导致突变小鼠的骨量减少,但成骨细胞数量正常,这表明,BMP在包括骨骼发育等多种发育过程中起重要作用。
外胚层中的BMP信号对于预期的轴旁中胚层的正确募集和体节的发育是必需的。BMPR1A编码骨形态发生蛋白(BMPs)的IA型BMP受体,包括BMP2和BMP4[25]。Miura等[26]在小鼠胚胎(Bmpr-More胚胎)的外胚层中使用BMPR1A的嵌合体失活来评估该基因在中胚层发育中的功能。与不能形成原肠胚的BMPR1A缺失型胚胎不同,Bmpr-More胚胎启动原肠形成,但预期的轴旁中胚层细胞向原始条纹的募集被推迟。抑制FGF信号可以恢复预期的轴旁中胚层募集的正常时间,促进体节的正常发育。这表明,BMP和FGF信号在近轴向中胚层发育过程中起拮抗作用,促进中胚层的正常发育。研究表明,FGFR3抑制了BMPR1A介导的成软骨细胞分化,FGFR3诱导骨形态发生蛋白Ⅰ型受体降解来调节骨骼发育[27]。
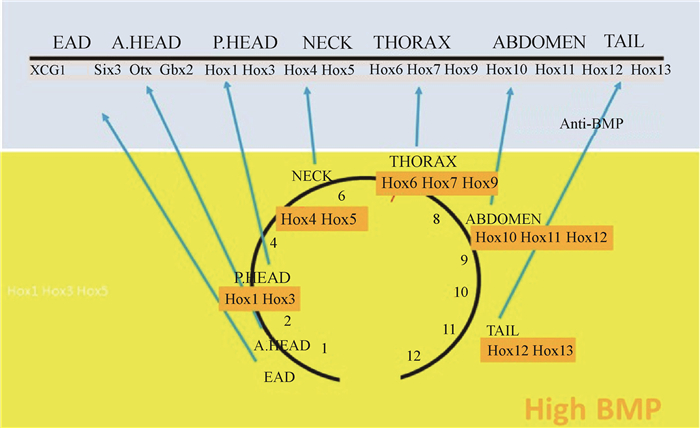
2.2 Hox基因家族及其信号通路同源异型复合基因(Hox)家族在脊椎模式形成和各部分属性决定过程中发挥了重要作用[28]。Hox基因家族可分为4个簇,即Hoxa、Hoxb、Hoxc和Hoxd,分别位于不同染色体上,每个簇又有13个旁系基因, 按顺序1~13排列,沿脊椎动物前后轴呈现出重叠的表达区域,形成了“HOX密码”,为每个椎骨分配属性形态(图 1)[21, 29]。
|
图 1 Hox基因家族调控脊椎属性模式示意图[21] Fig. 1 Schematic diagram of Hox gene family regulates the attribute pattern of the spine[21] |
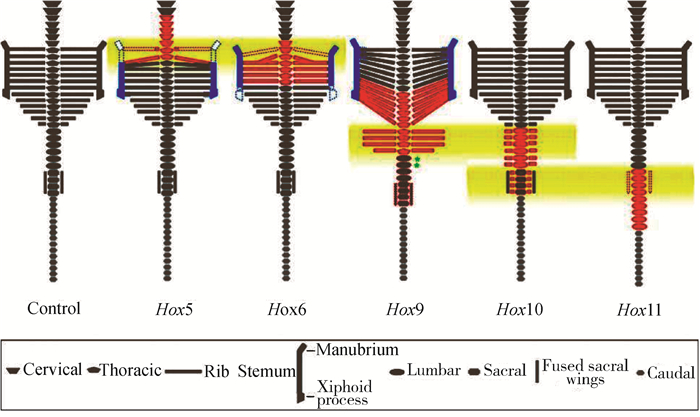
研究表明,Hox基因家族基因Hoxc8、Hox9、Hox10和Hox11均与脊椎骨同源异型转变相关。Hox基因在前后轴之间形成图案,并有助于确定沿该轴的位置。如果敲除Hox基因,可能会出现特定片段的畸形。同样,引起Hox基因异位表达可以改变体轴。Hoxc8在小鼠体内的表达失控会导致几种骨骼缺陷,如胸椎的同源异型转化和肋骨发育异常。Hoxc8控制最后一根胸椎的正常发育,删除Hoxc8的关键增强子会导致小鼠第一腰椎转化成额外的一节胸椎,相应的增加一对肋骨数。在胚胎晚期和出生后,Hoxc8通过调节BMP和Wnt信号通路中的关键基因参与成骨细胞的分化[30]。Hox9具有抑制肋骨过度发育的功能,敲除小鼠的Hox9基因,脊椎总数无变化,但是肋骨数明显增加。小鼠在没有Hox10基因功能的情况下,不会形成腰椎,取而代之的是从最后一节胸椎到荐椎均生出左右肋骨。在没有Hox11基因功能的情况下,不形成荐椎,并且这些椎骨转化成腰椎(图 2)[31-32]。Hox基因的表达受其上游FGF、Wnt、Notch和RA等信号通路的调控[11, 33]。
|
图 2 同源Hox轴向骨架表型的示意图[32] Fig. 2 Schematic representation of paralogous Hox axial skeleton phenotypes[32] |
Wnt信号传导途径是由配体蛋白质Wnt和膜蛋白受体结合激发的一组多下游通道的信号转导途径。经此途径,通过细胞表面受体胞内段的活化过程将细胞外的信号传递到细胞内。在调控体节形成方面,Wnt信号通路主要与Notch信号通路相互影响。椎骨源于前体节中胚层(PSM)分割产生的体节。Aulehla等[34]建立了Wnt/β-catenin信号和分节时钟之间的新型连接。Wnt通路的负调控因子Axin2直接受Wnt/β-catenin的控制,即使在Notch信号受损的情况下,PSM也会在LSMng表达中振荡表达。此外,Wnt3a是在PSM中振荡Notch信号活动所必需的,该研究表明,分节时钟是通过Wnt/β-catenin信号经由负反馈机制建立的,并且Wnt3a会控制脊椎动物的分节过程。当Wnt3a发生突变时,会影响体节分节,Wnt通路的负调控因子Axin2的表达相应降低。Chu等[35]从人类胚胎干细胞衍生的分节时钟模型在体内显示了哺乳动物分节时钟的许多标志,包括对Notch和Wnt信号通路的依赖性。Wnt的激活和转化生长因子β(TGF-β)的抑制是传播HES7振荡所必需的。这些结果表明,在发育中的新生人类体节中,TGF-β信号通路下调[36]。
2.4 FGF信号通路成纤维细胞生长因子(fibroblast growth factor, FGF)是一个18种分泌配体的家族[37],它可以促进中胚层细胞的生长,与体轴的建立和体节的形成等有关。中胚层定期切成体节(椎骨的前体)是由分子钟控制的,该钟在小鼠胚胎中存在FGF通路活性的超周期振荡(周期约2 h)[38]。Dubrulle等[39]研究表明,FGF8在后PSM中的表达产生了一个可以确定体节的边界位置和轴向同一性的移动波前,FGF8信号还可以影响体节的大小,呈负反馈调节。此外,通过控制鸡胚的边界位置发现,Hox基因的表达维持在适当编号的体节上,而不是绝对的轴向位置。这些结果表明,FGF8在确保分节过程和时空Hox基因激活的紧密协调中起作用,同时,FGF信号通路对体节分节的调控是通过与Notch、Wnt等信号通路共同作用完成的。Naoki等[40]发现,PSM中的形态发生子梯度起波前作用,其中,FGF和Wnt信号产生的梯度在前PSM处减弱,并随着尾巴伸长而不断向后端移动。
2.5 RA信号通路RA(视黄酸)指的是维生素A的体内活性代谢产物,可以调控基因表达,与神经系统的发育、体节的左右对称发生和脊椎动物体轴的延伸相关。研究表明,将小鼠妊娠第8天的胚胎暴露于RA中,第一或第二个腰椎被转变成胸椎(肋骨),而第一个荐椎通常变成腰椎。这些结构变化与Hox基因表达的位置相关。当Hoxa-10的表达向后移动,将会在第一节腰椎上形成额外一对肋骨[41]。Kawakami等[42]通过描述斑马鱼的胚胎发育,表明了阻止RA信号(在它到达侧板中胚层之前)会导致随机的左右不对称的体细胞。此外,揭示了一种由RA信号介导的机制,阻断RA信号或RA敲降后,会打破斑马鱼体节左右对称生成的模式,但RA信号的下降会影响6~13体节,从而表明,RA对两侧对称的体细胞发生方面起着至关重要的作用。Vermot和Pourquié[43]在鸡胚中研究报道,阻断鸡胚中RA的产生导致两个胚胎两侧的体节形成不同步,这表现在一个缩短的左节段区域。该缺陷与分节时钟振荡的协调丢失有关。在缺乏RA的鸡或小鼠胚胎[44]中,位置反转会导致体细胞发生偏侧缺陷的反转。RA以时间特定的方式调节对Wnt和BMP的响应,RA信号对于前肠祖细胞响应Wnt和BMP信号激活呼吸命运至关重要[45]。
2.6 Notch信号通路Notch信号通路广泛存在于脊椎动物和非脊椎动物,在进化上高度保守,在神经细胞的分化、脊椎动物体节的发育等过程中具有重要作用。Notch通路内基因的周期性表达是体节正常发育所必需的。Keskin等[46]研究表明,脊椎动物分节时钟控制着脊柱的精确时空模式。通过计数斑马鱼中的分节时钟基因的单个转录物表明,时钟基因具有较低的RNA幅度,并且表达变异性主要是由基因外源驱动的,而Notch信号通路则抑制了这种表达。Notch信号传导在控制体节从增殖到分化的转换中都起着至关重要的作用。Hes/her的家族基因是Notch信号传导的直接靶标。Carrieri等[47]通过先天性脊柱侧凸连锁分析显示6个基因(DLL3、MESP2、LFNG、HES7、TBX6和RIPPLY2)的突变,其中4个是Notch通路的组成部分(DLL3、MESP2、LFNG、HES7),Notch对小鼠的分节过程至关重要,因为在没有Notch信号的情况下,分节时钟停止并且没有9突节。在PSM的单个细胞水平上,通过不稳定时钟基因产物的正反馈和负反馈回路来建立振荡时钟基因表达,会增强或抑制激活它们的通路。相邻细胞之间时钟基因振荡的同步依赖于Notch信号10、11、12和13。其他与体节相关的通路内基因对Notch通路信号也会产生影响,如FGF及Wnt通路。
3 体节形成的经典模型研究表明,体节分节模型包括时钟波峰模型(clock and wavefront model)[48]、反应扩散模型(reaction-diffusion type model)[49]、沿PSM移动的细胞决定波(wave of cell determination)[50]和时钟诱导模型(clock and induction model)[51]等。但是部分模型具有一定的局限性,时钟波峰模型是目前被广泛接受的体节形成模型。
3.1 时钟波峰模型体节的形成具有严格的节律性,称为分节时钟。是由时钟基因Hes7(一种Hes家族转录因子)的周期性表达以及形态素Notch、FGF、Wnt、RA等的浓度梯度决定的周期性激活[48, 52-53],这种周期性被归纳为时钟波峰模型,由时钟和波前信号组成,时钟信号提供周期性定时的时间信息,而波前信号提供恒定距离的空间信息,当时钟和波前信号相遇时,PSM单元开始分化成体节。PSM细胞中的时钟基因振荡通过Delta-Notch信号同步[54]。体节的空间周期性来源于PSM中振荡基因表达的时间周期性。体节分割间隔因物种不同而异,例如,小鼠为120 min,雏鸡为90 min,斑马鱼为30 min[55],人类约5 h[56]。这些时间间隔与时钟基因的振荡周期一致:小鼠中的时钟基因是Hes7和Lfng[1],在小鼠胚胎中,此周期性受分段时钟基因Hes7的控制,该基因在PSM中表现出波状振荡表达[57]。雏鸡中是Hairy1和Hairy2,斑马鱼中是Her1和Her7和DeltaC(Notch配体)。Cooke和Zeeman[9]提出,时钟波峰模型是目前被广泛接受的体节形成模型,后来被试验确定为分节时钟[58],分节时钟(图 3)作为一种分子振荡器控制着脊椎动物的体细胞发生[59]。体节的周期性生成与前体节中胚层中同步基因表达的振荡模式密切相关,这种基因表达的模式即分节时钟。这种模型受到多种信号通路的共同调节,这些通路中的基因形成一个周期性表达的沿前体节中胚层传递的表达波谱,共同影响体节的延长和分节。当决定前沿经过前体节中胚层细胞后,该区域的前体节中胚层细胞就可以应答分节时钟传递的周期性分节信号[39]。以脊椎动物的分节为例,Goldbeter等[60]论证了RA和FGF沿卵裂期前中胚层(PSM)的拮抗梯度可能导致两个稳定的稳态共存。该研究认为,这种双稳态与FGF和RA信号水平的突变有关,这允许PSM细胞连续队列中的分节基因(如mesp2)响应于分节时钟而同步激活,从而影响体节的分节。研究表明,RA受体与其配体基因在轴旁中胚层显著表达,对体节形成过程中的分节时钟和Hox基因的表达模式进行调控。RA和FGF相互抑制产生的双稳态为时钟和波前体节发生模型中假定的全部或没有过渡提供了分子机制。鉴于相互对立的信号梯度在发育中很常见,因此,这种双稳态开关可能代表了胚胎模式背后的重要原理[60]。细胞通过激活片段宽域中的MESP基因来响应来自片段时钟的周期性信号[61],MESP基因的表达受到高FGF信号的抑制[62],并且需要RA信号才能转录,从而解释了它们在决定前沿水平的限制表达[63]。
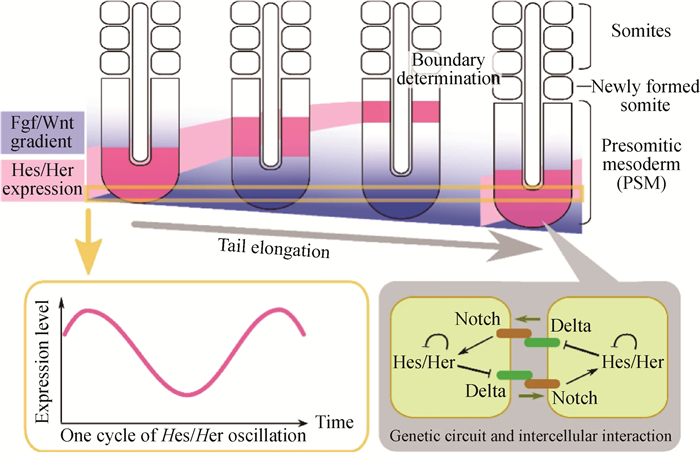
|
上图:PSM中Hes/Her基因的表达动态和Fgf/Wnt梯度。在神经管的两侧周期性地形成体节。左下图:Hes/Her基因在PSM中显示出振荡表达。右下图:Hes/Her基因的振荡是由细胞自主的负反馈回路驱动的,并通过Delta/Notch信号与细胞间相互作用耦合 Upper panel: Expression dynamics of Hes/Her genes and the Fgf/Wnt gradient in the PSM. Somites are periodically formed on both sides of the neural tube. Lower left panel: Hes/Her genes show oscillatory expression within the PSM. Lower right panel: the oscillation of Hes/Her genes is driven by the cell-autonomous negative feedback loop and is coupled to cell-cell interactions via Delta/Notch signaling 图 3 脊椎动物分节时钟的示意图[59] Fig. 3 Schematic view of the vertebrate segmentation clock[59] |
分节时钟控制着体节的周期性形成,体节是沿着身体轴线重复的结构,形成脊椎和肋骨。在体节分割过程中,时钟基因(如Hes7)的表达在早熟中胚层(PSM)中同步振荡。该同步振荡在前PSM处减慢,导致从后PSM到前PSM呈波状传播,这种动态表达取决于Notch信号传导,并且对于形成体节至关重要[64]。Sparrow等[65]研究表明,这个时钟系统的核心是Hes或Her基因(Hes7)的周期性表达,同时是PSM中Notch、FGF和Wnt信号通路的周期性激活[66-67]。Yoshioka-Kobayashi等[1]研究表明,个体的细胞活动是波动的但又不断地通过细胞-细胞耦合在种群水平上不断协调。典型的例子是体节分节时钟,其中时钟基因(如Hes7)的表达在组成分裂前中胚层的细胞之间同步振荡。这种同步化依赖于Notch信号通路;抑制这一通路会使振荡失去同步性,导致体节融合3~7节。Hubaud等[68]证明,PSM细胞可以以静止状态存在,并且可以响应于涉及Notch和Yap信号的细胞密度相关信号而切换到振荡状态。该研究发现了可激发振荡器模型,该模型超出了先前假定的耦合相位振荡器的范围,并成为分节时钟的更通用模型。在小鼠PSM中,转录阻遏物Hes7的表达同步振荡,像行波一样从后部传播到前部区域,并且每个波都导致体节分节[69-70]。Hes7振荡受延迟的负反馈调控:Hes7蛋白在延迟的时间抑制其自身的表达,而这种抑制作用下调Hes7的蛋白表达,从而导致表达重新激活[71-72]。Hes7的表达缺失和稳定表达都会导致严重的体节融合[73-74],而更快的Hes7振荡会加速分节的速度[75],表明Hes7振荡是小鼠分节时钟的基础。总之,Hes7基因缺失会导致体节分割紊乱,且其前后极性被破坏,诸如椎骨和肋骨之类的松节体衍生物严重地杂乱无章,因此Hes7基因对于协调节段分割至关重要。尽管进行了如此深入的研究,但振荡动力学的确切机理仍有待分析。例如,Hes7振荡在相邻的PSM单元之间同步,但是同步的确切机制未知。
4 展望探索脊椎的胚胎发育过程对脊椎数变异的研究有很大的帮助,目前研究表明,椎骨总数取决于中胚层体节数,体节数又受BMP、Hox、Notch、Wnt、FGF及RA信号通路调控。然而,脊椎动物体节的具体调控机制仍然未知,目前只有研究表明,Hox基因是调节体轴生长的候选基因,是脊椎各部分的形态属性的决定因子,体节数量固定后,脊椎模式的形成可由Hox基因的表达调控。把Hox基因表达图式和不同类型脊椎动物比较, 可以看出,脊椎动物类型是由Hox在体节中的表达格局(constellation)决定的。对于体节的具体影响因素和调控机理以及体节影响脊椎数变异的机理仍需要进一步研究。
| [1] | YOSHIOKA-KOBAYASHI K, MATSUMIYA M, NIINO Y, et al. Coupling delay controls synchronized oscillation in the segmentation clock[J]. Nature, 2020, 580(7801): 119–123. DOI: 10.1038/s41586-019-1882-z |
| [2] | DURSTON A J, PERES J, COHEN M H. Spiral waves and vertebrate embryonic handedness[J]. J Biosci, 2018, 43(2): 375–390. DOI: 10.1007/s12038-018-9756-3 |
| [3] | DUAN Y Y, ZHANG H, ZHANG Z, et al. Vrtn is required for the development of thoracic vertebrae in mammals[J]. Int J Biol Sci, 2018, 14(6): 667–681. DOI: 10.7150/ijbs.23815 |
| [4] | LI C Y, LI M, LI X Y, et al. Whole-genome resequencing reveals loci associated with thoracic vertebrae number in sheep[J]. Front Genet, 2019, 10: 674. DOI: 10.3389/fgene.2019.00674 |
| [5] | NELEMANS B K A, SCHMITZ M, TAHIR H, et al. Somite division and new boundary formation by mechanical strain[J]. iScience, 2020, 23(4): 100976. DOI: 10.1016/j.isci.2020.100976 |
| [6] | GOMEZ C, POURQUIÉ O. Developmental control of segment numbers in vertebrates[J]. J Exp Zool B Mol Dev Evol, 2009, 312(6): 533–544. |
| [7] | DEQUÉANT M L, POURQUIÉ O. Segmental patterning of the vertebrate embryonic axis[J]. Nat Rev Genet, 2008, 9(5): 370–382. DOI: 10.1038/nrg2320 |
| [8] | VROOMANS R M A, HOGEWEG P, TEN TUSSCHER K H W J. Around the clock: gradient shape and noise impact the evolution of oscillatory segmentation dynamics[J]. EvoDevo, 2018, 9: 24. DOI: 10.1186/s13227-018-0113-2 |
| [9] | COOKE J, ZEEMAN E C. A clock and wavefront model for control of the number of repeated structures during animal morphogenesis[J]. J Theor Biol, 1976, 58(2): 455–476. DOI: 10.1016/S0022-5193(76)80131-2 |
| [10] | PALLA A, BLAU H. The clock that controls spine development modelled in a dish[J]. Nature, 2020, 580(7801): 32–34. DOI: 10.1038/d41586-020-00322-y |
| [11] | ALEXANDER T, NOLTE C, KRUMLAUF R. Hox genes and segmentation of the hindbrain and axial skeleton[J]. Annu Rev Cell Dev Biol, 2009, 25: 431–456. DOI: 10.1146/annurev.cellbio.042308.113423 |
| [12] | ANDERSON M J, SCHIMMANG T, LEWANDOSKI M. An FGF3-BMP signaling axis regulates caudal neural tube closure, neural crest specification and anterior-posterior axis extension[J]. PLoS Genet, 2016, 12(5): e1006018. DOI: 10.1371/journal.pgen.1006018 |
| [13] | DIAZ-CUADROS M, WAGNER D E, BUDJAN C, et al. In vitro characterization of the human segmen-tation clock[J]. Nature, 2020, 580(7801): 113–118. DOI: 10.1038/s41586-019-1885-9 |
| [14] |
张红卫.
发育生物学[M]. 2版. 北京: 高等教育出版社, 2006.
ZHANG H W. Developmental biology[M]. 2nd ed. Beijing: Higher Education Press, 2006. (in Chinese) |
| [15] | AMINI Z, MAHDAVI-SHAHRI N, LARI R, et al. The effects of lead on the development of somites in chick embryos (Gallus gallus domesticus) under in vitro conditions: a histological study[J]. Toxicol Res (Camb), 2019, 8(3): 373–380. DOI: 10.1039/C8TX00340H |
| [16] | DURSTON A J. Two tier Hox collinearity mediates vertebrate axial patterning[J]. Front Cell Dev Biol, 2018, 6: 102. DOI: 10.3389/fcell.2018.00102 |
| [17] | HASHIGUCHI M, MULLINS M C. Anteroposterior and dorsoventral patterning are coordinated by an identical patterning clock[J]. Development, 2013, 140(9): 1970–1980. DOI: 10.1242/dev.088104 |
| [18] | DENANS N, IIMURA T, POURQUIÉ O. Hox genes control vertebrate body elongation by collinear Wnt repression[J]. Elife, 2015, 4: e04379. DOI: 10.7554/eLife.04379 |
| [19] | WYMEERSCH F J, SKYLAKI S, HUANG Y L, et al. Transcriptionally dynamic progenitor populations organised around a stable niche drive axial patterning[J]. Development, 2019, 146(1): dev168161. |
| [20] | MOREAU C, CALDARELLI P, ROCANCOURT D, et al. Timed collinear activation of Hox genes during gastrulation controls the avian forelimb position[J]. Curr Biol, 2019, 29(1): 35–50. DOI: 10.1016/j.cub.2018.11.009 |
| [21] | DURSTON A J. What are the roles of retinoids, other morphogens, and Hox genes in setting up the vertebrate body axis?[J]. Genesis, 2019, 57(7-8): e23296. |
| [22] | MISHINA Y, HANKS M C, MIURA S, et al. Generation of Bmpr/Alk3 conditional knockout mice[J]. Genesis, 2002, 32(2): 69–72. DOI: 10.1002/gene.10038 |
| [23] | YU P B, DENG D Y, LAI C S, et al. BMP type Ⅰ receptor inhibition reduces heterotopic ossification[J]. Nat Med, 2008, 14(12): 1363–1369. DOI: 10.1038/nm.1888 |
| [24] | MISHINA Y, STARBUCK M W, GENTILE M A, et al. Bone morphogenetic protein type ⅠA receptor signaling regulates postnatal osteoblast function and bone remodeling[J]. J Biol Chem, 2004, 279(26): 27560–27566. DOI: 10.1074/jbc.M404222200 |
| [25] | MISHINA Y. Function of bone morphogenetic protein signaling during mouse development[J]. Front Biosci, 2003, 8: d855–d869. DOI: 10.2741/1097 |
| [26] | MIURA S, DAVIS S, KLINGENSMITH J, et al. BMP signaling in the epiblast is required for proper recruitment of the prospective paraxial mesoderm and development of the somites[J]. Development, 2006, 133(19): 3767–3775. DOI: 10.1242/dev.02552 |
| [27] | QI H B, JIN M, DUAN Y Q, et al. FGFR3 induces degradation of BMP type Ⅰ receptor to regulate skeletal development[J]. Biochim Biophys Acta Mol Cell Res, 2014, 1843(7): 1237–1247. DOI: 10.1016/j.bbamcr.2014.03.011 |
| [28] | BÖHMER C. Correlation between Hox code and vertebral morphology in the mouse: towards a universal model for synapsida[J]. Zoological Lett, 2017, 3: 8. DOI: 10.1186/s40851-017-0069-4 |
| [29] | KESSEL M, GRUSS P. Homeotic transformations of murine vertebrae and concomitant alteration of Hox codes induced by retinoic acid[J]. Cell, 1991, 67(1): 89–104. DOI: 10.1016/0092-8674(91)90574-I |
| [30] | JUAN A H, LEI H, BHARGAVA P, et al. Multiple roles of Hoxc8 in skeletal development[J]. Ann N Y Acad Sci, 2006, 1068(1): 87–94. DOI: 10.1196/annals.1346.046 |
| [31] | WELLIK D M, CAPECCHI M R. Hox10 and Hox11 genes are required to globally pattern the mammalian skeleton[J]. Science, 2003, 301(5631): 363–367. DOI: 10.1126/science.1085672 |
| [32] | MCINTYRE D C, RAKSHIT S, YALLOWITZ A R, et al. Hox patterning of the vertebrate rib cage[J]. Development, 2007, 134(16): 2981–2989. DOI: 10.1242/dev.007567 |
| [33] | ZÁKÁNY J, KMITA M, ALARCON P, et al. Localized and transient transcription of Hox genes suggests a link between patterning and the segmentation clock[J]. Cell, 2001, 106(2): 207–217. DOI: 10.1016/S0092-8674(01)00436-6 |
| [34] | AULEHLA A, WEHRLE C, BRAND-SABERI B, et al. Wnt3a plays a major role in the segmentation clock controlling somitogenesis[J]. Dev Cell, 2003, 4(3): 395–406. DOI: 10.1016/S1534-5807(03)00055-8 |
| [35] | CHU L F, MAMOTT D, NI Z J, et al. An in vitro human segmentation clock model derived from embryonic stem cells[J]. Cell Rep, 2019, 28(9): 2247–2255. DOI: 10.1016/j.celrep.2019.07.090 |
| [36] | XI H B, FUJIWARA W, GONZALEZ K, et al. In vivo human somitogenesis guides somite development from hPSCs[J]. Cell Rep, 2017, 18(6): 1573–1585. DOI: 10.1016/j.celrep.2017.01.040 |
| [37] | MORGANI S M, SAIZ N, GARG V, et al. A Sprouty4 reporter to monitor FGF/ERK signaling activity in ESCs and mice[J]. Dev Biol, 2018, 441(1): 104–126. DOI: 10.1016/j.ydbio.2018.06.017 |
| [38] | SONNEN K F, LAUSCHKE V M, URAJI J, et al. Modulation of phase shift between Wnt and notch signaling oscillations controls mesoderm segmentation[J]. Cell, 2018, 172(5): 1079–1090. DOI: 10.1016/j.cell.2018.01.026 |
| [39] | DUBRULLE J, MCGREW M J, POURQUIÉ O. FGF signaling controls somite boundary position and regulates segmentation clock control of spatio-temporal Hox gene activation[J]. Cell, 2001, 106(2): 219–232. DOI: 10.1016/S0092-8674(01)00437-8 |
| [40] | NAOKI H, AKIYAMA R, SARI D W K, et al. Noise-resistant developmental reproducibility in vertebrate somite formation[J]. PLoS Comput Biol, 2019, 15(2): e1006579. DOI: 10.1371/journal.pcbi.1006579 |
| [41] | GRUSS P, KESSEL M. Axial specification in higher vertebrates[J]. Curr Opin Genet Dev, 1991, 1(2): 204–210. DOI: 10.1016/S0959-437X(05)80071-1 |
| [42] | KAWAKAMI Y, RAYA Á, RAYA R M, et al. Retinoic acid signalling links left-right asymmetric patterning and bilaterally symmetric somitogenesis in the zebrafish embryo[J]. Nature, 2005, 435(7039): 165–171. DOI: 10.1038/nature03512 |
| [43] | VERMOT J, POURQUIÉ O. Retinoic acid coordinates somitogenesis and left-right patterning in vertebrate embryos[J]. Nature, 2005, 435(7039): 215–220. DOI: 10.1038/nature03488 |
| [44] | BRUECKNER M, D'EUSTACHIO P, HORWICH A L. Linkage mapping of a mouse gene, iv, that controls left-right asymmetry of the heart and viscera[J]. Proc Natl Acad Sci U S A, 1989, 86(13): 5035–5038. DOI: 10.1073/pnas.86.13.5035 |
| [45] | RANKIN S A, MCCRACKEN K W, LUEDEKE D M, et al. Timing is everything: reiterative Wnt, BMP and RA signaling regulate developmental competence during endoderm organogenesis[J]. Dev Biol, 2018, 434(1): 121–132. DOI: 10.1016/j.ydbio.2017.11.018 |
| [46] | KESKIN S, DEVAKANMALAI G S, KWON S B, et al. Noise in the vertebrate segmentation clock is boosted by time delays but tamed by notch signaling[J]. Cell Rep, 2018, 23(7): 2175–2185. DOI: 10.1016/j.celrep.2018.04.069 |
| [47] | CARRIERI F A, MURRAY P J, DITSOVA D, et al. CDK1 and CDK2 regulate NICD1 turnover and the periodicity of the segmentation clock[J]. EMBO Rep, 2019, 20(7): e46436. |
| [48] | NAOKI H, MATSUI T. Somite boundary determination in normal and clock-less vertebrate embryos[J]. Dev Growth Differ, 2020, 62(3): 177–187. DOI: 10.1111/dgd.12655 |
| [49] | HORMUTH D A, WEIS J A, BARNES S L, et al. A mechanically coupled reaction-diffusion model that incorporates intra-tumoural heterogeneity to predict in vivo glioma growth[J]. J R Soc Interface, 2017, 14(128): 20161010. DOI: 10.1098/rsif.2016.1010 |
| [50] | LI D L, HALLACK A, CLEVELAND R O, et al. 3D multicellular model of shock wave-cell interaction[J]. Acta Biomater, 2018, 77: 282–291. DOI: 10.1016/j.actbio.2018.04.041 |
| [51] | SCHNELL S, MAINI P K. Clock and induction model for somitogenesis[J]. Dev Dyn, 2000, 217(4): 415–420. DOI: 10.1002/(SICI)1097-0177(200004)217:4<415::AID-DVDY8>3.0.CO;2-3 |
| [52] | LIAO B K, OATES A C. Delta-Notch signalling in segmentation[J]. Arthropod Struct Dev, 2017, 46(3): 429–447. DOI: 10.1016/j.asd.2016.11.007 |
| [53] | PAIS-DE-AZEVEDO T, MAGNO R, DUARTE I, et al. Recent advances in understanding vertebrate segmentation [version 1; peer review: 3 approved][J]. F1000Res, 2018, 7: 97. DOI: 10.12688/f1000research.12369.1 |
| [54] | VENZIN O F, OATES A C. What are you synching about? Emerging complexity of notch signaling in the segmentation clock[J]. Dev Biol, 2020, 460(1): 40–54. DOI: 10.1016/j.ydbio.2019.06.024 |
| [55] | MORRISS-KAY G M, MURPHY P, HILL R E, et al. Effects of retinoic acid excess on expression of Hox-2.9 and Krox-20 and on morphological segmentation in the hindbrain of mouse embryos[J]. EMBO J, 1991, 10(10): 2985–2995. DOI: 10.1002/j.1460-2075.1991.tb07849.x |
| [56] | MATSUDA M, YAMANAKA Y, UEMURA M, et al. Recapitulating the human segmentation clock with pluripotent stem cells[J]. Nature, 2020, 580(7801): 124–129. DOI: 10.1038/s41586-020-2144-9 |
| [57] | MATSUMIYA M, TOMITA T, YOSHIOKA-KOBAYASHI K, et al. ES cell-derived presomitic mesoderm-like tissues for analysis of synchronized oscillations in the segmentation clock[J]. Development, 2018, 145(4): dev156836. |
| [58] | PALMEIRIM I, HENRIQUE D, ISH-HOROWICZ D, et al. Avian hairy gene expression identifies a molecular clock linked to vertebrate segmentation and somitogenesis[J]. Cell, 1997, 91(5): 639–648. DOI: 10.1016/S0092-8674(00)80451-1 |
| [59] | YOSHIOKA-KOBAYASHI K, KAGEYAMA R. Imaging and manipulating the segmentation clock[J]. Cell Mol Life Sci, 2021, 78(4): 1221–1231. DOI: 10.1007/s00018-020-03655-z |
| [60] | GOLDBETER A, GONZE D, POURQUIÉO. Sharp developmental thresholds defined through bistability by antagonistic gradients of retinoic acid and FGF signaling[J]. Dev Dyn, 2007, 236(6): 1495–1508. DOI: 10.1002/dvdy.21193 |
| [61] | MORIMOTO M, TAKAHASHI Y, ENDO M, et al. The Mesp2 transcription factor establishes segmental borders by suppressing Notch activity[J]. Nature, 2005, 435(7040): 354–359. DOI: 10.1038/nature03591 |
| [62] | DELFINI M C, DUBRULLE J, MALAPERT P, et al. Control of the segmentation process by graded MAPK/ERK activation in the chick embryo[J]. Proc Natl Acad Sci U S A, 2005, 102(32): 11343–11348. DOI: 10.1073/pnas.0502933102 |
| [63] | MORENO T A, KINTNER C. Regulation of segmental patterning by retinoic acid signaling during Xenopus somitogenesis[J]. Dev Cell, 2004, 6(2): 205–218. DOI: 10.1016/S1534-5807(04)00026-7 |
| [64] | KAGEYAMA R, SHIMOJO H, ISOMURA A. Oscillatory control of notch signaling in development[M]//BORGGREFE T, GIAIMO B D. Molecular Mechanisms of Notch Signaling. Cham: Springer, 2018: 265-277. |
| [65] | SPARROW D B, GUILLÉN-NAVARRO E, FATKIN D, et al. Mutation of HAIRY-AND-ENHANCER-OF-SPLIT-7 in humans causes spondylocostal dysostosis[J]. Hum Mol Genet, 2008, 17(23): 3761–3766. DOI: 10.1093/hmg/ddn272 |
| [66] | HUBAUD A, POURQUIÉ O. Signalling dynamics in vertebrate segmentation[J]. Nat Rev Mol Cell Biol, 2014, 15(11): 709–721. DOI: 10.1038/nrm3891 |
| [67] | OATES A C, MORELLI L G, ARES S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock[J]. Development, 2012, 139(4): 625–639. DOI: 10.1242/dev.063735 |
| [68] | HUBAUD A, REGEV I, MAHADEVAN L, et al. Excitable dynamics and Yap-dependent mechanical cues drive the segmentation clock[J]. Cell, 2017, 171(3): 668–682. DOI: 10.1016/j.cell.2017.08.043 |
| [69] | BESSHO Y, SAKATA R, KOMATSU S, et al. Dynamic expression and essential functions of Hes7 in somite segmentation[J]. Genes Dev, 2001, 15(20): 2642–2647. DOI: 10.1101/gad.930601 |
| [70] | NIWA Y, MASAMIZU Y, LIU T X, et al. The initiation and propagation of Hes7 oscillation are cooperatively regulated by Fgf and notch signaling in the somite segmentation clock[J]. Dev Cell, 2007, 13(2): 298–304. DOI: 10.1016/j.devcel.2007.07.013 |
| [71] | BESSHO Y, HIRATA H, MASAMIZU Y, et al. Periodic repression by the bHLH factor Hes7 is an essential mechanism for the somite segmentation clock[J]. Genes Dev, 2003, 17(12): 1451–1456. DOI: 10.1101/gad.1092303 |
| [72] | TAKASHIMA Y, OHTSUKA T, GONZÁLEZ A, et al. Intronic delay is essential for oscillatory expression in the segmentation clock[J]. Proc Natl Acad Sci U S A, 2011, 108(8): 3300–3305. DOI: 10.1073/pnas.1014418108 |
| [73] | HIRATA H, BESSHO Y, KOKUBU H, et al. Instability of Hes7 protein is crucial for the somite segmentation clock[J]. Nat Genet, 2004, 36(7): 750–754. DOI: 10.1038/ng1372 |
| [74] | SPARROW D B, CHAPMAN G, SMITH A J, et al. A mechanism for gene-environment interaction in the etiology of congenital scoliosis[J]. Cell, 2012, 149(2): 295–306. DOI: 10.1016/j.cell.2012.02.054 |
| [75] | HARIMA Y, TAKASHIMA Y, UEDA Y, et al. Accelerating the tempo of the segmentation clock by reducing the number of introns in the Hes7 gene[J]. Cell Rep, 2013, 3(1): 1–7. DOI: 10.1016/j.celrep.2012.11.012 |



