2. 山东农业大学动物科学技术学院, 泰安 271018
2. College of Animal Science and Technology, Shandong Agricultural University, Tai'an 271018, China
金黄色葡萄球菌(简称金葡菌)是奶牛乳腺炎常见和主要的致病菌之一,属于机会性环境致病菌,感染率较其他致病菌更高且存在多种表面致病因子[1-3],这些表面致病因子通常能够诱发免疫反应,是疫苗研发的关键[4-6]。其中,细胞壁锚定表面蛋白作为黏附素,在金葡菌的黏附过程中发挥重要作用。目前已知金葡菌能表达24种不同的细胞壁锚定表面蛋白[7]。与肽聚糖连接的表面蛋白前体经加工后从细菌生物被膜中释放出来,扩散到宿主组织中协助金葡菌定植[8-9],金葡菌能同时产生包括溶细胞毒素在内的多种外毒素,导致宿主体内强烈的炎症反应,引发奶牛乳腺炎,对奶牛养殖行业造成极大的影响[10]。
不同种的细菌黏附素种类存在差异,其中,丝氨酸富集重复蛋白(SRRP)是革兰阳性菌最常见的一种表面黏附素蛋白[11-12]。该家族蛋白前体N端均有1段非典型的信号肽,与信号肽相邻的片段依次为短丝氨酸重复区域(SRR1)、非重复序列区域(NRR)、长丝氨酸重复区域(SRR2)和1段相对保守的细胞壁锚定序列(LPXTG)(该序列被分选酶识别并在成熟过程中被剪接)[3, 13-16]。其中,NRR结构域对于金葡菌黏附性至关重要[17-18],而SRR2可能参与将SRR1及NRR延伸到细菌表面的过程[19]。SasA蛋白(又称为Srap)是金葡菌表面蛋白之一,属于SRRP家族,相对分子质量较大,由2 271个氨基酸组成。SasA的功能目前尚未完全研究清楚,但有研究结果表明,SasA能够黏附人血小板[20-21],且金葡菌表面蛋白A突变菌株与野生菌相比,侵染能力显著减弱[22]。同时,在家兔模型中观察到,该蛋白发挥血管感染毒力决定因子的作用[23]。目前,牛源金葡菌中也发现编码SasA蛋白的基因,这提示SasA蛋白作为表面抗原可成为哺乳动物金葡菌疫苗的新靶点。然而关于牛源金葡菌SasA蛋白与宿主细胞互作及在奶牛乳腺炎中致病作用的研究较少,因此,本试验重点研究该蛋白对奶牛乳腺上皮细胞的黏附作用。
本研究主要分为两部分,首先鉴定SasA基因在牛源金葡菌中的携带情况及序列相似性,再利用流式细胞仪测定SasA蛋白重组片段对奶牛乳腺上皮细胞的黏附性高低。
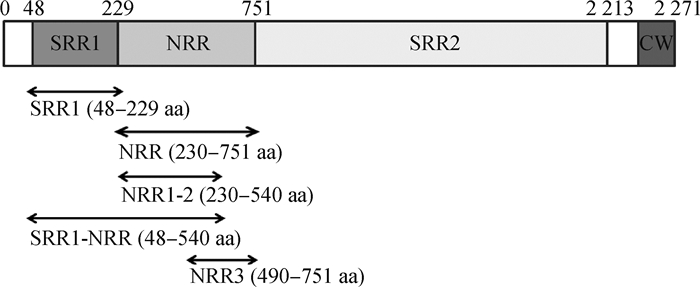
1 材料与方法 1.1 材料1.1.1 试验菌株与细胞 试验采用中国北方5个荷斯坦牛场随机取样分离纯化的73株牛源金葡菌菌株。目标片段表达载体为pET21a+重组质粒转化的BL21大肠杆菌菌株,由军事科学医学院生物工程研究所疫苗与抗体工程研究室筛选馈赠。NRR的各片段(NRR、NRR1-2、NRR3)及SRR1片段构建如图 1所示。体外培养的Mac-T细胞系由山东农业大学动物科学技术学院提供。
|
图 1 金黄色葡萄球菌不同区段NRR片段 Fig. 1 S. aureus NRR of different domains |
1.1.2 主要仪器 流式细胞仪(Life Technology公司), 核酸分析仪GE公司(Nano Drop), 普通PCR扩增仪(美国Bio-Rad公司)。
1.1.3 主要试剂 纯化用的Q柱、Ni柱Buffer以及PBS、NaOH、LB液体培养基、100 mg·mL-1氨苄青霉素、IPTG贮存液、30%丙烯酰胺、10%过硫酸铵、pH为6.8和8.0的1 mol·L-1 Tris-HCl、SDS-PAGE缓冲液、上样缓冲液、凝胶脱色液等由实验室配制;DMEM细胞培养基、胰酶消化液和胎牛血清白蛋白(BSA)购自Gibco公司,Cy3荧光染料购自GE Healthcare公司,溶解Cy3的甲基亚砜购自Sigma公司,PCR扩增用的Ex Taq、dNTPs、Ex Taq Buffer购自TaKaRa大连宝生物有限公司,细菌基因组提取使用Promega细胞基因组DNA样品提取试剂盒,Western blot采用的抗体来自于小鼠的抗His单抗。
1.2 方法1.2.1 Mac-T细胞的培养 细胞在37 ℃条件下复苏,离心后,弃掉上清液,加入完全培养液,置5% CO2、37 ℃条件下恒温过夜培养。培养至细胞汇合度达90%,进行细胞传代,连续培养3代后,用于后续目标蛋白的黏附试验。
1.2.2 细菌基因组提取 取20 μL菌液置于2 mL TSB培养基37 ℃培养,收集1 mL培养液,利用Promega细胞基因组DNA样品提取试剂盒按照说明进行金葡菌基因组DNA的提取。提取出的DNA置于2~8 ℃暂时保存,用于后续SasA基因鉴定及序列同源性分析。
1.2.3 SasA基因鉴定 使用Oligo6.0软件设计上下游引物,序列:5′-AGTCATAGTTTAGTGAGT-CAAGATAATCAAA-3′ (上游引物);5′-ATTTCTT-GTTACTTCATATTTAAAAGTTGTCG -3′ (下游引物)。
PCR反应体系:上下游引物各0.25 μL,10×buffer 2.5 μL,dNTP 2 μL, Taq酶0.125 μL,用双蒸水将反应体系补充到25 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,51.2 ℃退火90 s,72 ℃延伸35 s,35个循环;72 ℃延伸10 min;4 ℃保存。扩增产物经凝胶电泳鉴定SasA基因,对经鉴定携带SasA基因的菌株进行二代测序,并挑选其中测序结果较好的45株菌株利用Geneious软件进行保守性分析,构建进化树,具体方法参考文献[24]。
1.2.4 目标片段表达试验 37 ℃条件下,将冻存菌株放置在加入100 μg·mL-1氨苄青霉素的LB液体培养基中过夜复苏。将比例稀释至1∶100,移至用平底烧瓶盛装的含有100 μg·mL-1氨苄青霉素的LB培养基,37 ℃ 220 r·min-1条件下培养。当测定到细菌悬浮液OD600 nm达0.6~0.8时,加入IPTG,使其浓度达到0.5 mmol·L-1,同样条件下,培养6 h以充分诱导蛋白表达。将悬浊液在6 000 r·min-1的转速下离心10 min收集菌体,并用80 mL Ni柱Binding Buffer重悬菌体,然后以超声波5 s后间隔5 s的频率破碎菌体,时长为30 min。当观察到细菌悬浊液清亮时,12 000 g离心20 min后,取上清液,利用目标片段构建时带有的his标签和蛋白等电点先后用Ni柱和Source 30Q阴离子层析柱纯化,每次纯化后均用SDS-PAGE分离目标片段。
1.2.5 目标蛋白纯化及诱导效果检测 柱纯化出现蛋白峰时,收集峰后蛋白质,用12% SDS-PAGE进行鉴定,浓缩胶80 V分离15 min,分离胶180 V分离45 min,考马斯亮蓝染液染色30 min后脱色,观察结果。取未诱导表达的菌体破碎后的上清液与纯化的NRR片段作对照,Western blot检验结果。
1.2.6 目标蛋白Cy3荧光标记 将待荧光染色的蛋白置于0.1 mol·L-1 pH = 9.3的Na2CO3透析液中,并调节蛋白质的最终浓度为1 mg·mL-1, 所得的蛋白与Cy3染料结合,在避光条件下,孵育30~60 min,反应后的混合物通过凝胶过滤柱,除去未反应的染料,目标蛋白被洗脱。通过全波长扫描测得A280 nm以及A552 nm的值,计算Cy3标记程度。
1.2.7 目标片段黏附性检测 当奶牛乳腺上皮细胞(Mac-T)的细胞汇合度达到90%时,收集细胞并用PBS清洗,加入胰酶对细胞进行消化,37 ℃放置5 min,加入3倍体积的完全培养液终止消化。将细胞悬浮液置于1 000 r·min-1条件下离心5 min,弃去上清液,并将剩余部分用PBS反复清洗。细胞固定液重悬细胞,37 ℃固定30 min。固定后的细胞用PBS清洗3遍,并调整细胞数至106个·mL-1,将荧光标记的NRR片段、SRR1片段、BSA与细胞置于流式管中共同避光孵育1 h。SasA蛋白黏附性检测试验中,加入的BSA与NRR浓度如表 1,共设置7个浓度以保证最大浓度时黏附性达到饱和,进入平台期。NRR黏附性验证试验中,选取的NRR以及SRR1浓度为50和250 nmol·L-1。由于NRR浓度为50和100 nmol·L-1时, 完整的NRR与细胞结合的比例大约为40%,但100 nmol·L-1条件下略高,为使流式细胞检测结果更直观,各片段间黏附性差异更加明显,主要黏附片段定位试验中将各目标片段浓度调整至100 nmol·L-1。以上每个浓度各设置3个生物学重复。孵育完成后, 用PBS清洗2次后, 再另加500 μL PBS重悬细胞。对细胞进行流式细胞仪检测。各组黏附效果差异显著分析用Excel软件进行t-test检验,使用Graphpad Prism软件绘图。
|
|
表 1 BSA与NRR浓度 Table 1 The concentration of BSA and NRR |
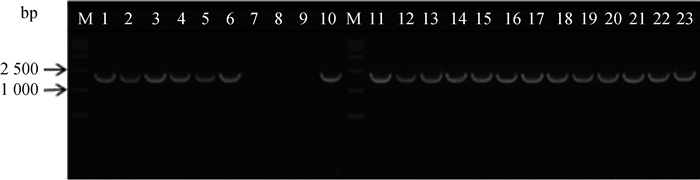
SasA基因鉴定主要采用PCR扩增编码SRR1-NRR段的基因。结果显示,73株金葡菌中有63株含SasA基因(部分样本电泳检测结果如图 2所示),占比86.3%。对SasA基因携带菌株测序并将选中的45株的序列通过软件比对构建进化树(图 3),得出基因序列一致性为97.1%。
|
M. DNA相对分子质量标准;1~23.73株金黄色葡萄球菌中的前23株 M. DNA marker; 1-23. The first 23 strains among 73 strains of S. aureus 图 2 SasA基因扩增后电泳检测结果 Fig. 2 Electrophoretic result of SasA amplification |

|
图 3 SasA基因携带菌株的基因进化树 Fig. 3 Phylogenetic tree of SasA gene-carrying strains |
图 3中,0.006表示该长度的分支代表的基因变异度为0.006,由此可看出,这些菌株SasA基因普遍变异程度较小,基因序列具有高度同源性。
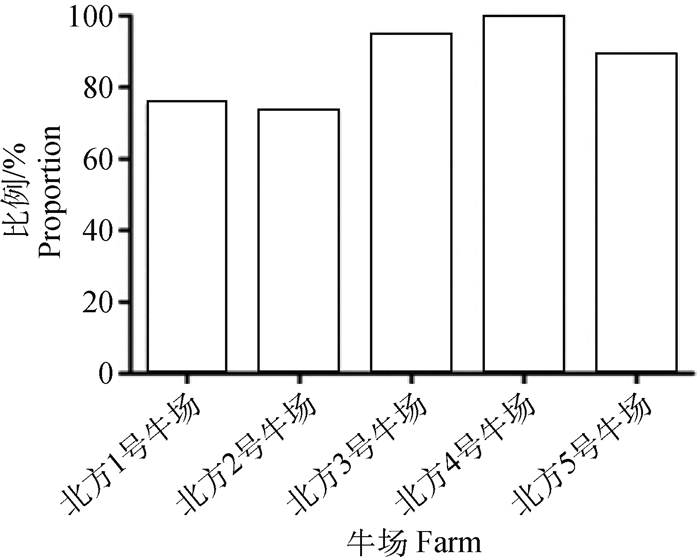
携带SasA基因的63株金葡菌菌株在采样的5个牛场中的来源分布如图 4所示。图 4的结果显示,SasA基因携带菌株在5个牛场中所占比例均高于60%,表明携带SasA基因的金葡菌存在一定的普遍性。
|
图 4 携带SasA基因的金葡菌菌株在5个牛场的分布 Fig. 4 Proportion of SasA gene-carrying strains in 5 dairy farms |
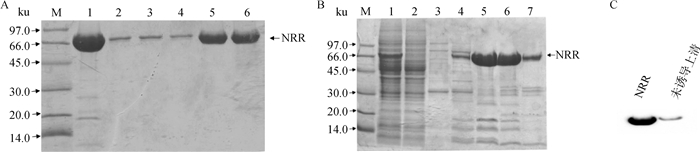
最后1次纯化所得蛋白,用PBS调整蛋白浓度至1 mg·mL-1,由ImageJ软件分析电泳图(图 5A、B),得到NRR纯化度为98%。最后Western blot检测诱导表达效果如图 5,图 5结果显示,经过诱导且纯化后获得的NRR量显著高于未诱导样。
|
M. 蛋白相对分子质量标准;1. 未纯化NRR; 2. 纯化NRR; 3~6(或7).收集的蛋白峰 M. Protein marker; 1. Unpurified NRR; 2. Purified NRR; 3-6 (or 7). Protein collected at peak 图 5 Ni柱纯化(A)和Q柱纯化(B)的电泳结果及NRR Western blot检验结果(C) Fig. 5 SDS-PAGE of NRR purified by Ni column (A), Q column (B) and Western blot of NRR (C) |
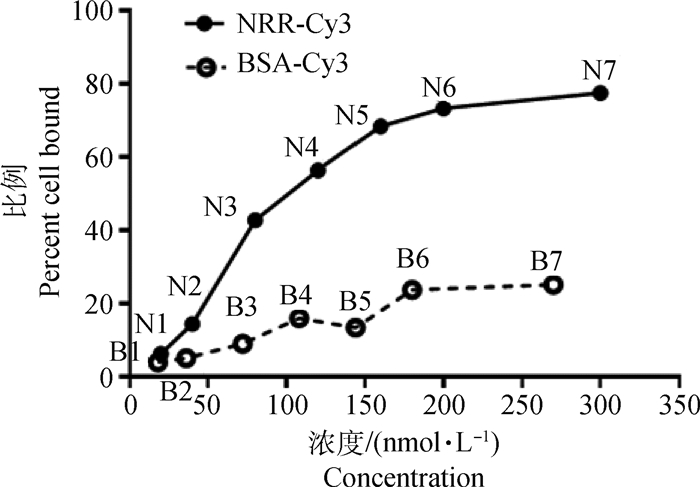
通过流式细胞仪检测呈阳性的细胞数,结果如图 6所示。结合NRR的Mac-T细胞数量显著高于结合BSA的细胞数量(图 7),说明NRR对奶牛乳腺上皮细胞存在较强的黏附性。
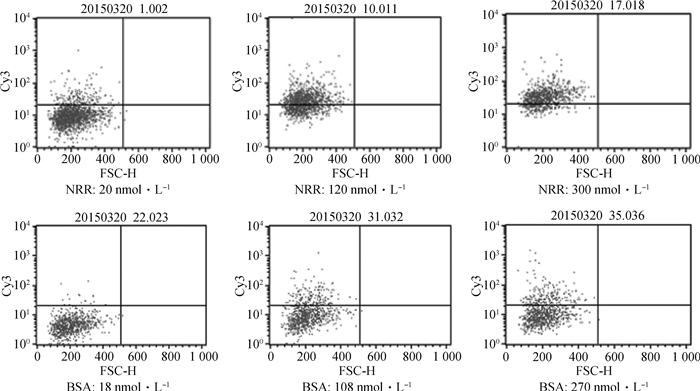
|
以上只展示该浓度下第1个生物学重复的流式检测结果, 下同 The above figures only show the flow cytometry test results of the first repeat at different concentrations, the same as below 图 6 不同浓度下与Mac-T结合的NRR-Cy3或BSA-Cy3的流式检测结果 Fig. 6 Flow cytometry tests of different concentrations of BSA-Cy3 or NRR-Cy3 binding to Mac-T |
|
图 7 黏附于细胞上的NRR-Cy3与BSA-Cy3比率 Fig. 7 Percent of NRR-Cy3 and BSA-Cy3 binding to Mac-T |
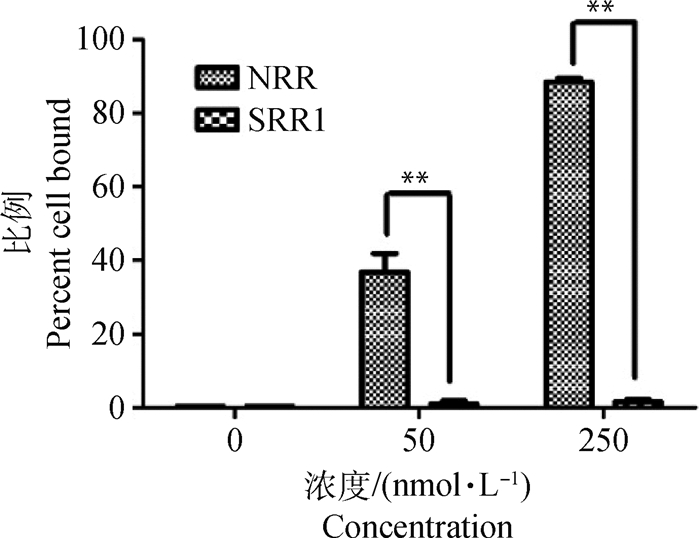
先后选取浓度为50和250 nmol·L-1的NRR及SRR1片段,荧光Cy3标记,与Mac-T孵育1 h,流式细胞仪检测。结果显示,NRR黏附的细胞比率极显著高于SRR1(P<0.01,图 8、9),表明NRR是奶牛乳源金葡菌的主要黏附片段。
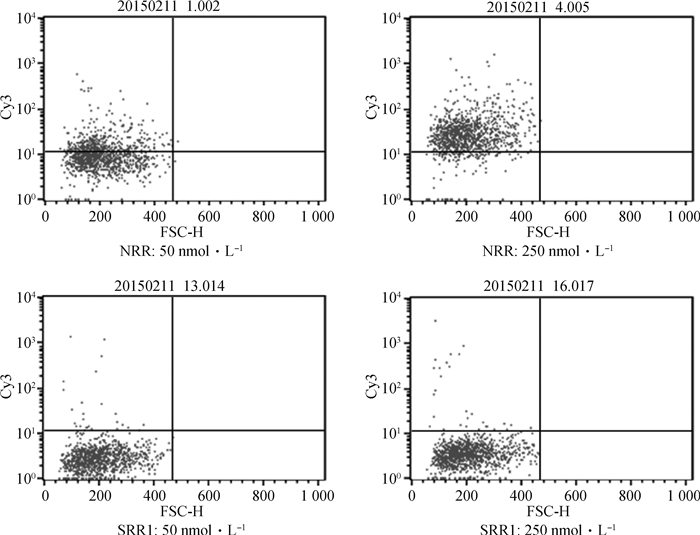
|
图 8 不同浓度下与Mac-T结合的NRR-Cy3或SRR1-Cy3的流式检测结果 Fig. 8 Flow cytometry tests of different concentrations of NRR-Cy3 or SRR1-Cy3 binding to Mac-T |
|
*. P<0.05;**.P<0.01;***.P<0.001 图 9 黏附于Mac-T细胞上的NRR-Cy3与SRR1-Cy3比率 Fig. 9 The proportion of NRR-Cy3 and SRR1-Cy3 binding to Mac-T |
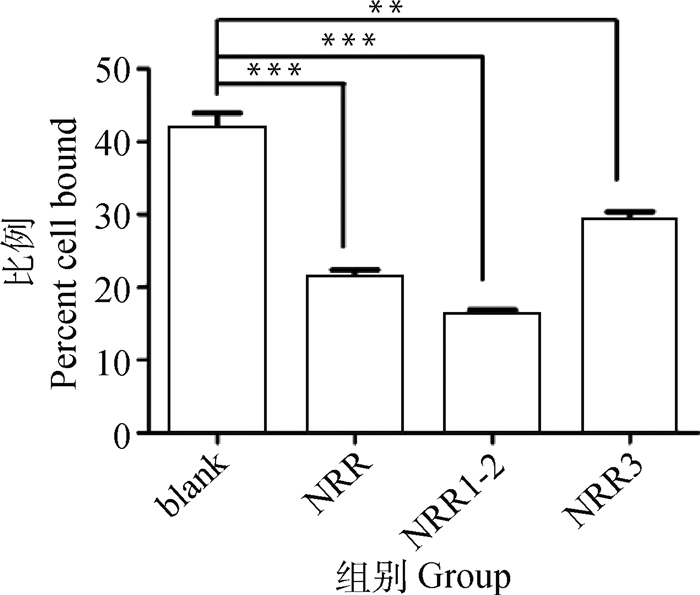
为进一步确定NRR片段中起主要黏附作用的区段,分别将浓度为100 nmol·L-1的不用荧光标记的NRR1-2、NRR3、NRR(图 1)与100 nmol·L-1NRR-Cy3混合后与细胞孵育。通过流式细胞仪检测发现,未标记的NRR(P<0.001)、NRR1-2(P<0.001)、NRR3(P<0.01) 均对NRR-Cy3黏附Mac-T存在抑制作用。但相较之下,NRR1-2抑制作用更显著,即发挥主要黏附作用的区域大致定位在NRR1-2区段(图 10)。
|
*. P<0.05;**.P<0.01;***.P<0.001 图 10 不同区段NRR片段抑制NRR-Cy3黏附 Fig. 10 Inhibition of NRR-Cy3 binding to Mac-T by different NRR domains |
金葡菌与宿主细胞受体结合主要依靠一系列黏附素,黏附是金葡菌定植于奶牛乳腺上皮细胞的关键步骤,深入研究金葡菌的黏附作用,找到作用的受体,有利于人为干扰或抑制金葡菌黏附宿主细胞从而减少感染细胞数[25-27]。此外,有关表面黏附和免疫逃逸的基因是金葡菌疫苗重要的候选基因,因此,SasA基因分布和功能的研究也极为重要[28-29]。
有关于SasA基因人流行病学调查研究结果显示,人体分离纯化得到的42株金葡菌菌株中84%均含SasA基因。本研究中,针对牛源金葡菌SasA基因的携带情况进行调查,采集到的73个样本中86.3%含该基因,这说明SasA基因广泛存在于在人和牛源金葡菌中[24]。
Yang等[30]对NRR结构域的晶体结构分析将整个片段划分为4个部分,包括两个不同的串联钙黏蛋白样的模块,1个β-折叠和1个豆科凝集素样模块(L-Lectin,245—751 aa),因此SasA也被归为类豆科植物凝集素细胞壁锚定表面蛋白。已有的研究发现,SasA蛋白黏附宿主细胞是通过L-Lectin结构的识别实现的,这可能是导致本试验中NRR与SRR1黏附性存在显著差异的重要原因。
本研究在参考前人对于L-lectin及NRR-SRR1 (48—540 aa) [19]黏附性探究的基础上,单独挑选NRR与SRR1片段(图 1)进行黏附性差异分析验证了NRR的黏附性,并通过竞争抑制黏附试验来定位主要黏附片段,发现NRR1-2 (230—540 aa)竞争抑制完整NRR片段黏附的作用更显著,在本研究中的所有片段中黏附力最强,这与结构中L-Lectin(245—492 aa)基本相符,也解释了NRR3的竞争抑制黏附能力较弱的原因。豆科凝集素样模块富含碱性氨基酸,可与N-乙酰神经氨酸(Neu5Ac)特异性结合,与调节宿主细胞的唾液酸化受体的黏附过程有关[22, 30],进一步证实了NRR在SasA黏附性调节中的重要作用,也揭示了相关受体的潜在特性。在人唾液中分离出的gp340受体能与SasA通过Neu5Ac结合,故SasA可成为人与奶牛疫苗研究共同的候选靶点[22]。
为确定NRR1-2片段对奶牛乳腺细胞的具体影响和定位该片段与奶牛乳腺上皮细胞结合的位点,进一步的研究拟寻找奶牛乳腺上皮细胞表面含有Neu5Ac的糖蛋白,制备抗NRR1-2单抗并与NRR1-2共孵育侵染纯化后的该糖蛋白,通过对比单抗组与非单抗组感染程度确定NRR1-2的黏附效应和验证该受体。
4 结论SasA基因在奶牛乳源金葡菌菌株中普遍存在,各菌株该基因的序列具有高度的保守性。除此之外,在SasA蛋白中,对奶牛乳腺上皮细胞起主要黏附作用的为NRR片段而非SRR1,大致定位在其结构域的230—540位氨基酸。本研究为确定人与奶牛金葡菌疫苗研究共同的候选靶点提供了重要试验数据,为定位牛乳腺上皮细胞SasA蛋白受体提供了参考信息。
致谢: 本研究得到中国人民解放军军事科学院军事医学研究院徐俊杰研究员及团队成员的大力支持和指导。
| [1] | CÔTÉ-GRAVEL J, MALOUIN F. Symposium review: features of Staphylococcus aureus mastitis pathogenesis that guide vaccine development strategies[J]. J Dairy Sci, 2019, 102(5): 4727–4740. DOI: 10.3168/jds.2018-15272 |
| [2] | TAKAMATSU D, HATA E, OSAKI M, et al. Role of SraP in adherence of Staphylococcus aureus to the bovine mammary epithelia[J]. J Vet Med Sci, 2008, 70(7): 735–738. DOI: 10.1292/jvms.70.735 |
| [3] | ROCHE F M, MASSEY R, PEACOCK S J, et al. Characterization of novel LPXTG-containing proteins of Staphylococcus aureus identified from genome sequences[J]. Microbiology (Reading), 2003, 149(Pt 3): 643–654. |
| [4] | YI S Q, ZHANG X Y, YANG Y L, et al. Immunity induced by Staphylococcus aureus surface protein A was protective against lethal challenge of Staphylococcus aureus in BALB/c mice[J]. Microbiol Immunol, 2012, 56(6): 406–410. DOI: 10.1111/j.1348-0421.2012.00451.x |
| [5] | FOSTER T J, GEOGHEGAN J A, GANESH V K, et al. Adhesion, invasion and evasion: the many functions of the surface proteins of Staphylococcus aureus[J]. Nat Rev Microbiol, 2014, 12(1): 49–62. DOI: 10.1038/nrmicro3161 |
| [6] | STRANGER-JONES Y K, BAE T, SCHNEEWIND O. Vaccine assembly from surface proteins of Staphylococcus aureus[J]. Proc Natl Acad Sci U S A, 2006, 103(45): 16942–16947. DOI: 10.1073/pnas.0606863103 |
| [7] | GEOGHEGAN J A, FOSTER T J. Cell wall-anchored surface proteins of Staphylococcus aureus: many proteins, multiple functions[J]. Curr Top Microbiol Immunol, 2017, 409: 95–120. |
| [8] |
周宏, 李韩平, 姜永强. 金黄色葡萄球菌表面蛋白研究进展[J]. 生物技术通讯, 2004, 15(1): 73–75.
ZHOU H, LI H P, JIANG Y Q. The progress of Staphylococcus aureus surface proteins[J]. Letters in Biotechnology, 2004, 15(1): 73–75. DOI: 10.3969/j.issn.1009-0002.2004.01.025 (in Chinese) |
| [9] | MAZMANIAN S K, TON-THAT H, SCHNEEWIND O. Sortase-catalysed anchoring of surface proteins to the cell wall of Staphylococcus aureus[J]. Mol Microbiol, 2001, 40(5): 1049–1057. DOI: 10.1046/j.1365-2958.2001.02411.x |
| [10] | PEREYRA E A L, PICECH F, RENNA M S, et al. Detection of Staphylococcus aureus adhesion and biofilm-producing genes and their expression during internalization in bovine mammary epithelial cells[J]. Vet Microbiol, 2016, 183: 69–77. DOI: 10.1016/j.vetmic.2015.12.002 |
| [11] | SANCHEZ C J, SHIVSHANKAR P, STOL K, et al. The pneumococcal serine-rich repeat protein is an intra-species bacterial adhesin that promotes bacterial aggregation in vivo and in biofilms[J]. PLoS Pathog, 2010, 6(8): e1001044. DOI: 10.1371/journal.ppat.1001044 |
| [12] | MAGRO G, BIFFANI S, MINOZZI G, et al. Virulence genes of S. aureus from dairy cow mastitis and contagiousness risk[J]. Toxins (Basel, 2017, 9(6): 195. DOI: 10.3390/toxins9060195 |
| [13] | RAMBOARINA S, GARNETT J A, ZHOU M X, et al. Structural insights into serine-rich fimbriae from gram-positive bacteria[J]. J Biol Chem, 2010, 285(42): 32446–32457. DOI: 10.1074/jbc.M110.128165 |
| [14] | SIEGEL S D, REARDON M E, TON-THAT H. Anchoring of LPXTG-like proteins to the gram-positive cell wall envelope[J]. Curr Top Microbiol Immunol, 2017, 404: 159–175. |
| [15] | FISCHETTI V A. Surface proteins on gram-positive bacteria[J]. Microbiol Spectr, 2019, 7(4): GPP3-0012-2018. doi: 10.1128/microbiolspec.GPP3-0012-2018. |
| [16] | BOWDEN M G, CHEN W, SINGVALL J, et al. Identification and preliminary characterization of cell-wall-anchored proteins of Staphylococcus epidermidis[J]. Microbiology (Reading), 2005, 151(Pt 5): 1453–1464. |
| [17] | LIZCANO A, SANCHEZ C J, ORIHUELA C J. A role for glycosylated serine-rich repeat proteins in Gram-positive bacterial pathogenesis[J]. Mol Oral Microbiol, 2012, 27(4): 257–269. DOI: 10.1111/j.2041-1014.2012.00653.x |
| [18] | PYBURN T M, BENSING B A, XIONG Y Q, et al. A structural model for binding of the serine-rich repeat adhesin GspB to host carbohydrate receptors[J]. PLoS Pathog, 2011, 7(7): e1002112. DOI: 10.1371/journal.ppat.1002112 |
| [19] | YANG Y L, QIAN M Y, YI S Q, et al. Monoclonal antibody targeting Staphylococcus aureus Surface Protein A (SasA) protect against Staphylococcus aureus sepsis and peritonitis in mice[J]. PLoS One, 2016, 11(2): e0149460. DOI: 10.1371/journal.pone.0149460 |
| [20] | SIBOO I R, CHAMBERS H F, SULLAM P M. Role of SraP, a serine-rich surface protein of Staphylococcus aureus, in binding to human platelets[J]. Infect Immun, 2005, 73(4): 2273–2280. DOI: 10.1128/IAI.73.4.2273-2280.2005 |
| [21] | ZHU L, INOUE K, YOSHIZUMI S, et al. Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP[J]. J Bacteriol, 2009, 191(10): 3248–3255. DOI: 10.1128/JB.01815-08 |
| [22] | KUKITA K, KAWADA-MATSUO M, OHO T, et al. Staphylococcus aureus SasA is responsible for binding to the salivary agglutinin gp340, derived from human saliva[J]. Infect Immun, 2013, 81(6): 1870–1879. DOI: 10.1128/IAI.00011-13 |
| [23] | YANG Y L, YU R, YANG X X, et al. Protection against Staphylococcus aureus and tetanus infections by a combined vaccine containing SasA and TeNT-Hc in mice[J]. Mol Med Rep, 2017, 15(4): 2369–2373. DOI: 10.3892/mmr.2017.6227 |
| [24] |
杨益隆. 金黄色葡萄球菌SasA蛋白免疫保护性的研究[D]. 北京: 中国人民解放军军事医学科学院, 2016.
YANG Y L. Protective immunity of targeting Staphylococcus aureus surface protein A (SasA)[D]. Beijing: Academy of Military Medical Sciences, 2016. (in Chinese) |
| [25] | CHAVAKIS T, HUSSAIN M, KANSE S M, et al. Staphylococcus aureus extracellular adherence protein serves as anti-inflammatory factor by inhibiting the recruitment of host leukocytes[J]. Nat Med, 2002, 8(7): 687–693. DOI: 10.1038/nm728 |
| [26] |
丁进东, 彭程, 贺奋义. 奶牛乳房炎金葡菌黏附素亚单位疫苗的基础研究[J]. 畜牧兽医杂志, 2014, 33(3): 1–4.
DING J D, PENG C, HE F Y. The study of subunit vaccine targeting adhesins of S. aureus induced mastitis in dairy cows[J]. Journal of Animal Science and Veterinary Medicine, 2014, 33(3): 1–4. DOI: 10.3969/j.issn.1004-6704.2014.03.001 (in Chinese) |
| [27] | LEONARD A C, PETRIE L E, COX G. Bacterial anti-adhesives: inhibition of Staphylococcus aureus nasal colonization[J]. ACS Infect Dis, 2019, 5(10): 1668–1681. DOI: 10.1021/acsinfecdis.9b00193 |
| [28] | DEGO O K, VAN DIJK J E, NEDERBRAGT H. Factors involved in the early pathogenesis of bovine Staphylococcus aureus mastitis with emphasis on bacterial adhesion and invasion. A review[J]. Vet Q, 2002, 24(4): 181–198. DOI: 10.1080/01652176.2002.9695135 |
| [29] | MCCARTHY A J, LINDSAY J A. Genetic variation in Staphylococcus aureus surface and immune evasion genes is lineage associated: implications for vaccine design and host-pathogen interactions[J]. BMC Microbiol, 2010, 10: 173. DOI: 10.1186/1471-2180-10-173 |
| [30] | YANG Y H, JIANG Y L, ZHANG J, et al. Structural insights into SraP-mediated Staphylococcus aureus adhesion to host cells[J]. PLoS Pathog, 2014, 10(6): e1004169. DOI: 10.1371/journal.ppat.1004169 |



