奇异变形杆菌是革兰阴性菌,属于肠杆菌科变形杆菌属,广泛分布于动物的肠道中,是一种条件致病菌。奇异变形杆菌具有鞭毛、菌毛、尿素酶、外膜蛋白等毒力因子,可以黏附到宿主黏膜表面对宿主产生损伤[1]。自1989年江益民等[2]首次从鸡中分离出奇异变形杆菌以来,我国陆续有鸡[3]、水貂[4]、山羊[5]、猪[6]等感染的报道。奇异变形杆菌会引起猪腹泻、体温升高、肠道和肺部病变,并且有一定致死性[7]。除此之外,奇异变形杆菌还是一种人畜共患病病原,会引起人的尿路感染[8]。由于会给畜牧生产和人类健康带来不利影响,造成经济损失,对奇异变形杆菌的防控不容忽视。
噬菌体(bacteriophage)是能够感染细菌的病毒,广泛分布于细菌存在之处,例如,土壤和动物的肠道中。依据遗传物质可将噬菌体分为双股DNA群(dsDNA)、单股DNA群(ssDNA)、双股RNA(dsRNA)、单股RNA群(ssRNA)。依据形态,双股DNA群下将噬菌体分为肌尾噬菌体(Myoviridae)、短尾噬菌体(Podoviridae)、长尾噬菌体(Siphoviridae)等[9]。在短尾噬菌体中,C3样的噬菌体较为少见,在此前的电镜观察中,5 500株噬菌体中只观察到了0.5%的C3样噬菌体[10]。C3样噬菌体中最具代表性的是大肠杆菌噬菌体phiEco32[11],并以此命名了phiEco32属。目前,根据国际病毒分类委员会主要物种列表2019.v1(ICTV Master Species List 2019.v1),以phiEco32为代表种的phiEco32属已被命名为Kuravirus属。Kuravirus属下还包括了大肠杆菌噬菌体172-1[12]、ECMP2[13]、NJ01[14]、Septima11(NC_023593)、SU10[15]等种。除此之外,沙门菌噬菌体7-11[16]、阪崎肠杆菌噬菌体vB_CsaP_GAP52(NC_019402.1)、副溶血性弧菌噬菌体Vp_R1[17]也被归为Kuravirus属,但未被收录到国际病毒分类委员会主要物种列表中。目前,仅发现1株C3样的革兰阳性菌噬菌体:乳酸乳球菌噬菌体KSY1,它的基因组与其他C3样噬菌体没有同源性[18]。
目前,对于奇异变形杆菌感染仍多采用抗生素进行预防和治疗,但广谱抗生素的使用已导致严重的负面效应。近年来,已有研究报道奇异变形杆菌对不同抗生素产生耐药性,并且产超广谱β-内酰胺酶(ESBLs)菌株比例逐渐上升[19],产生多重耐药性。此外,2020年,我国已全面禁止在饲料中添加抗生素,寻找安全有效的抗生素替代品成为当务之急。烈性噬菌体作为感染细菌的病毒,有严格的宿主特异性、能够自我复制等优势,成为最有潜力的抗生素的替代品之一。
本文从断奶前仔猪和健康断奶仔猪粪样中分离到1株C3样的奇异变形杆菌噬菌体,对其生物学特性和基因组进行研究,为防治奇异变形杆菌感染提供一定研究基础。
1 材料与方法 1.1 材料1.1.1 菌株及噬菌体来源 用于噬菌体分离的菌株奇异变形杆菌(Proteus mirabilis),为南京农业大学消化道微生物实验室从健康猪粪样中分离得到,NCBI登录号为MH643694。
用于噬菌体分离的10份样品来源于安徽滁州某猪场5头断奶前与5头断奶后仔猪粪样。
1.1.2 主要试剂与培养基 LB培养基中酵母膏及胰蛋白胨购自OXOID,琼脂粉购自Solarbio,NaCl购自南京强盛功能化学股份有限公司;SM缓冲液购自生工生物工程(上海)股份有限公司;0.22 μm一次性针头滤器购自Millipore;限制性核酸内切酶购自TaKaRa公司。
1.2 噬菌体分离纯化采用双层平板法对噬菌体进行分离。将1 g粪样用20 mL SM液稀释,12 000 r·min-1离心10 min去除杂质,将上清过滤除菌。将100 μL滤液与100 μL处于对数期的宿主菌混合后,加入5 mL LB半固体培养基中,倒双层平板。将双层平板置于37 ℃培养5~6 h,观察有无噬菌斑。挑取单个噬菌斑反复纯化3次,即可获得单一噬菌体。
1.3 噬菌体基因组的提取和酶切鉴定参照《分子克隆实验指南》[20]提取噬菌体基因组。使用限制性核酸内切酶Hind Ⅲ、BamHⅠ、XhoⅠ、EcoRⅠ、SacⅠ酶切后,用1%凝胶电泳观察成像并分析。
1.4 噬菌体形态观察噬菌体培养液经0.22 μm滤器过滤后,用100 kDa超滤管浓缩至浓度为109~1010PFU·mL-1,送南京农业大学电镜室采用磷钨酸溶液染色法进行形态观察。
1.5 噬菌体生物特性分析1.5.1 噬菌体最佳感染复数(multiplicity of infection,MOI) 测定 将噬菌体以感染复数100∶1、10∶1、1∶1、1∶10、1∶100、1∶1 000、1∶10 000、1∶100 000分别接种至处于对数期的宿主菌中,37 ℃培养4.5 h,得到噬菌体滴度最高的MOI为最佳MOI。
1.5.2 噬菌体一步生长曲线及抑菌曲线绘制 按照最佳MOI将噬菌体和宿主菌接入LB培养基中,37 ℃孵育15 min后4 000 r·min-1离心5 min,倒掉上清以去除未吸附的噬菌体,再用LB培养基重悬菌体。迅速置于37 ℃、120 r·min-1震荡培养6 h。在一定时间点取样,以时间为横坐标,以噬菌体滴度为纵坐标绘制噬菌体生长曲线。以时间为横坐标,OD600 nm为纵坐标,并以宿主菌作为对照绘制抑菌曲线。
1.5.3 噬菌体热稳定性及pH稳定性 将0.9 mL SM缓冲液置于30、40、50、60、70、80 ℃预热后,加入0.1 mL滴度为109PFU·mL-1噬菌体,使终浓度为108 PFU·mL-1。在30、60 min时分别取样,以双层平板法检测效价。
取0.1 mL滴度为109PFU·mL-1的噬菌体培养液与0.9 mL pH为2~13的SM缓冲液混合,37 ℃培养3 h后,以双层平板法进行计数。
1.6 噬菌体全基因组测序及组学研究噬菌体序列测定由上海凌恩科技公司完成。
噬菌体基因组分析使用GeneMarkS(版本:4.6b,http://topaz.gatech.edu/GeneMark/)预测编码基因。使用BLAST在线工具(http://blast.ncbi.nlm.nih.gov/) 进行碱基序列相似性比对及编码基因的注释。最后将基因组上传至NCBI获得序列号。
使用ARAGORN (http://130.235.244.92/ARAGORN/)确定基因组中是否存在tRNA。使用VFDB(http://www.mgc.ac.cn/VFs/main.htm)进行毒力基因分析,使用CARD数据库(https://card.mcmaster.ca/home)进行抗生素抗性分析。使用MEGA X绘制进化树。
2 结果 2.1 噬菌体的分离鉴定2.1.1 噬菌体的分离纯化及鉴定 从2份断奶前粪样以及4份断奶后粪样中均分离得到噬菌体,将从这6份样品中分离得到的噬菌体分别提取基因组,进行酶切鉴定,酶切结果显示6个噬菌体酶切条带一致,酶切结果如图 1A所示,故判定为同一株噬菌体,将其命名为奇异变形杆菌噬菌体vB_PmiP_P59(Proteus mirabilis phage vB_PmiP_P59),简称为P59。噬菌斑大小均一,直径约为1 mm,边缘不整齐,如图 1B。
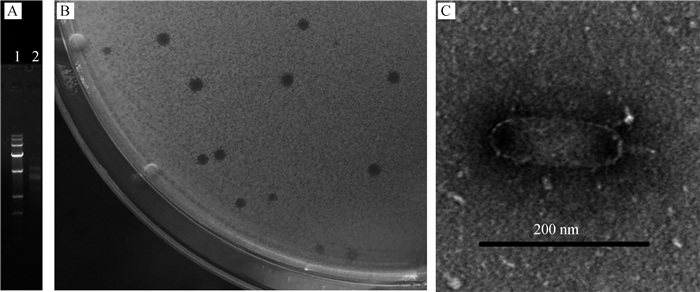
|
A.噬菌体P59基因组酶切图(泳道1和2分别为15000 marker和粪中分离得到的噬菌体);B. 噬菌体P59噬菌斑形态;C.噬菌体P59电镜形态 A. Electrophoresis of phage P59's genome digested with restriction endonucleases (Lane 1. 15000 marker; Lane 2. phage isolated from fecal); B. Plaques of phage P59; C. Electron micrograph of phage P59 图 1 噬菌体P59的分离鉴定 Fig. 1 Isolation and identification of P59 |
2.1.2 噬菌体分类鉴定 电镜结果(图 1C)表明,P59头部为扁平的椭圆形,长152 nm,宽54 nm;尾部大小约40 nm×12 nm,尾部长度小于头部宽度,推断P59属于短尾病毒科(Podoviridae);头部长度超过宽度数倍,根据Ackermann和Eisenstark[21]的分类方法推断其为C3样噬菌体。
2.2 噬菌体的生物学特性2.2.1 最佳感染复数(multiplicity of infection,MOI) 测定 如图 2A,噬菌体P59在MOI为10-3时获得的噬菌体效价最高,为2.2×1010PFU·mL-1。
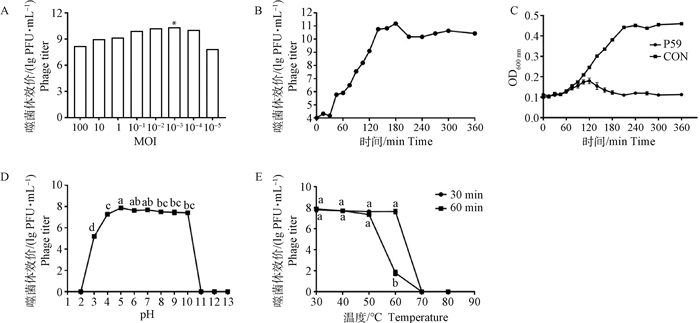
|
A. 最佳MOI;B.一步生长曲线;C.抑菌曲线;D. pH稳定性;E. 热稳定性。图中所标字母相异代表各处理组差异显著(P < 0.05),字母相同代表各处理组差异不显著(P≥0.05)。下图同 A. Optimal MOI of phage P59; B. One-step growth curve of phage P59; C. Bactericidal curve of phage P59; D.Thermal stability of phage P59; E. pH stability of phage P59. Different letters denote significant difference between the treatments(P < 0.05), the same letter denotes no significant difference between treatments(P≥0.05). The same as below 图 2 噬菌体P59的生物学特性 Fig. 2 Biological characteristics of phage P59 |
2.2.2 噬菌体一步生长曲线及抑菌曲线绘制 以时间为横坐标,噬菌体滴度为纵坐标绘制噬菌体一步生长曲线。生长曲线如图 2B所示,P59的潜伏期为30 min,爆发时间为105 min,裂解量=爆发末期噬菌体滴度/感染初期宿主菌浓度=2 754 PFU·cell-1。
抑菌曲线如图 2C所示,P59对奇异变形杆菌有显著抑菌效果(P < 0.05)。
2.2.3 噬菌体的热稳定性及pH稳定性 如图 2D,噬菌体P59在pH 5~10效价保持稳定;在pH为4时,存活率下降了一个数量级;pH为11时,完全失去活性。
如图 2E,噬菌体P59经过30~50 ℃处理30、60 min时效价均保持稳定;60 ℃处理30 min时效价保持稳定,处理60 min后效价显著降低(P < 0.05),70 ℃处理30 min后已失去活性。
2.3 噬菌体基因组学研究2.3.1 噬菌体全基因组概况 噬菌体基因组为线形双链DNA,长度为90 187 bp,(G+C)%含量为34.65%。将噬菌体全基因组核酸序列提交GenBank,获取的登录号为MT664722。根据BLASTn比对,与1株奇异变形杆菌噬菌体Privateer(90 710 bp,MT028297.1)和1株阪崎肠杆菌vB_CsaP_009(92 122 bp,NC_048664.1)相似性较高,分别为95.06%、96.52%,覆盖率分别为96%、91%。
噬菌体基因组中tRNA的作用是编码宿主中较少出现的密码子以增加噬菌体蛋白的表达[22]。经过ARAGORN分析,P59基因组中有4个tRNA基因存在,分别为tRNA-Trp(CCA)、tRNA-Arg(TCT)、tRNA-Met(CAT)、tRNA-Arg(CCT)。其他Kuravirus属噬菌体基因组均含有Arg-tRNA (TCT),但该tRNA在大肠杆菌噬菌体基因组中较为罕见。
经过VFDB对比分析噬菌体P59中没有发现毒力因子的编码基因,经过CARD分析未发现携带耐药基因,表明该噬菌体具有较好的生物安全性。
2.3.2 噬菌体功能性ORF分析 利用GeneMarkS(版本:4.6b,http://topaz.gatech.edu/GeneMark/)进行初步预测,共发现136个开放阅读框(ORF)。与其他Kuravirus属噬菌体相似,P59的ORF绝大多数以AUG作为起始密码子,达到90.44%;12个ORF以GUG开头(8.82%);1个以UUG开头(7.35%)。
使用BLASTp中的Nr数据库将预测基因的蛋白序列进行对比,获得预测基因的注释信息。其中,已知功能的32个ORF如表 1所示。噬菌体基因组可分为4个功能模块,包括噬菌体结构蛋白,DNA复制和修饰、核苷酸修饰与调节,以及其他功能基因(图 3)。与噬菌体形态相关的8个ORF包括ORF17(末端酶大亚基)、ORF18(门户蛋白)、ORF20(支架蛋白)、ORF21(衣壳和支架蛋白)、ORF22(淀粉结合蛋白)、ORF28(结构蛋白)、ORF30(宿主特异性蛋白J)、ORF34(DNA注射蛋白)。与DNA复制相关的8个ORF包括ORF41(核酸内切酶)、ORF54(DNA连接酶)、ORF56(核酸外切酶)、ORF58、72、86(DNA聚合酶)、ORF82(DNA结合蛋白)、ORF87(引物酶/解旋酶)。与核苷酸合成和修饰相关的7个ORF包括ORF43(腺嘌呤甲基转移酶)、ORF50(烟酰胺核糖核酸)、ORF81、94、96(脱氧核糖核苷酸还原酶)、ORF85(dCTP脱氨酶)、ORF128(烟酰胺磷酸核糖基转移酶)、ORF129(磷酸核糖焦磷酸激酶)。ORF81、94、96参与编码的脱氧核糖核苷酸还原酶(RNR),是辅助代谢基因(AMGs)的一种,能支持宿主被感染过程中的代谢,从而促进噬菌体的复制和修复[23]。
|
|
表 1 噬菌体P59编码的功能蛋白 Table 1 Genes with proposed functions in the genome of phage P59 |
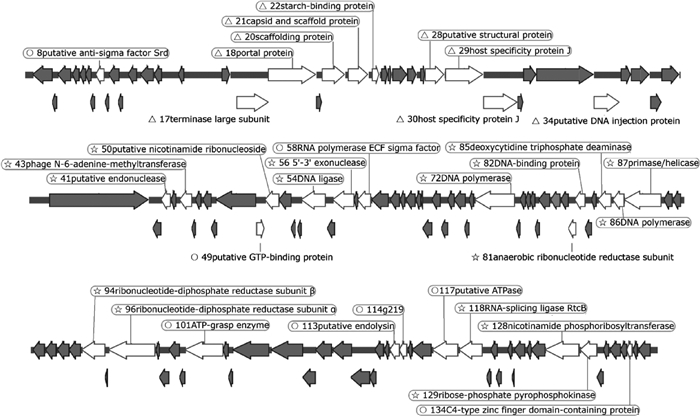
|
箭头方向代表转录方向。△代表噬菌体结构蛋白;☆代表DNA复制和核苷酸代谢蛋白;○代表其他功能蛋白;灰色代表假想蛋白 The direction of each arrow represents the direction of transcription. △ represent the phage structural proteins. ☆ represent phage DNA metabolism and modification proteins. ○ represent other proteins. Grey arrows represent hypothetical proteins 图 3 P59的基因组结构示意图 Fig. 3 Genome structure of phage P59 |
ORF58编码RNA聚合酶ECF sigma因子,该因子在phiEco32、7-11、Vp_R1、SU10等其他Kuravirus属噬菌体中均有发现,能够识别病毒中晚期基因的启动子[24]。ORF113可以产生活性物质内溶素,内溶素是一种裂解酶,可与穿孔素共同作用于细胞壁和细胞膜使宿主菌破裂,释放子代噬菌体[25]。除此之外,未在P59基因组中发现整合酶序列,说明P59是1株烈性噬菌体。
2.4 进化树构建支架蛋白是C3样的噬菌体基因组中较为保守的部分,通常用于分类[15]。使用MEGA X软件,采用邻接法对P59以及其他9株C3样噬菌体的支架蛋白构建进化树。如图 4,进化树演化出了两个分支,在支架蛋白上与P59进化距离最近的是沙门菌噬菌体7-11和阪崎肠杆菌噬菌体GAP52。

|
进化树基于邻接法构建。数字代表置信水平,标尺代表遗传距离。黑色圆点.P59。括号内.序列号 The tree was inferred using the Neighbor-Joining method. Numbers at the branch point represent the confidence level. Scale represents the genetic distance. Black ball. P59. In brackets. Accession No. 图 4 基于P59和其他C3样噬菌体支架蛋白构建同源进化树 Fig. 4 Phylogenetic tree based on the scaffolding proteins between P59 and other C3 phages |
作者从健康仔猪的粪样中分离出1株奇异变形杆菌的噬菌体P59。动物和人的肠道内存在大量噬菌体,这些内源性噬菌体能够消灭宿主菌,同时,在肠道内发挥免疫作用,帮助控制局部炎症和自身免疫反应[26]。因此,推测内源性的P59在保护仔猪肠道健康方面也发挥了积极的作用。
P59核酸类型为线形双链DNA,基因组大小为90 187 bp。基因组中含有136个ORF,编码4个tRNA。P59的支架蛋白、末端酶大亚基、门户蛋白等与噬菌体形态相关的蛋白均与C3样噬菌体蛋白有较高的同源性。C3样噬菌体代表株phiEco32含有的关于核苷酸合成和DNA复制的蛋白,除ATP结合蛋白、胸苷酸合成酶、HNH核酸内切酶之外,在P59中也均有发现。根据Lavigne等[27]的分类方法,可以依据全基因组蛋白质组学分析的结果对短尾噬菌体进行分类,当40%的蛋白质具有同源性时,可以将2株噬菌体归为同一属。使用CoreGenes3.5将P59的蛋白质组与phiEco32进行比对。将BLASTp分数设置为50时,有53个蛋白(41.41%)有同源性。将BLASTp分数设置为75时,P59与phiEco32有39个蛋白(30.47%)具有同源性,说明这两株噬菌体可能属于同一亚科而非同一属。基于形态学特征和基因组特征,噬菌体P59属于短尾噬菌体科,具有C3样形态特征。
噬菌体在自然界中广泛存在,可以特异性地裂解宿主菌,还具有高度穿透性,可以通过体内的肠道黏膜屏障、血脑屏障等[28]。随着抗生素的滥用,致病菌的耐药菌株大量出现,噬菌体的治疗价值开始得到重视。但噬菌体的宿主特异性使其抑菌谱狭窄,应用上存在局限性,因此多采用噬菌体鸡尾酒(phage cocktail)疗法制成混合制剂来拓展抑菌谱, 也降低所有噬菌体都产生突变的可能性[29]。目前,一些公司已有噬菌体鸡尾酒产品用于畜牧生产上细菌感染的防治,但相关研究多针对沙门菌[30]、大肠杆菌[31]、空肠弯曲杆菌[32]等致病菌的噬菌体,对于条件致病菌噬菌体的关注较少。条件致病菌在宿主抵抗力低下时会产生致病性,危害宿主健康。仔猪在断奶期间易产生应激,抵抗力较差,易被条件致病菌感染,导致腹泻。因此,可在这些鸡尾酒制剂中再加入一些条件致病菌的噬菌体,可能会有更好的防治细菌感染的效果。生物学特性分析表明P59有较好的抑菌能力,同时最佳感染复数小,裂解量大,爆发时间长,有一定的温度和pH稳定性,未携带抗生素抗性基因和毒力基因,说明P59有一定的应用前景,可用于奇异变形杆菌感染的预防和治疗。除此之外,有研究表明,噬菌体内溶素可作用的范围比噬菌体的宿主谱更广[33],不易使细菌产生抗性突变株,具有广阔的应用前景。噬菌体P59可产生内溶素,其内溶素纯化后可能也具备应用于治疗细菌感染的潜力。
4 结论分离到1株C3样奇异变形杆菌噬菌体vB_PmiP_ P59,最佳感染复数为0.001,潜伏期为30 min,爆发期为105 min,裂解量为2 754 PFU·cell-1,在温度小于50 ℃,pH为5~10时稳定。P59对宿主菌有较好的抑菌效果,有一定的应用潜力。
| [1] | SOSA V, ZUNINO P. Effect of Ibicella lutea on uropathogenic Proteus mirabilis growth, virulence, and biofilm formation[J]. J Infect Dev Ctries, 2009, 3(10): 762–770. DOI: 10.3855/jidc.232 |
| [2] |
江益民, 龙塔, 侯玉泽, 等. 鸡奇异变形杆菌病病原的分离与鉴定[J]. 河南农业科学, 1990(7): 31–33.
JIANG Y M, LONG T, HOU Y Z, et al. Isolation and identification of pathogenic Proteus mirabilis from chickens[J]. J Henan Agri Sci, 1990(7): 31–33. (in Chinese) |
| [3] |
朱瑞良, 张绍学, 牛钟相. AA肉种鸡奇异变形杆菌病流行病学调查[J]. 中国预防兽医学报, 1994(4): 46–47.
ZHU R L, ZHANG S X, NIU Z X. Epidemiological investigation on AA broiler breeder Proteus mirabilis[J]. Chinese Journal of Preventive Veterinary Medicine, 1994(4): 46–47. (in Chinese) |
| [4] |
徐春厚, 卫广森, 李殿凯, 等. 水貂变形杆菌的分离鉴定[J]. 中国兽医科学, 1994, 24(12): 33–34.
XU C H, WEI G S, LI D K, et al. Isolation and identification of Proteus mirabilis from mink[J]. Chinese Veterinary Science, 1994, 24(12): 33–34. (in Chinese) |
| [5] |
崔国林, 钟世勋, 杨世发, 等. 山羊奇异变形杆菌分离鉴定及其16S-23S rRNA ISR序列RFLP分析[J]. 畜牧兽医学报, 2013, 44(6): 919–924.
CUI G L, ZHONG S X, YANG S F, et al. Isolation and identification of Proteus mirabilis from goat and the analysis of its 16S-23S rRNA ISR sequence by RFLP[J]. Acta Veterinaria Et Zootechnica Sinica, 2013, 44(6): 919–924. (in Chinese) |
| [6] |
叶树株, 黄言钧, 张理谟, 等. 猪变形杆菌感染症的调查研究[J]. 畜牧兽医学报, 1991, 22(1): 74–77.
YE S Z, HUANG Y J, ZHANG L M, et al. An investigative study on infection of proteus in pigs[J]. Acta Veterinaria et Zootechnica Sinica, 1991, 22(1): 74–77. DOI: 10.3321/j.issn:0366-6964.1991.01.014 (in Chinese) |
| [7] |
刘泽文, 袁芳艳, 田永祥, 等. 猪变形杆菌病的研究进展[J]. 安徽农业科学, 2014, 42(12): 3578–3579.
LIU Z W, YUAN F Y, TIAN Y X, et al. Research progress of porcine Proteus mirabilis disease[J]. Journal of Anhui Agricultural Sciences, 2014, 42(12): 3578–3579. DOI: 10.3969/j.issn.0517-6611.2014.12.041 (in Chinese) |
| [8] |
李欣南, 夏欣, 李永才, 等. 奇异变形杆菌研究进展[J]. 现代畜牧兽医, 2011, 21(12): 73–75.
LI X N, XIA X, LI Y C, et al. The research progress of Proteus mirabilis animal origin[J]. Modern Journal of Animal Husbandry and Veterinary Medicine, 2011, 21(12): 73–75. DOI: 10.3969/j.issn.1672-9692.2011.12.051 (in Chinese) |
| [9] |
冯烨, 刘军, 孙洋, 等. 噬菌体最新分类与命名[J]. 中国兽医学报, 2013, 33(12): 1954–1958.
FENG Y, LIU J, SUN Y, et al. An introduction to current classification and nomenclature of bacterial viruses[J]. Chinese Journal of Veterinary Science, 2013, 33(12): 1954–1958. (in Chinese) |
| [10] | ACKERMANN H W. 5500 phages examined in the electron microscope[J]. Arch Virol, 2007, 152(2): 227–243. DOI: 10.1007/s00705-006-0849-1 |
| [11] | SAVALIA D, WESTBLADE L F, GOEL M, et al. Genomic and proteomic analysis of phiEco32, a novel Escherichia coli bacteriophage[J]. J Mol Biol, 2008, 377(3): 774–789. DOI: 10.1016/j.jmb.2007.12.077 |
| [12] |
陈绵绵. 大肠杆菌噬菌体宿主特异性形成的尾丝蛋白分子基础[D]. 南京: 南京农业大学, 2017.
CHEN M M. Molecular mechanism of tail fiber protein for host specificity in Escherichia coli phage[D]. Nanjing: Nanjing Agricultural University, 2017. (in Chinese) |
| [13] | NHO S W, HA M A, KIM K S, et al. Complete Genome sequence of the bacteriophages ECBP1 and ECBP2 isolated from two different Escherichia coli strains[J]. J Virol, 2012, 86(22): 12439–12440. DOI: 10.1128/JVI.02141-12 |
| [14] | LI Y Z, CHEN M M, TANG F, et al. Complete genome sequence of the novel lytic avian pathogenic coliphage NJ01[J]. J Virol, 2012, 86(24): 13874–13875. DOI: 10.1128/JVI.02727-12 |
| [15] | MIRZAEI M K, ERIKSSON H, KASUGA K, et al. Genomic, proteomic, morphological, and phylogenetic analyses of vB_EcoP_SU10, a Podoviridae phage with C3 morphology[J]. PLoS One, 2014, 9(12): e116294. DOI: 10.1371/journal.pone.0116294 |
| [16] | KROPINSKI A M, LINGOHR E J, ACKERMANN H W. The genome sequence of enterobacterial phage 7-11, which possesses an unusually elongated head[J]. Arch Virol, 2011, 156(1): 149–151. DOI: 10.1007/s00705-010-0835-5 |
| [17] | REN H Y, LI Z, XU L, et al. Genome sequence analysis of Vibrio parahaemolyticus lytic phage Vp_R1 with a C3 morphotype[J]. Arch Virol, 2019, 164(11): 2865–2871. DOI: 10.1007/s00705-019-04364-0 |
| [18] | CHOPIN A, DEVEAU H, EHRLICH S D, et al. KSY1, a lactococcal phage with a T7-like transcription[J]. Virology, 2007, 365(1): 1–9. DOI: 10.1016/j.virol.2007.03.044 |
| [19] | YAZDI M, BOUZARI M, GHAEMI E A. Genomic analyses of a novel bacteriophage (VB_PmiS-Isfahan) within Siphoviridae family infecting Proteus mirabilis[J]. Genomics, 2019, 111(6): 1283–1291. DOI: 10.1016/j.ygeno.2018.08.008 |
| [20] |
SAMBROOK J, RUSSELL D W. 分子克隆实验指南[M]. 黄培堂, 王嘉玺, 朱厚础, 译. 3版. 北京: 科学出版社, 2002: 185-187, 195-197.
SAMBROOK J, RUSSELL D W. Molecular cloning: a laboratory manual[M]. HUANG P T, WANG J X, ZHU H C, trans. 3rd ed. Beijing: Science Press, 2002: 185-187, 195-197. (in Chinese) |
| [21] | ACKERMANN H W, EISENSTARK A. The present state of phage taxonomy[J]. Intervirology, 1974, 3(4): 201–219. DOI: 10.1159/000149758 |
| [22] | BAILLY-BECHET M, VERGASSOLA M, ROCHA E. Causes for the intriguing presence of tRNAs in phages[J]. Genome Res, 2007, 17(10): 1486–1495. DOI: 10.1101/gr.6649807 |
| [23] | DWIVEDI B, XUE B J, LUNDIN D, et al. A bioinformatic analysis of ribonucleotide reductase genes in phage genomes and metagenomes[J]. BMC Evol Biol, 2013, 13: 33. DOI: 10.1186/1471-2148-13-33 |
| [24] | PAGET M S B, KANG J G, ROE J H, et al. sigma R, an RNA polymerase sigma factor that modulates expression of the thioredoxin system in response to oxidative stress in Streptomyces coelicolor A3(2)[J]. EMBO J, 1998, 17(19): 5776–5782. DOI: 10.1093/emboj/17.19.5776 |
| [25] | FISCHETTI V A. Bacteriophage lytic enzymes: novel anti-infectives[J]. Trends Microbiol, 2005, 13(10): 491–496. DOI: 10.1016/j.tim.2005.08.007 |
| [26] | GÓRSKI A, WEBER-DABROWSKA B. The potential role of endogenous bacteriophages in controlling invading pathogens[J]. Cell Mol Life Sci, 2005, 62(5): 511–519. DOI: 10.1007/s00018-004-4403-6 |
| [27] | LAVIGNE R, SETO D, MAHADEVAN P, et al. Unifying classical and molecular taxonomic classification: analysis of the Podoviridae using BLASTP-based tools[J]. Res Microbiol, 2008, 159(5): 406–414. DOI: 10.1016/j.resmic.2008.03.005 |
| [28] | HUH H, WONG S, ST JEAN J, et al. Bacteriophage interactions with mammalian tissue: Therapeutic applications[J]. Adv Drug Deliv Rev, 2019, 145: 4–17. DOI: 10.1016/j.addr.2019.01.003 |
| [29] | KORTRIGHT K E, CHAN B K, KOFF J L, et al. Phage therapy: A renewed approach to combat antibiotic-resistant bacteria[J]. Cell Host Microbe, 2019, 25(2): 219–232. DOI: 10.1016/j.chom.2019.01.014 |
| [30] | HOOTON S P T, ATTERBURY R J, CONNERTON I F. Application of a bacteriophage cocktail to reduce Salmonella typhimurium U288 contamination on pig skin[J]. Int J Food Microbiol, 2011, 151(2): 157–163. DOI: 10.1016/j.ijfoodmicro.2011.08.015 |
| [31] | SMITH H W, HUGGINS M B. Effectiveness of phages in treating experimental Escherichia coli diarrhoea in calves, piglets and lambs[J]. J Gen Microbiol, 1983, 129(8): 2659–2675. |
| [32] | WAGENAAR J A, VAN BERGEN M A P, MUELLER M A, et al. Phage therapy reduces Campylobacter jejuni colonization in broilers[J]. Vet Microbiol, 2005, 109(3-4): 275–283. DOI: 10.1016/j.vetmic.2005.06.002 |
| [33] | NELSON D, LOOMIS L, FISCHETTI V A. Prevention and elimination of upper respiratory colonization of mice by group A streptococci by using a bacteriophage lytic enzyme[J]. Proc Natl Acad Sci U S A, 2001, 98(7): 4107–4112. DOI: 10.1073/pnas.061038398 |



