2. 省部共建农产品质量安全危害因子与风险防控国家重点实验室, 杭州 310021;
3. 浙江农业科学院农产品质量标准研究所, 杭州 310021;
4. 金华市农业科学研究院畜牧兽医研究所, 金华 321017
2. State Key Laboratory for Managing Biotic and Chemical Threats to the Quality and Safety of Agro-products, Hangzhou 310021, China;
3. Institute of Quality and Standard for Agro-products, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;
4. Institute of Animal Husbandry and Veterinary Medicine, Jinhua Academy of Agricultural Sciences, Jinhua 321017, China
猪肠道菌群是一个复杂且动态变化的微生态系统,成年猪肠道细菌数量约为1014个[1]。但猪肠道菌群会受到多种因素的影响,如日龄等,仔猪刚出生时,肠道菌群组成不稳定,随着日龄增加,猪肠道菌群从单一向复杂逐渐演化,并逐渐形成以厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)为主的菌群结构[2]。Wang等[1]对不同生长阶段猪肠道菌群结构分析发现,布劳特氏菌属(Blautia)和密螺旋体菌属(Treponema)的相对丰度随着日龄的增加而增加,大肠杆菌(Escherichia)和瘤胃球菌属(Ruminococcus)相对丰度逐渐减少。刘颖[3]发现,梅山猪肠道中拟杆菌属(Bacteroides)和梭杆菌属(Fusobacterium)随着日龄的增加而减少。肠道菌群与脂肪沉积存在一定的相关性[1],张丽萍[4]采用荧光定量PCR检测发现,太湖猪和长白猪回肠内容物的拟杆菌属-普雷沃氏菌属细菌数量随着日龄增加而逐渐增加,与背膘厚变化呈正相关。肠道菌群对脂肪沉积的调控作用主要是通过影响其代谢活动和调节肠道中与脂肪代谢相关基因的表达来实现的[5]。脂肪酸结合蛋白(fatty acidbinding proteins, FABPs)存在于肠黏膜和其他利用脂肪酸的组织中,其中,FABP1主要在哺乳动物的肝和小肠中表达,FABP2特异性地在小肠中表达,FABP4主要在脂肪细胞中表达[6],参与细胞脂肪酸的吸收和运输以及脂肪酸代谢平衡的调节,维持细胞脂质稳态,在调节脂肪储存和分布中发挥重要作用[7]。Zimmerman和Veerkamp[8]研究表明,无菌小鼠接种拟杆菌后回肠中FABP4表达量增加,增强了回肠细胞对脂肪酸吸收转运的能力,从而促进了脂肪的吸收和机体脂肪沉积。Huang等[9]研究结果表明,低出生体重(LBW)仔猪回肠中FABP1、FABP2和FABP4表达水平和脂肪合成能力降低,且FABP1表达量与回肠微生物瘤胃菌属UCG-008(Ruminococcaceae UCG-008)和霍氏真杆菌([Eubacterium]hallii group)丰度呈正相关;FABP2和FABP4表达量与回肠微生物布劳特氏菌属(Blautia)和双歧杆菌属(Bifidobacterium)丰度呈负相关。
金华猪是我国重要的地方优良品种,具有体脂含量高、肉质好等特点,也是重要的肥胖或者脂肪沉积研究模型动物[10-11]。本实验室前期就金华猪脂肪沉积与肠道菌群的关系开展了相关研究,发现金华猪的肠道菌群结构显著不同于瘦肉型长白猪[12],并且通过粪菌移植试验也证明了金华猪的梭菌属(Clostridium)、颤螺菌属(Oscillibacter)和普雷沃菌(Prevoella)属等可以促进脂肪的沉积[13];通过对金华猪不同肠段微生物功能分析发现,回肠微生物可能有助于金华猪摄入较多脂肪从而促进体脂沉积[11],但目前, 关于猪小肠(例如回肠)中肠道菌群结构与脂肪沉积的研究较少。因此,本试验选择不同日龄的金华猪为研究对象,重点分析金华猪回肠菌群结构的发育性变化以及与脂肪酸结合蛋白和背膘厚的相关性,为调节猪的脂肪沉积和改善肉质提供理论依据。
1 材料与方法 1.1 试验动物和样品采集在金华市农业科学研究院试验牧场选择同批次的60头金华猪进行饲养,饲养期间,自由采食和饮水,预防免疫和饲养管理按常规方法进行。分别在45(D45)、90(D90)、150(D150)和270(D270)日龄各选择4头金华猪公猪。日粮组成及主要营养成分见表 1。屠宰前禁食12 h,称重屠宰,并测定其背膘厚,具体参照王富国等[14]的测定方法,分别测定猪前部(肩部最厚处)、6~7肋骨处、中部(胸腰椎结合处)和后部(腰荐结合处)背膘厚,然后取平均值。迅速采集回肠中段内容物用于菌群结构分析;参照Wang等[15]的方法采集回肠黏膜,具体方法:取回肠中段2~3 cm肠道组织,剪开后在预冷的生理盐水中漂洗,滤纸吸干,于平皿内刮取回肠黏膜,转移至1.5 mL离心管内,置于液氮速冻,于干冰中转运至实验室,-80 ℃保存。
|
|
表 1 基础日粮组成及营养水平 Table 1 Composition and nutrient levels of basal diets |
使用TRIzol® Plus RNA Purification Kit提取回肠黏膜RNA,按照逆转录试剂盒SuperScriptTMⅢ First-Strand Synthesis SuperMix for qRT-PCR(Invitrogen)说明书逆转录成cDNA。参照Huang等[9]的引物序列(表 2)对金华猪回肠黏膜的脂肪酸结合蛋白基因FABP1、FABP2和FABP4进行荧光定量PCR分析,引物由生工生物工程(上海)股份有限公司合成。qRT-PCR使用Power SYBR®Green PCR Master Mix在CFX384多重实时荧光定量PCR仪(美国Bio-Rad)中进行,qRT-PCR扩增体系为20 μL:SDW 8.0 μL, Power SYBR® Green Master Mix 10.0 μL, 上、下游引物各0.5 μL,cDNA 1.0 μL。反应条件为:95 ℃ 1 min;95 ℃ 15 s, 63 ℃ 25 s,40个循环。每个样品重复三次,各个基因的相对表达水平以2-ΔΔCt进行统计分析。
|
|
表 2 qRT-PCR引物序列 Table 2 Primer sequnence for qRT-PCR |
采用QIAamp Stool Mini Kit (QIAGEN, CA)试剂盒提取金华猪回肠内容物微生物基因组DNA,使用Nanodrop 2000测定所提取DNA的纯度和浓度。
1.4 PCR反应和高通量测序分析测序由明科生物技术(杭州)有限公司完成,使用引物515F(GTGCCAGCMGCCGCGG)和907R(CCGTCAATTCMTTTRAGTTT)对细菌16S rRNA基因V4~V5进行PCR扩增,采用Illumina Hiseq高通量测序平台进行测序,按照97%相似性对非重复序列进行OTU聚类,使用Sliva和RPD数据库对所有OTU的代表性序列进行物种匹配。采用主坐标分析(principal coordinate analysis, PCoA)进行样品间beta多样性分析。
1.5 统计分析不同日龄金华猪的体重、背膘厚、菌群相对丰度和脂肪酸结合蛋白基因表达数据均采用SPSS 18.0软件进行单因素方差分析(one-way ANOVA),使用GraphPad Prism 8软件作图,结果以“平均值±标准误”表示,P<0.05为差异显著,P<0.01为差异极显著。金华猪回肠菌群丰度与脂肪酸结合蛋白和背膘厚的相关性采用Spearman相关系数进行分析。
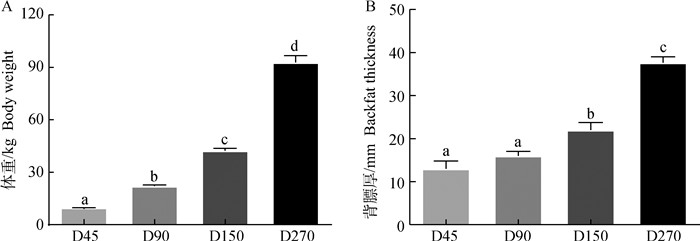
2 结果 2.1 不同日龄金华猪体重和背膘厚由图 1可知,随着日龄的增加,金华猪体重和背膘厚均显著增加(P<0.05)。在45、90、150和270日龄时金华猪的平均体重分别为9.79、22、42.35和92.60 kg(图 1A),背膘厚分别为13.0、16.0、22.0和37.6 mm(图 1B)。
|
图中值均为“平均值±标准误”;数据柱上小写字母不同表示差异显著(P<0.05),相同表示差异不显著(P>0.05)。图 2同 All exprimental data are expressed as the "mean±standard error"; Value columns with different lowercase letters mean significant difference (P < 0.05), while the same lowercase letter means no sigificant differences(P > 0.05). The same as Figure 2 图 1 不同日龄金华猪体重和背膘厚 Fig. 1 Body weight and backfat thickness of Jinhua pigs at different ages |
通过荧光定量PCR发现,随着日龄的增加,金华猪回肠脂肪酸结合蛋白FABP1、FABP2和FABP4基因表达均显著上升(P<0.05)(图 2)。与45日龄相比,FABP1表达量在270日龄升高了123.09%(P=0.014);FABP2表达量在150和270日龄分别升高了65.04%(P=0.021)和93.52%(P=0.002);FABP4表达量在150和270日龄分别升高了64.5%(P=0.012)和102.81%(P<0.001)。

|
图 2 不同日龄金华猪回肠脂肪酸结合蛋白基因表达水平 Fig. 2 Expression of fatty acid binding protein genes in the ileum of Jinhua pigs at different ages |
随着金华猪日龄的增加,菌群OTU数目、香农指数和Chao1指数均呈先降低后升高的趋势(表 3);各日龄覆盖度指数也均大于0.99,说明了该测序结果可以代表样本中菌群的真实情况。
|
|
表 3 不同日龄金华猪回肠菌群样品测序结果 Table 3 Sequencing result of ileal microbiota samples of Jinhua pigs at different ages |
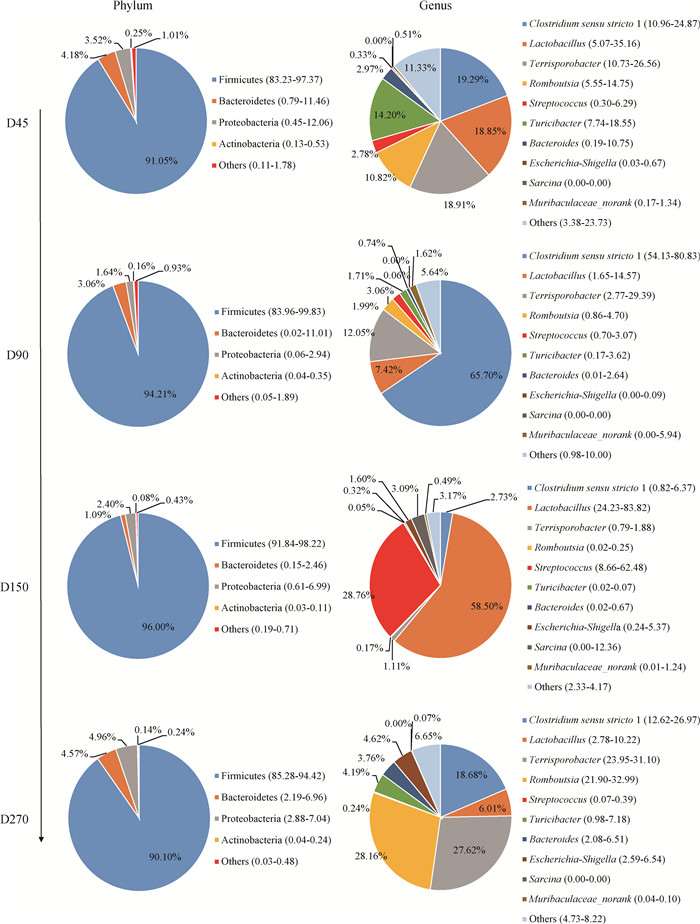
对不同日龄金华猪的回肠菌群结构进行分析发现,在门水平上(图 3),优势菌门主要是厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria);其中,厚壁菌门相对丰度在各日龄均大于90%,为绝对优势菌门。
|
图 3 不同日龄金华猪回肠菌群结构 Fig. 3 Microbial community structure in the ileum of Jinhua pigs at different ages |
在属水平上,相对丰度最高的10个优势菌属分别为狭义梭菌属1(Clostridium sensu stricto 1)、乳酸菌属(Lactobacillus)、土孢杆菌属(Terrisporobacter)、Romboutsia、链球菌属(Streptococcus)、苏黎世杆菌属(Turicibacter)、拟杆菌属(Bacteroides)、埃希氏菌-志贺氏菌属(Escherichia-Shigella)、八叠球菌(Sarcina)和Muribaculaceae_norank;在45、90日龄时狭义梭菌属1(Clostridium sensu stricto 1)和乳酸菌属(Lactobacillus)为主要优势菌属;而在150日龄时乳酸菌属(Lactobacillus)取代了狭义梭菌属1(Clostridium sensu stricto 1)成为相对丰度最高的优势属,其相对丰度为58.50%,显著高于其他日龄(P<0.05);270日龄时,Romboutsia成为肠道的第一优势菌属,其相对丰度为28.16%。
通过对相对丰度最高的50个菌属进行热图分析发现,不同日龄金华猪回肠中的优势菌属不同,具有明显的动态演替特征(图 4)。

|
图 4 金华猪回肠中相对丰度最高的50个菌属热图 Fig. 4 Heatmap of 50 genera with the highest relative abundance in the ileum of Jinhua pigs |
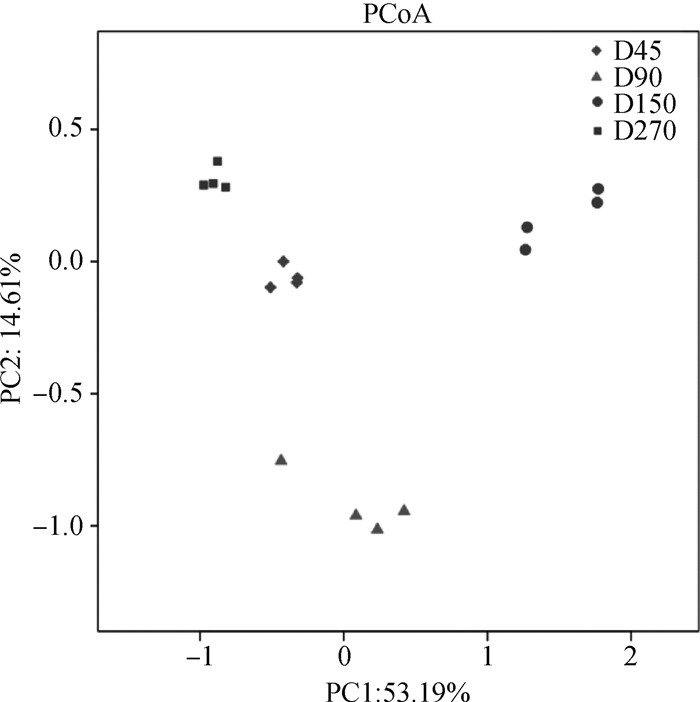
对不同日龄金华猪回肠菌群结构进行PCoA(图 5)发现,各日龄有显著不同的聚类,说明不同日龄金华猪回肠菌群结构存在较大差异。
|
图 5 不同日龄金华猪回肠菌群结构主坐标分析 Fig. 5 PCoA plot of microbial community structure in the ileum of Jinhua pigs at different ages |
对金华猪回肠脂肪酸结合蛋白表达和背膘厚的相关性分析发现(表 4),金华猪回肠脂肪酸结合蛋白FABP1、FABP2和FABP4表达与背膘厚呈正相关,相关系数分别为0.480 1,0.597 9和0.795 3。
|
|
表 4 脂肪酸结合蛋白基因表达与背膘厚的相关性 Table 4 Correlation between fatty acid binding protein genes expression and backfat thickness |
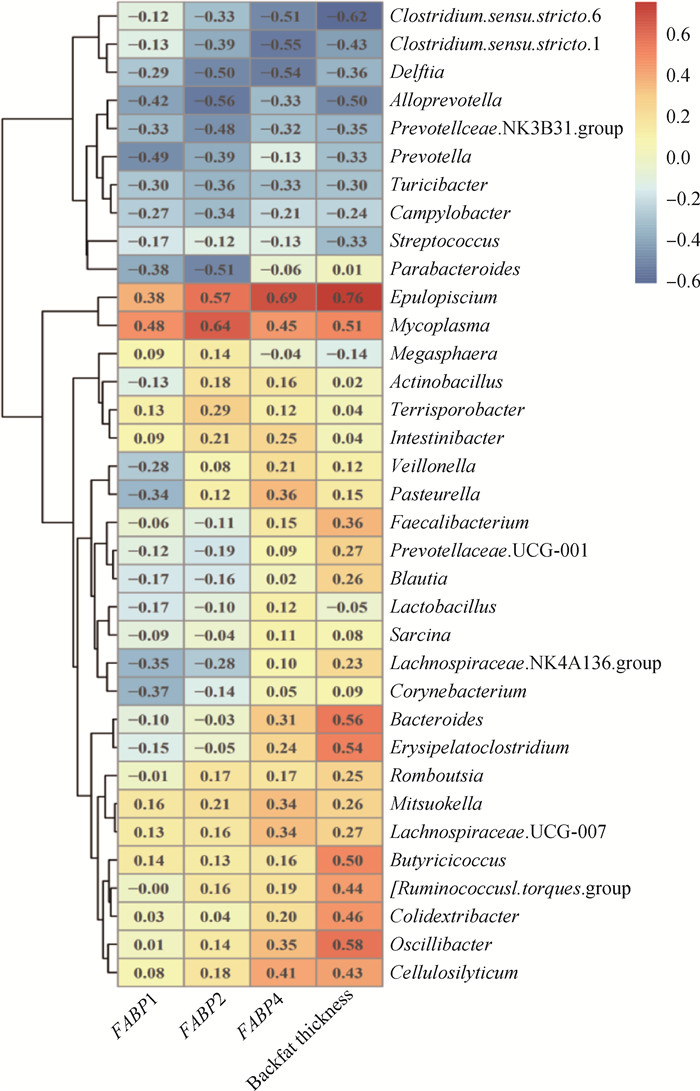
对金华猪回肠菌群与脂肪酸结合蛋白表达和背膘厚的相关性分析发现(图 6),Epulopiscium和Mycoplasma相对丰度与FABP1、FABP2和FABP4表达量和背膘厚均呈正相关(ρ范围0.38~0.76);丁酸球菌属(Butyricicoccus)、Erysipelatoclostridium、拟杆菌属(Bacteroides)和颤螺菌属(Oscillibacter)相对丰度与背膘厚呈正相关(ρ范围0.50~0.58);Delftia和拟普雷沃菌属(Alloprevo- tella)相对丰度与FABP2表达量呈负相关(ρ范围-0.50~-0.56);狭义梭菌属1(Clostridium sense stricto 1)和狭义梭菌属6(Clostridium sense stricto 6)相对丰度与FABP4表达量和背膘厚呈负相关(ρ范围-0.43~-0.62)。
|
图 6 金华猪回肠菌群与脂肪酸结合蛋白基因表达和背膘厚的相关性分析 Fig. 6 Correlation analysis of ileal microbial community structures with fatty acid binding protein genes expression and backfat thickness in Jinhua pigs |
不同生长阶段猪肠道的菌群多样性、结构和组成不同,可能与饲粮或肠道生理相关[1]。本研究中,alpha多样性分析表明,金华猪回肠菌群的香农指数、Chao1指数随着日龄的增加均呈先降低后增加的趋势。刘颖[3]对梅山猪不同发育阶段肠道微生物变化的研究发现,小肠菌群多样性呈先降低后增加的趋势,与本研究结果相一致。对金华猪回肠菌群结构分析发现,在门水平上,不同日龄阶段回肠的优势菌门均以厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)为主,二者占金华猪回肠菌群的相对丰度为95%左右,其中厚壁菌门占回肠菌群的90%以上,与本实验室前期[13]和Isaacson和Kim[16]研究结果一致。在属水平上,金华猪回肠中狭义梭菌属1(Clostridium sensu stricto 1)、乳酸菌属(Lactobacillus)是相对丰度最高的菌属。Looft等[17]研究发现,杜长大猪回肠中相对丰度最高的菌属是苏黎世杆菌属(Turicibacter)和厌氧杆菌属;Yang等[18]研究发现,在莱芜猪回肠中相对丰度最高的是梭菌属,不同研究的肠道菌群丰度差异可能是由于品种、日龄和环境等造成的。在270日龄时,Romboutsia和土孢杆菌属(Terrisporobacter)相对丰度显著升高说明了金猪猪肠道中的共生益生菌从前期狭义梭菌属1、乳酸菌属为主演替成Romboutsia和土孢杆菌属(Terrisporobacter)为主。有研究表明,Romboutsia具有益生性,与肠道健康密切相关[19]。
随着猪日龄的增加,其肠道菌群发生变化,脂肪沉积或与脂肪沉积相关基因的表达也发生相应变化[18]。FABPs是脂肪储存与分配的关键介质,在调节脂肪的储存和分布中发挥重要作用[6-7],且Zimmerman和Veerkamp[8]与Huang等[9]研究表明,回肠FABPs的表达水平与肠道菌群的变化密切相关。本研究表明,Epulopiscium和Mycoplasma与FABP1、FABP2和FABP4表达水平呈正相关。Epulopiscium可以编码碳水化合物酶如A.sohal和Naso,促进纤维多糖类的降解与消化[20];Mycoplasma属于产短链脂肪酸菌属,可为肠上皮细胞提供能量并降低肠道炎症[21]。因此,金华猪回肠Epulopiscium和Mycoplasma相对丰度与FABP1、FABP2和FABP4表达水平和背膘厚呈正相关可能是其通过促进纤维多糖类的降解和产生短链脂肪酸、降低肠道炎症反应等促进了FABPs的表达,增强了回肠细胞吸收和转运脂肪酸的能力[22],从而导致金华猪脂肪沉积量能力增强。此外,本研究也发现,拟杆菌属(Bacteroides)和颤螺菌属(Oscillospira)与背膘厚呈正相关;拟杆菌属(Bacteroides)可促进与回肠脂类物质吸收相关的酶类mRNA表达[23];颤螺菌属(Oscillospira)能促进宿主能量吸收从而导致脂肪沉积[24]。这部分解释了这些菌属与脂肪酸结合蛋白的发育性变化与脂肪沉积呈现相关性的原因。
4 结论金华猪回肠优势菌门为厚壁菌门,优势菌属为狭义梭菌属1、乳酸菌属、土孢杆菌属、Romboutsia和链球菌属;在属水平上菌群结构呈现发育性变化。回肠菌属Epulopiscium和Mycoplasma相对丰度与脂肪酸结合蛋白FABP1、FABP2和FABP4基因表达水平和背膘厚呈正相关,推测Epulopiscium和Mycoplasma可能通过调控脂肪酸结合蛋白表达来调节猪的脂肪沉积。
| [1] | WANG X F, TASI T, DENG F L, et al. Longitudinal investigation of the swine gut microbiome from birth to market reveals stage and growth performance associated bacteria[J]. Microbiome, 2019, 7(1): 109. DOI: 10.1186/s40168-019-0721-7 |
| [2] | LI N, HUANG S M, JIANG L L, et al. Differences in the gut microbiota establishment and metabolome characteristics between low-and normal-birth-weight piglets during early-life[J]. Front Microbiol, 2018, 9: 1798. DOI: 10.3389/fmicb.2018.01798 |
| [3] |
刘颖. 梅山猪不同发育阶段肠道微生物变化及免疫调控机制分析[D]. 扬州: 扬州大学, 2018.
LIU Y. The dynamics of gut microbime and immune regulatory mechanism analysis of Meishan pigs at different growth stages[D]. Yangzhou: Yangzhou University, 2018. (in Chinese) |
| [4] |
张丽萍. 不同品种猪肠道微生物与体脂、ANGPTL4基因关系的研究[D]. 成都: 四川农业大学, 2007.
ZHANG L P. The relationship of gut microbiota with body fat ANGPTL4 gene in different breeds of pigs[D]. Chengdu: Sichuan Agricultural University, 2007. (in Chinese) |
| [5] |
郭秀兰. 猪肠道硬壁菌门和拟杆菌门数量的检测及其相对丰度与脂肪沉积的相关性研究[D]. 成都: 四川农业大学, 2009.
GUO X L. Detection of Firmicutes and Bacteroidetes in the pig gut and the correlation between their abundance and fat deposit[D]. Chengdu: Sichuan Agricultural University, 2009. (in Chinese) |
| [6] | FISHER R M, ERIKSSON P, HOFFSTEDT J, et al. Fatty acid binding protein expression in different adipose tissue depots from lean and obese individuals[J]. Diabetologia, 2001, 44(10): 1268–1273. DOI: 10.1007/s001250100643 |
| [7] | ZHANG Y R, ZHANG J M, REN Y H, et al. Tracing the evolution of fatty acid-binding proteins (FABPs) in organisms with a heterogeneous fat distribution[J]. FEBS Open Bio, 2020, 10(5): 861–872. DOI: 10.1002/2211-5463.12840 |
| [8] | ZIMMERMAN A W, VEERKAMP J H. New insights into the structure and function of fatty acid-binding proteins[J]. Cell Mol Life Sci, 2002, 59(7): 1096–1116. DOI: 10.1007/s00018-002-8490-y |
| [9] | HUANG S M, WU Z H, LI T T, et al. Perturbation of the lipid metabolism and intestinal inflammation in growing pigs with low birth weight is associated with the alterations of gut microbiota[J]. Sci Total Environ, 2020, 719: 137382. DOI: 10.1016/j.scitotenv.2020.137382 |
| [10] | GONG Y J, ZOU X T, XIA W R, et al. Comparative metabolomic analysis of caecal digesta between Jinhua pig and Landrace pig[J]. Czech J Anim Sci, 2019, 64(8): 332–342. DOI: 10.17221/43/2018-CJAS |
| [11] | XIAO Y P, KONG F L, XIANG Y, et al. Comparative biogeography of the gut microbiome between Jinhua and Landrace pigs[J]. Sci Rep, 2018, 8(1): 5985. DOI: 10.1038/s41598-018-24289-z |
| [12] | YANG H, XIAO Y P, WANG J J, et al. Core gut microbiota in Jinhua pigs and its correlation with strain, farm and weaning age[J]. J Microbiol, 2018, 56(5): 346–355. DOI: 10.1007/s12275-018-7486-8 |
| [13] | YANG H, XIANG Y, ROBINSON K, et al. Gut microbiota is a major contributor to adiposity in pigs[J]. Front Microbiol, 2018, 9: 3045. DOI: 10.3389/fmicb.2018.03045 |
| [14] |
王国富, 高树新, 张立凡, 等. 大白猪二脂酰甘油酰基转移酶1和2基因(DGAT1和DGAT2)发育性表达特点及其与背膘厚的关联分析[J]. 农业生物技术学报, 2011, 19(4): 705–710.
WANG G F, GAO S X, ZHANG L F, et al. Developmental expression characteristics of Acyl CoA: Diacylglycerol Acyltransferase Genes 1 and 2 (DGAT1 and DGAT2) and their association analysis with backfat thickness in Large White pig[J]. Journal of Agricultural Biotechnology, 2011, 19(4): 705–710. DOI: 10.3969/j.issn.1674-7968.2011.04.016 (in Chinese) |
| [15] | WANG J J, CHEN L X, LI D F, et al. Intrauterine growth restriction affects the proteomes of the small intestine, liver, and skeletal muscle in newborn pigs[J]. J Nutr, 2008, 138(1): 60–66. DOI: 10.1093/jn/138.1.60 |
| [16] | ISAACSON R, KIM H B. The intestinal microbiome of the pig[J]. Anim Health Res Rev, 2012, 13(1): 100–109. DOI: 10.1017/S1466252312000084 |
| [17] | LOOFT T, ALLEN H K, CANTAREL B L, et al. Bacteria, phages and pigs: the effects of in-feed antibiotics on the microbiome at different gut locations[J]. ISME J, 2014, 8(8): 1566–1576. DOI: 10.1038/ismej.2014.12 |
| [18] | YANG H, HUANG X C, FANG S M, et al. Uncovering the composition of microbial community structure and metagenomics among three gut locations in pigs with distinct fatness[J]. Sci Rep, 2016, 6(1): 27427. DOI: 10.1038/srep27427 |
| [19] | GERRITSEN J. The genus Romboutsia: genomic and functional characterization of novel bacteria dedicated to life in the intestinal tract[D]. Netherlands: Wageningen University, 2015. |
| [20] | NGUGI D K, MIYAKE S, CAHILL M, et al. Genomic diversification of giant enteric symbionts reflects host dietary lifestyles[J]. Proc Natl Acad Sci USA, 2017, 114(36): E7592–E7601. DOI: 10.1073/pnas.1703070114 |
| [21] | PARK S K, KIM M S, ROH S W, et al. Blautia stercoris sp. nov., isolated from human faeces[J]. Int J Syst Evol Microbiol, 2012, 62(Pt4): 776–779. |
| [22] | MATHUR R, BARLOW G M. Obesity and the microbiome[J]. Exp Rev Gastroenterol Hepatol, 2015, 9(8): 1087–1099. DOI: 10.1586/17474124.2015.1051029 |
| [23] | HOOPER L V, WONG M H, THELIN A, et al. Molecular analysis of commensal host-microbial relationships in the intestine[J]. Science, 2001, 291(5505): 881–884. DOI: 10.1126/science.291.5505.881 |
| [24] | DUCA F A, SAKAR Y, LEPAGE P, et al. Replication of obesity and associated signaling pathways through transfer of microbiota from obese-prone rats[J]. Diabetes, 2014, 63(5): 1624–1636. DOI: 10.2337/db13-1526 |



