2. 青藏高原动物遗传资源保护与利用教育部重点实验室, 成都 610041
2. Key Laboratory of Qinghai-Tibetan Plateau Animal Genetic Resource Reservation and Utilization, Chengdu 610041, China
牛凸隆病毒(bovine torovirus, BToV)是套式病毒目(Nidovirales)托巴套氏病毒科(Tobaniviridae)凸隆病毒亚科(Torovirinae)凸隆病毒属(Torovirus)的成员,凸隆病毒属的成员还包括马凸隆病毒(equine torovirus, EToV)和猪凸隆病毒(porcine torovirus, PToV)(国际病毒分类委员会:https://talk.ictvonline.org/)。BToV主要引起犊牛腹泻甚至死亡,同时,也可引起成年牛腹泻[1-2]。近年来,有学者从牛呼吸道样本中检测到BToV,证明其具有双重组织嗜性,推测也是潜在的呼吸道致病因子[3-4]。自1982年在美国首次报道以来,目前,世界上已有17个国家报道BToV的存在或流行,其已经具有广泛的地域分布[3-10]。
BToV为有囊膜单股正链RNA病毒,目前, GenBank中仅有5条BToV基因组序列,除加拿大BToV基因组为原型毒株Breda 1以外,其余均为基因Ⅱ型毒株。这5条BToV基因组长度为28 297~ 28 475 bp,基因组由5′UTR和ORF1a、ORF1b、S、M、HE、N 6个ORF区以及3′UTR组成[11]。ORF1a、ORF1b为两个大的重叠ORF区,分别编码聚合酶pp1a和pp1b,在病毒的复制过程中起着重要作用[11]。S基因编码纤突蛋白(S蛋白),是诱导机体产生中和抗体的重要抗原[12],并且与病毒的致病性、组织嗜性和宿主范围密切相关[13-14]。S蛋白可在“类胰蛋白酶”裂解位点(1 003—1 007 aa)裂解为S1和S2两个亚基[15-16],S1亚基负责与宿主细胞表面的受体结合,S2亚基则介导病毒与宿主细胞的膜融合,在病毒的感染过程中起着重要作用[17]。M基因编码的膜蛋白在病毒的组装、成熟和核衣壳识别过程中起作用[18]。HE基因编码血凝素酯酶蛋白(HE蛋白),具有凝集素结构域(R)、酯酶结构域(E)和膜近端结构域(MP)3个结构域[19]。病毒通过HE蛋白实现与唾液酸的可逆性黏附,从而避免由于结合“假”受体而失去感染力,在启动感染的初期发挥重要作用[19]。N基因编码的核衣壳蛋白在病毒转录和翻译中发挥作用[18]。
青藏高原的高寒高海拔地理气候特点孕育了其独特的生态系统[20]。牦牛是青藏高原特有物种,全世界约有1 700万头牦牛,90%以上的牦牛都分布在我国青藏高原[21]。牦牛腹泻给牦牛养殖业带来巨大的经济损失[22]。目前,已知引起牦牛腹泻的病毒主要有A群牛轮状病毒(BRV)[23-24]、牛冠状病毒(BCoV)[25]、牛病毒性腹泻病毒(BVDV)[26]、纽布病毒(NeV)[27]和星状病毒(AstV)[22]等,但目前还没有在牦牛中检测到BToV的报道。因此,本研究旨在证实青藏高原牦牛BToV的存在并研究其分子特征,为牦牛腹泻的防控提供参考。
1 材料与方法 1.1 临床样本145份3月龄以内牦牛腹泻粪便样本于2018年5—7月采集于西藏(3个牧场)、青海(3个牧场)、四川藏区(6个牧场),样本冻存于-80 ℃冰箱备用。
1.2 主要试剂及仪器TrizolTM、pMD19-T克隆载体、反转录试剂盒、DH5α感受态细胞等购于宝生物公司;PCR预混酶购于TOYOBO公司;1640培养基购自Hyclone公司,胰蛋白酶、胎牛血清购自BI公司;HRT-18细胞系由本实验室保存;凝胶成像系统Doc2000(Bio-Rad公司,美国);荧光倒置显微镜(Olympus Corporation公司,日本);基因扩增仪、CO2 细胞培养箱(Thermo公司,美国)。
1.3 RNA提取与cDNA合成取粪便0.2 g与PBS缓冲液(1∶5)混匀后以8 000 r·min-1离心8 min,取上清液400 μL用Trizol法提取RNA,然后反转录合成cDNA冻存于-20 ℃待用。
1.4 临床样本BToV检测采用Ito等[28]的RT-PCR方法进行BToV检测。PCR阳性产物送生物工程(上海)股份有限公司测序以验证序列正确性并用于遗传进化分析。
1.5 病毒的分离培养参照Aita等[2]的BToV分离培养条件对本研究中BToV阳性样本进行BToV分离培养。
1.6 阳性样本中其他肠道病毒共感染调查对BToV阳性样本采用先前报道的方法进一步检测BRV、BCoV、BVDV 3种病毒的感染情况[23, 29-30]。PCR阳性产物送生物工程(上海)股份有限公司测序以验证序列正确性。
1.7 基因组扩增根据GenBank中现有的5条完整BToV基因组序列,采用Primer Premier 5.0设计27对引物(表 1),用于扩增基因组序列(包括5′和3′末端序列),所有BToV阳性样本均进行全基因组扩增。PCR扩增条件:94 ℃ 4 min,94 ℃ 30 s,48 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。反应体系:PCR预混酶12.5 μL,上、下游引物各1.5 μL,cDNA 2 μL,ddH2O补足25 μL。阳性PCR产物使用胶回收试剂盒回收后连接于pMD19-T载体,再转入DH5α感受态细胞,增菌后送生工生物工程(上海)有限公司进行测序。对获得的序列采用SeqMan7.0 (version 7.0; DNASTAR, USA) 拼接,拼接后的序列进行BLAST比对验证。再基于拼接后的序列设计10对引物(表 2),参照上述相同方法进行序列复核。
|
|
表 1 BToV基因组序列测定引物信息 Table 1 Primer information of BToV genome |
|
|
表 2 BToV基因组复核引物信息 Table 2 Review primer information of BToV genome |
使用ORF在线预测工具(https://www.ncbi.nlm.nih.gov/orffinder/)预测基因组的ORF位置;使用MEGA 7.0.26进行多序列比对,并采用最大似然法建立核苷酸及氨基酸系统发育树;使用MegAlign (DNASTAR Inc. , WI, USA)计算核苷酸和氨基酸相似性;使用RDP 4.97中的RDP、GeneConv、Chimaera、MaxChi、BootScan、SiScan、3Seq 7种方法进行重组分析。
2 结果 2.1 临床样本检测及病毒分离培养145份样本检出10份BToV阳性,平均阳性率约为6.9%(10/145)。其中,西藏、青海、四川藏区的阳性率分别约为11.8%(4/34)、8.9%(4/45)、3.0%(2/66)。这10份BToV阳性样本分别来自4个不同牧场,阳性率分别约为36.4%(4/11)、5.3%(1/19)、2.9%(1/35)、16.0%(4/25)。10份BToV阳性样本中有5份为BToV单独感染,3份为BToV与BRV混合感染,1份为BToV与BCoV混合感染,1份为BToV、BRV、BCoV 3种病原混合感染。将10份阳性样本接种HRT-18细胞系盲传6代后均无细胞病变产生,并检测细胞培养物中BToV,无阳性。
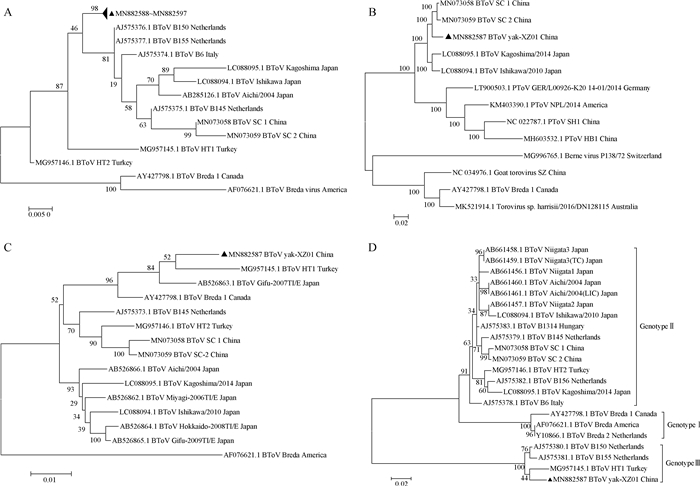
2.2 M基因遗传进化分析10份BToV阳性PCR产物测序成功(635 bp)(GenBan登录号:MN882588~MN882597),其核苷酸相似性为99.7%~100%,与GenBank中已有的13个BToV-M基因片段(≥635 bp)的核苷酸相似性为94.8%~98.6%。系统发育分析表明,本研究中的10个M基因片段聚为独立的一支,与荷兰毒株的亲缘关系最近,与美国毒株亲缘关系最远(图 1A)。进一步分析发现,这10个M基因片段与所有13个BToV-M基因片段相比,它们均在138位碱基由T突变为C,但该突变并未引起氨基酸改变。
|
▲表示本研究中的序列 ▲ marks the sequences in this study 图 1 基于BToV阳性M基因片段(635 bp)(A)、BToV yak-XZ01基因组核苷酸序列(B)、BToV yak-XZ01完整S基因氨基酸序列(C)、BToV yak-XZ01完整HE基因氨基酸序列(D)的进化树 Fig. 1 Phylogenetic tree based on partial M gene (635 bp) in positive samples (A), nucleotides sequence of genome of BToV yak-XZ01(B), complete amino acid sequence of S gene of BToV yak-XZ01 (C), and complete amino acid sequence of HE gene of BToV yak-XZ01 (D) |
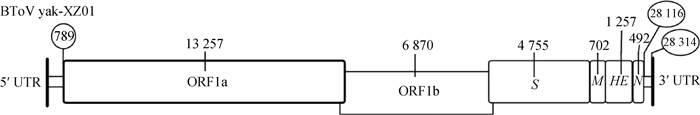
本研究成功从1份西藏阳性样本中拼接得一条牦牛源BToV基因组,命名为yak-XZ01(GenBank登录号:MN882587)。基因组全长28 314 bp,GC含量为36.31%,由5′UTR和ORF1a、ORF1b、S、M、HE、N 6个ORF区以及3′UTR组成,长度分别为788、13 257、6 870、4 755、702、1 257、492、198 bp(图 2)。该基因组与GenBank中其余5条BToV基因组相比,核苷酸相似性为82%~97%,与国内奶牛源BToV基因组SC2基因组相似性最高(97%)。基于GenBank中已有的5个BToV基因组、7个哺乳动物ToV基因组和yak-XZ01基因组的系统发育分析表明,BToV yak-XZ01与国内奶牛源基因组SC1和SC2共同聚为一支,亲缘关最近,但也有一定的遗传距离,与澳大利亚毒株亲缘关系最远(图 1B)。
|
圈中数字代表基因组中位置,剩下数字代表每个基因长度 The numbers in the circle represent the location in the genome, and the remaining numbers represent the length of each gene 图 2 BToV yak-XZ01基因组示意图 Fig. 2 Genomic organization of BToV yak-XZ01 |
BToV yak-XZ01的S基因全长4 755 bp,编码1 584个氨基酸。该S基因与GenBank中已有的14条完整BToV S基因的核苷酸相似性为91.4%~ 97.3%,氨基酸相似性为89.97%~97.85%,与其中的日本毒株AB526863.1氨基酸相似性最高。基于完整S基因氨基酸序列的系统发育分析表明,BToV yak-XZ01与土耳其毒株MG957145.1共同聚为一支,亲缘关系最近,与美国毒株AF076621.1亲缘关系最远(图 1C)。进一步分析表明,与GenBank中已有的14条完整BToV S基因序列相比,本研究中的S基因具有8个独特的氨基酸变异(S35F、T54I、I132V、Q614R、T801I、E885Q、T1 335I、K1 583R),其中,前6个氨基酸突变位点位于S1区,后2个氨基酸突变位点位于S2区。
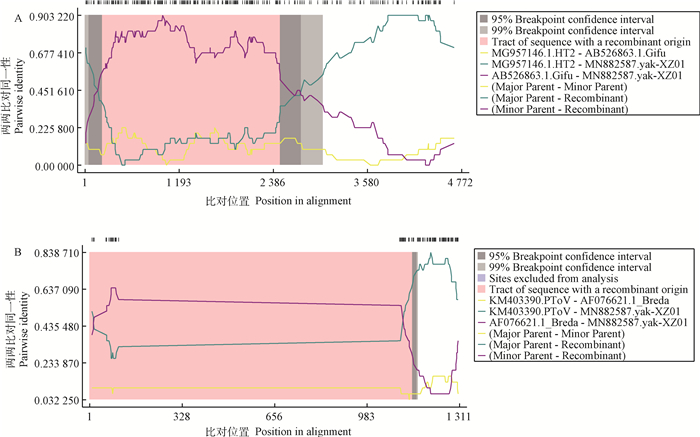
重组分析表明本研究的S基因发生区域重组事件,RDP、GeneConv、Chimaera、MaxChi、BootScan、SiScan、3Seq 7种方法支持,得分为0.623,重组区域推测为222—2 473 bp区间。预测的主要亲本毒株是土耳其MG957146.1,次要亲本毒株是日本AB526863.1(图 3A)。
|
图 3 BToV yak-XZ01 S基因(A)、HE基因(B)重组分析 Fig. 3 Recombinant analysis the S gene and HE gene of BToV yak-XZ01 |
BToV yak-XZ01的HE基因全长1 257 bp,编码418个氨基酸。该HE基因与GenBank中已有的21条完整BToV-HE基因的核苷酸相似性为71.4%~96.9%,氨基酸相似性为75.7%~96.9%,与其中的荷兰毒株AJ575380.1氨基酸相似性最高。基于完整HE基因氨基酸序列的系统发育分析表明,BToV yak-XZ01株属于BToV基因Ⅲ型毒株,与土耳其毒株MG957145.1同聚为一支,亲缘关系最近(图 1D)。进一步分析表明,与GenBank中已有的21个完整BToV HE基因序列相比,本研究的HE基因在酯酶结构域具有1个独特的氨基酸变异(P44S)。
重组分析表明本研究的HE基因发生点重组事件,RDP、GeneConv、Chimaera、MaxChi、BootScan、SiScan、3Seq 7种方法支持,得分为0.453,重组断点推测位于1 067 bp,预测的主要亲本毒株是美国PToV KM403390.1,次要亲本毒株是美国BToV AF076621.1(图 3B)。
3 讨论BToV是导致牛腹泻的一种重要病原[1-2],目前已有包括中国在内的17个国家报道该病毒[3-10]。本研究首次从牦牛中检测到BToV,阳性样本来自3个省(区)的4个不同牧场,丰富了牦牛腹泻病原谱,虽然阳性率不高,但是具有广泛的地域分布,这与土耳其的报道类似[8]。M基因检测片段的系统发育分析表明,所有牦牛源BToV毒株在遗传进化树上单独聚为一大支,表现出独特的进化趋势,提示有必要进一步开展牦牛源BToV的流行病学调查。值得注意的是,这10份阳性样本中有5份存在混合感染,这会增加临床诊断的困难性,需要引起重视。
本研究从1份西藏阳性样本中拼接得到1条牦牛源BToV基因组,与GenBank中现有的5条BToV基因组相比,该基因组具有以下4个特点:(1)这是第1个牦牛源BToV基因组;(2)该基因组也是第1个基因Ⅲ型基因组;(3)该基因组的S、HE基因存在重组事件; (4)该基因组的S、HE基因均具有独特的氨基酸突变。这些发现对进一步了解BToV的分子特征和遗传进化具有重要意义。
本研究基因组与国内奶牛源基因组SC2同源性最高,亲缘关系最近,这表明牦牛BToV有可能由国内奶牛BToV传播而来。但是,与国内奶牛源BToV SC1、SC2相比,牦牛源BToV的各ORF区域均存在氨基酸变异,这可能是BToV在感染牦牛的过程中对新宿主以及青藏高原环境适应所发生的变异[31]。本研究的BToV基因组为基因Ⅲ型,而国内关于BToV的分子流行病学资料较少[9, 32],目前只检测到基因Ⅱ型的存在,与本研究中BToV的基因型不同。前期研究表明,基因Ⅱ型和Ⅲ型毒株在荷兰[33] (GenBank登录号: AJ575380.1)和土耳其(GenBank登录号MG957145.1、MG957146.1)同时存在流行,因此有必要进一步调查基因Ⅱ型和Ⅲ型毒株在我国牛群中的流行情况。
BToV的S蛋白与BCoV类似,该蛋白是引起机体产生中和抗体的重要抗原[12],并且目前普遍认为S蛋白与病毒的致病性、组织嗜性和宿主范围的变化有关[13-14]。在病毒的感染过程中,S蛋白通过“类胰蛋白酶”裂解位点(1 003—1 007aa)裂解为S1和S2两个亚基[15-16],S1亚基负责与宿主细胞表面的受体结合,S2亚基则介导病毒与宿主细胞的膜融合[17]。本研究的S蛋白上存在8个独特的氨基酸变异(S35F、T54I、I132V、Q614R、T801I、E885Q、T1 335I、K1 583R),其中前6个变异位点位于“S1”亚基,后2个变异位点位于“S2”亚基,这可能就会改变病毒与宿主细胞表面受体结合以及膜融合的能力[34]。然而,目前关于BToV S蛋白研究资料较少,这些独特的氨基酸变异的生物学意义还需要进一步研究。
BCoV的S、HE基因重组事件十分常见[35-36]。基因重组在冠状病毒的进化中起着重要作用,病毒可通过重组来有效地改变组织嗜性和宿主范围,从而产生新的致病毒株[37-38]。BToV与BCoV基因组结构相似,两者S蛋白功能类似[39]。本研究首次报道了凸隆病毒属成员S基因的区域重组事件,进一步分析发现土耳其毒株MG957145.1也具有相同的重组事件。鉴于S基因在病毒的致病性、组织嗜性和宿主范围上起着重要作用,这种S基因重组毒株的生物学特性需要进一步研究。
目前,GenBank中有21条完整HE基因序列,根据BToV-HE基因序列特征可将其划分为3种基因型[33],最早发现的3个BToV毒株均为基因Ⅰ型毒株,来自加拿大[11]、美国[16]和荷兰(GenBank登录号: Y10866.1);基因Ⅱ型毒株共有15个,主要报道于日本[2]、中国[9]和荷兰[33]等国;基因Ⅲ型毒株共有3个,仅在土耳其(GenBank登录号MG957145.1)和荷兰[33]有报道。近年来流行的毒株均为基因Ⅱ型和Ⅲ型毒株。2003年Smits等[33]对GenBank中HE基因进行重组分析,认为基因Ⅱ型毒株是由基因Ⅰ型原型毒株与PToV重组而来,而基因Ⅲ型毒株则被认为是由基因Ⅱ型毒株与一种未知的ToV重组而来。由于该重组分析所用序列都是2003年之前上传,因此可能由于当时HE基因序列的数量和年限限制,未能预测到基因Ⅲ型毒株的亲本株。而本研究中HE基因重组事件的亲本毒株预测为美国BToVⅠ型毒株与美国PToV毒株(该序列为2014年上传),进一步分析发现GenBank中已有的3株Ⅲ型毒株也存在相同的重组事件。因此,BToV基因Ⅲ型毒株有可能是由BToVⅠ毒株与PToV毒株重组而来,这对了解Ⅲ型毒株的遗传进化提供了参考。
HE蛋白的E结构域负责行使受体破坏酶的功能,从而使病毒能够可逆地结合唾液酸[19]。而本研究的HE蛋白在E结构域中的氨基酸突变(P44S)可能会影响HE蛋白的酯酶活性,从而影响病毒对唾液酸的可逆性结合。R结构域负责行使受体结合的功能,相关的受体结合位点包括Phe207、Leu170、Leu168、Phe264、Trp109以及形成疏水袋内发夹结构的206—213位氨基酸残基,已有研究证明当受体结合位点的氨基酸残基经丙氨酸替代可明显降低HE蛋白与受体结合的能力[19]。而包括本研究毒株在内的所有基因Ⅲ型毒株均在这些关键的结合位点上有4个氨基酸变异(L170V、W109Y、G210D、T209A),这些变异可能会直接影响基因Ⅲ型毒株HE蛋白与受体结合的能力,然而并不是所有的氨基酸突变都是突变为丙氨酸,这些基因Ⅲ型毒株共有的氨基酸突变对受体结合能力的影响,还需要进一步研究予以证明。
4 结论本研究证实了BToV在牦牛中的存在,丰富了牦牛腹泻病原谱,为牦牛腹泻防控提供了参考。获得了牦牛源BToV基因Ⅲ型基因组,报道了BToV中的S基因重组事件,为进一步研究BToV的流行和遗传进化提供了参考。
| [1] | HOET A E, SMILEY J, THOMAS C, et al. Association of enteric shedding of bovine torovirus (Breda virus) and other enteropathogens with diarrhea in veal calves[J]. Am J Vet Res, 2003, 64(4): 485–490. DOI: 10.2460/ajvr.2003.64.485 |
| [2] | AITA T, KUWABARA M, MURAYAMA K, et al. Characterization of epidemic diarrhea outbreaks associated with bovine torovirus in adult cows[J]. Arch Virol, 2012, 157(3): 423–431. DOI: 10.1007/s00705-011-1183-9 |
| [3] | ITO T, OKADA N, OKAWA M, et al. Detection and characterization of bovine torovirus from the respiratory tract in Japanese cattle[J]. Vet Microbiol, 2009, 136(3-4): 366–371. DOI: 10.1016/j.vetmic.2008.11.014 |
| [4] | HOET A E, CHO K O, CHANG K O, et al. Enteric and nasal shedding of bovine torovirus (Breda virus) in feedlot cattle[J]. Am J Vet Res, 2002, 63(3): 342–348. DOI: 10.2460/ajvr.2002.63.342 |
| [5] | WOODE G N, REED D E, RUNNELS P L, et al. Studies with an unclassified virus isolated from diarrheic calves[J]. Vet Microbiol, 1982, 7(3): 221–240. DOI: 10.1016/0378-1135(82)90036-0 |
| [6] | DUCKMANTON L, CARMAN S, NAGY É, et al. Detection of bovine torovirus in fecal specimens of calves with diarrhea from Ontario farms[J]. J Clin Microbiol, 1998, 36(5): 1266–1270. DOI: 10.1128/JCM.36.5.1266-1270.1998 |
| [7] | NOGUEIRA J S, ASANO K M, DE SOUZA S P, et al. First detection and molecular diversity of Brazilian bovine torovirus (BToV) strains from young and adult cattle[J]. Res Vet Sci, 2013, 95(2): 799–801. DOI: 10.1016/j.rvsc.2013.04.006 |
| [8] | GÜLAÇTI I·, IVIDAN H, SÖZDUTMAZ I·. Detection of bovine torovirus in fecal specimens from calves with diarrhea in Turkey[J]. Arch Virol, 2014, 159(7): 1623–1627. DOI: 10.1007/s00705-014-1977-7 |
| [9] | LI H, ZHANG B, YUE H, et al. First detection and genomic characteristics of bovine torovirus in dairy calves in China[J]. Arch Virol, 2020, 165(7): 1577–1583. DOI: 10.1007/s00705-020-04657-9 |
| [10] | LOJKIĆ I, KREŠIC' N, ŠIMIC' I, et al. Detection and molecular characterisation of bovine corona and toroviruses from Croatian cattle[J]. BMC Vet Res, 2015, 11(1): 202. DOI: 10.1186/s12917-015-0511-9 |
| [11] | DRAKER R, ROPER R L, PETRIC M, et al. The complete sequence of the bovine torovirus genome[J]. Virus Res, 2006, 115(1): 56–68. DOI: 10.1016/j.virusres.2005.07.005 |
| [12] | KOOPMANS M, EDERVEEN J, WOODE G N, et al. Surface proteins of Breda virus[J]. Am J Vet Res, 1986, 47(9): 1896–1900. |
| [13] | GALLAGHER T M, BUCHMEIER M J. Coronavirus spike proteins in viral entry and pathogenesis[J]. Virology, 2001, 279(2): 371–374. DOI: 10.1006/viro.2000.0757 |
| [14] | PENG G Q, XU L Q, LIN Y L, et al. Crystal structure of bovine coronavirus spike protein lectin domain[J]. J Biol Chem, 2012, 287(50): 41931–41938. DOI: 10.1074/jbc.M112.418210 |
| [15] | HORZINEK M C, EDERVEEN J, KAEFFER B, et al. The peplomers of Berne virus[J]. J Gen Virol, 1986, 67(Pt 11): 2475–2483. |
| [16] | DUCKMANTON L M, TELLIER R, LIU P, et al. Bovine torovirus: sequencing of the structural genes and expression of the nucleocapsid protein of Breda virus[J]. Virus Res, 1998, 58(1-2): 83–96. DOI: 10.1016/S0168-1702(98)00104-X |
| [17] | LI F. Structure, function, and evolution of coronavirus spike proteins[J]. Annu Rev Virol, 2016, 3(1): 237–261. DOI: 10.1146/annurev-virology-110615-042301 |
| [18] | HOET A E, SAIF L J. Bovine torovirus (Breda virus) revisited[J]. Anim Health Res Rev, 2004, 5(2): 157–171. DOI: 10.1079/AHR200498 |
| [19] | LANGEREIS M A, ZENG Q H, GERWIG G J, et al. Structural basis for ligand and substrate recognition by torovirus hemagglutinin esterases[J]. Proc Natl Acad Sci U S A, 2009, 106(37): 15897–15902. DOI: 10.1073/pnas.0904266106 |
| [20] |
张玉波, 杜金鸿, 李俊生, 等. 青藏高原生态系统发育与生物多样性[J]. 科技导报, 2017, 35(12): 14–18.
ZHANG Y B, DU J H, LI J S, et al. Ecosystem development and biodiversity of Tibetan Plateau[J]. Science & Technology Review, 2017, 35(12): 14–18. (in Chinese) |
| [21] |
孟庆辉, 陈永杏, 董红敏, 等. 牦牛分布特点及其种群数量[J]. 家畜生态学报, 2017, 38(3): 80–85.
MENG Q H, CHEN Y X, DONG H M, et al. The distribution characteristics and populations of yak[J]. Acta Ecologae Animalis Domastici, 2017, 38(3): 80–85. DOI: 10.3969/j.issn.1673-1182.2017.03.017 (in Chinese) |
| [22] | CHEN X, ZHANG B, YUE H, et al. A novel astrovirus species in the gut of yaks with diarrhoea in the Qinghai-Tibetan Plateau, 2013[J]. J Gen Virol, 2015, 96(12): 3672–3680. DOI: 10.1099/jgv.0.000303 |
| [23] |
周芳, 岳华, 张斌, 等. 牦牛轮状病毒VP6基因序列分析及RT-PCR检测方法的建立与应用[J]. 畜牧兽医学报, 2016, 47(7): 1465–1473.
ZHOU F, YUE H, ZHANG B, et al. Establishment and application of an RT-PCR assay for yak rotavirus based on the sequence analysis of yak rotavirus VP6 gene[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(7): 1465–1473. (in Chinese) |
| [24] | YAN N, LI R, WANG Y W, et al. High prevalence and genomic characteristics of G6P[1] Bovine Rotavirus A in yak in China[J]. J Gen Virol, 2020, 101(7): 701–711. DOI: 10.1099/jgv.0.001426 |
| [25] |
何琪富, 汤承, 郭紫晶, 等. 牦牛源牛冠状病毒部分基因的扩增、序列分析及病毒分离鉴定[J]. 畜牧兽医学报, 2019, 50(2): 343–353.
HE Q F, TANG C, GUO Z J, et al. Amplification, sequence analysis of bovine coronavirus genes and isolation of the viruses from yak[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 343–353. (in Chinese) |
| [26] |
陈新诺, 肖敏, 阮文强, 等. 川藏地区牦牛牛病毒性腹泻病毒分子流行病学调查及分离鉴定[J]. 畜牧兽医学报, 2018, 49(3): 606–613.
CHEN X N, XIAO M, RUAN W Q, et al. Molecular epidemiological investigation and isolation of bovine viral diarrhea virus in yak in Sichuan-Tibet Plateau region[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 606–613. (in Chinese) |
| [27] | GUO Z J, HE Q F, ZHANG B, et al. First detection of neboviruses in yak (Bos grunniens) and identification of a novel neboviruses based on complete genome[J]. Vet Microbiol, 2019, 236: 108388. DOI: 10.1016/j.vetmic.2019.108388 |
| [28] | ITO T, OKADA N, FUKUYAMA S I. Epidemiological analysis of bovine torovirus in Japan[J]. Virus Res, 2007, 126(1-2): 32–37. DOI: 10.1016/j.virusres.2007.01.013 |
| [29] |
何琪富, 郭紫晶, 李然, 等. 牛冠状病毒RT-PCR检测方法的建立及应用[J]. 畜牧兽医学报, 2018, 49(10): 2292–2298.
HE Q F, GUO Z J, LI R, et al. Establishment and application of a RT-PCR assay for detecting bovine coronavirus[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(10): 2292–2298. DOI: 10.11843/j.issn.0366-6964.2018.10.026 (in Chinese) |
| [30] | GONG X W, LIU L H, ZHENG F Y, et al. Molecular investigation of bovine viral diarrhea virus infection in yaks (Bos gruniens) from Qinghai, China[J]. Virol J, 2014, 11: 29. DOI: 10.1186/1743-422X-11-29 |
| [31] | QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J]. Nat Genet, 2012, 44(8): 946–949. DOI: 10.1038/ng.2343 |
| [32] |
师志海, 徐照学, 兰亚莉, 等. 牛环曲病毒河南分离株的遗传进化分析[J]. 动物医学进展, 2019, 40(10): 28–31.
SHI Z H, XUE Z X, LAN Y L, et al. Phylogenetic analysis of bovine torovirus Henan strains[J]. Progress in Veterinary Medicine, 2019, 40(10): 28–31. (in Chinese) |
| [33] | SMITS S L, LAVAZZA A, MATIZ K, et al. Phylogenetic and evolutionary relationships among torovirus field variants: evidence for multiple intertypic recombination events[J]. J Virol, 2003, 77(17): 9567–9577. DOI: 10.1128/JVI.77.17.9567-9577.2003 |
| [34] | LI W H, ZHANG C S, SUI J H, et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2[J]. EMBO J, 2005, 24(8): 1634–1643. DOI: 10.1038/sj.emboj.7600640 |
| [35] | KEHA A, XUE L, YAN S, et al. Prevalence of a novel bovine coronavirus strain with a recombinant hemagglutinin/esterase gene in dairy calves in China[J]. Transbound Emerg Dis, 2019, 66(5): 1971–1981. DOI: 10.1111/tbed.13228 |
| [36] | MARTÍNEZ N, BRANDĀO P E, DE SOUZA S P, et al. Molecular and phylogenetic analysis of bovine coronavirus based on the spike glycoprotein gene[J]. Infect Genet Evol, 2012, 12(8): 1870–1878. DOI: 10.1016/j.meegid.2012.05.007 |
| [37] | GRAHAM R L, BARIC R S. Recombination, reservoirs, and the modular spike: mechanisms of coronavirus cross-species transmission[J]. J Virol, 2010, 84(7): 3134–3146. DOI: 10.1128/JVI.01394-09 |
| [38] | LI F. Receptor recognition and cross-species infections of SARS coronavirus[J]. Antiviral Res, 2013, 100(1): 246–254. DOI: 10.1016/j.antiviral.2013.08.014 |
| [39] | DE VRIES A A F, HORZINEK M C, ROTTIER P J M, et al. The genome organization of the Nidovirales: similarities and differences between arteri-, toro-, and coronaviruses[J]. Semin Virol, 1997, 8(1): 33–47. DOI: 10.1006/smvy.1997.0104 |



