先天性免疫系统是机体抵御病原体感染的第一道防线。先天性免疫系统的模式识别受体(pattern-recognition receptors, PRRs)可识别病原相关分子模式(pathogen-associated molecular patterns, PAMPs)和损伤相关分子模式(damage-associated molecular patterns, DAMPs),病原相关分子模式包括病原微生物的结构组分、核酸以及蛋白质等[1]。PRRs主要分为4个家族:Toll样受体(toll-like receptors, TLRs)、NOD样受体(NOD-like receptors, NLRs)、C型凝集素受体(C-type lectin receptors, CLRs)以及RIG-Ⅰ样受体(RIG-Ⅰ like receptors, RLRs)[2-3]。配体结合可激活PRRs从而激活下游的炎性复合体、干扰素调节因子(interferon regulatory factors, IRFs)、核转录因子κB (nuclear factor kappa-B,NF-κB)及丝裂原激活蛋白激酶(mitogen-activated protein kinases, MAPKs)等信号,引起炎性细胞因子、趋化因子和Ⅰ型干扰素等的产生。近年来,研究表明线粒体与先天性免疫反应之间存在串扰。
线粒体是动态的、具有双膜结构的细胞器,起源于15亿~25亿年前的一种古老的变形杆菌[4-6]。线粒体含有自己的遗传物质线粒体DNA,线粒体DNA是一种小的、双链的环状分子,可以编码13种氧化磷酸化系统必需的蛋白亚基、2个核糖体RNA以及22个转运RNA,驱动线粒体呼吸和ATP产生[7]。线粒体在细胞中执行多种功能,是细胞的能量工厂。除产生能量外,它们在代谢中的重要作用还包括氨基酸和脂肪酸代谢以及血红素、激素和铁硫团簇的生物合成。此外,线粒体参与调节细胞凋亡、钙稳态以及活性氧信号[8]。同时,线粒体被证明在宿主免疫反应中有着多种作用,如发挥信号和效应功能、促进免疫细胞激活和抗菌防御,并在细胞和组织损伤时触发炎症反应[9]。当受到内源性或外源性刺激时,线粒体会发生应激,释放线粒体DAMPs,如线粒体DNA、线粒体活性氧等进入细胞质或细胞外环境[10]。线粒体DNA与细菌DNA相似,包含大量可被TLR受体识别的去甲基化CpG基序。近年来,越来越多的研究证据表明,释放进入细胞质或细胞外的线粒体DNA可通过激活细胞分子信号通路参与不同类型的先天性免疫调节,在病原感染和炎症性疾病中发挥关键的作用。因此,本文就线粒体DNA自身的独特性、线粒体DNA如何激活先天性免疫反应以及其在病原感染和炎症性疾病中的作用进行讨论。
1 线粒体DNA的独特性线粒体DNA自身具有的几个独特特征与其在先天性免疫反应和炎症中的作用有关。首先,每一个真核细胞中都存在成百上千的线粒体DNA,线粒体DNA的丰度以一种细胞类型特异性的方式受到细胞严格调控,并且当受到内源性和外源性刺激时,线粒体DNA数量会发生变化,这与核DNA形成鲜明对比[11]。例如,人的骨骼肌和心肌细胞比神经元细胞含有更多的线粒体DNA来满足这些细胞更高的能量需求[12]。小鼠巨噬细胞在受到各种PAMPs(如脂多糖)刺激时会启动线粒体DNA的复制,激活含吡啶结构域的Nod样受体蛋白3(nod-like receptor pyrin domain containing 3,NLRP3)的炎性小体,从而引起促炎细胞因子的分泌和引发炎症[13]。最新的研究发现牛分枝杆菌感染小鼠巨噬细胞后线粒体DNA拷贝数也会显著增加[14]。这表明细胞线粒体DNA丰度的改变可能是一种真正的应激反应。其次,线粒体DNA除含有细菌核酸序列的残基外,其甲基化方式也与核DNA不同,这使其看起来更像是外来的而不是自身的DNA。目前,对于线粒体DNA甲基化的精确程度(无甲基化或有一定程度的甲基化)、甲基化的本质(CpG甲基化还是N6腺嘌呤甲基化)以及负责催化甲基化的酶的种类尚未达成共识[15]。然而,虽然线粒体DNA甲基化的分子细节很重要,但无论甲基化程度是高是低、甲基化本质为何,PRRs都可能识别线粒体DNA。第三,线粒体DNA仅编码小于1%的线粒体蛋白,约99%的线粒体蛋白都是核DNA编码的,这些蛋白包括参与维持线粒体结构和动力学的因子、线粒体信号转导的介质、线粒体代谢途径的成分,甚至负责线粒体DNA复制、转录和翻译的酶[16]。第四,相对于核DNA, 线粒体DNA的修复机制是有限的。线粒体缺乏核苷酸切除修复和核DNA的其他修复机制。从能量和代谢的观点来看,同一细胞中同时存在多个野生型线粒体DNA拷贝可以耐受线粒体DNA突变[17]。然而,突变或受损的线粒体DNA仍可被细胞内的先天性免疫传感器检测到并激活炎症[15]。第五,线粒体DNA转录和复制过程中产生的多种中间核酸种类(如双链RNA、无帽mRNA以及DNA: RNA杂合体)也被证明具有免疫刺激潜力[18]。线粒体DNA的独特性使其更容易与PRRs结合,从而激发先天性免疫反应,这也突显了完整的线粒体膜在阻止健康细胞和组织中先天性免疫激活的重要屏障作用。线粒体作为高度敏感的细胞器,在各种类型刺激作用下极易发生应激反应,从而失去线粒体膜的完整性,使得线粒体DNA进入细胞质,被PRRs识别,最终引发先天性免疫反应。这表明线粒体DNA可能作为病理条件下启动炎性反应的中枢环节。
另外,线粒体DNA并不是裸露存在的,而是被线粒体DNA结合蛋白[如线粒体转录因子A (mitochondrial transcription factor A,TFAM)] 包装成拟核样结构。由于线粒体DNA与呼吸复合物产生的线粒体活性氧非常接近,因此,TFAM起到保护性外壳的作用并压缩线粒体DNA,从而保护其免受氧化损伤[19]。细胞和组织中的TFAM浓度及其与线粒体DNA结合密度可能被调控,从而实现不同的包装模式和对线粒体DNA转录的精确调控[20]。此外,TFAM与其他高迁移率族蛋白类似,具有免疫调节潜能,这也同样支持了线粒体DNA及其相关分子作为先天性免疫系统激动剂的观点[21-22],更为重要的是,TFAM表达以及其与线粒体DNA的结合紧密度受到细胞和组织的自主调控,表明线粒体DNA激发的先天性免疫或炎性反应不仅存在于病理过程中,而且还可能是细胞和组织主动控制的生理过程的重要环节。
2 线粒体DNA是天然免疫反应的激活剂2004年,Collins等[23]首次报道线粒体DNA具有免疫刺激的潜能。他们发现将线粒体DNA注射到小鼠关节中会导致局部炎症,引起关节炎。进一步的研究表明,线粒体DNA引起的炎症依赖于线粒体DNA中氧化损伤的碱基的存在,注射相同序列但没有氧化碱基的寡聚脱氧核苷酸则不会引起炎症。线粒体DNA能引起有效免疫反应的观察开辟了一个全新的研究领域。此后,许多研究证实了这些早期观察,并表明线粒体DNA可以直接激活先天性免疫系统的PRRs以增强促炎反应[1](图 1)。
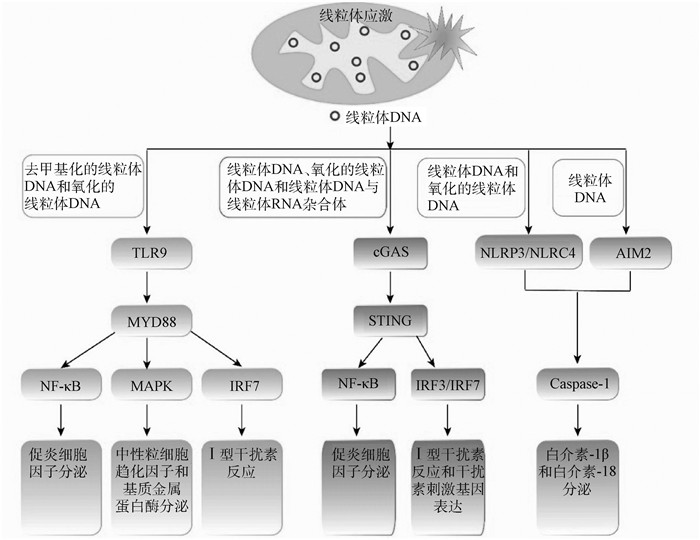
|
线粒体发生应激后,线粒体DNA释放进入细胞质。去甲基化的线粒体DNA和氧化的线粒体DNA可激活TLR9及其下游信号,从而引起促炎细胞因子、中性粒细胞趋化因子以及基质金属蛋白酶分泌,并诱导Ⅰ型干扰素反应;线粒体DNA、氧化的线粒体DNA以及线粒体DNA和线粒体RNA杂合体可激活cGAS及其下游信号,引起促炎细胞因子分泌、Ⅰ型干扰素反应以及干扰素刺激基因表达;线粒体DNA和氧化的线粒体DNA可激活NLRP3或NLRC4炎性小体,线粒体DNA可激活AIM2炎性小体,从而引起含半胱氨酸的天冬氨酸蛋白水解酶1(caspase-1)活化,诱导白介素-1β和白介素-18分泌 Upon mitochondrial stress, mitochondrial DNA (mtDNA) is released into the cytoplasm. Hypomethylated CpG mtDNA and oxidized mtDNA can activate TLR9 and its downstream signals, resulting in secretion of pro-inflammatory cytokines, neutrophil chemoattraction and matrix metalloproteinase secretion, and induce type Ⅰ interferon response. Mitochondrial DNA, oxidized mtDNA and mtDNA-mtRNA hybrid trigger the activation of cGAS and its downstream signals, causing the secretion of pro-inflammatory cytokines, type Ⅰ interferon response and interferon-stimulated gene expression. Mitochondrial DNA and oxidized mtDNA activate NLRP3 or NLRC4 inflammasomes, and mtDNA engages AIM2 inflammasomes activation, eliciting activation of caspase-1 and inducing secretion of interleukin-1 and interleukin-18 图 1 线粒体DNA激活先天性免疫系统的模式识别受体 Fig. 1 Mitochondrial DNA triggers the activation of pattern recognition receptors of innate immune system |
Toll样受体家族可通过识别大量不同的细菌特征进而激发先天性免疫。Toll样受体9(toll-like receptor 9,TLR9)是第一个被证明可以识别核酸的Toll样受体,它可识别DNA的低甲基化CpG基序[24]。TLR9主要在单核细胞、巨噬细胞、浆细胞样树突状细胞和B淋巴细胞中表达。在生理状态下,TLR9驻留在内质网中,当受到CpG DNA刺激后,TLR9转运至内体和溶酶体,识别DNA并通过衔接蛋白髓样分化初级反应蛋白88(myeloid differentiation primary response protein 88,MYD88)进行信号传导[25]。MYD88可激活MAPKs和NF-κB以触发炎症反应或激活干扰素调节因子7(interferon regulatory factor 7, IRF7)以增强树突状细胞或其他免疫细胞中的Ⅰ型干扰素反应(图 1)。2010年,Hauser实验室的两项研究证明来自线粒体DNA的CpG基序可以激活TLR9信号,从而激活p38和p42-44 MAPK活性、CXC趋化因子配体8分泌以及中性粒细胞的趋化性[26-27]。此外,他们还报道在创伤患者和其他非感染性损伤患者的血浆中存在循环的线粒体DNA,这表明线粒体DNA是一种DAMPs。线粒体DNA不仅可以通过细胞外释放机制激活TLR9,而且还可通过以细胞内部结合机制激活TLR9来促进炎症和疾病的发生。Oka等[28]研究表明,在脱氧核糖核酸酶Ⅱ(DNase Ⅱ)缺失的小鼠心肌细胞中,线粒体DNA不能通过自噬有效降解,从而在胞内被TLR9识别,诱发心肌细胞炎性反应和扩张型心肌病。此外,氧化的线粒体DNA已被证明可诱导TLR9和IRF7依赖的干扰素α表达,从而增强浆细胞样树突细胞的Ⅰ型干扰素反应[29]。综上所述,线粒体DNA释放进入细胞质后可激活TLR9受体,引起炎性反应。在风湿性关节炎、动脉粥样硬化、高血压、急性肝损伤和NASH等疾病的发生发展中扮演着重要的角色。
2.2 线粒体DNA与炎性小体激活哺乳动物的炎性小体是宿主先天性免疫系统的重要组成部分。它们是一组胞质蛋白聚集物,由至少一种先天性免疫传感器分子[如NLRP3、含CARD结构域的Nod样受体4(nod-like receptor CARD domain containing 4, NLRC4)或黑素瘤缺失蛋白2 (absence in melanoma 2, AIM2)]和一种效应分子(即caspase-1前体)组成。传感器分子和效应分子之间通过匹配的结构域结合,某些炎性小体不存在这样的结构域,所以需要额外的衔接分子(如凋亡相关斑点样蛋白)介导结合。衔接分子可连接传感器分子和效应分子,诱导炎性小体的组装。在病原体感染或无菌损伤后,炎性小体传感器、衔接分子和效应分子受到刺激后会迅速组装成一个高度有组织的、大的胞质蛋白复合物,导致caspase-1前体自我剪切和激活。活化的caspase-1可将未成熟的细胞因子[即白介素-1β(interleukin-1β, IL-1β)和白介素-18前体]分别蛋白水解为具有生物活性的形式,从而引发炎症[30]。
炎性小体可被外源性PAMPs以及在细胞坏死或应激时释放的内源性DAMPs激活[31]。虽然线粒体DAMPs的释放和线粒体动力学的改变与炎性小体的激活有关,但线粒体参与NLRP3和其他炎症小体激活的确切机制尚不清楚[32]。
近年来,大量研究证据表明线粒体DNA是炎性小体的内源性激动剂。2010年,Nakahira等[33]的研究率先将线粒体DNA与NLRP3炎性小体激活联系起来。该研究显示,当小鼠骨髓源巨噬细胞经脂多糖(lipopolysaccharide,LPS)和ATP刺激后,线粒体DNA会释放进入细胞质,促进NLRP3炎性小体的激活,从而使IL-1β和IL-18分泌增加。2012年,研究表明细胞凋亡过程中释放到细胞质内的氧化线粒体DNA可与NLRP3炎性小体结合,从而使后者活化。该项研究增进了人们对线粒体DNA和NLRP3炎性小体之间相互作用的理解[34]。利用敲除TFAM基因的方法使小鼠巨噬细胞中95%以上的线粒体DNA耗尽,导致NLRP3炎性小体的激活被抑制。然而在敲除TFAM的巨噬细胞中外源转染氧化的线粒体DNA可促进受抑制的NLRP3炎性小体的激活[13],这表明氧化的线粒体DNA可以有效激活NLRP3炎性小体。除了NLRP3炎性小体外, 线粒体DNA也可能影响其他炎性小体的激活,例如转染线粒体DNA可直接激活NLRC4炎性小体[35]。此外,Ⅱ型糖尿病患者血浆中的线粒体DNA可触发AIM2炎性小体依赖的caspase-1活化以及巨噬细胞中IL-1β和IL-18的分泌[36]。此外,由线粒体DNA释放引起的相关炎性小体的激活可能参与多种疾病过程,包括动脉粥样硬化、年龄相关性黄斑变性和某些细菌感染[35, 37]。上述研究表明,线粒体DNA作为炎性小体激动剂与疾病的致病机制有关。
虽然有明确的证据表明线粒体DNA参与炎性小体的激活,但仍有许多机制相关的问题有待研究。首先,目前线粒体DNA是如何进入细胞质的尚不清楚,这是一个主动和程序化的过程,还是一个线粒体损伤后的被动事件? 如果是主动过程,线粒体DNA的释放是否依赖一个共同的潜在机制?抑或在很大程度上依赖于细胞内外的环境因素? 尽管有人提出Bcl-2相关X蛋白(Bcl-2-associated X protein,BAX)/Bcl-2同源拮抗剂(Bcl-2 homologous antagonist/killer,BAK)依赖性线粒体外膜孔的形成和线粒体内膜向外挤出是介导细胞凋亡过程中的线粒体DNA释放的机制[38],但尚不清楚炎症激活因子触发的线粒体损伤是否利用相同的机制释放线粒体DNA。同样地,线粒体通透性转换孔(mitochondrial permeability transition pore, MPTP)在炎性小体激活剂诱导的线粒体DNA释放中的作用仍存在争议,需要进一步阐明[39]。2020年,有研究报道成孔蛋白Gasdermin D可将线粒体DNA释放到细胞质中[40]。研究表明,细菌内毒素脂多糖可激活成孔蛋白Gasdermin D,而活化的Gasdermin D可在内皮细胞中形成线粒体孔,从而释放线粒体DNA进入细胞质中。其次,尚不清楚是整个线粒体DNA基因组被释放到细胞质中,还是只有小片段受损的线粒体DNA从线粒体中泄漏出来,从而激活炎性小体。第三,尽管在免疫共沉淀试验中NLRP3和NLRC4复合物似乎与线粒体DNA或氧化的线粒体DNA结合,但NLRs本身是否可直接结合线粒体DNA还是需要其他因子尚不清楚。此外,需要进一步研究自噬或线粒体自噬通过去除受损的线粒体DNA来限制炎性小体激活的机制,以及线粒体损伤和线粒体DNA释放与炎性小体依赖的caspase-1激活的上下游关系[41-42]。最后,还需要更多的努力来解开NLRs与线粒体的动态联系以及这种联系是如何特异性地激活NLRs信号通路的[43]。
2.3 线粒体DNA与Ⅰ型干扰素反应线粒体DNA除可触发促炎反应外,释放进入细胞质和细胞外的线粒体DNA也可激活PRRs触发Ⅰ型干扰素和干扰素刺激基因表达。虽然一些机制细节仍有待研究,但明确线粒体DNA作为诱导Ⅰ型干扰素的DAMPs对理解涉及线粒体应激和以Ⅰ型干扰素表达为特征的感染性和非感染性疾病的病理机制具有重要意义。在本节内容中,将讨论线粒体DNA作为细胞质中或质膜上PRRs的配体,如何调节TLR9、循环GMP-AMP合酶(cyclic GMP-AMP synthase, cGAS)以及干扰素基因刺激蛋白(stimulator of interferon genes,STING)介导的Ⅰ型干扰素反应。
2.3.1 细胞内线粒体DNA激活cGAS-STING信号通路 cGAS-STING信号轴是Ⅰ型干扰素对外源性和内源性DNA反应的关键调节通路[44]。DNA感受器cGAS可检测细胞质DNA并生成环鸟苷一磷酸-腺苷一磷酸(cyclic guanosine monophosphate-adenosine monophosphate,cGAMP),cGAMP作为第二信使激活STING。活化的STING与TANK结合激酶1(TANK-binding kinase 1, TBK1)结合,后者使干扰素调节因子3 (interferon regulatory factor 3, IRF3) 磷酸化,以促进其同源二聚化并易位至细胞核,从而诱导干扰素β和干扰素刺激基因的表达。一些研究小组已证明cGAS是感染细胞胞质中病毒和细菌DNA的主要传感器[45-46]。此外,cGAS-STING信号在协调Ⅰ型干扰素和对自身DNA的炎症反应、诱导Ⅰ型干扰素病理过程(如Aicardi Goutieres综合征)以及调节肿瘤微环境中的炎症反应方面发挥了重要作用[47-48]。虽然大多数cGAS内源性配体被认为来源于核DNA,但一些证据表明线粒体DNA在某些情况下也可以作为细胞内源性的cGAS配体。
2014年,研究表明在Bax/Bak介导的细胞凋亡过程中释放的线粒体DNA可以激活cGAS-STING-IRF3信号通路,并触发Ⅰ型干扰素反应和干扰素刺激基因表达。在caspase-9缺失或caspase-3和caspase-7缺失的情况下,线粒体DNA激活cGAS,增强Ⅰ型干扰素反应和干扰素刺激基因表达,这表明凋亡caspase级联功能会抑制凋亡过程中细胞内在的、线粒体DNA依赖的Ⅰ型干扰素反应[49-50]。White等[50]利用免疫沉淀分析发现在Bax和Bak使线粒体外膜通透性增强后,cGAS不加区别地与线粒体DNA结合,这表明在凋亡过程中整个线粒体基因组有可能都暴露在cGAS可结合的环境中,这就引发了对有关线粒体外膜通透性增强后线粒体DNA释放机制以及cGAS是否有可能进入线粒体内部等问题的思考。
线粒体通透性转换孔的激活与长度达700 bp的线性线粒体DNA片段的释放有关,因此,在某些情况下,MPTP可能在细胞凋亡或线粒体应激期间负责线粒体DNA片段的释放[51-52]。与MPTP依赖的线粒体DNA释放机制一致,研究表明疫苗佐剂壳聚糖刺激会引起线粒体应激、线粒体活性氧的产生和胞质DNA的积累,从而激活树突细胞中的cGAS-STING信号传导和Ⅰ型干扰素应答。尽管该研究未明确表明线粒体DNA是激活cGAS的配体,但通过加入环孢菌素A抑制了壳聚糖刺激诱导的Ⅰ型干扰素和干扰素刺激基因表达,环孢菌素A以前被证明可阻断MPTP和线粒体DNA片段的释放[53]。但是,必须谨慎解释这些发现,因为环孢菌素A可以直接抑制PRR和NF-κB信号传导,因此其在该研究条件下对Ⅰ型干扰素反应的调节可能与线粒体通透性的改变无关[54-55]。MPTP作为线粒体DNA释放的直接通道的假设最近受到挑战,需要进行更多的研究才能阐明[56]。线粒体ATP合酶似乎是MPTP的主要成分,甚至可能是实际的孔[57-58]。结合质粒R388的细菌Ⅳ型偶联蛋白TrwB作为细菌DNA移位酶FtsK/SpoIIIE家族成员之一,是一种DNA依赖性的F1 ATP酶,参与结合过程中细菌之间的DNA转移[59]。TrwB在结构上与线粒体F1 ATP酶相关,而且考虑到线粒体ATP合酶在MPTP形成以及MPTP在线粒体DNA释放中的作用,有理由相信受调控的线粒体DNA释放可能是该细胞器细菌起源的进化遗迹。未来的研究应该阐明MPTP在线粒体DNA释放中的作用,或许更重要的是明确能被cGAS而不是其他DNA传感器特异性识别的线粒体DNA种类。
2015年,又一项研究为线粒体DNA依赖的cGAS-STING信号通路的激活提供了证据[60]。该研究发现TFAM杂合子(TFAM+/-)小鼠的细胞和组织中的线粒体拟核形态发生了改变,推测是由于线粒体DNA包装的改变导致干扰素刺激基因和抗病毒因子表达增加,并且对病毒感染具有明显的抵抗能力。TFAM+/-细胞中的抗病毒因子表达的增加是由cGAS-STING-TBK1信号传导到IRF3所驱动的,并依赖于异常的拟核结构的存在及其传递的应激。用双脱氧胞嘧啶处理TFAM+/-细胞可特异性地抑制线粒体DNA复制并促进线粒体DNA消耗,显著降低干扰素刺激基因表达和病毒抗性,这表明在此研究背景下cGAS的激活是线粒体DNA依赖性的。虽然该研究并没有揭示线粒体DNA片段从线粒体中释放的确切机制,但TFAM+/-细胞中线粒体的形态发生了改变,并且线粒体融合蛋白的敲除可抑制干扰素刺激基因表达,这表明线粒体形态或线粒体动力学的改变也有助于线粒体DNA的释放。总之,该研究的发现进一步支持了线粒体应激时释放到胞质的线粒体DNA可作为内源性cGAS配体的观点。2019年,Aarreberg等[61]报道炎性细胞因子IL-1β可引起线粒体DNA释放,从而激活cGAS-STING先天性免疫信号通路。该研究表明外源性IL-1β可诱导人髓样细胞、成纤维细胞和上皮细胞中干扰素β(interferon-β, IFN-β)产生和激活干扰素信号,从而诱导一种抑制登革病毒感染的强大先天性免疫反应。这与先前关于IL-1β和IFN-β信号之间是相互抑制的研究结果是相矛盾的[62]。先前的研究报道在结核分枝杆菌感染的小鼠和巨噬细胞中,IL-1β和IFN-β信号之间存在相互平衡的机制,然而在该研究中并未考虑到线粒体DNA也可引起IFN-β的产生。上述研究说明阐明线粒体DNA激活cGAS-STING信号通路的机制在相关病原的研究中具有关键作用。
2.3.2 细胞外线粒体DNA在Ⅰ型干扰素反应中的作用 除了细胞内线粒体DNA在触发Ⅰ型干扰素反应中的作用外,从活化的中性粒细胞释放的线粒体DNA也可参与邻近免疫细胞上的cGAS-STING途径或TLR9途径[29, 63]。中性粒细胞胞外诱捕网(neutrophil extracellular trap, NET)的形成与细菌清除和无菌性炎症疾病(如系统性红斑狼疮)有关,导致细胞死亡和中性粒细胞DNA或蛋白质复合物进入细胞外空间。NET最近被证明含有线粒体DNA,因此,增加了细胞外线粒体DNA可能参与Ⅰ型干扰素介导的系统性红斑狼疮患者发病机制的可能性[64]。与这一假设相一致,Lood等[63]的研究表明,含有核糖核蛋白的免疫复合物刺激中性粒细胞,可增加线粒体活性氧的生成和线粒体易位到细胞表面,以支持富含氧化的线粒体DNA的网状物的形成。DNA的氧化程度是决定其免疫刺激潜能的关键因素,人外周血单核细胞和脾细胞的Ⅰ型干扰素和促炎细胞因子的表达被氧化作用增强,经线粒体活性氧清除剂治疗的小鼠发生类似狼疮的疾病减少。研究发现对线粒体DNA富集的NETs的反应需要STING,而不是TLR9-MYD88,这表明cGAS在这种反应中起着至关重要的作用[65]。氧化线粒体DNA也被证明可以与浆细胞样树突细胞上的TLR9结合[66]。Caielli等[29]进一步指出中性粒细胞释放的氧化线粒体DNA是浆细胞样树突细胞分泌TLR9依赖性干扰素α的驱动因素。他们发现由于线粒体自噬减少,中性粒细胞将线粒体DNA-TFAM复合物(以及某些特异性的线粒体组分)释放到细胞外空间。虽然有研究表明线粒体来源的囊泡(mitochondria-derived vesicles, MDVs)通常会将氧化线粒体DNA导向溶酶体进行降解, 然而在系统性红斑狼疮患者的中性粒细胞中,这一过程被打乱,导致氧化线粒体DNA-TFAM复合物向细胞外的释放。然后,这些复合物激活晚期糖基化终末产物受体(receptor for advanced glycosylation end products, RAGE)和TLR9,促使浆细胞样树突细胞产生干扰素α,从而参与诱导系统性红斑狼疮的发生。虽然这些观察结果提示了中性粒细胞特异的线粒体自噬和线粒体DNA降解机制的可能性,但是控制MDVs运输和氧化的线粒体DNA包装机制仍不清楚。上述研究表明,细胞外线粒体DNA在炎症和Ⅰ型干扰素介导的系统性红斑狼疮以及其他自身免疫性疾病的诱导中起作用。
2.4 线粒体DNA特异性激活先天性免疫传感器的决定因素由于细胞中存在多种先天性免疫传感器(如cGAS、NLRP3、AIM2或TLR9),一个重要的问题是: 线粒体DNA从应激线粒体中逃逸后如何决定激活哪个传感器?到目前为止,已经提出了几个决定因素。首先,DNA长度很重要。虽然AIM2对短的DNA片段具有一定的结合活性,但最近的研究表明要脱离AIM2-DNA相互作用的“迟缓期”需要长度至少为70 bp的DNA片段,而当DNA大于250 bp时,产生稳定的AIM2-DNA复合物的效率达到最佳[67-68]。这些发现与早期的研究报道一致,即80 bp,但不是50 bp或更短的DNA能在体外诱导人外周血单核细胞产生AIM2炎性体依赖的IL-1β[69]。同样,cGAS也更倾向于结合长DNA,而不是短DNA[70]。相比之下,NLRP3的激活似乎与DNA片段长度无关[13]。其次,线粒体DNA的氧化还原状态似乎是决定免疫传感器激活特异性的另一个重要因素。具体来说,已被证明转染氧化的线粒体DNA可激活NLRP3,而非氧化的线粒体DNA与AIM2相结合。事实上,几乎所有的NLRP3(而不是AIM2)激活剂都能诱导线粒体损伤和随后的线粒体活性氧产生,从而促进线粒体DNA的氧化损伤[32]。第三,似乎TFAM结合的拟核结构也在某种程度上决定了线粒体DNA激活哪个免疫传感器。例如,裸露的氧化线粒体DNA完全能够在TFAM缺陷型巨噬细胞中诱导NLRP3炎性小体激活[13]。相反,TFAM似乎是线粒体DNA诱导的cGAS激活所必需的[21]。第四,免疫传感器的不同亚细胞定位在确定线粒体DNA激活哪种先天性免疫传感器方面也起着至关重要的作用。例如,TLR9位于内质网,因此需要被运输到内体和溶酶体中才可识别线粒体DNA[71]。在巨噬细胞中,尽管AIM2被认为普遍分布于整个细胞[72],但NLRP3存在于静息状态下巨噬细胞的内质网中,在NLRP3激活剂的刺激下才被转移到内质网线粒体相关膜和内质网高尔基体接触位点[73]。与这些炎性小体传感器相比,cGAS之前被认为是一种胞质蛋白,但cGAS也存在于树突状细胞的细胞核中,它利用其非酶促的N末端结构域与着丝粒DNA结合并启动先天性免疫反应[74]。然而,人和小鼠巨噬细胞中的cGAS驻留在细胞膜上,通过其N端磷酸肌醇结合结构域选择性地与PI(4, 5)P2结合。cGAS定位在细胞膜上被认为有助于抵抗病毒感染,但无助于检测自我DNA。支持这一观点的是,脂质结合有缺陷的cGAS突变体被错误地定位到细胞质和核腔,在基因毒性应激下可诱导一种强有力的Ⅰ型干扰素反应,但是对病毒感染表现出更弱的反应[75]。活化的caspase-1被证明可以裂解巨噬细胞中的cGAS,从而抑制DNA病毒感染小鼠后Ⅰ型干扰素的产生[76]。这表明巨噬细胞可能有一个精密的机制严格调控线粒体DNA等自身DNA激活cGAS的反应,以防止不可控的Ⅰ型干扰素的产生,否则可能诱发自身免疫性疾病。与上述caspase-1拮抗cGAS的作用相反,cGAS-STING通路的组分在某些情况下可能会促进炎性小体的激活。例如,STING的激活配体cGAMP被证明可以促进炎性小体的激活,从而诱导人类和小鼠的骨髓源巨噬细胞和树突细胞产生IL-1β[77]。综上所述,虽然线粒体DNA可激活多种先天性免疫信号通路,但其具体机制仍有待进一步探索。
由于相对于核DNA,线粒体DNA的修复机制是有限的,线粒体缺乏核苷酸切除修复和核DNA的其他修复机制,因此,线粒体DNA极易发生氧化损伤[17]。据报道,线粒体DNA的D-loop和Ori-L区域可发生不同种类的氧化损伤,如单链断裂(single strand breaks, SSBs)、双链断裂(double strand breaks, DSBs)、脱碱基位点(abasic sites, AP sites)和氧化碱基(如7, 8-dihydro-8-oxoguanine, 8 oxoG)[78]。氧化损伤的线粒体DNA会优先被TFAM结合[79],TFAM结合会使线粒体DNA上形成U型结构,从而有利于被cGAS识别[21]。然而,TFAM的结合对于线粒体DNA被NLRP3识别不是所需要的[13]。同时,单链断裂或双链断裂的损伤会使氧化损伤的线粒体DNA长度比非损伤线粒体DNA的长度更短,这表明氧化的线粒体DNA可能更容易被cGAS和NLRP3所识别,而不是AIM2。TLR9对于线粒体DNA的识别与线粒体DNA在细胞内的位置有关,位于内体和溶酶体中的氧化线粒体DNA或线粒体DNA可能更容易被TLR9识别。与TFAM解离的氧化线粒体DNA会被直接送到溶酶体,位于溶酶体的线粒体DNA则更容易被TLR9所识别[29]。然而,PRRs识别线粒体DNA的机制是高度复杂的,需要更多的体内和体外研究去阐释。最新的研究报道了一种可用于线粒体DNA碱基编辑的方法[80],细菌胞苷脱氨酶毒素DddA可实现线粒体的碱基编辑,这对于进一步研究线粒体DNA的特性和线粒体相关疾病的治疗提供了新的途径和方法。
3 线粒体DNA在病原感染中的作用虽然线粒体被称为细胞的能量工厂,但它也调节程序性细胞死亡途径,并在先天性免疫系统中发挥中央枢纽的作用。线粒体可通过诱导抗病毒信号通路诱发炎症[1]。胞质传感器RIG-Ⅰ和黑色素瘤分化相关蛋白5(melanoma differentiation-associated protein 5,MDA5)识别不同的病毒RNA种类并通过线粒体抗病毒信号蛋白(mitochondrial anti-viral signalling protein, MAVs)激活下游信号,引起Ⅰ型和Ⅲ型干扰素等细胞因子的表达,从而限制病毒复制[81]。除线粒体蛋白外,线粒体DNA也可通过cGAS-STING-IRF3信号通路诱导Ⅰ型干扰素产生,引发抗病毒免疫反应[60]。尽管cGAS是DNA特异性的模式识别受体,但感染登革热病毒等RNA病毒也会引起cGAS-STING反应[82]。登革热病毒感染会引起氧化的线粒体DNA释放进入细胞质中,从而激活cGAS[83]和TLR9受体[84],而且,登革热病毒已进化出免疫逃逸策略,通过编码靶向cGAS和STING降解的蛋白酶来逃避感染过程中胞质线粒体DNA诱导的cGAS信号传导,从而确保病毒感染的持久性[85]。2019年,研究表明,甲型流感病毒M2蛋白会引起线粒体DNA以MAVs依赖的方式释放进入细胞质,胞质线粒体DNA可被cGAS和DDX41(D-E-A-D-box polypeptide 41)识别,从而激活STING依赖的天然免疫反应,限制病毒在宿主体内的复制。然而,流感病毒的非结构蛋白1 (nonstructural protein 1, NS1) 也可与线粒体DNA结合以逃避STING依赖的抗病毒免疫[86]。这表明线粒体DNA可能在RNA病毒感染中发挥着关键的作用。
据报道,线粒体产生的活性氧在巨噬细胞相关的抗菌免疫反应中起关键作用。同线粒体活性氧一样, 线粒体DNA也参与抗菌反应。某些宿主免疫细胞(如中性粒细胞和肥大细胞)利用基于核DNA的胞外诱捕网(extracellular traps, ET) (由核DNA、组蛋白和抗菌肽等组成)捕获并杀死微生物病原体[87]。嗜酸性粒细胞可以特异性地释放线粒体DNA,而线粒体DNA作为胞外诱捕网的组分之一可以协助固定微生物病原体,因此,嗜酸性粒细胞可以识别并杀死微生物[88]。像中性粒细胞和肥大细胞的胞外诱捕网一样,嗜酸性粒细胞的胞外诱捕的过程依赖于活性氧的产生,但重要的是,不会导致细胞死亡,因为在线粒体DNA被释放后嗜酸性粒细胞仍然存活。尽管明显缺乏吞噬活性,但嗜碱性细胞也可以通过形成含有线粒体DNA和颗粒蛋白的嗜碱性细胞胞外诱捕网杀死细菌[89]。如上所述,在嗜酸性粒细胞和嗜碱性粒细胞中,线粒体DNA在病原体诱导的细胞外诱捕网形成等抗菌免疫反应中发挥重要作用。
关于病原体感染诱导的线粒体DNA应激,已有研究表明单纯疱疹病毒1 (herpes simplex virus 1, HSV-1)和HSV-2感染细胞会引起线粒体DNA应激和线粒体DNA拷贝数的迅速下降。缺失靶向线粒体DNA基因的HSV-1突变株在触发抗病毒反应和干扰素刺激基因表达方面效率较低[60, 90],这可能说明细胞对线粒体DNA稳态的监测是一种进化上有益的机制,它与病毒核酸的典型感应相配合,充分参与抗病毒先天性免疫。然而,宿主Ⅰ型干扰素反应也可增强某些微生物(如结核分枝杆菌)的发病机制。结核分枝杆菌感染可触发cGAS-STING信号和线粒体应激,诱导Ⅰ型干扰素和干扰素刺激基因表达,从而有利于自身存活[45, 91-92]。此外,与单纯疱疹病毒感染不同,牛分枝杆菌感染小鼠巨噬细胞会增加线粒体DNA的拷贝数,引起线粒体DNA释放进入细胞质内,参与牛分枝杆菌诱导的干扰素β产生。通过siRNA干扰TFAM表达可抑制线粒体DNA拷贝数的增加,减少进入细胞质的线粒体DNA的量,从而抑制细胞内细菌存活[14]。这些结果表明,线粒体损伤和线粒体DNA释放可能是结核分枝杆菌或牛分枝杆菌以及其他微生物用于促进cGAS激活、增加Ⅰ型干扰素反应和增强细胞内存活的一种策略。
综上所述,线粒体DNA在不同类型病原感染中可以扮演有益或有害的双重作用。然而,受损线粒体的异常积累和线粒体DNA泄漏到细胞质中也可能引起炎症性疾病。
4 线粒体DNA与炎性疾病线粒体DNA在激活炎性小体、TLR9和cGAS-STING途径中发挥重要作用,因此线粒体DNA作为一种重要的炎症驱动因子,促进许多人类疾病的发生发展也就不足为奇了。例如,线粒体DNA被认为是人和小鼠系统性红斑狼疮发病机制的关键因素。中性粒细胞是系统性红斑狼疮的关键致病性免疫细胞, 用含核糖核蛋白的免疫复合物刺激中性粒细胞导致相对于基线的线粒体活性氧产生增加,然后线粒体迁移到细胞表面,有助于形成富含氧化线粒体DNA的中性粒细胞胞外诱捕网。这些氧化的线粒体DNA被邻近的免疫细胞(例如浆细胞样树突细胞)识别,然后诱导Ⅰ型干扰素和促炎细胞因子的产生,从而促进小鼠狼疮样疾病的发展[29, 63]。中性粒细胞胞外诱捕网中的线粒体DNA的免疫刺激作用似乎依赖于STING和TLR9。然而,炎性小体介导的线粒体DNA识别是否也在系统性红斑狼疮发病机制中发挥作用还需要进一步研究。
此外,线粒体DNA还参与心血管疾病的致病进程。例如,已证明释放的线粒体DNA可激活心肌细胞中TLR9,从而诱发心肌炎和扩张型心肌病,最终导致心力衰竭。与野生型小鼠相比,DNase II缺陷小鼠心脏炎性细胞浸润增加,炎性细胞因子分泌过剩以及心肌自噬溶酶体中线粒体DNA沉积增加。使用TLR9抑制剂寡聚脱氧核苷酸处理或将TLR9基因敲除,可显著减缓DNase II缺陷小鼠的心肌病的发展。这表明线粒体DNA-TLR9信号轴可能有助于该模型小鼠心肌慢性炎症和心力衰竭的发生[28]。除诱导心力衰竭外,氧化线粒体DNA还被证明会促进动脉粥样硬化发展。死亡细胞释放的线粒体DNA可以与抗菌肽LL-37形成复合物。这种线粒体DNA: LL-37复合物可逃避DNase II的降解,并能激活浆细胞样树突细胞、中性粒细胞和内皮细胞上的TLR9受体,从而加剧动脉粥样硬化[93]。
线粒体DNA也可驱动非酒精性脂肪肝炎(non-alcoholic steatohepatitis, NASH)的致病进程[94],NASH是工业化国家最常见的肝病[95]。NASH患者和小鼠血浆中含有更高水平的氧化线粒体DNA。线粒体DNA的氧化可增加其激活TLR9的能力。血浆线粒体DNA主要存在于肝细胞来源的微颗粒(microparticles, MPs)中。从血浆中去除这些MPs导致TLR9激活的显著降低。在小鼠中,高脂饮食引起的NASH发展需要骨髓细胞中TLR9的表达,给予TLR9拮抗剂的小鼠表现出NASH症状的减轻,证明线粒体DNA释放和TLR9信号在NASH的发病机制中具有重要作用[94]。在另一项研究中,STING缺失在NASH小鼠模型中减轻了肝脂肪变性、纤维化和炎症。高脂饮食喂养小鼠的肝细胞线粒体DNA可以诱导STING依赖的枯否氏细胞(肝巨噬细胞)激活,这表明线粒体DNA诱导的STING激活可能促进NASH的肝脏炎症[96]。如以前的研究所述[97],研究线粒体DNA在NASH患者中是否也能诱导炎性小体的激活将会是一个有意义的工作。
5 小结线粒体是多功能的细胞器,是细胞代谢和信号传递的关键枢纽,但越来越多的证据表明,线粒体作为内源性损伤相关分子模式的重要来源,参与了对病原体感染和细胞损伤的先天性免疫反应。线粒体DNA相对的低甲基化、独特的结构特征和对氧化损伤的高度敏感性等特性,使其成为一种强有力的线粒体损伤相关分子模式,可以激活TLR9、炎性小体、cGAS和其他先天性免疫传感器,从而触发促炎过程和Ⅰ型干扰素反应,在病原感染和炎性疾病的发生发展中扮演着重要角色。然而,目前,关于线粒体DNA是如何释放进入细胞质或细胞外以及其如何特异性地激活不同先天性免疫传感器的机制尚不清楚。因此,进一步阐明线粒体DNA释放和感应机制、异常的线粒体DNA释放参与炎性疾病发展的机制以及线粒体DNA在病原体感染中发挥的作用,可为控制感染和相关疾病的治疗干预开辟新的途径。
| [1] | WEST A P, SHADEL G S, GHOSH S. Mitochondria in innate immune responses[J]. Nat Rev Immunol, 2011, 11(6): 389–402. DOI: 10.1038/nri2975 |
| [2] | FUKATA M, VAMADEVAN A S, ABREU M T. Toll-like receptors (TLRs) and nod-like receptors (NLRs) in inflammatory disorders[J]. Semin Immunol, 2009, 21(4): 242–253. DOI: 10.1016/j.smim.2009.06.005 |
| [3] | MARAKALALA M J, NDLOVU H. Signaling C-type lectin receptors in antimycobacterial immunity[J]. PLoS Pathog, 2017, 13(6): e1006333. DOI: 10.1371/journal.ppat.1006333 |
| [4] | GALLUZZI L, KEPP O, KROEMER G. Mitochondria: master regulators of danger signalling[J]. Nat Rev Mol Cell Biol, 2012, 13(12): 780–788. DOI: 10.1038/nrm3479 |
| [5] | LANE N, MARTIN W. The energetics of genome complexity[J]. Nature, 2010, 467(7318): 929–934. DOI: 10.1038/nature09486 |
| [6] | MCINERNEY J, PISANI D, O'CONNELL M J. The ring of life hypothesis for eukaryote origins is supported by multiple kinds of data[J]. Philos Trans R Soc Lond B Biol Sci, 2015, 370(1678): 20140323. DOI: 10.1098/rstb.2014.0323 |
| [7] | SHADEL G S, CLAYTON D A. Mitochondrial DNA maintenance in vertebrates[J]. Annu Rev Biochem, 1997, 66: 409–435. DOI: 10.1146/annurev.biochem.66.1.409 |
| [8] | SHADEL G S, HORVATH T L. Mitochondrial ROS signaling in organismal homeostasis[J]. Cell, 2015, 163(3): 560–569. DOI: 10.1016/j.cell.2015.10.001 |
| [9] | WEINBERG S E, SENA L A, CHANDEL N S. Mitochondria in the regulation of innate and adaptive immunity[J]. Immunity, 2015, 42(3): 406–417. DOI: 10.1016/j.immuni.2015.02.002 |
| [10] | WEST A P. Mitochondrial dysfunction as a trigger of innate immune responses and inflammation[J]. Toxicology, 2017, 391: 54–63. DOI: 10.1016/j.tox.2017.07.016 |
| [11] | WANG L Y. Mitochondrial purine and pyrimidine metabolism and beyond[J]. Nucleosides Nucleotides Nucleic Acids, 2016, 35(10-12): 578–594. DOI: 10.1080/15257770.2015.1125001 |
| [12] | THEILEN N T, KUNKEL G H, TYAGI S C. The role of exercise and TFAM in preventing skeletal muscle atrophy[J]. J Cell Physiol, 2017, 232(9): 2348–2358. DOI: 10.1002/jcp.25737 |
| [13] | ZHONG Z Y, LIANG S, SANCHEZ-LOPEZ E, et al. New mitochondrial DNA synthesis enables NLRP3 inflammasome activation[J]. Nature, 2018, 560(7717): 198–203. DOI: 10.1038/s41586-018-0372-z |
| [14] | SONG Y J, HUSSAIN T, WANG J, et al. Mitochondrial transcription factor a regulates Mycobacterium bovis-induced IFN-β production by modulating mitochondrial DNA replication in macrophages[J]. J Infect Dis, 2020, 221(3): 438–448. |
| [15] | WEST A P, SHADEL G S. Mitochondrial DNA in innate immune responses and inflammatory pathology[J]. Nat Rev Immunol, 2017, 17(6): 363–375. DOI: 10.1038/nri.2017.21 |
| [16] | MEHTA M M, WEINBERG S E, CHANDEL N S. Mitochondrial control of immunity: beyond ATP[J]. Nat Rev Immunol, 2017, 17(10): 608–620. DOI: 10.1038/nri.2017.66 |
| [17] | YASUKAWA T, KANG D C. An overview of mammalian mitochondrial DNA replication mechanisms[J]. J Biochem, 2018, 164(3): 183–193. DOI: 10.1093/jb/mvy058 |
| [18] | DHIR A, DHIR S, BOROWSKI L S, et al. Mitochondrial double-stranded RNA triggers antiviral signalling in humans[J]. Nature, 2018, 560(7717): 238–242. DOI: 10.1038/s41586-018-0363-0 |
| [19] | KANG D C, KIM S H, HAMASAKI N. Mitochondrial transcription factor A (TFAM): roles in maintenance of mtDNA and cellular functions[J]. Mitochondrion, 2007, 7(1-2): 39–44. DOI: 10.1016/j.mito.2006.11.017 |
| [20] | BESTWICK M L, SHADEL G S. Accessorizing the human mitochondrial transcription machinery[J]. Trends Biochem Sci, 2013, 38(6): 283–291. DOI: 10.1016/j.tibs.2013.03.006 |
| [21] | ANDREEVA L, HILLER B, KOSTREWA D, et al. cGAS senses long and HMGB/TFAM-bound U-turn DNA by forming protein-DNA ladders[J]. Nature, 2017, 549(7672): 394–398. DOI: 10.1038/nature23890 |
| [22] | JULIAN M W, SHAO G H, BAO S Y, et al. Mitochondrial transcription factor a serves as a danger signal by augmenting plasmacytoid dendritic cell responses to DNA[J]. J Immunol, 2012, 189(1): 433–443. DOI: 10.4049/jimmunol.1101375 |
| [23] | COLLINS L V, HAJIZADEH S, HOLME E, et al. Endogenously oxidized mitochondrial DNA induces in vivo and in vitro inflammatory responses[J]. J Leukoc Biol, 2004, 75(6): 995–1000. DOI: 10.1189/jlb.0703328 |
| [24] | BARBALAT R, EWALD S E, MOUCHESS M L, et al. Nucleic acid recognition by the innate immune system[J]. Annu Rev Immunol, 2011, 29: 185–214. DOI: 10.1146/annurev-immunol-031210-101340 |
| [25] | LATZ E, SCHOENEMEYER A, VISINTIN A, et al. TLR9 signals after translocating from the ER to CpG DNA in the lysosome[J]. Nat Immunol, 2004, 5(2): 190–198. DOI: 10.1038/ni1028 |
| [26] | ZHANG Q, ITAGAKI K, HAUSER C J. Mitochondrial DNA is released by shock and activates neutrophils via p38 map kinase[J]. Shock, 2010, 34(1): 55–59. DOI: 10.1097/SHK.0b013e3181cd8c08 |
| [27] | ZHANG Q, RAOOF M, CHEN Y, et al. Circulating mitochondrial DAMPs cause inflammatory responses to injury[J]. Nature, 2010, 464(7285): 104–107. DOI: 10.1038/nature08780 |
| [28] | OKA T, HIKOSO S, YAMAGUCHI O, et al. Mitochondrial DNA that escapes from autophagy causes inflammation and heart failure[J]. Nature, 2012, 485(7397): 251–255. DOI: 10.1038/nature10992 |
| [29] | CAIELLI S, ATHALE S, DOMIC B, et al. Oxidized mitochondrial nucleoids released by neutrophils drive type I interferon production in human lupus[J]. J Exp Med, 2016, 213(5): 697–713. DOI: 10.1084/jem.20151876 |
| [30] | WEN H T, MIAO E A, TING J P Y. Mechanisms of NOD-like receptor-associated inflammasome activation[J]. Immunity, 2013, 39(3): 432–441. DOI: 10.1016/j.immuni.2013.08.037 |
| [31] | MAN S M, KANNEGANTI T D. Converging roles of caspases in inflammasome activation, cell death and innate immunity[J]. Nat Rev Immunol, 2016, 16(1): 7–21. DOI: 10.1038/nri.2015.7 |
| [32] | ELLIOTT E I, SUTTERWALA F S. Initiation and perpetuation of NLRP3 inflammasome activation and assembly[J]. Immunol Rev, 2015, 265(1): 35–52. DOI: 10.1111/imr.12286 |
| [33] | NAKAHIRA K, HASPEL J A, RATHINAM V A K, et al. Autophagy proteins regulate innate immune responses by inhibiting the release of mitochondrial DNA mediated by the NALP3 inflammasome[J]. Nat Immunol, 2011, 12(3): 222–230. DOI: 10.1038/ni.1980 |
| [34] | SHIMADA K, CROTHER T R, KARLIN J, et al. Oxidized mitochondrial DNA activates the NLRP3 inflammasome during apoptosis[J]. Immunity, 2012, 36(3): 401–414. DOI: 10.1016/j.immuni.2012.01.009 |
| [35] | JABIR M S, HOPKINS L, RITCHIE N D, et al. Mitochondrial damage contributes to Pseudomonas aeruginosa activation of the inflammasome and is downregulated by autophagy[J]. Autophagy, 2015, 11(1): 166–182. DOI: 10.4161/15548627.2014.981915 |
| [36] | BAE J H, JO Ⅱ S, KIM S J, et al. Circulating cell-free mtDNA contributes to AIM2 inflammasome-mediated chronic inflammation in patients with type 2 diabetes[J]. Cells, 2019, 8(4): 328. DOI: 10.3390/cells8040328 |
| [37] | TUMURKHUU G, SHIMADA K, DAGVADORJ J, et al. Ogg1-dependent DNA repair regulates NLRP3 inflammasome and prevents atherosclerosis[J]. Circ Res, 2016, 119(6): e76–e90. |
| [38] | MCARTHUR K, WHITEHEAD L W, HEDDLESTON J M, et al. BAK/BAX macropores facilitate mitochondrial herniation and mtDNA efflux during apoptosis[J]. Science, 2018, 359(6378): eaao6047. DOI: 10.1126/science.aao6047 |
| [39] | ALLAM R, LAWLOR K E, YU E C W, et al. Mitochondrial apoptosis is dispensable for NLRP3 inflammasome activation but non-apoptotic caspase-8 is required for inflammasome priming[J]. EMBO Rep, 2014, 15(9): 982–990. DOI: 10.15252/embr.201438463 |
| [40] | HUANG L S, HONG Z G, WU W, et al. mtDNA activates cGAS signaling and suppresses the YAP-mediated endothelial cell proliferation program to promote inflammatory injury[J]. Immunity, 2020, 52(3): 475–486. DOI: 10.1016/j.immuni.2020.02.002 |
| [41] | YU J J, NAGASU H, MURAKAMI T, et al. Inflammasome activation leads to Caspase-1-dependent mitochondrial damage and block of mitophagy[J]. Proc Natl Acad Sci U S A, 2014, 111(43): 15514–15519. DOI: 10.1073/pnas.1414859111 |
| [42] | ZHONG Z Y, UMEMURA A, SANCHEZ-LOPEZ E, et al. NF-κB restricts inflammasome activation via elimination of damaged mitochondria[J]. Cell, 2016, 164(5): 896–910. DOI: 10.1016/j.cell.2015.12.057 |
| [43] | SUBRAMANIAN N, NATARAJAN K, CLATWORTHY M R, et al. The adaptor MAVS promotes NLRP3 mitochondrial localization and inflammasome activation[J]. Cell, 2013, 153(2): 348–361. DOI: 10.1016/j.cell.2013.02.054 |
| [44] | CHEN Q, SUN L J, CHEN Z J J. Regulation and function of the cGAS-STING pathway of cytosolic DNA sensing[J]. Nat Immunol, 2016, 17(10): 1142–1149. DOI: 10.1038/ni.3558 |
| [45] | COLLINS A C, CAI H C, LI T, et al. Cyclic GMP-AMP synthase is an innate immune DNA sensor for Mycobacterium tuberculosis[J]. Cell Host Microbe, 2015, 17(6): 820–828. DOI: 10.1016/j.chom.2015.05.005 |
| [46] | GAO D X, WU J X, WU Y T, et al. Cyclic GMP-AMP synthase is an innate immune sensor of HIV and other retroviruses[J]. Science, 2013, 341(6148): 903–906. DOI: 10.1126/science.1240933 |
| [47] | BARBER G N. STING: infection, inflammation and cancer[J]. Nat Rev Immunol, 2015, 15(12): 760–770. DOI: 10.1038/nri3921 |
| [48] | CROW Y J, MANEL N. Aicardi-Goutières syndrome and the type I interferonopathies[J]. Nat Rev Immunol, 2015, 15(7): 429–440. DOI: 10.1038/nri3850 |
| [49] | RONGVAUX A, JACKSON R, HARMAN C C D, et al. Apoptotic caspases prevent the induction of type I interferons by mitochondrial DNA[J]. Cell, 2014, 159(7): 1563–1577. DOI: 10.1016/j.cell.2014.11.037 |
| [50] | WHITE M J, MCARTHUR K, METCALF D, et al. Apoptotic caspases suppress mtDNA-induced STING-mediated type I IFN production[J]. Cell, 2014, 159(7): 1549–1562. DOI: 10.1016/j.cell.2014.11.036 |
| [51] | GARCIA N, CHÁVEZ E. Mitochondrial DNA fragments released through the permeability transition pore correspond to specific gene size[J]. Life Sci, 2007, 81(14): 1160–1166. DOI: 10.1016/j.lfs.2007.08.019 |
| [52] | PATRUSHEV M, KASYMOV V, PATRUSHEVA V, et al. Mitochondrial permeability transition triggers the release of mtDNA fragments[J]. Cell Mol Life Sci, 2004, 61(24): 3100–3103. DOI: 10.1007/s00018-004-4424-1 |
| [53] | CARROLL E C, JIN L, MORI A, et al. The vaccine adjuvant chitosan promotes cellular immunity via DNA sensor cGAS-STING-dependent induction of type I interferons[J]. Immunity, 2016, 44(3): 597–608. DOI: 10.1016/j.immuni.2016.02.004 |
| [54] | HOWELL J, SAWHNEY R, TESTRO A, et al. Cyclosporine and tacrolimus have inhibitory effects on toll-like receptor signaling after liver transplantation[J]. Liver Transpl, 2013, 19(10): 1099–1107. DOI: 10.1002/lt.23712 |
| [55] | NISHIYAMA S, MANABE N, KUBOTA Y, et al. Cyclosporin A inhibits the early phase of NF-κB/RelA activation induced by CD28 costimulatory signaling to reduce the IL-2 expression in human peripheral T cells[J]. Int Immunopharmacol, 2005, 5(4): 699–710. DOI: 10.1016/j.intimp.2004.11.018 |
| [56] | KANNEGANTI T D, KUNDU M, GREEN D R. Innate immune recognition of mtDNA—an undercover signal?[J]. Cell Metab, 2015, 21(6): 793–794. DOI: 10.1016/j.cmet.2015.05.019 |
| [57] | ALAVIAN K N, BEUTNER G, LAZROVE E, et al. An uncoupling channel within the c-subunit ring of the F1FO ATP synthase is the mitochondrial permeability transition pore[J]. Proc Natl Acad Sci U S A, 2014, 111(29): 10580–10585. DOI: 10.1073/pnas.1401591111 |
| [58] | BERNARDI P, RASOLA A, FORTE M, et al. The mitochondrial permeability transition pore: channel formation by F-ATP synthase, integration in signal transduction, and role in pathophysiology[J]. Physiol Rev, 2015, 95(4): 1111–1155. DOI: 10.1152/physrev.00001.2015 |
| [59] | MATILLA I, ALFONSO C, RIVAS G, et al. The conjugative DNA translocase TrwB is a structure-specific DNA-binding protein[J]. J Biol Chem, 2010, 285(23): 17537–17544. DOI: 10.1074/jbc.M109.084137 |
| [60] | WEST A P, KHOURY-HANOLD W, STARON M, et al. Mitochondrial DNA stress primes the antiviral innate immune response[J]. Nature, 2015, 520(7548): 553–557. DOI: 10.1038/nature14156 |
| [61] | AARREBERG LD, ESSER-NOBIS K, DRISCOLL C, et al. Interleukin-1β induces mtDNA release to activate innate immune signaling via cGAS-STING[J]. Mol Cell, 2019, 74(4): 801–815. DOI: 10.1016/j.molcel.2019.02.038 |
| [62] | MAYER-BARBER K D, ANDRADE B B, OLAND S D, et al. Host-directed therapy of tuberculosis based on interleukin-1 and type I interferon crosstalk[J]. Nature, 2014, 511(7507): 99–103. DOI: 10.1038/nature13489 |
| [63] | LOOD C, BLANCO L P, PURMALEK M M, et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease[J]. Nat Med, 2016, 22(2): 146–153. DOI: 10.1038/nm.4027 |
| [64] | MCILROY D J, JARNICKI A G, AU G G, et al. Mitochondrial DNA neutrophil extracellular traps are formed after trauma and subsequent surgery[J]. J Crit Care, 2014, 29(6): 1133. e1–1133. e5. DOI: 10.1016/j.jcrc.2014.07.013 |
| [65] | WEST A P, BRODSKY I E, RAHNER C, et al. TLR signalling augments macrophage bactericidal activity through mitochondrial ROS[J]. Nature, 2011, 472(7344): 476–480. DOI: 10.1038/nature09973 |
| [66] | PAZMANDI K, AGOD Z, KUMAR B V, et al. Oxidative modification enhances the immunostimulatory effects of extracellular mitochondrial DNA on plasmacytoid dendritic cells[J]. Free Radic Biol Med, 2014, 77: 281–290. DOI: 10.1016/j.freeradbiomed.2014.09.028 |
| [67] | MATYSZEWSKI M, MORRONE S R, SOHN J. Digital signaling network drives the assembly of the AIM2-ASC inflammasome[J]. Proc Natl Acad Sci U S A, 2018, 115(9): E1963–E1972. DOI: 10.1073/pnas.1712860115 |
| [68] | MORRONE S R, MATYSZEWSKI M, YU X, et al. Assembly-driven activation of the AIM2 foreign-dsDNA sensor provides a polymerization template for downstream ASC[J]. Nat Commun, 2015, 6: 7827. DOI: 10.1038/ncomms8827 |
| [69] | JIN T C, PERRY A, JIANG J S, et al. Structures of the HIN domain: DNA complexes reveal ligand binding and activation mechanisms of the AIM2 inflammasome and IFI16 receptor[J]. Immunity, 2012, 36(4): 561–571. DOI: 10.1016/j.immuni.2012.02.014 |
| [70] | LUECKE S, HOLLEUFER A, CHRISTENSEN M H, et al. cGAS is activated by DNA in a length-dependent manner[J]. EMBO Rep, 2017, 18(10): 1707–1715. DOI: 10.15252/embr.201744017 |
| [71] | WANG L Y, LIEBMEN M N, WANG X D. Roles of mitochondrial DNA signaling in immune responses[M]//SUN H Z, WANG X D. Mitochondrial DNA and Diseases. Singapore: Springer, 2017, 1038: 39-53. |
| [72] | HU B, JIN C C, LI H B, et al. The DNA-sensing AIM2 inflammasome controls radiation-induced cell death and tissue injury[J]. Science, 2016, 354(6313): 765–768. DOI: 10.1126/science.aaf7532 |
| [73] | CHEN J Q, CHEN Z J J. PtdIns4P on dispersed trans-Golgi network mediates NLRP3 inflammasome activation[J]. Nature, 2018, 564(7734): 71–76. DOI: 10.1038/s41586-018-0761-3 |
| [74] | GENTILI M, LAHAYE X, NADALIN F, et al. The N-terminal domain of cGAS determines preferential association with centromeric DNA and innate immune activation in the nucleus[J]. Cell Rep, 2019, 26(9): 2377–2393. e13. DOI: 10.1016/j.celrep.2019.01.105 |
| [75] | BARNETT K C, CORONAS-SERNA J M, ZHOU W, et al. Phosphoinositide interactions position cGAS at the plasma membrane to ensure efficient distinction between self- and viral DNA[J]. Cell, 2019, 176(6): 1432–1446. e11. DOI: 10.1016/j.cell.2019.01.049 |
| [76] | WANG Y T, NING X H, GAO P F, et al. Inflammasome activation triggers caspase-1-mediated cleavage of cGAS to regulate responses to DNA virus infection[J]. Immunity, 2017, 46(3): 393–404. DOI: 10.1016/j.immuni.2017.02.011 |
| [77] | SWANSON K V, JUNKINS R D, KURKJIAN C J, et al. A noncanonical function of cGAMP in inflammasome priming and activation[J]. J Exp Med, 2017, 214(12): 3611–3626. DOI: 10.1084/jem.20171749 |
| [78] | CHIMIENTI G, PICCA A, SIRAGO G, et al. Increased TFAM binding to mtDNA damage hot spots is associated with mtDNA loss in aged rat heart[J]. Free Radic Biol Med, 2018, 124: 447–453. DOI: 10.1016/j.freeradbiomed.2018.06.041 |
| [79] | YOSHIDA Y, IZUMI H, ISE T, et al. Human mitochondrial transcription factor A binds preferentially to oxidatively damaged DNA[J]. Biochem Biophys Res Commun, 2002, 295(4): 945–951. DOI: 10.1016/S0006-291X(02)00757-X |
| [80] | MOK B Y, DE MORAES M H, ZENG J, et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing[J]. Nature, 2020, 583(7817): 631–637. DOI: 10.1038/s41586-020-2477-4 |
| [81] | YONEYAMA M, ONOMOTO K, JOGI M, et al. Viral RNA detection by RIG-I-like receptors[J]. Curr Opin Immunol, 2015, 32: 48–53. DOI: 10.1016/j.coi.2014.12.012 |
| [82] | SCHOGGINS J W, MACDUFF D A, IMANAKA N, et al. Pan-viral specificity of IFN-induced genes reveals new roles for cGAS in innate immunity[J]. Nature, 2014, 505(7485): 691–695. DOI: 10.1038/nature12862 |
| [83] | SUN B, SUNDSTRÖM K B, CHEW J J, et al. Dengue virus activates cGAS through the release of mitochondrial DNA[J]. Sci Rep, 2017, 7(1): 3594. DOI: 10.1038/s41598-017-03932-1 |
| [84] | LAI J H, WANG M Y, HUANG C Y, et al. Infection with the dengue RNA virus activates TLR9 signaling in human dendritic cells[J]. EMBO Rep, 2018, 19(8): e46182. |
| [85] | AGUIRRE S, MAESTRE A M, PAGNI S, et al. DENV inhibits type I IFN production in infected cells by cleaving human STING[J]. PLoS Pathog, 2012, 8(10): e1002934. DOI: 10.1371/journal.ppat.1002934 |
| [86] | MORIYAMA M, KOSHIBA T, ICHINOHE T. Influenza A virus M2 protein triggers mitochondrial DNA-mediated antiviral immune responses[J]. Nat Commun, 2019, 10(1): 4624. DOI: 10.1038/s41467-019-12632-5 |
| [87] | VON KÖCKRITZ-BLICKWEDE M, NIZET V. Innate immunity turned inside-out: antimicrobial defense by phagocyte extracellular traps[J]. J Mol Med (Berlin, Germany), 2009, 87(8): 775–783. DOI: 10.1007/s00109-009-0481-0 |
| [88] | YOUSEFI S, GOLD J A, ANDINA N, et al. Catapult-like release of mitochondrial DNA by eosinophils contributes to antibacterial defense[J]. Nat Med, 2008, 14(9): 949–953. DOI: 10.1038/nm.1855 |
| [89] | YOUSEFI S, MORSHED M, AMINI P, et al. Basophils exhibit antibacterial activity through extracellular trap formation[J]. Allergy, 2015, 70(9): 1184–1188. DOI: 10.1111/all.12662 |
| [90] | SAFFRAN H A, PARE J M, CORCORAN J A, et al. Herpes simplex virus eliminates host mitochondrial DNA[J]. EMBO Rep, 2007, 8(2): 188–193. DOI: 10.1038/sj.embor.7400878 |
| [91] | WATSON R O, BELL S L, MACDUFF D A, et al. The cytosolic sensor cGAS detects Mycobacterium tuberculosis DNA to induce type I interferons and activate autophagy[J]. Cell Host Microbe, 2015, 17(6): 811–819. DOI: 10.1016/j.chom.2015.05.004 |
| [92] | WIENS K E, ERNST J D. The mechanism for type I interferon induction by Mycobacterium tuberculosis is bacterial strain-dependent[J]. PLoS Pathog, 2016, 12(8): e1005809. DOI: 10.1371/journal.ppat.1005809 |
| [93] | ZHANG Z Y, MENG P, HAN Y J, et al. Mitochondrial DNA-LL-37 complex promotes atherosclerosis by escaping from autophagic recognition[J]. Immunity, 2015, 43(6): 1137–1147. DOI: 10.1016/j.immuni.2015.10.018 |
| [94] | GARCIA-MARTINEZ I, SANTORO N, CHEN Y L, et al. Hepatocyte mitochondrial DNA drives nonalcoholic steatohepatitis by activation of TLR9[J]. J Clin Invest, 2016, 126(3): 859–864. DOI: 10.1172/JCI83885 |
| [95] | YOUNOSSI Z M, KOENIG A B, ABDELATIF D, et al. Global epidemiology of nonalcoholic fatty liver disease—Meta-analytic assessment of prevalence, incidence, and outcomes[J]. Hepatology, 2016, 64(1): 73–84. DOI: 10.1002/hep.28431 |
| [96] | YU Y S, LIU Y, AN W S, et al. STING-mediated inflammation in Kupffer cells contributes to progression of nonalcoholic steatohepatitis[J]. J Clin Invest, 2019, 129(2): 546–555. |
| [97] | MRIDHA A R, WREE A, ROBERTSON A A B, et al. NLRP3 inflammasome blockade reduces liver inflammation and fibrosis in experimental NASH in mice[J]. J Hepatol, 2017, 66(5): 1037–1046. DOI: 10.1016/j.jhep.2017.01.022 |



