2. 内蒙古阿拉善盟畜牧研究所, 巴彦浩特 750306
2. Inner Mongolia Alxa League Livestock Research Institute, Bayanhot 750306, China
骆驼科动物包含3个属6种动物,分别为单峰驼、双峰驼、美洲驼、羊驼、原驼和骆马,其中,单峰驼和双峰驼属于骆驼属[1]。双峰驼是我国固有的畜种,主要分布在内蒙古、新疆、青海、甘肃、宁夏等地区,研究双峰驼抗逆性的分子机制对探究人类及其他动物的的抗逆性具有重要作用。恶劣的生存环境使得骆驼在长期的进化过程中形成了耐高温、耐干旱、耐盐、耐高血糖的独特的适应荒漠草原的生物学特征[2-3]。沙漠地区的水源和植被含盐量较高,双峰驼可以利用这些水源和植物,维持正常的生理活动[4]。骆驼为适应沙漠环境而进化出特殊的生理结构,包括进化出驼峰,储存大量脂肪,可以调节体温,并在缺少食物时提供能量;形成只有瘤胃、网胃和皱胃而没有瓣胃的独特的消化系统,以适应沙漠环境。研究者基于全基因组、转录组和宏基因组对双峰驼沙漠环境适应性开展了相关研究。基于全基因组数据的种间分析发现,双峰驼在长期进化的过程中形成了独特的免疫系统,并且具有胰岛素抵抗的作用[5]。对驼峰脂肪与皮下脂肪和内脏脂肪进行转录组分析,结果显示,驼峰除了有渗透调节的作用外,还具有内分泌以及免疫的功能[6]。消化系统对动物能量代谢和水分的吸收以及新陈代谢具有至关重要的作用,前人对双峰驼结肠组织的转录组研究结果显示,双峰驼通过减少RNA合成,降低新陈代谢和增强免疫能力以适应缺水和高盐的环境[7-8]。对双峰驼瘤胃微生物的宏基因组学研究显示,双峰驼瘤胃微生物种类丰富,且受季节变化的影响[9]。对羊瘤胃组织的研究表明,反刍动物体内与脂质代谢相关基因表达模式的改变可能与瘤胃合成挥发性脂肪酸有关[10]。目前,对骆驼适应性研究多针对成年期开展,较少开展胚胎期组织发育和分化研究,基于瘤胃的转录组研究尚未见相关报道。
本试验选取成年期和胚胎期各3峰阿拉善双峰驼,对其瘤胃进行组织学以及转录组学分析,筛选双峰驼瘤胃发育相关的候选基因,从消化的角度探究双峰驼沙漠适应性的分子机制。
1 材料与方法 1.1 试验动物与样品采集屠宰3峰10~12岁的健康成年期阿拉善双峰驼(M1、M2、M3)以及3峰9~10月龄的健康胚胎期阿拉善双峰驼(T1、T2、T3),采集此6峰双峰驼的瘤胃组织样品各两份,分别存于RNA-latter和4%多聚甲醛中,用于提取RNA和固定组织以制备石蜡组织切片。
1.2 双峰驼瘤胃组织石蜡组织切片的制备快速取瘤胃组织约1 cm3,生理盐水清洗后放到4%多聚甲醛溶液中固定24 h,然后进行脱水、透明、浸蜡、包埋,修整蜡块后对蜡块进行片厚为4 μm的切片后,进行烤片、脱蜡、复水和苏木素伊红(hematoxylin eosin,H.E)染色4 min,中性树脂胶封固,切片镜检拍照观察[11]。
1.3 双峰驼瘤胃组织RNA-Seq检测1.3.1 双峰驼瘤胃组织总RNA的提取和质量检测 使用Qiagen公司的组织总RNA提取试剂盒,采集6峰阿拉善双峰的瘤胃组织于液氮中充分研磨,按照试剂盒说明书提取组织总RNA。使用Agilent 2100 bioanalyzer检测RNA总量、浓度、完整度(RIN值)及OD值,并控制RNA总量>2 μg,RNA浓度>100 ng·μL-1,RIN值≥7,且28S/18S≥ 0.7,以用于后续的测序分析。
1.3.2 双峰驼瘤胃组织cDNA文库构建与RNA测序 取1 μg RNA样品,严格按照Illumina®Tru SeqTM RNA样品制备操作试剂盒说明书进行cDNA文库的构建。主要过程包括:用oligo dT将mRNA从总RNA中分离富集,使用带有二价阳离子的片段缓冲液在高温下使mRNA片段化,cDNA的合成,衔接测序接头,片段的纯化分离,PCR扩增等[12]。使用Agilent 2100 bioanalyzer检测cDNA文库大小和浓度及质量,最终得到cDNA文库。
严格按照TruSeq PE Cluster Kit v3-cBot-HS试剂盒说明,使用cBot Cluster Generation系统对含有索引接头(index-coded)的样品进行聚类。形成聚类后,在Illumina Hiseq 2000测序平台上分别对3峰成年期双峰驼试验样品和3峰胚胎期双峰驼试验样品进行读长为125 bp的双末端测序,获得原始测序数据raw reads。
1.3.3 双峰驼瘤胃组织转录组测序原始数据的质控 用NGSQC Toolkit v2.3.3软件[13]对raw reads进行过滤, 去除接头序列,切割3′末端低质量以及N碱基的序列,以及去除过滤后长度小于25 bp的reads,质控后最终得到clean reads。
1.3.4 测序数据的比对和转录本的组装 从NCBI网站下载双峰驼的基因组,版本号为Ca_bactrianus_MBC_1.0,以及基因组注释文件。使用TopHat v2.0.11软件[14]将质控后的clean data比对到参考基因组上。用Cufflink软件[15]进行转录本的组装。使用cuffmerge将组装后的转录本以及参考基因组组成新的注释文件,使用FPKM(fragments per kilobase of exon model per million mapped reads)值评估转录本的表达量。
1.3.5 差异基因的鉴定 用Cufflink软件的Cuffdiff包对成年期和胚胎期双峰驼瘤胃组织的差异基因进行鉴定和分析。以胚胎期为对照组,成年期为试验组,用FDR法进行多重校正,选取同时满足FPKM值>1,FDR<0.05以及Fold change>2的基因作为显著差异表达基因。
1.3.6 差异表达基因层次聚类分析 用R软件中的聚类分析包对上述筛选的成年期和胚胎期双峰驼瘤胃组织的显著差异表达基因进行层次聚类,并使用R软件中的pheatmap包进行热图的绘制。
1.3.7 差异表达基因的GO和KEGG富集分析 基因本体论(gene ontology,GO)[16]是基因功能国际分类标准,在生物信息领域广泛使用,包含细胞成分(cellular component)、分子功能(molecular function)和生物过程(biological process)3个方面。京都基因和基因组百科全书(kyoto encyclopedia of genes and genomes)数据库[17]主要用于富集基因的生物通路分析。使用g:profiler[18]在线网站对差异表达基因进行GO和KEGG富集分析。使用超几何检验方法计算差异表达基因显著富集的GO条目和KEGG信号通路。校正P-value<0.05时,认为该GO条目或KEGG信号通路为显著富集。
1.4 差异表达基因实时荧光定量PCR验证随机选取6个差异表达基因,采用实时荧光定量PCR(RT-qPCR)验证基因的表达水平。
1.4.1 瘤胃组织RNA的提取与反转录 RNA提取方法步骤同“1.3.1”。每个RNA样品取1 μL使用RT-PCR Kit试剂盒(TaKaRa,大连,中国)反转录后得到cDNA。
1.4.2 引物的设计与合成 下载NCBI网站上的基因mRNA序列,选用β-actin作为内参基因,利用NCBI网站中的primer-BLAST在线工具设计引物(表 1)。引物由北京天一辉远生物科技有限公司合成。
|
|
表 1 本试验采用的qRT-PCR鉴定引物 Table 1 qRT-PCR primers used in this study |
1.4.3 RT-qPCR检测 使用ABI Q7实时荧光定量仪(applied Biosystems,Forest City, CA, SA)进行荧光定量试验,每个样品设置3个技术性重复。使用2-△△Ct方法计算基因的相对表达量,使用t检验对相对表达量进行分析,若P-value<0.05,则为差异显著。
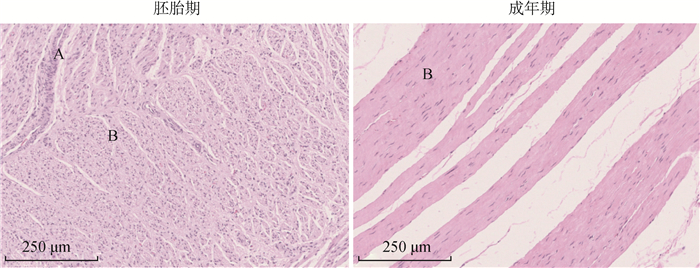
2 结果 2.1 双峰驼瘤胃组织切片分析瘤胃组织切片H.E染色结果(图 1)显示,胚胎期的瘤胃中上皮细胞和肌纤维清晰可见,分布密集,在成年期的瘤胃组织中,可见明显的肌纤维,肌纤维直径较宽,肌纤维间的空隙较大。说明与胚胎期相比,瘤胃在成年期有了极大程度的发育和分化,为适应沙漠的极端环境提供条件。
|
A.上皮细胞; B.肌纤维 A. Epithelial cells; B. Muscle fiber 图 1 瘤胃组织切片(H.E染色10×) Fig. 1 Rumen tissue section(H.E staining 10×) |
通过Illumina Hiseq 2000测序平台得到各样品的测序原始数据,如表 2所示,每个样品产生不低于10G的数据量,各样本的质控率都在90%以上,Q30数据都在88%以上,测得数据的总量和质量都满足后续分析的条件。
|
|
表 2 转录组测序数据质量检测分析 Table 2 Transcriptome sequencing data quality inspection and analysis |
使用TopHat将质控后的clean data比对到参考基因组,结果如表 3所示,在6个双峰驼瘤胃组织样品中,有78.90%~86.60%的Reads可以比对到双峰驼的参考基因组上,满足后续分析的质量要求。
|
|
表 3 转录组测序结果比对参考基因组 Table 3 Transcriptome sequencing results alignment to reference genome |
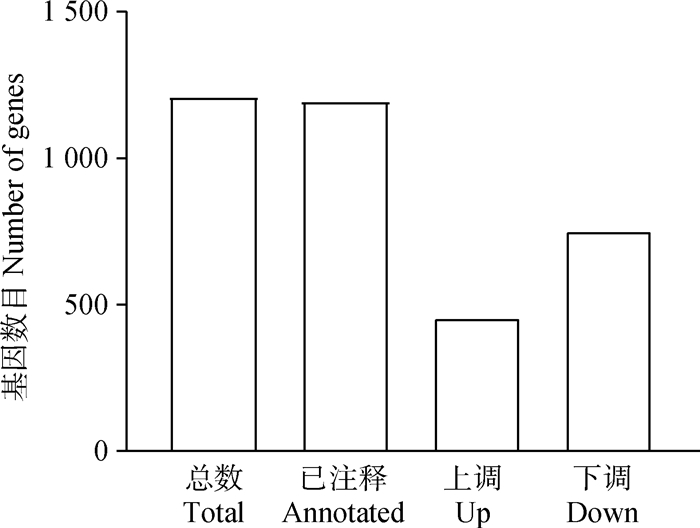
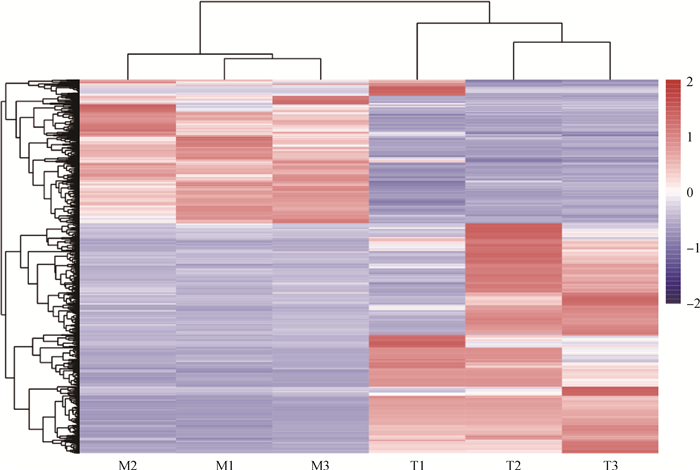
研究共鉴定到24 198个基因,以胚胎期为对照组,以FDR<0.05,Fold Change>2为条件筛选差异表达基因,共筛选到1 207个差异表达基因,其中,注释的基因1 195个,未知的基因12个,上调基因456个,下调基因751个(图 2)。对筛选出来的成年期双峰驼瘤胃组织与胚胎期双峰驼瘤胃组织差异表达基因进行聚类分析,结果表明,M1和M3首先聚到一起,再和M2聚为一类;T2和T3首先聚到一起,再和T1聚为一类(图 3)。说明成年期3个个体(M1、M2、M3)的瘤胃组织差异基因表达模式更接近,胚胎期3个个体(T1、T1、T3)的瘤胃组织差异基因表达模式更接近,进一步证明了转录组测序数据的可靠性。
|
图 2 差异表达基因统计 Fig. 2 Statistics of differentially expressed genes |
|
图 3 1 207个差异表达基因的聚类结果 Fig. 3 The clustering results of 1 207 differentially expressed genes |
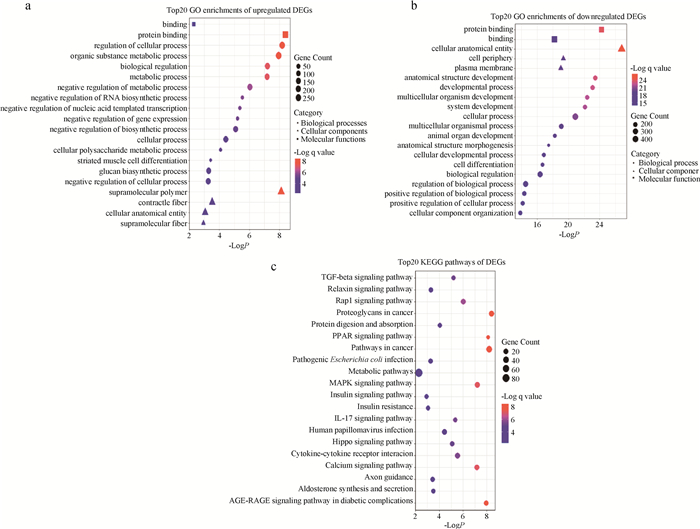
使用g:profiler在线软件对双峰驼成年期瘤胃组织和胚胎期瘤胃组织上调差异表达基因和下调差异表达基因分别进行GO富集分析以注释基因功能,以P-value<0.05为筛选显著富集的条件,上调差异表达基因显著富集到了62个GO条目,其中包括3个分子功能条目,17个细胞成分条目和42个生物过程条目;下调差异表达基因显著富集于366个GO条目中,其中包括29个分子功能条目,52个细胞成分条目和285个生物过程条目, 上调差异表达基因主要富集在蛋白质结合、生物代谢过程的负调控、细胞多糖代谢过程、葡聚糖生物合成过程等GO条目中(图 4a),下调差异表达基因主要富集在代谢进程、生物过程的正向调控、生物条件多细胞生物发展等GO条目中(图 4b)。通过筛选到的这些显著富集的GO条目可以发现,双峰驼成年期和胚胎期瘤胃组织差异表达基因在生物代谢、蛋白质合成以及糖代谢方面发挥了重要的作用。
|
a.上调表达基因显著富集的前20个GO条目;b.下调表达基因显著富集的前20个GO条目;c.差异基因显著富集的前20个KEGG信号通路 a.The top 20 GO terms enriched by upregulated differentially expressed genes; b. The top 20 GO terms enriched by downregulated differentially expressed genes; c. The top 20 KEGG pathways enriched by differentially expressed genes 图 4 差异表达基因GO与KEGG富集分析结果 Fig. 4 Results of GO and KEGG enrichment analysis of differentially expressed genes |
使用g:profiler在线软件对双峰驼成年期和胚胎期瘤胃组织差异表达基因进行KEGG富集分析,以筛选基因所在的信号通路,以P-value<0.05为筛选显著富集的条件,共筛选到73条显著的KEGG信号通路,主要富集在MAPK信号通路、癌症中的蛋白聚糖信号通路、糖尿病并发症中的AGE-RAGE信号通路、胰岛素抵抗等通路中(图 4c),其中,被富集到的主要基因有丝裂原活化蛋白激酶12(mitogen-activated protein kinase 12,MAPK12)、丝裂原活化蛋白激酶12(mitogen-activated protein kinase 13,MAPK13)、转化生长因子-β3(transforming growth factor beta3,TGF-β3)、骨形态发生蛋白2(bone morphogenetic protein 2,BMP2)、脂肪酸结合蛋白5(fatty acid binding protein 5,FABP5)、过氧化物酶体增殖剂激活受体γ(peroxisome proliferator activated receptor gamma,PPARγ)、钙/钙调蛋白依赖性蛋白激酶1(calcium/calmodulin dependent protein kinase I,CaMK1)等。
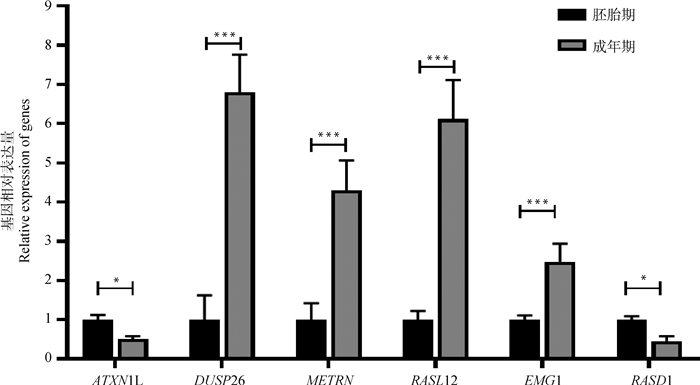
2.6 RT-qPCR验证随机选取6个差异表达基因进行RT-qPCR验证,结果如图 5所示,6个差异表达基因的RT-qPCR表达模式与RNA测序结果一致,进一步证明了转录组测序数据的可靠性。
|
*.P<0.05;***. P<0.001 图 5 6个差异表达基因的RT-qPCR验证 Fig. 5 RT-qPCR verification of 6 differentially expressed genes |
从组织切片的结果来看,成年期双峰驼瘤胃组织与胚胎期瘤胃组织差异明显,与胚胎期相比,瘤胃在成年期有了极大程度的发育和分化,以往的研究中还尚未见对双峰驼瘤胃组织的研究,基于此,本研究分别对成年期和胚胎期的瘤胃组织进行了进一步的转录组学研究。
本研究以胚胎期为对照组,分别对双峰驼成年期和胚胎期瘤胃组织上调和下调差异表达基因进行GO富集分析,以识别差异表达基因所在细胞成分、分子功能、生物过程分类。结果显示,上调差异表达基因显著富集于代谢过程的负调控、RNA生物合成过程的负调控、核酸模板转录的负调控、基因表达的负调控、细胞多糖代谢、葡聚糖生物合成等生物过程中。说明为了适应沙漠极端环境,双峰驼瘤胃组织的基因表达、RNA合成等活动减少。之前也有研究表明[6],在禁水条件下,双峰驼会通过降低结肠组织的RNA合成来降低新陈代谢,以适应缺水环境,与本研究结果相似。双峰驼具有异于其他哺乳动物的血糖耐受能力[19-20],瘤胃作为消化系统的重要组成部分,对糖的吸收和代谢起到了不可忽视的作用。本研究中,上调差异表达基因显著富集到细胞多糖代谢过程和葡聚糖生物合成过程,说明成年期的双峰驼瘤胃组织中糖代谢活动更加丰富,有利于双峰驼高血糖的耐受。下调差异表达基因显著富集到多细胞生物发育、细胞分化、生物过程的积极调控等生物过程,说明在沙漠环境中,双峰驼瘤胃组织的生物发育和细胞分化等活动处于下调趋势,有助于降低组织的代谢,以适应干燥、缺水的恶劣环境。
KEGG富集主要是超几何检验方法,计算差异表达基因所富集的信号通路和代谢通路。本研究中,差异表达基因主要显著富集于糖尿病并发症中的MAPK信号通路、AGE-RAGE信号通路、胰岛素抵抗、胰岛素信号通路、PI3K-Akt信号通路、钙信号通路、蛋白质消化吸收、醛固酮的合成与分泌通路等。同时,在这些通路中筛选到了MAPK12、MAPK13、FABP5、PPARγ、CaMK1等可能与双峰驼沙漠适应性相关的基因。
FABP5脂肪酸结合蛋白是一类小的、高度保守的细胞质蛋白,能结合长链脂肪酸和其他疏水配体。FABPs可能在脂肪酸摄取、运输和代谢中发挥作用。该基因的多态性与2型糖尿病有关。过氧化物酶体增殖物激活受体(PPAR)是被脂肪酸及其衍生物激活的核激素受体[21]。这些相关基因在本研究中显著上调并被显著富集到PPAR信号通路(PPAR signaling pathway)中,PPARγ可促进脂肪细胞分化[22]。也有研究表明,MAPKs的下调表达对于脂肪细胞分化起到了很重要的作用[23], 本研究中,MAPK13显著下调,可能也对促进脂肪细胞分化有重要作用,与2014年Jiang等[10]对绵羊瘤胃的研究结果相符。说明双峰驼的瘤胃对脂肪分化可能也起到了重要作用。
胰岛素抵抗是指胰岛素靶组织不能对胰岛素产生适当反应的情况[24]。胰岛素与其受体结合会引起受体自身磷酸化并激活受体酪氨酸激酶,导致胰岛素受体底物(insulin receptor substrates,IRSs)的酪氨酸磷酸化, IRS的磷酸化会导致磷脂酰肌醇3-激酶(PI3K)活化,进而导致丝氨酸/苏氨酸激酶(serine/threonine kinase,Akt)及其下游介体AS160活化,这些是胰岛素诱导的葡萄糖转运的重要步骤。如果依赖胰岛素的PI3K激活受损,下游的信号转导也无法进行,进而影响葡萄糖的转运[25]。现有的研究也表明,双峰驼能够维持高于其他哺乳动物的血糖水平且能正常生活,与胰岛素抵抗作用有很大关系[26]。同时,脂肪细分化也可以提高血糖摄取,从而达到对高血糖的耐受[10]。双峰驼可能是以这种方式调节血糖含量,以达到对血糖的高耐受性。
晚期糖基化终产物(advanced glycation end products, AGEs)是由美拉德反应产生的,是蛋白质的氨基和糖的醛基之间非酶性糖基化反应最终的产物[27-28]。AGEs的主要受体为高级糖基化终产物受体(RAGE或AGER),已被定义为模式识别受体[29]。AGE/RAGE信号传导引起涉及NADPH氧化酶、蛋白激酶C和MAPKs的多个细胞内信号途径的激活,然后导致NF-κB具有活性。NF-κB促进促炎性细胞因子(如IL-1、IL-6和TNF-α)以及多种与动脉粥样硬化相关基因(包括VCAM-1、组织因子VEGF和RAGE)的表达[30]。另外,其经由RAGE诱导的JAK-STAT介导通路和PI3K-Akt依赖性途径,参与细胞增殖和凋亡[31]。缺氧介导的Egr-1的诱导激活也需要AGE-RAGE相互作用,与糖代谢具有十分紧密的关系[32]。
研究表明,MAPK信号通路、PPAR信号通路、AGE-RAGE信号通路、PI3K-Akt信号通路均与糖尿病机制有关[31]。本研究中,MAPK信号通路、PPAR信号通路、糖尿病并发症中的AGE-RAGE信号通路、PI3K-Akt信号通路都被显著富集,说明它们可能共同调控了双峰驼血糖的代谢,使得双峰驼对血糖的耐受性提高,不易受到高血糖的危害,以适应沙漠恶劣的环境。
盐是调控生物体内水分均衡的主要因素。沙漠地区多为高含盐量的水源和植被,有研究表明,高盐饮食会导致钠钙交换增加,从而引起血管平滑肌收缩,导致血压升高[33-34]。双峰驼由于生存于恶劣的环境,采食条件受到很大的限制,被迫食用大量含盐分很高的植物和水源[35],但是依然能够维持正常的生理活动,这是区别于其他哺乳动物非常重要的特点,说明双峰驼可能具有其独特的血压调节机制。醛固酮吸收钠和水在调节全身血压中起重要作用。血管紧张素(Ang)、钾(K+)和促肾上腺皮质激素(ACTH)是促进醛固酮分泌的主要生因子[36]。本研究中,CaMK1显著上调,CaMK1表达量的升高会诱导固醇激素合成急性调节蛋白(steroidogenic acute regulatory protein,STAR)和细胞色素P450家族11亚家族B成员2(cytochrome P450 family 11 subfamily B member 2,CYP11B2)基因的表达,以解除对醛固酮生物合成的限制。双峰驼可能是以这种方式促进醛固酮的合成和分泌,以达到调节血压的作用[37]。
蛋白质是生物体营养动态平衡必不可少的饮食成分。通常,摄入的蛋白质在胃、胰腺和小肠酶的作用下经历一系列复杂的降解过程,得到氨基酸和小肽的混合物,再由特异的氨基酸转运蛋白将对应的酸性氨基酸、中性氨基酸和碱性氨基酸转运到小肠上皮细胞[38]。小肽被PEPT1转运蛋白吸收进入小肠上皮细胞。小肠细胞内的肽段被水解,得到的氨基酸与氨基酸转运蛋白一起通过多个基底外侧氨基酸转运蛋白释放到血液中[39],完成蛋白质的消化与吸收。瘤胃作为重要的消化器官,在蛋白质的吸收与消化上起到了非常重要的作用。有研究表明[40],骆驼会利用激素调节不同饮水量时体内和驼乳中的水分含量,以确保在饮水量受到限制的情况下保证胎儿和幼驼的水分供应,使胚胎时期的双峰驼胎儿在母体环境中水分和营养充足,受外界环境的影响较小。本研究中,成年期低表达基因显著富集于蛋白质消化与吸收途径,说明为了适应成年后食物匮乏、水分稀少的恶劣沙漠环境,双峰驼瘤胃细胞内蛋白质消化吸收活动降低,意味着与胚胎期相比,成年期的双峰驼瘤胃减少了蛋白质的消化吸收,这可能与RNA的合成减少有关。
4 结论从组织学水平看,在成年期的瘤胃组织中,可见明显的肌纤维,肌纤维直径较宽,肌纤维间的空隙较大,与胚胎期相比,瘤胃在成年期有了极大程度的发育和分化。RNA-seq结果显示,成年期双峰驼瘤胃组织与胚胎期双峰驼瘤胃组织共筛选到1 207个差异表达基因,其中上调差异表达基因456个,下调差异表达基因751个。差异表达基因的GO和KEGG富集分析结果表明,差异表达基因多参与MAPK信号通路、胰岛素抵抗、胰岛素信号通路、AGE-RAGE信号通路、PI3K-Akt信号通路、蛋白质消化吸收、醛固酮的合成与分泌等过程。这些功能有利于双峰驼在沙漠环境中促进脂肪细胞的分化,降低代谢效率、提高血糖耐受性以及调节血压平衡,以更好的在沙漠干燥缺水的环境中生存。
| [1] |
哈斯高娃, 乌日罕, 伊茹汗, 等. 雌性骆驼繁殖研究进展[J]. 草食家畜, 2019(3): 6–12.
HASI G W, WU R H, YI R H, et al. Research on female camel breeding[J]. Grass-Feeding Livestock, 2019(3): 6–12. (in Chinese) |
| [2] | TIBARY A, EL ALLALI K. Dromedary camel:a model of heat resistant livestock animal[J]. Theriogenology, 2020, 154: 203–211. DOI: 10.1016/j.theriogenology.2020.05.046 |
| [3] | CAO Y, ZHANG D, ZHOU H M. Key genes differential expressions and pathway involved in salt and water-deprivation stresses for renal cortex in camel[J]. BMC Mol Biol, 2019, 20(1): 11. DOI: 10.1186/s12867-019-0129-8 |
| [4] |
曾献春, 郑李娟, 庄伟伟. 骆驼消化道内喹啉降解菌的分离、筛选及降解能力[J]. 云南大学学报:自然科学版, 2017, 39(6): 1073–1081.
ZENG X C, ZHENG L J, ZHUANG W W. Isolation and screening of quinoline-degrading bacteria and their biodegradation capability in the digestive tract of camels[J]. Journal of Yunnan University:Natural Sciences Edition, 2017, 39(6): 1073–1081. (in Chinese) |
| [5] | The Bactrian Camels Genome Sequencing and Analysis Consortium. Genome sequences of wild and domestic Bactrian camels[J]. Nat Commun, 2012, 3: 1202. DOI: 10.1038/ncomms2192 |
| [6] | GUO F C, SI R D L, HE J, et al. Comprehensive transcriptome analysis of adipose tissue in the Bactrian camel reveals fore hump has more specific physiological functions in immune and endocrine systems[J]. Livest Sci, 2019, 228: 195–200. DOI: 10.1016/j.livsci.2019.09.003 |
| [7] |
凌宇, 齐昱, 曹俊伟, 等. 转录组揭示禁水环境下双峰驼结肠组织适应机制[J]. 畜牧兽医学报, 2018, 49(11): 2359–2370.
LING Y, QI Y, CAO J W, et al. Transcriptome reveals the adapting mechanism of colon tissues of Bactrian camel in water-deficient environment[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(11): 2359–2370. DOI: 10.11843/j.issn.0366-6964.2018.11.007 (in Chinese) |
| [8] |
凌宇, 齐昱, 孟凡华, 等. 盐胁迫下双峰驼结肠组织环境适应能力研究[J]. 农业生物技术学报, 2018, 26(10): 1723–1736.
LING Y, QI Y, MENG F H, et al. Study on environmental adaptability of Bactrian camel (Camelidae bactrianus) colon tissue under salt stress[J]. Journal of Agricultural Biotechnology, 2018, 26(10): 1723–1736. (in Chinese) |
| [9] | HE J, LI G W, HAI L, et al. An analysis of the forestomach bacterial microbiota in the Bactrian camel[J]. J Camel Pract Res, 2019, 26(1): 71–79. DOI: 10.5958/2277-8934.2019.00010.9 |
| [10] | JIANG Y, XIE M, CHEN W B, et al. The sheep genome illuminates biology of the rumen and lipid metabolism[J]. Science, 2014, 344(6188): 1168–1173. DOI: 10.1126/science.1252806 |
| [11] |
邱名浩, 厉洁, 刘娟. 家鸽石蜡组织切片质量的优化研究[J]. 德州学院学报, 2019, 35(4): 37–39, 43.
QIU M H, LI J, LIU J. The study on the quality optimization of paraffin tissue section of pigeon[J]. Journal of Dezhou University, 2019, 35(4): 37–39, 43. (in Chinese) |
| [12] |
董坤哲.藏猪高海拔环境适应性分子机制探讨[D].北京: 中国农业科学院, 2015.
DONG K Z.Analysis of molecular mechanisms of high-altitude adaptation in Tibetan pig[D]. Beijing: Chinese Academy of Agricultural Sciences, 2015.(in Chinese) |
| [13] | PATEL R K, JAIN M. NGS QC toolkit:a toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7(2): e30619. DOI: 10.1371/journal.pone.0030619 |
| [14] | TRAPNELL C, PACHTER L, SALZBERG S L. TopHat:discovering splice junctions with RNA-Seq[J]. Bioinformatics, 2009, 25(9): 1105–1111. DOI: 10.1093/bioinformatics/btp120 |
| [15] | TRAPNELL C, WILLIAMS B A, PERTEA G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nat Biotechnol, 2010, 28(5): 511–515. DOI: 10.1038/nbt.1621 |
| [16] | CAMON E, MAGRANE M, BARRELL D, et al. The gene ontology annotation (GOA) database:sharing knowledge in uniprot with gene ontology[J]. Nucleic Acids Res, 2004, 32(S1): D262–D266. |
| [17] | KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Res, 2008, 36(S1): D480–D484. |
| [18] | REIMAND J, ARAK T, ADLER P, et al. g:profiler-a web server for functional interpretation of gene lists (2016 update)[J]. Nucleic Acids Res, 2016, 44(W1): W83–W89. DOI: 10.1093/nar/gkw199 |
| [19] | ELMAHDI B, SALLMANN H P, FUHRMANN H, et al. Comparative aspects of glucose tolerance in camels, sheep, and ponies[J]. Comp Biochem Physiol A Physiol, 1997, 118(1): 147–151. |
| [20] | KASKE M, ELMAHDI B, ENGELHARDT W, et al. Insulin responsiveness of sheep, ponies, miniature pigs and camels:results of hyperinsulinemic clamps using porcine insulin[J]. J Comp Physiol B, 2001, 171(7): 549–556. DOI: 10.1007/s003600100205 |
| [21] | FUJⅡ H. PPARs-mediated intracellular signal trans-duction[J]. Nihon Rinsho, 2005, 63(4): 565–571. |
| [22] | SAVAGE D B. PPARγ as a metabolic regulator:insights from genomics and pharmacology[J]. Expert Rev Mol Med, 2005, 7(1): 1–16. |
| [23] | SAKAUE H, OGAWA W, NAKAMURA T, et al. Role of MAPK phosphatase-1 (MKP-1) in adipocyte differentiation[J]. J Biol Chem, 2004, 279(38): 39951–39957. DOI: 10.1074/jbc.M407353200 |
| [24] | CHOI K, KIM Y B. Molecular mechanism of insulin resistance in obesity and type 2 diabetes[J]. Korean J Intern Med, 2010, 25(2): 119–129. DOI: 10.3904/kjim.2010.25.2.119 |
| [25] | LAN D C, XU N G, SUN J, et al. Electroacupuncture mitigates endothelial dysfunction via effects on the Pi3K/Akt signalling pathway in high fat diet-induced insulin-resistant rats[J]. Acupunct Med, 2018, 36(3): 162–169. DOI: 10.1136/acupmed-2016-011253 |
| [26] | WU H G, GUANG X M, AL-FAGEEH M B, et al. Camelid genomes reveal evolution and adaptation to desert environments[J]. Nat Commun, 2014, 5: 5188. DOI: 10.1038/ncomms6188 |
| [27] | QUAN W, LI Y, JIAO Y, et al. Simultaneous generation of acrylamide, β-carboline heterocyclic amines and advanced glycation ends products in an aqueous Maillard reaction model system[J]. Food Chem, 2020, 332: 127387. DOI: 10.1016/j.foodchem.2020.127387 |
| [28] | BEDOUI S A, BARBIROU M, STAYOUSSEF M, et al. Identification of novel advanced glycation end products receptor gene variants associated with colorectal cancer in Tunisians:a case-control study[J]. Gene, 2020, 754: 144893. DOI: 10.1016/j.gene.2020.144893 |
| [29] | NEEPER M, SCHMIDT A M, BRETT J, et al. Cloning and expression of a cell surface receptor for advanced glycosylation end products of proteins[J]. J Biol Chem, 1992, 267(21): 14998–15004. DOI: 10.1016/S0021-9258(18)42138-2 |
| [30] | YAMAGISHI S I. Role of advanced glycation end products (AGEs) and receptor for AGEs (RAGE) in vascular damage in diabetes[J]. Exp Gerontol, 2011, 46(4): 217–224. DOI: 10.1016/j.exger.2010.11.007 |
| [31] | KANWAR Y S, SUN L, XIE P, et al. A glimpse of various pathogenetic mechanisms of diabetic nephro-pathy[J]. Annu Rev Pathol Mech Dis, 2011, 6: 395–423. DOI: 10.1146/annurev.pathol.4.110807.092150 |
| [32] | CALCUTT N A, COOPER M E, KERN T S, et al. Therapies for hyperglycaemia-induced diabetic complications:from animal models to clinical trials[J]. Nat Rev Drug Discov, 2009, 8(5): 417–430. |
| [33] | BKAILY G, SIMON Y, MENKOVIC I, et al. High salt-induced hypertrophy of human vascular smooth muscle cells associated with a decrease in glycocalyx[J]. J Mol Cell Cardiol, 2018, 125: 1–5. DOI: 10.1016/j.yjmcc.2018.10.006 |
| [34] | BEAINI S, SALIBA Y, HAJAL J, et al. VEGF-C attenuates renal damage in salt-sensitive hypertension[J]. J Cell Physiol, 2019, 234(6): 9616–9630. DOI: 10.1002/jcp.27648 |
| [35] | KACZENSKY P, ADIYA Y, VON WEHRDEN H, et al. Space and habitat use by wild Bactrian camels in the Transaltai Gobi of southern Mongolia[J]. Biol Conserv, 2014, 169: 311–318. |
| [36] | HATTANGADY N G, OLALA L O, BOLLAG W B, et al. Acute and chronic regulation of aldosterone production[J]. Mol Cell Endocrinol, 2012, 350(2): 151–162. |
| [37] | BOLLAG W B. Regulation of aldosterone synthesis and secretion[J]. Compr Physiol, 2011, 4(3): 1017–1055. |
| [38] | BRÖER S. Amino acid transport across mammalian intestinal and renal epithelia[J]. Physiol Rev, 2008, 88(1): 249–286. |
| [39] | WU G. Intestinal mucosal amino acid catabolism[J]. J Nutr, 1998, 128(8): 1249–1252. |
| [40] | FARAH Z. Camel milk properties and products[M]. St. Gallen: SKAT, 1996. |



