肠道是机体的消化器官,用以维持机体的健康生长及发育[1],肠道菌群在其中发挥了重要作用[2]。菌群可以通过增加小肠毛细血管密度、改变肠道生理和运动状态来影响肠上皮的作用机制,从而促进日粮中的能量利用[3],帮助消化吸收。反刍动物肠道内的细菌在降解膳食纤维方面起主要作用,日粮的消化也依赖于肠道中的纤维素水解细菌[4]。有研究表明,通过调控日粮组成,使藏绵羊在日增重升高的同时,瘤胃菌群的丰富度也发生了显著变化,主要表现为普雷沃特氏菌属、Ruminococcus属和Succiniclasticum属等物种丰度的升高[5]。另有研究表明,通过饲喂黑牛高谷物日粮,显著增加肌内脂肪的同时,发现瘤胃内菌群丰度显著下降[6];相似的情况也发生在绵羊瘤胃研究中,高谷物的日粮饲喂在改变瘤胃内细菌群落的组成及丰度的同时对菌群的代谢途径也产生影响[7]。在本研究的前期结果中发现,日粮的改变主要影响阿勒泰羊的体重及体脂沉积的变化,同时又有研究表明,空肠菌群对机体免疫和体内稳态起重要作用[8],其次还对脂肪的生成具有调控作用[9]。因此,本试验首先研究不同生长速度的阿勒泰羊空肠菌群的差异;其次,通过日粮调控阿勒泰羊的生长速度,来探讨生长速度的改变对菌群的影响,从而探讨肠道菌群与绵羊生长发育之间的关系,为通过肠道菌群调控动物生长提供参考依据。
1 材料与方法 1.1 动物饲喂和分组根据试验设计打耳标记录300只阿勒泰羔羊初生体重,正常饲喂至6月龄时,筛选分组,将300只雄性羔羊中日增重为前10%的定义为生长速度快的高体重组,日增重为后10%的定义为生长速度慢的低体重组。两组各随机选择12只体重相近羔羊,将高体重组((32.88±1.80) kg)随机分为高体重能量日粮限饲组(HR)和高体重标准日粮对照组(HC);低体重组((22.90±4.05) kg)随机分为低体重能量日粮增加组(LA)和低体重标准日粮对照组(LC),每组各6只。按照中国肉羊饲养标准(2004)设计日粮(能量摄入为营养需要量的100%),HR、HC、LC和LA组分别饲喂能量摄入为营养需要量的75%、100%、100%和125%的日粮[10],不同能量水平日粮组成见表 1。于每日09:00和18:00饲喂,自由饮水,预试期7 d,正试期30 d。
|
|
表 1 不同能量水平日粮组成 Table 1 Dietary composition of different energy levels |
饲喂试验结束后,称体重,计算日增重。屠宰后取每只阿勒泰羊空肠相同部位的内容物,根据TIANamp Stool DNA Kit (天根生化科技有限责任公司,北京)说明书进行总DNA抽提,DNA浓度和纯度利用单滴分光光度计(ONE Drop,OD 1000+,中国)进行检测(浓度≥50 ng·μL-1,A260/280在1.7~1.9之间),利用1%琼脂糖凝胶电泳检测DNA的提取质量,质检合格后DNA样品保存于-80 ℃待检。
1.3 PCR扩增及Illumina Miseq测序按指定测序区域(V3-V4区),合成带有barcode的特异引物,对V3-V4可变区进行PCR扩增。每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris-HCl洗脱,2%琼脂糖电泳检测,利用QuantiFluorTM-ST (Promega, USA)进行定量检测。根据Illumina MiSeq平台(Illumina, SanDiego, USA)标准操作规程将纯化后的扩增片段构建PE 2*300的文库(上海美吉生物医药科技有限公司)。在Usearch(vsesion 7.0 http://drive5.com/usearch/)平台上对样本进行聚类分析,根据不同的相似度水平,对所有序列进行OTU划分,在Silva128/16 s-bacteria数据库比对下,采用RDP classifier贝叶斯算法对97%的相似水平下的OTU进行生物信息统计分析。
1.4 统计分析阿勒泰羊体重数据应用GraphPad Prism 6软件对试验数据进行统计分析,采用T检验(t-test-unpaired)分析进行差异显著性检验,P<0.05表示差异显著,P<0.01表示差异极显著。
2 结果 2.1 阿勒泰羊肠道菌群物种分类统计本研究从所有阿勒泰羊空肠内容物提取的DNA中检测出2 057个操作分类单元(OTU),25个门水平细菌群落,835个属水平细菌群落(见表 2)。经统计结果后,剔除门分类水平丰度小于1%及未有明确分类(p_unclassified_k_norank)的细菌群落后按整体样本的最小序列数进行质量抽平,抽平是样本均一化的过程,可保证各个样本在相同测序量的情况下进行比较;对数据进行抽平后,本研究得到1 978个高质量OTUs,20个门水平细菌群落,818个属水平细菌群落。
|
|
表 2 物种分类统计表 Table 2 Species classification statistics |
由图 1可知,在标准日粮饲喂条件下,7月龄阿勒泰羊体重较6月龄均有升高,且生长速度快的HC组体重仍显著高于LC组(P<0.05)。

|
数据表示为“平均数±标准误”,**表示差异极显著(P < 0.01),*表示差异显著(P < 0.05)。HC.高体重标准日粮对照组;LC.低体重标准日粮对照组。下同 The data are represented as "mean±standard error", ** means extremely significant difference (P < 0.01), * means significant difference (P < 0.05). HC. High body weight control group; LC. Low body weight control group. The same as below 图 1 不同生长速度阿勒泰羊体重差异 Fig. 1 Body weight difference of Altay sheep with different growth rates |
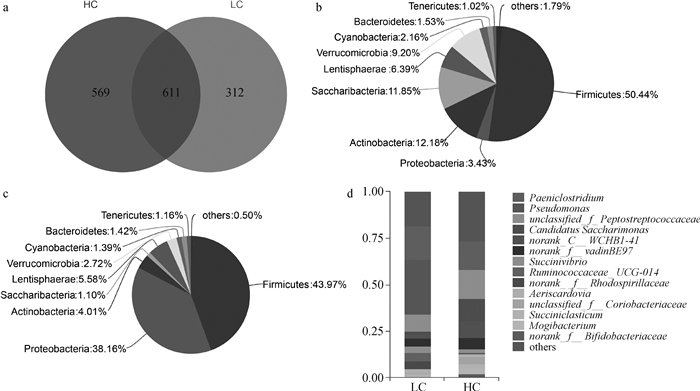
通过图 2a可看出,在HC、LC组阿勒泰羊空肠内容物提取的DNA中分别获得了1 180和923个OTUs,其中HC、LC组中共有OTU数为611个,HC组特有OTU数为569个,LC组特有OTU数为312个(图 2a)。在门分类水平上,HC组厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、Saccharibacteria门的物种丰度高于LC组,它们分别占菌群数量的50.44%、12.18%、11.85%(图 2b);LC组中变形菌门(Proteobacteria)丰度高于HC组,它占菌群数量的38.16%(图 2c)。在属分类水平上,LC组中变形菌门下的绿脓杆菌属(Pseudomonas)和红螺菌科未分类属(norank_f_Rhodospirillaceae)丰度高于HC组,HC组中疣微菌门下的WCHB1-41属、厚壁菌门下的Candidatus-Saccharimonas 属和消化链球菌科未明确信息属(unclassified_f_Peptostreptococcaceae)、放线菌门下的红蝽杆菌科未明确信息属(unclassified_f_Coriobacteriaceae)和双歧杆菌科未明确信息属(norank_f_Bifidobacteriaceae)丰度高于LC组(图 2d)。
|
a. Venn图(OTU); b. HC组群落组成(门水平); c. LC组群落组成(门水平); d.群落组成(属水平) a. Venn diagram(OTU); b. HC group community composition (phylum level); c. LC group community composition(phylum level); d. Community composition (genus level) 图 2 不同生长速度阿勒泰羊肠道菌群组成差异 Fig. 2 Composition differences of intestinal microbiota of Altay sheep with different growth rates |
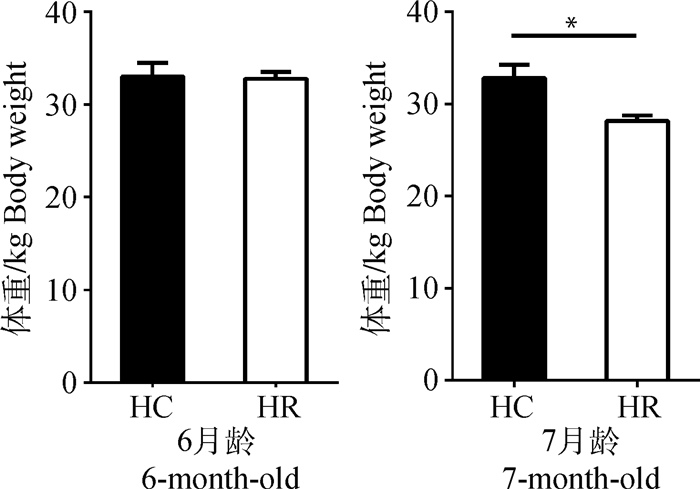
2.3.1 日粮能量限饲对高体重组阿勒泰羊生长速度的影响 由图 3可知,对生长快速组阿勒泰羊饲喂能量摄入量为机体所需的75%的能量饲料后,HR组羊由初始体重(32.77±0.751) kg减重至末体重(28.17±0.601) kg,每只羊平均减重4.60 kg;HC羊由初始体重(33.00±1.457) kg减重至末体重(32.83± 1.424) kg,每只羊平均减重0.17 kg;HR组日增重-0.15 kg,低于HC组日增重0.021 kg。低能量水平日粮抑制了生长速度快羔羊的生长,HR组体重显著低于HC组(P<0.05)。
|
HR.高体重能量日粮限饲组。下同 HR.High body weight restriction feed group. The same as below 图 3 日粮能量限饲对阿勒泰羊体重的影响 Fig. 3 Effect of dietary energy restriction on body weight of Altay Sheep |
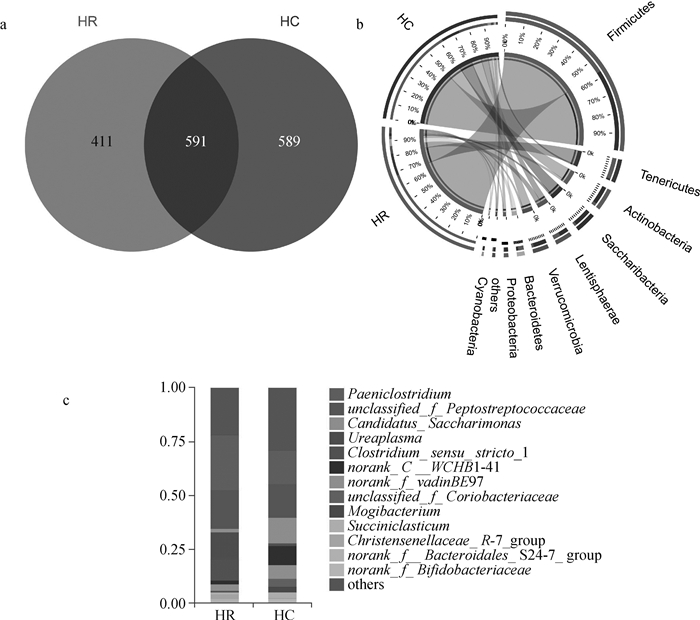
2.3.2 减缓生长速度对肠道菌群组成的影响 通过图 4a可看出,在HR、HC组阿勒泰羊空肠内容物提取的DNA中分别获得了1 002和1 180个OTUs,其中HR、HC组中共有OTU数为591个,HR组特有OTU数为411个,HC组特有OTU数为589个。减缓高体重阿勒泰羊的生长速度后,在门分类水平上(图 4b),厚壁菌门、软壁菌门(Tenericutes)和拟杆菌门(Bacteroidetes)菌群的丰度有所升高,变形菌门、放线菌门、Saccharibacteria门、疣微菌门(Verrucomicrobia)、蓝藻门(Cyanobacteria)等菌群丰度降低。在属分类水平上(图 4c),厚壁菌门下的拟乳菌属(Paeniclostridium)、消化链球菌科未明确信息属和梭菌属(Clostridium-Sensu-Stricto-1),黏胶球形菌门下的Ureaplasma 属丰度升高,黏胶球形菌门下的norank_f_ vadinBE97 属、厚壁菌门下的Candidatus_Saccharimonas 属、疣微菌门下的norank_c_WCHB1-41属丰度降低。
|
a. Venn图(OTU); b.样本与物种关系(门水平); c.群落组成(属水平) a. Venn diagram(OTU); b. Relationship of sample and species (phylum level); c. Community composition (genus level) 图 4 减缓生长速度对阿勒泰羊肠道菌群组成的影响 Fig. 4 Effect of slowing down growth rate on intestinal microbiota of Altay sheep |
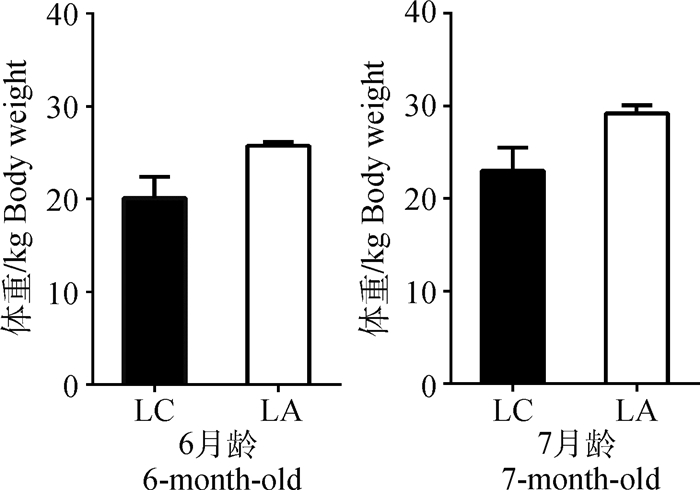
2.4.1 日粮能量增加对低体重组阿勒泰羊生长速度的影响 生长慢速组阿勒泰羊饲喂能量摄入量为机体所需的125%的能量饲料后,由图 5可知,LA组羊由初始体重(25.73±0.418) kg增重至末体重(29.20±0.851) kg,每只羊平均增长3.47 kg;LC羊由初始体重(20.07±2.334) kg增重至末体重(23.00±2.500)kg,每只羊平均增长2.93 kg;LA组日增重0.116 kg高于LC组日增重0.098 kg。高能量水平日粮加快了生长速度慢羔羊的生长,7月龄时,LA组体重高于LC组。
|
LA.低体重能量日粮增加组。下同 LA.Low body weight add feed group. The same as below 图 5 日粮能量增加对阿勒泰羊体重的影响 Fig. 5 Effect of dietary energy add feed on body weight of Altay sheep |
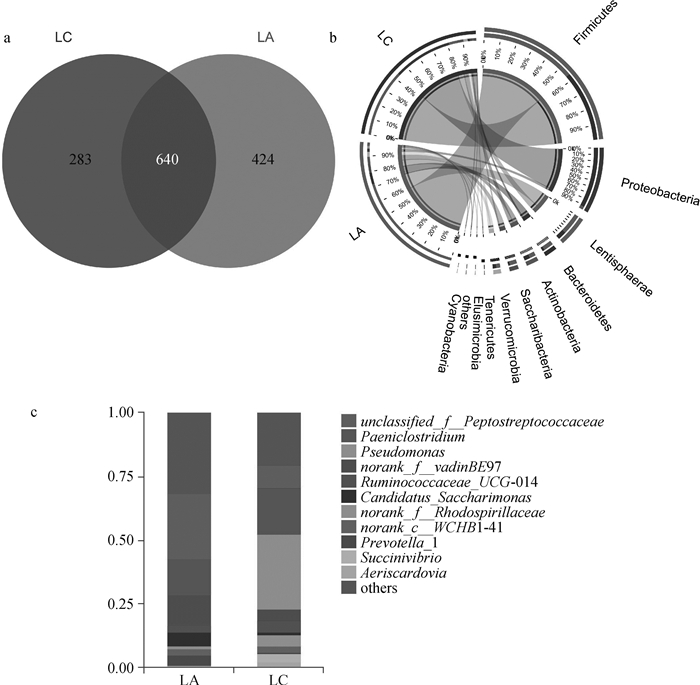
2.4.2 加快生长速度对肠道菌群物种组成的影响 通过图 6a可看出,在LC、LA组阿勒泰羊空肠内容物提取的DNA中分别获得了923和1 064个OTUs,其中LC、LA中共有OTU数为640个,LC组特有OTU数为283个,LC组特有OTU数为424个。增加低体重组阿勒泰羊的生长速度后,在门分类水平上(图 6b),厚壁菌门、黏胶球形菌门(Lentisphaerae)、Saccharibacteria门、拟杆菌门(Bacteroidetes)、迷踪菌门(Elusimicrobia)等菌群丰度升高,变形菌门、蓝藻门(Cyanobacteria)等菌群丰度降低。在属分类水平上(图 6c),变形菌门下的绿脓杆菌属、红螺菌科未分类属(norank_f_Rhodospirillaceae)和琥珀酸弧菌属(Succinivibrio)丰度降低,厚壁菌门下的消化链球菌科未明确信息属、Candidatus-Saccharimonas属、黏胶球形菌门下的的norank_f_vadinBE97属、拟杆菌门下的普氏菌属(Prevotella_1)丰度升高。
|
a. Venn图(OTU); b.样本与物种关系(门水平); c.群落组成(属水平) a. Venn diagram(OTU); b. Relationship of sample and species (phylum level); c. Community composition (genus level) 图 6 加快生长速度对阿勒泰羊肠道菌群组成的影响 Fig. 6 Effect of accelerating growth rate on intestinal microbiota of Altay sheep |
肠道微生物群被称为“第二基因组”,在动物体内发挥重要作用,它可维持机体的生长发育及健康。在对肠道菌群的研究中,空肠菌群的研究相比于其它肠道部位较少。有报道称,日粮中脂质成分的吸收和消化主要在空肠中完成[11],空肠菌群又对脂肪的生成具有促进作用[9];其次,在本研究的前期试验中证明, 日粮中能量水平的改变对体脂沉积有显著影响,因此,本研究采用Illumina Miseq测序技术,重点探讨了空肠部位肠道菌群的组成及变化情况。
本研究表明,阿勒泰羊肠道中厚壁菌门、放线菌门和变形菌门为优势物种,与美利奴羊[12]、藏绵羊[5]及蒙古绵羊[13]的研究一致。首先对相同饲喂条件下,不同生长速度的阿勒泰羊肠道菌群组成进行表征,结果表明,在门分类水平上,生长快速组的羊肠道中厚壁菌门、放线菌门和Saccharibacteria门的物种丰度高于生长慢速组的羊。有研究称,反刍动物肠道中的厚壁菌门可以降解食物中的纤维[14-15],有助于食物消化及动物的生长发育,同时它也是反刍动物肠道中丰度最高、最具代表性的门水平物种[16]。生长慢速组羊中变形菌门丰度较高,研究表明,变形菌门具有重要的食物降解作用,并且在牛[17]和熊猫[18]的肠道菌群中占主导地位。有研究发现,绵羊肠道中拟杆菌门也是优势物种[13],但本试验结果显示,阿勒泰羊空肠中的优势物种中没有拟杆菌门,可能是由于羊的品种、地理环境或者饲喂草料的不同引起。
在属分类水平上,生长快速组羊肠道中unclassified_f_Peptostreptococcaceae 属的物种丰度高于生长慢速组,有研究表明,链球菌有助于食物的消化,具有产生机体所需维生素和调节免疫力等功能,有助于机体健康生长[19]。本研究发现,生长慢速组的羊肠道中拟乳菌属、瘤胃菌属、绿脓杆菌属、琥珀酸弧菌属等物种的丰度高于生长快速组。有研究表明,瘤胃菌属是牛肠道厚壁菌门中丰度最高的属水平物种[20];但本研究显示,拟乳菌属是厚壁菌门中丰度最高的物种,相反,瘤胃菌属丰度排名靠后。有研究证实,瘤胃菌属是典型的纤维降解菌[21],其中大部分可降解蛋白质,帮助食物在肠道中分解消化[22],有助于机体生长发育。本研究推测,拟乳菌属丰度过高可能是造成阿勒泰羊生长速度慢的原因。
3.2 日粮改变对于阿勒泰羊生长速度的影响分析本研究通过改变日粮中能量的摄入量来改变阿勒泰羊的生长速度,以期探讨生长速度对肠道菌群的影响。有研究表明,饲喂较多的谷物和较少的精料可显著增加奶牛的产奶量[23],并且不会对机体造成不利影响。因此,本试验选用玉米作为谷物饲料提供能量来源,在研究饲料能量供给对阿勒泰羊生长的影响时,饲喂过程中未出现对其健康产生的不利影响,而仅仅是改变了生长速度的快慢。给生长快速组的羊饲喂能量摄入为营养需要量75%的日粮,结果显示,其日增重低于对照组羊的日增重,并且试验组与对照组之间体重差异显著(P<0.05);给生长慢速组羊饲喂能量摄入为营养需要量125%的日粮后,结果显示,其日增重水平高于对照组羊的日增重,试验组与对照组之间体重差异不显著(P>0.05)。
3.3 阿勒泰羊生长速度的变化对肠道菌群物种组成的影响分析本试验结果表明,加快低体重组阿勒泰羊的生长速度后,肠道菌群物种组成发生了变化。在门分类水平上,LA组厚壁菌门、拟杆菌门和迷踪菌门丰度均有升高;迷踪菌门首先在白蚁肠道中被发现,其次在沉积物、地下水和土壤中发现的较多[24],但是其在动物肠道中的研究还很少,这一点有望在后续研究中继续探讨。厚壁菌门中大多数物种可促进日粮中热量的利用,进而促进机体体重的增加[25];拟杆菌门的含量与体重指数呈正相关,含量越高,体重增加越多[26];在本研究中,生长慢速组的羊在生长速度变快后,也表现出厚壁菌门和拟杆菌门的增加,与前人研究结果一致,表明厚壁菌门和拟杆菌门可促进动物的生长。在属分类水平上,LA组中瘤胃菌属和绿脓杆菌属的物种丰度降低,其中,瘤胃菌属是厚壁菌门中最具代表性的物种,也是降解抗性多糖(如淀粉和纤维素)的物质[22],还可提供一系列酶降解系统[27]。本研究显示,厚壁菌门的整体丰度升高,但是下分的属水平物种有所降低,说明相同门水平下的不同属的物种,在动物生长发育中所发挥的作用是不同的,它们的相互协作促进机体的健康生长。
减缓高体重组阿勒泰羊的生长速度后,肠道菌群丰度也发生了变化。HR组厚壁菌门和拟杆菌门丰度均升高,放线菌门丰度降低。有研究证明,拟杆菌有助于食物中复杂碳水化合物的消化[28],在人的双胞胎肠道菌群研究中发现,体瘦的双胞胎肠道菌群中拟杆菌门的丰度较高,并且含有丰富的与碳水化合物代谢有关的基因,放线菌门水平较低[29];这一研究结果与本试验HR组的结果一致,表明高丰度的拟杆菌门和低丰度的放线菌门与体重的增长呈负相关。随着体重的减轻,厚壁菌门与拟杆菌门的比例逐渐降低[30],这一结论与减缓高体重羊的生长速度结果一致,厚壁菌门比拟杆菌门的比率从34%降低至15.46%;在属分类水平上,HR组拟乳菌属丰度升高,与第一部分的试验结果一致,拟乳菌属丰度的变化与生长速度呈负相关关系。
本试验结果表明,无论是正常生长状态下,还是通过日粮调控生长速度后,都表现出生长速度较快的组中,Saccharibacteria门和消化链球菌科未明确信息属的菌群丰度较高,拟乳菌属丰度较低。依据本试验的结果,推测肠道菌群中物种丰度的变化与生长速度的变化有相关性。
4 结论不同生长速度组中,肠道菌群的组成有差异;在改变生长速度后,肠道菌群组成也相应地发生变化。综合分析两部分试验结果可得,阿勒泰羊肠道中厚壁菌门和拟杆菌门的物种丰度变化可能与生长速度有关,Saccharibacteria门的物种丰度变化与生长速度呈正相关,拟乳菌属的物种丰度变化与生长速度呈负相关。研究成果为通过调控特定肠道菌群的丰度从而促进羔羊健康生长提供了理论依据。
| [1] |
郭晓红, 郭玉龙, 刘亚丹, 等. 仔猪不同发育阶段结肠微生物菌群特征分析[J]. 畜牧兽医学报, 2019, 50(9): 1759–1774.
GUO X H, GUO Y L, LIU Y D, et al. Characteristics of intestinal microflora of colon at different developmental stages in piglets[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(9): 1759–1774. (in Chinese) |
| [2] | BRESTOFF J R, ARTIS D. Commensal bacteria at the interface of host metabolism and the immune system[J]. Nat Immunol, 2013, 14(7): 676–684. DOI: 10.1038/ni.2640 |
| [3] | BOULANGÉ C L, NEVES A L, CHILLOUX J, et al. Impact of the gut microbiota on inflammation, obesity, and metabolic disease[J]. Genome Med, 2016, 8: 42. DOI: 10.1186/s13073-016-0303-2 |
| [4] | WANG J, FAN H, HAN Y, et al. Characterization of the microbial communities along the gastrointestinal tract of sheep by 454 pyrosequencing analysis[J]. Asian-Australas J Anim Sci, 2017, 30(1): 100–110. |
| [5] | CUI X X, WANG Z F, YAN T H, et al. Rumen bacterial diversity of Tibetan sheep (ovis aries) associated with different forage types on the Qinghai-Tibetan Plateau[J]. Can J Microbiol, 2019, 65(12): 859–869. DOI: 10.1139/cjm-2019-0154 |
| [6] | OGATA T, MAKINO H, ISHIZUKA N, et al. Long-term high-grain diet altered the ruminal pH, fermentation, and composition and functions of the rumen bacterial community, leading to enhanced lactic acid production in Japanese Black beef cattle during fattening[J]. PLoS One, 2019, 14(11): e0225448. DOI: 10.1371/journal.pone.0225448 |
| [7] | SEDDIK H, XU L, WANG Y, et al. A rapid shift to high-grain diet results in dynamic changes in rumen epimural microbiome in sheep[J]. Animal, 2019, 13(8): 1614–1622. DOI: 10.1017/S1751731118003269 |
| [8] | JIN J P, ZHANG L P, JIA J L, et al. Effects of maternal low-protein diet on microbiota structure and function in the jejunum of Huzhu Bamei suckling piglets[J]. Animal, 2019, 9(10): 713. DOI: 10.3390/ani9100713 |
| [9] | KLEEREBEZEM M. Microbial metabolic gatekeeping in the jejunum[J]. Nat Microbiol, 2018, 3(6): 650–651. DOI: 10.1038/s41564-018-0172-0 |
| [10] |
张涛.不同能量水平日粮对绵羊脂肪酸合成酶基因和肥胖基因表达的影响[D].兰州: 甘肃农业大学, 2007.
ZHANG T.Effects of different energy intake on the expression of fatty acid synthase gene and obese gene in sheep[D]. Lanzhou: Gansu Agricultural University, 2007.(in Chinese) |
| [11] | KASTL A J JR, TERRY N A, WU G D, et al. The structure and function of the human small intestinal microbiota:current understanding and future directions[J]. Cell Mol Gastroenterol Hepatol, 2019, 9(1): 34–35. |
| [12] | MAMUN M A A, SANDEMAN M, RAYMENT P, et al. The composition and stability of the faecal microbiota of Merino sheep[J]. J Appl Microbiol, 2020, 128(1): 280–291. |
| [13] | ZENG Y, ZENG D, NI X Q, et al. Microbial community compositions in the gastrointestinal tract of Chinese Mongolian sheep using Illumina MiSeq sequencing revealed high microbial diversity[J]. AMB Express, 2017, 7(1): 75. DOI: 10.1186/s13568-017-0378-1 |
| [14] | EVANS N J, BROWN J M, MURRAY R D, et al. Characterization of novel bovine gastrointestinal tract Treponema isolates and comparison with bovine digital dermatitis treponemes[J]. Appl Environ Microbiol, 2011, 77(1): 138–147. |
| [15] | THOETKIATTIKUL H, MHUANTONG W, LAOTHANACHAREON T, et al. Comparative analysis of microbial profiles in cow rumen fed with different dietary fiber by tagged 16S rRNA gene pyrosequencing[J]. Curr Microbiol, 2013, 67(2): 130–137. DOI: 10.1007/s00284-013-0336-3 |
| [16] | TANCA A, FRAUMENE C, MANGHINA V, et al. Diversity and functions of the sheep faecal microbiota :a multi-omic characterization[J]. Microb Biotechnol, 2017, 10(3): 541–554. |
| [17] | MAO S Y, ZHANG M L, LIU J H, et al. Characterising the bacterial microbiota across the gastrointestinal tracts of dairy cattle:membership and potential function[J]. Sci Rep, 2015, 5: 16116. DOI: 10.1038/srep16116 |
| [18] | FANG W, FANG Z M, ZHOU P, et al. Evidence for lignin oxidation by the giant panda fecal microbiome[J]. PLoS One, 2012, 7(11): e50312. DOI: 10.1371/journal.pone.0050312 |
| [19] | BULL M J, PLUMMER N T. Part 1:the human gut microbiome in health and disease[J]. Integr Med (Encinitas), 2014, 13(6): 17–22. |
| [20] | KIM M, KIM J, KUEHN L A, et al. Investigation of bacterial diversity in the feces of cattle fed different diets[J]. J Anim Sci, 2014, 92(2): 683–694. |
| [21] |
李琼, 王立志, 王之盛, 等. 不同氮利用率山羊瘤胃细菌结构与组成的差异[J]. 动物营养学报, 2019, 31(8): 3651–3663.
LI Q, WANG L Z, WANG Z S, et al. Differences of bacterial structure and composition in rumen of goats with different nitrogen utilization[J]. Chinese Journal of Animal Nutrition, 2019, 31(8): 3651–3663. (in Chinese) |
| [22] | HAN X F, YANG Y X, YAN H L, et al. Rumen bacterial diversity of 80 to 110-day-old goats using 16S rRNA sequencing[J]. PLoS One, 2015, 10(2): e0117811. DOI: 10.1371/journal.pone.0117811 |
| [23] | TAO S Y, TIAN P, LUO Y W, et al. Microbiome-metabolome responses to a high-grain diet associated with the hind-gut health of goats[J]. Front Micro-biol, 2017, 8: 1764. DOI: 10.3389/fmicb.2017.01764 |
| [24] | MÉHEUST R, CASTELLE C J, CARNEVALI P B M, et al.Aquatic Elusimicrobia are metabolically diverse compared to gut microbiome Elusimicrobia and some have novel nitrogenase-like gene clusters[J/OL]. bioRxiv, 2019, (2019-09-11)[2020-03-02]. http://www.biorxiv.org/content/10.1101/765248v2. |
| [25] | KRAJMALNIK-BROWN R, ILHAN Z E, KANG D W, et al. Effects of gut microbes on nutrient absorption and energy regulation[J]. Nutr Clin Pract, 2012, 27(2): 201–214. DOI: 10.1177/0884533611436116 |
| [26] | IGNACIO A, FERNANDES M R, RODRIGUES V A A, et al. Correlation between body mass index and faecal microbiota from children[J]. Clin Microbiol Infect, 2016, 22(3): 258.e1–258.e8. DOI: 10.1016/j.cmi.2015.10.031 |
| [27] | COLLIER C T, HOFACRE C L, PAYNE A M, et al. Coccidia-induced mucogenesis promotes the onset of necrotic enteritis by supporting Clostridium perfringens growth[J]. Vet Immunol Immunopathol, 2008, 122(1-2): 104–115. DOI: 10.1016/j.vetimm.2007.10.014 |
| [28] | SPENCE C, WELLS W G, SMITH C J. Characterization of the primary starch utilization operon in the obligate anaerobe Bacteroides fragilis:regulation by carbon source and oxygen[J]. J Bacteriol, 2006, 188(13): 4663–4672. DOI: 10.1128/JB.00125-06 |
| [29] | KOLIADA A, SYZENKO G, MOSEIKO V, et al. Association between body mass index and Firmicutes/Bacteroidetes ratio in an adult Ukrainian population[J]. BMC Microbiol, 2017, 17(1): 120. DOI: 10.1186/s12866-017-1027-1 |
| [30] | SWEENEY T E, MORTON J M. The human gut microbiome:a review of the effect of obesity and surgically induced weight loss[J]. JAMA Surg, 2013, 148(6): 563–569. DOI: 10.1001/jamasurg.2013.5 |



