随着人类基因组计划的完成,生命科学开始进入后基因组时代,科学家们发现在基因组中仅有2%的核苷酸编码序列,其余98%的非编码RNA(non-coding RNA, ncRNA)被认为是基因组转录的“噪声”[1]。1990年,有学者在哺乳动物中发现了第1个lncRNA-H19,随后发现lncRNA Xist在哺乳动物X染色体失活和剂量补偿效应的作用[2-3]。2002年,Okazaki等[4]基于cDNA文库分析了小鼠的转录组,鉴定出11 665个lncRNAs。至此,开启了lncRNAs研究的先河。2003年,美国国立人类基因组研究院启动“DNA元件百科全书”计划,旨在解析人类基因组非编码RNA的功能[5]。LncRNAs作为ncRNA家族的新成员之一,从表观遗传学、转录调控及转录后调控3个层面对基因进行调控,在人与动物疾病中起着重要的作用。在拟南芥、线虫、果蝇、小鼠和人类等多种生物中已发现数以万计的lncRNAs分子,截至2020年1月,GENCODE (Version 32)数据库(www.gencodegenes)显示,在人和小鼠中分别鉴定到17 910和13 201个lncRNAs[6]。随着LncRNAs的发现及其功能的深入研究,蕴育着一个新层次基因表达调控方式的发现。近年来研究发现lncRNAs的调控作用几乎贯穿于整个生命活动,并可通过天然免疫、改变细胞代谢等方式参与抗病毒感染[7-9]。
天然免疫应答是机体抵御病毒感染的第一道防线,在抵抗病毒入侵过程中发挥重要作用[10]。免疫系统通过模式识别受体有效监控入侵病毒在宿主细胞内的复制增殖,而lncRNAs可以在天然免疫应答中通过复杂的信号通路调控基因表达来发挥抗病毒感染的作用[11]。笔者系统梳理了近年来参与调控抗病毒天然免疫应答lncRNAs的研究成果,重点关注lncRNAs表达调控网络,以期为从事lncRNAs介导抗病毒研究的学者提供参考和依据。
1 lncRNA鉴定、特征及功能 1.1 lncRNA鉴定lncRNAs是一类转录本长度大于200 nt的非编码RNA,不编码蛋白,可编码小肽[12-13]。绝大多数lncRNAs由RNA聚合酶Ⅱ转录而来,具有mRNA的特征,如5′端帽子、聚腺苷酸化、剪切等,也有一些是RNA聚合酶Ⅲ转录生成无polyA修饰的lncRNAs。对于未知lncRNAs转录本的鉴定是利用Cufflinks对Tophat的比对结果进行转录本重构,将组装的转录本mapping到参考基因组,去除已知的mRNA和小于200 bp的转录本,再利用CPC[14]和CNCI[15]对转录本进行lncRNAs预测。最后通过转录本覆盖度、外显子数目、与非lncRNAs序列比对等一系列过滤条件,鉴定新型候选lncRNAs。
lncRNAs广泛存在于各种生物体内,其产生方式主要有几种:(1)编码基因发生框插入,转成一个与先前编码序列合成的lncRNAs;(2)染色体重排,两个不转录且距离很远的片段合并在一起,产生含有多个外显子的lncRNAs;(3)非编码基因通过反转录转座作用复制,形成有功能或无功能的反转录基因;(4)在ncRNA内部形成相邻的重复序列,从而产生lncRNAs;(5)通过转位基因插入,产生有功能的lncRNAs[16]。
1.2 lncRNA特征迄今为止,在Noncode数据库(http://www.noncode.org/)中共收录548 640个lncRNAs转录本和354 855个lncRNAs基因。由于lncRNAs种类多、数量大和分类方式复杂,人们对其分类尚缺乏统一标准。lncRNAs基于其在基因组上的位置,可分为正义型(sense)、反义型(antisense)、双向型(bidirectional)、内含子型(intronic)和基因间型(intergenic)[17]。目前,研究较多的是Intergenic lncRNAs,其具有较低的编码潜能且不与注释的编码基因重叠。lncRNAs具有不同的保守性[18-19],表达具有组织及时空特异性,Ransohoff等[20]研究表明,在lncRNAs中78%具有组织特异性,而mRNA中只有19%具有组织特异性。相比于mRNA,lncRNAs具有较少的外显子且长度较短,并且以平均低于mRNA 10倍水平表达[21]。
1.3 lncRNA功能尽管lncRNAs表达量低,但其表达模式却具有细胞、组织和发育阶段的特异性,在整个生命活动具有举足轻重的作用。研究表明,lncRNAs作为功能蛋白质的信号分子、诱饵分子、指导分子、骨架分子等[22]多种方式(图 1),在基因转录、翻译、蛋白修饰及染色质重构等多重水平调控基因表达网络[23]。其实一些lncRNAs是miRNA的前体,与miRNA不同的是,lncRNAs没有固定的作用模式,而是通过多种调控方式介导基因表达。表观遗传学调控[24],lncRNAs会招募染色质重构和修饰复合体到特定位点,改变甲基化状态、染色体结构和修饰状态,进而调控基因表达。转录调控[25],lncRNAs可作为配基与转录因子结合,控制基因转录活性。转录后调控[26],编码蛋白的基因上游启动子区转录,干扰下游基因表达;抑制RNA聚合酶Ⅱ和介导蛋白修饰及染色质重构,影响下游基因表达;与编码蛋白基因的转录本结合,干扰mRNA剪切,同时在酶的作用下形成内源性siRNA等。lncRNAs还可以像海绵一样与mRNA竞争结合miRNA的MREs,从而形成ceRNA阻止miRNA同靶基因mRNA结合,进而调控基因表达[27]。
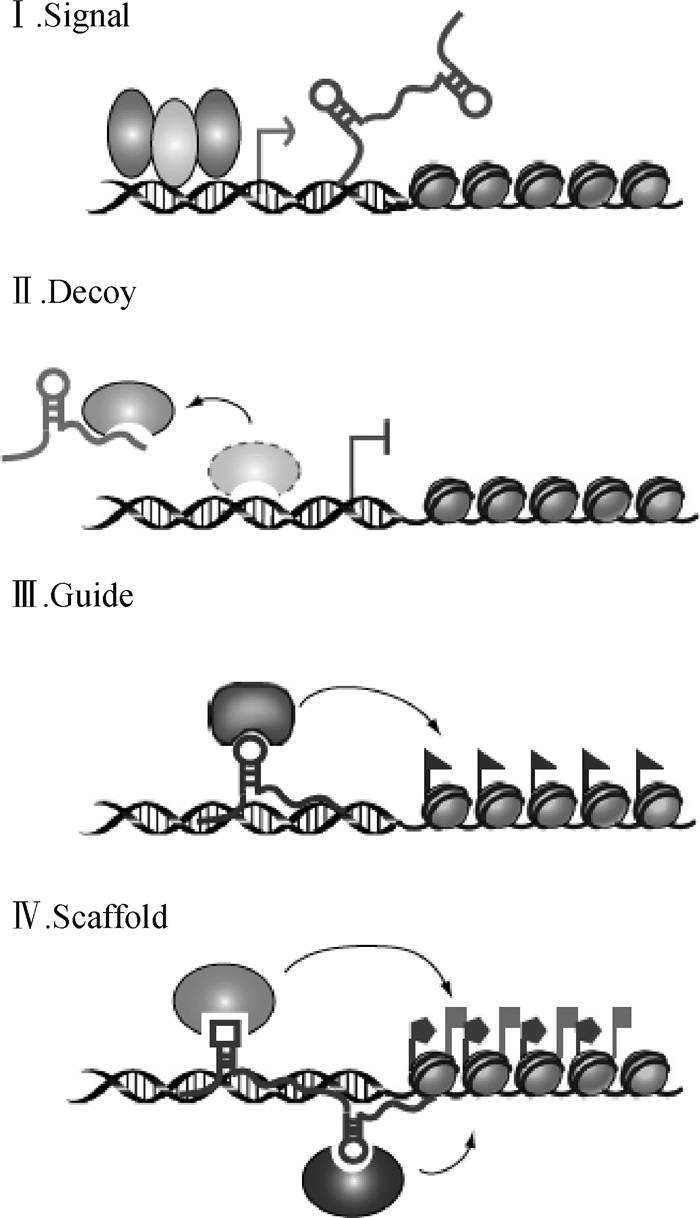
|
Ι.信号分子(singnal archetype),lncRNA可以作为转录活性指示剂进一步调控基因表达;Ⅱ.诱饵分子(decoy archetype),lncRNA能与调控RNA结合并将其隔离,从而对靶基因进行调控;Ⅲ.指导分子(guide archetype),lncRNA能指导蛋白复合物定位到特定的调控位点,从而调节靶基因;Ⅳ.骨架分子(scaffold archetype),lncRNA可以作为蛋白或RNA的组装平台,招募复合物,调控靶基因 图 1 LncRNA机制的4种原型的示意图[22] Fig. 1 Schematic diagram of the four archetypes of lncRNA mechanism |
天然免疫应答是机体抵抗病毒入侵的第一道防线,而模式识别受体(pattern recognition receptor,PRR)是天然免疫识别病原体的基础[7]。免疫系统会通过维甲酸诱导基因(retinoic acid inducible gene,RIG)样受体(RIG-like receptors,RLR)和Toll样受体(toll like receptor,TLR)等模式识别受体有效监测病毒感染宿主细胞[28]。激活的PRR通过启动激酶活化级联反应将信号传递给MyD88(myeloid differentiation primary response 88)等配体分子,然后通过干扰素调节因子(interferon regulator factor,IRF)和核因子κB(nuclear factor-kappa B,NF-κB)等诱导干扰素(interferon, IFN)、干扰素刺激因子(interferon stimulated gene, ISGs)及肿瘤坏死因子(tumor necrosis factor, TNF)来抑制病毒复制。近年来研究表明,在病毒与宿主共进化过程中,病毒利用宿主lncRNAs靶向调控分子表达,促进病毒复制,宿主利用自身lncRNAs靶向调控分子,通过免疫应答来抑制病毒感染[9]。
2.1 病毒诱导lncRNA差异性转录病毒的感染可以导致宿主lncRNAs的差异转录,在这些差异的lncRNAs中,有很多在免疫信号通路中发挥着抗病毒作用。例如在人类疾病方面,Peng等[29]构建严重急性呼吸综合征(severe acute respiratory syndrome coronavirus, SARS-CoV)感染的小鼠模型,对其肺进行高通量测序分析,发现504个注释和1 406个未注释的lncRNAs发生差异表达,表明lncRNAs为响应SARS-CoV感染而发生差异性转录。同时证明部分差异性转录lncRNAs在病毒感染过程中调控天然免疫。甲型流感病毒(influenza A virus,IAV)严重威胁全球公共卫生安全,Hu等[30]在H5N8病毒感染的小鼠中鉴定出差异表达的lncRNAs,并根据在小鼠基因组中的染色体位置,命名为lncRNA(NONMMUT011061),在其互作靶基因中发现与细胞凋亡(CPSF3、THOC1和PARN)和免疫调节(FXR1)等显著相关,尽管未直接靶向与流感相关的分子,但是作者推测lncRNA(NONMMUT011061)在流感病毒入侵过程中起到了关键的调控作用。随后有研究者在H3N2猪流感病毒感染的A549细胞表达谱中筛选出6 129个差异表达的lncRNAs,其中,上调4 963个,下调1 166个,差异表达lncRNAs的靶基因能富集到细胞代谢和自噬等生物过程,表明lncRNAs在流感病毒复制中起到关键的调控作用[31]。在众多差异表达的lncRNAs中,只有少数部分lncRNAs主要介导病毒复制,Pan等[32]研究表明IFN-β能够诱导lncRNA-ISR表达来抑制IAV在小鼠体内复制,干扰lncRNA-ISR后可导致IAV复制显著增加,过表达lncRNA-ISR后可抑制流感病毒复制。Maarouf等[33]构建细胞系和小鼠的IAV感染模型,发现lncRNA-155的表达显著上调,在敲除lncRNA-155的小鼠中显著增加IAV复制和毒力,而过表达lncRNA-155后显著抑制了IAV复制。Chai等[34]通过对IAV感染和未感染的细胞系进行lncRNAs表达谱分析,发现lnc-ISG20在感染后显著上调表达,从而发挥抑制病毒复制的功能,而敲低lnc-ISG20后增加了病毒复制。这些研究表明lncRNA-ISR、lncRNA-155和Lnc-ISG20在IAV复制过程中均发挥着重要的调控作用,与IAV感染诱导的宿主抗病毒先天免疫有关。
动物疫病防控与人类公共健康息息相关,在动物病毒感染过程中同样受lncRNA差异性转录调控。例如,Ma等[35]通过对牛病毒性腹泻病毒(BVDV)感染2、6和18 h后的MDBK细胞进行测序,分析lncRNAs和mRNA表达谱,共鉴定1 236个lncRNAs转录本和3 261个mRNA转录本差异性转录调节。猪丁型冠状病毒(porcine delt acoronavirus, PDCoV)是一种新型猪冠状病毒,可引起仔猪的肠道坏死,肠壁变薄以及肠绒毛萎缩。Tang等[36]对PDCoV感染不同阶段新生仔猪中lncRNAs进行分析,在感染的早期、中期和后期鉴定出99、41和33个差异表达的lncRNAs,表明lncRNAs在PDCoV感染不同阶段发生差异性转录。据报道,lncRNAs在马立克病病毒[37]、猪流行性腹泻病毒[38]、狂犬病病毒[39]等多种动物病毒感染中都存在差异性转录。越来越多的证据表明,病毒感染后可使宿主lncRNAs发生差异性转录,其调控的分子机制仍值得深入探讨。
2.2 lncRNA调控IFN介导天然免疫应答当病毒入侵宿主后,一方面诱导宿主lncRNAs的差异性转录,另一方面与PRR结合,启动PRR信号级联反应,活化下游免疫相关基因转录,诱导IFN等细胞因子的表达。IFN等抗病毒细胞因子分泌到细胞内外,与其受体(IFNR)结合激活JAK-STAT信号通路,诱导一系列ISGs表达,这些ISGs直接或间接发挥IFN的抗病毒和免疫调节功能[40],从而为机体建立起抗病毒感染的第一道防线(图 2)。IFN应答过程包括PRR激活、IFN产生和ISGs表达等,已有的研究表明,lncRNAs可在不同环节调控IFN介导天然免疫应答[7]。
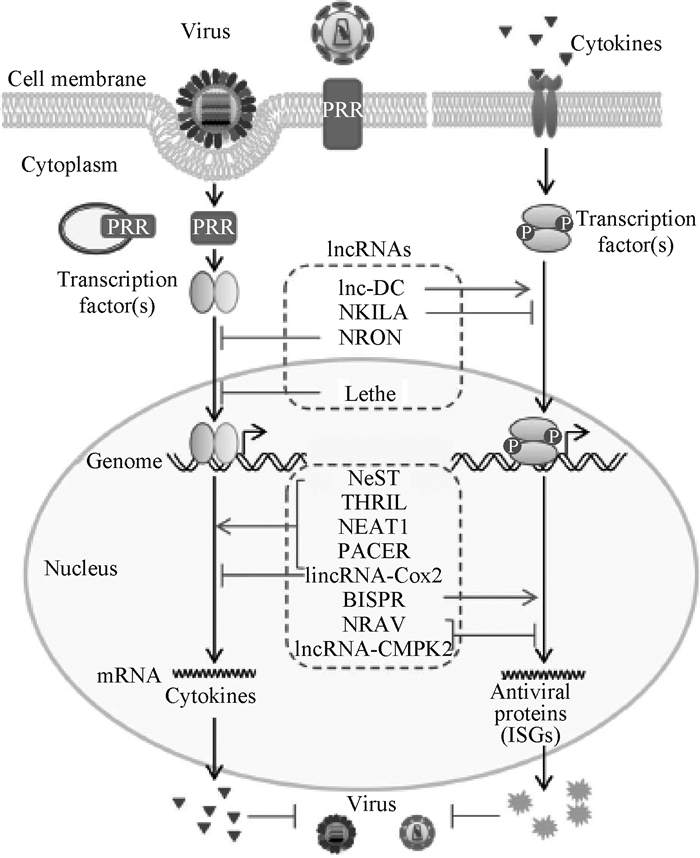
|
图 2 lncRNA调控干扰素免疫应答的机制[7] Fig. 2 Mechanisms of lncRNA in regulating interferon immune responses |
lnc-Lsm3b是一种源于宿主自身的lncRNA,可被IFN诱导表达,它与病毒RNA竞争结合RIG-I,是免疫应答晚期的负反馈调控因子。RIG-I的分子包含C端CTD结构域和N端的CARDs、DECH解旋酶结构域,病毒未感染时RIG-I分子以自身抑制状态存在,其中CARD2结构域与DECH解旋酶结构域的插入区结合,而当RNA病毒入侵后,病毒的5′三磷酸双链与CTD结构域结合,改变RIG-I结构并释放CARDs结构域,介导下游信号传递与免疫应答。曹雪涛课题组利用免疫共沉淀(IP)获取与RIG-I结合的RNA,将拉下来的所有RNA进行RNA-seq测序发现,当使用水疱性口炎病毒(vesi-cular stomatitis virus, VSV)刺激后,目的lnc-Lsm3b表达量明显增加。然后,构建lnc-Lsm3b缺陷小鼠进行体外刺激和体内感染VSV,发现lnc-Lsm3b缺陷后,IFN表达量显著升高,表明lnc-Lsm3b抑制机体抗病毒过程。然后, 利用紫外交联免疫共沉淀测序技术(CLIP-seq)发现,lnc-Lsm3b与RIG-I结合并改变其空间构象,使RIG-I无法识别RNA,导致IFN产生减少。同时,lnc-Lsm3b在RNA病毒感染的巨噬细胞内可抑制RIG-I介导的免疫应答,阻抑IFN与抗炎因子的产生,抗病毒信号受到抑制[41]。Lin等[9]通过对VSV全转录组测序发现Lnczc3 h7a能够分别结合TRIM25和RIG-I,并以脚手架的方式促进TRIM25和RIG-I的结合,增强TRIM25对RIG-I的K63泛素化,从而正向调控RIG-I介导的信号通路和抗病毒天然免疫反应。
hnRNPA2B1是一种新型DNA感受器蛋白,当DNA病毒入侵宿主后,将自身基因组DNA注射进细胞核,hnRNPA2B1能够识别病毒DNA并通过自身二聚化产生IFN,这一系列过程导致hnRNPA2B1从细胞核到细胞质的转移,在细胞质中它启动TBK1-IRF3信号通路诱导IFN-α/β表达。与此同时,hnRNPA2B1通过STING信号通路增强IFN-α/β的活化以应答DNA病毒的感染[42]。lncLrrc55-AS是病毒通过IFN-JAK-STAT途径在宿主细胞中被诱导产生的一种lncRNA,该分子可有效诱导IFN-I表达,在病毒感染过程中通过正反馈调节促进干扰素调节因子3(IRF3)信号传导和IFN-I的产生,从而介导先天免疫应答[8]。
ISGs是一类可被IFN诱导表达的免疫相关因子,在天然免疫应答中发挥着重要的抗病毒作用[43],一些研究表明lncRNAs是重要的ISGs表达调控因子。Ouyang等[44]通过检测感染和未感染IAV肺泡上皮细胞的lncRNAs表达变化发现,lncRNA-NRAV(negative regulator of antiviral response)在IAV感染的细胞中表达水平显著降低。然后通过在小鼠和细胞上体内、体外过表达NRAV,IAV复制能力及毒力都显著增强,敲除NRAV后则减弱IAV的复制及毒力,说明NRAV是抗病毒天然免疫应答的负调控因子。进一步研究NRAV促进IAV复制的机制,在病毒感染的细胞中过表达NRAV,然后通过全基因组表达谱芯片分析细胞中基因表达谱,基因本体(gene ontology,GO)显示众多差异表达基因与病毒复制相关,并且多种ISGs表达发生显著变化,包括IFIT2、IFIT3、OASL、MxA和IFITM3等,这些ISGs可通过不同的途径抑制病毒复制。研究表明,NRAV改变关键ISGs中IFITM3和MxA启动子区的组蛋白修饰水平:激活H3K4me3,抑制H3K27me3,从而负调控IFITM3和MxA的基因转录,同时通过RNA pull-down和RIP试验还发现,NRAV特异性与转录因子ZONAB相互作用,从而影响MxA的转录调控。Huang等[45]通过对感染和未感染传染性法氏囊病病毒的DF-1细胞进行高通量测序,筛选出48个显著差异表达基因,并发现Loc107051710通过调节IRF8促进了IFN-α/β的产生,从而促进ISGs的抗病毒能力。
此外,王品等[46]发现病毒诱导干扰素非依赖的独特lncRNA-ACOD1,该分子在体内外均能响应病毒感染,显著上调表达,从而促进多种病毒复制。在敲低GOT2后,lncRNA-ACOD1不能促进病毒的感染,表明lncRNA-ACOD1通过GOT2来增强病毒的感染能力。干扰素非依赖的lncRNAs通过调控胞内代谢促进病毒复制是一种促进病毒感染新作用模式,使细胞内代谢向有利于病毒复制的方向转变,促进了病毒复制和病毒逃逸。
2.3 lncRNA调控NF-κB信号通路先天性免疫应答是机体抵抗病毒感染的生理性防御反应,在免疫应答过程中首先通过PRR识别病毒的特定成分,尽管不同的PRR介导免疫应答的方式不同,但都要通过启动NF-κB信号通路。NF-κB信号通路不仅能调控IFN转录,还能调控细胞因子、趋化因子和黏附分子等众多免疫相关因子的表达。在哺乳动物中,已经鉴定出NF-κB家族中的6个转录因子:RelA (p65)、RelB、c-Rel、p50、p52和Relish,其中p50/p65研究最多。LncRNA-lethe可通过NF-κB信号转导器的p65亚基招募转录激活因子3 (STAT3)途径诱导表达,并且通过下调Ⅰ型干扰素反应,提高Lethe的表达促进丙型肝炎病毒的复制[47]。内源性逆转录病毒(endogenous retroviruses,ERVs)是基因组转座子元件之一,Zhou等[48]通过RNA-seq发现小鼠基因组中存在大量ERV的转录元件,并鉴定了一个内源性逆转录病毒来源的lncRNA-EPAV (ERV-derived lncRNA positively regulates antiviral responses),该分子可与SFPQ结合正反馈NF-κB促进免疫关键转录因子转录,提高宿主抗病毒天然免疫应答能力。此外,lncRNA ISR(interferon-stimulated lncRNA)可以抑制IAV复制,IAV感染NF-κB通路干扰的小鼠后,lncRNA ISR表达水平显著降低,表明lncRNA ISR在IAV感染过程中通过IFN-β依赖性信号抑制病毒复制[32]。
lncRNA-MALAT1是一种高度保守的转移相关肺腺癌转录物,与NF-κB相互作用以依赖的方式表达,在细胞核中MALAT1与p50/p65负调控NF-κB,起介导先天免疫的炎症作用[49]。HOTAIR(HOX transcript antisense intergenic RNA)除了可以抑制NF-κB以外,还可以通过翻译后修饰其亚基p65来调节NF-κB的活性。Wu等[50]研究发现,HOTAIR在脓毒症小鼠心肌细胞中因TNF-α和p65磷酸化水平升高而上调表达。Jin等[51]通过MIR31HG与NF-κB通路相互作用的研究,表明下调MIR31HG可抑制NF-κB通路,MIR31HG通过IκBα激活NF-jB信号,因此MIR31HG-NF-κB在炎症条件下抑制骨细胞分化。LncRNA-NKILA是一个与NF-κB信号通路相互作用的lncRNA,该分子以NF-κB、TNFα和IL1β依赖的方式诱导表达,NKILA可与NF-κB/IκB结合形成稳定的三元复合体,掩盖IκB的磷酸化位点,抑制IKK诱导的IκB磷酸化和NF-κB活化,负反馈调节NF-κB信号在炎症刺激的乳腺上皮细胞中过度激活[52]。
尽管上述部分lncRNAs未涉及病毒感染,但由于MALAT1/NKILA等lncRNAs可以结合宿主基因组上数百个活跃的染色质位点,同时考虑到NF-κB信号转导途径在介导病毒感染的炎症反应及免疫应答过程中起着十分重要的作用,因此调控NF-κB信号通路的lncRNAs在介导病毒感染中的作用仍值得深入探讨。
3 ceRNA调控网络在病毒与宿主相互作用中,研究人员发现基因的转录后调控并不是简单的miRNA-mRNA的沉默机制,而是一个复杂的调控网络,竞争性内源RNA(competing endogenous RNA, ceRNA)作为一种更为精细和复杂的基因表达调控模式拓宽了人们的认知[53]。Salmena等[54]提出新的假说,在ceRNA网络中,lnRNA和mRNA分子可以通过miRNA应答元件(miRNA reaction elements, MREs)吸附miRNA,这也是lncRNA参与转录后调控机制之一。研究表明,HSUR1和HSUR2可以特异性靶向宿主细胞来源的miR-16、miR-142-3p和miR-27,从而作为miRNA“海绵”调控宿主基因表达和病毒活性[55]。Chen等[56]通过对慢性乙型肝炎病毒感染样本进行lncRNAs表达谱测序分析,共鉴定出5 550个差异表达的lncRNAs,其中lncRNA T039096与表达量差异最大的基因mRNA-ZFP57共表达,两者可能在ceRNA调控网络相互作用,共同影响靶基因的表达水平,从而参与乙型肝炎病毒的发生发展。Wang等[57]首次构建埃博拉病毒感染人类细胞后诱导的效应的ceRNA网络,其中circRNA-chr1能9靶向miR-30b-3p来调控CLDN18表达,参与病毒的调控。Dai等[58]通过对不同时间点感染禽白血病病毒巨噬细胞进行高通量测序,在感染3 hpi时鉴定出差异表达128个lncRNAs和15个miRNA,ceRNA机制表明,lncRNA-miRNA通过Jak-STAT信号通路调控CCND3和SOCS5影响病毒复制。与此同时,XLOC_672329、ALDBGALG0000001429、XLOC_016500和ALDBGALG0000000253也响应了宿主抗病毒免疫应答。Wang等[59]对感染马立克病病毒(MDV)鸡的脾进行表达谱分析,发现ceRNA网络参与MDV发生。Gga-miR-155不仅与circGTDC1和circMYO1B相互作用,而且还靶向免疫相关基因,这表明非编码RNA在介导免疫应答基因中的作用。
另外,随着m6A甲基化测序技术的发展,lncRNAs的m6A修饰成为ceRNA中新的调控机制。Zheng等[60]发现lncRNA-FAM225A中m6A修饰水平显著高于正常细胞,并且能够提高其转录水平稳定性。ceRNA机制发现FAM225A能够竞争性结合miR-590-3p和miR-1275激活FAK/PI3K/Akt信号通路上调靶基因ITGB3介导细胞增殖、迁移和侵袭等过程。lncNRA作为宿主和病毒相互作用的新型调控因子,除了现有的调控作用外,是否还存在多种机制?这还需要人们不断地去探索。
4 展望虽然关于lncRNAs的研究进展迅速,但由于lncRNAs种类多、数量大以及作用机制复杂,目前, 对绝大部分lncRNAs在免疫调控中的功能仍然未知。随着研究工具和技术手段的不断进步,病毒与宿主相互作用的关键lncRNAs不断被鉴定,宿主利用自身lncRNAs靶向调控天然免疫应答抑制病毒感染,病毒利用宿主及自身编码的lncRNAs靶向调控分子表达来干扰宿主天然免疫应答,为病毒在细胞内复制塑造适宜的微环境。研究发现,越来越多的病毒能够编码lncRNAs,但对病毒编码lncRNAs的鉴定还有一定的困难,迄今为止,发现的这种进化保守lncRNA的功能和作用机制可能只是冰山一角。随着对lncRNAs的深入研究,使人们逐渐认识到基因组存在人类知之甚少的“暗物质”,这一领域将会给人们带来持续不断的惊喜,研究结果不仅有助于深入洞悉病毒-宿主间的互作机制,同时,为设计抗病毒药物提供新的靶点和思路。
| [1] | CARNINCI P, KASUKAWA T, KATAYAMA S, et al. The transcriptional landscape of the mammalian genome[J]. Science, 2005, 309(5740): 1559–1563. DOI: 10.1126/science.1112014 |
| [2] | BRANNAN C I, DEES E C, INGRAM R S, et al. The product of the H19 gene may function as an RNA[J]. Mol Cell Biol, 1990, 10(1): 28–36. DOI: 10.1128/MCB.10.1.28 |
| [3] | BROWN C J, BALLABIO A, RUPERT J L, et al. A gene from the region of the human X inactivation centre is expressed exclusively from the inactive X chromosome[J]. Nature, 1991, 349(6304): 38–44. DOI: 10.1038/349038a0 |
| [4] | OKAZAKI Y, FURUNO M, KASUKAWA T, et al. Analysis of the mouse transcriptome based on functional annotation of 60, 770 full-length cDNAs[J]. Nature, 2002, 420(6915): 563–573. DOI: 10.1038/nature01266 |
| [5] | HANGAUER M J, VAUGHN I W, MCMANUS M T. Pervasive transcription of the human genome produces thousands of previously unidentified long intergenic noncoding RNAs[J]. PLoS Genet, 2013, 9(6): e1003569. DOI: 10.1371/journal.pgen.1003569 |
| [6] | DERRIEN T, JOHNSON R, BUSSOTTI G, et al. The GENCODE v7 catalog of human long noncoding RNAs:analysis of their gene structure, evolution, and expression[J]. Genome Res, 2012, 22(9): 1775–1789. DOI: 10.1101/gr.132159.111 |
| [7] | OUYANG J, HU J Y, CHEN J L. lncRNAs regulate the innate immune response to viral infection[J]. Wiley Interdiscip Rev RNA, 2016, 7(1): 129–143. DOI: 10.1002/wrna.1321 |
| [8] | ZHOU Y M, LI M X, XUE Y Q, et al. Interferon-inducible cytoplasmic lncLrrc55-AS promotes antiviral innate responses by strengthening IRF3 phosphorylation[J]. Cell Res, 2019, 29(8): 641–654. DOI: 10.1038/s41422-019-0193-0 |
| [9] | LIN H Y, JIANG M H, LIU L, et al. The long noncoding RNA Lnczc3 h7a promotes a TRIM25-mediated RIG-I antiviral innate immune response[J]. Nat Immunol, 2019, 20(7): 812–823. DOI: 10.1038/s41590-019-0379-0 |
| [10] | AKIRA S, UEMATSU S, TAKEUCHI O. Pathogen recognition and innate immunity[J]. Cell, 2006, 124(4): 783–801. DOI: 10.1016/j.cell.2006.02.015 |
| [11] | MENG X Y, LUO Y Z, ANWAR M N, et al. Long non-coding RNAs:emerging and versatile regulators in host-virus interactions[J]. Front Immunol, 2017, 8: 1663. DOI: 10.3389/fimmu.2017.01663 |
| [12] | COLLINS F S. The case for a US prospective cohort study of genes and environment[J]. Nature, 2004, 429(6990): 475–477. DOI: 10.1038/nature02628 |
| [13] | FITZGERALD K A, CAFFREY D R. Long noncoding RNAs in innate and adaptive immunity[J]. Curr Opin Immunol, 2014, 26: 140–146. DOI: 10.1016/j.coi.2013.12.001 |
| [14] | KONG L, ZHANG Y, YE Z Q, et al. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine[J]. Nucl Acids Res, 2007, 35(S2): W345–W349. |
| [15] | SUN L, LUO H T, BU D C, et al. Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts[J]. Nucl Acids Res, 2013, 41(17): e166. DOI: 10.1093/nar/gkt646 |
| [16] | PONTING C P, OLIVER P L, REIK W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629–641. DOI: 10.1016/j.cell.2009.02.006 |
| [17] | DAHARIYA S, PADDIBHATLA I, KUMAR S, et al. Long non-coding RNA: Classification, biogenesis and functions in blood cells[J]. Mol Immunol, 2019, 112: 82–92. DOI: 10.1016/j.molimm.2019.04.011 |
| [18] | ULITSKY I, KARTEL D P. lincRNAs: genomics, evolution, and mechanisms[J]. Cell, 2013, 154(1): 26–46. DOI: 10.1016/j.cell.2013.06.020 |
| [19] | KRETZ M, SIPRASHVILI Z, CHU C, et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR[J]. Nature, 2013, 493(7431): 231–235. DOI: 10.1038/nature11661 |
| [20] | RANSOHOFF J D, WEI Y N, KHAVARI P A. The functions and unique features of long intergenic non-coding RNA[J]. Nat Rev Mol Cell Biol, 2018, 19: 143–157. DOI: 10.1038/nrm.2017.104 |
| [21] | MELÉ M, MATTIOLI K, MALLARD W, et al. Chromatin environment, transcriptional regulation, and splicing distinguish lincRNAs and mRNAs[J]. Genome Res, 2017, 27(1): 27–37. DOI: 10.1101/gr.214205.116 |
| [22] | WANG K C, CHANG H Y. Molecular mechanisms of long noncoding RNAs[J]. Mol Cell, 2011, 43(6): 904–914. DOI: 10.1016/j.molcel.2011.08.018 |
| [23] | ZHAO X, SU L, HE X Y, et al. Long noncoding RNA CA7-4 promotes autophagy and apoptosis via sponging MIR877-3P and MIR5680 in high glucose-induced vascular endothelial cells[J]. Autophagy, 2020, 16(1): 70–85. DOI: 10.1080/15548627.2019.1598750 |
| [24] | ZHAO J, SUN B K, ERWIN J A, et al. Polycomb proteins targeted by a short repeat RNA to the mouse X-chromosome[J]. Science, 2008, 322(5902): 750–756. DOI: 10.1126/science.1163045 |
| [25] | MARTIANOV I, RAMADASS A, BARROS A S, et al. Repression of the human dihydrofolate reductase gene by a non-coding interfering transcript[J]. Nature, 2007, 445(7128): 666–670. DOI: 10.1038/nature05519 |
| [26] | CESANA M, CACCHIARELLI D, LEGNINI I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2): 358–369. DOI: 10.1016/j.cell.2011.09.028 |
| [27] | HUANG Y, XU Y, LU Y H, et al. lncRNA Gm10451 regulates PTIP to facilitate iPSCs-derived β-like cell differentiation by targeting miR-338-3p as a ceRNA[J]. Biomaterials, 2019, 216: 119266. DOI: 10.1016/j.biomaterials.2019.119266 |
| [28] | XIAN H F, YANG S A, JIN S H, et al. LRRC59 modulates type Ⅰ interferon signaling by restraining the SQSTM1/p62-mediated autophagic degradation of pattern recognition receptor DDX58/RIG-I[J]. Autophagy, 2020, 16(3): 408–418. DOI: 10.1080/15548627.2019.1615303 |
| [29] | PENG X X, GRALINSKI L, ARMOUR C D, et al. Unique signatures of long noncoding RNA expression in response to virus infection and altered innate immune signaling[J]. mBio, 2010, 1(5): e00206–10. |
| [30] | HU J, HU Z L, WANG X Q, et al. Deep sequencing of the mouse lung transcriptome reveals distinct long non-coding RNAs expression associated with the high virulence of H5N1 avian influenza virus in mice[J]. Virulence, 2018, 9(1): 1092–1111. DOI: 10.1080/21505594.2018.1475795 |
| [31] | ZHANG Y N, YU T Q, DING Y G, et al. Analysis of expression profiles of long noncoding RNAs and mRNAs in A549 cells infected with H3N2 swine influenza virus by RNA sequencing[J]. Virol Sin, 2020, 35(2): 171–180. |
| [32] | PAN Q D, ZHAO Z H, LIAO Y, et al. Identification of an interferon-stimulated long noncoding RNA (lncRNA ISR) involved in regulation of influenza A virus replication[J]. Int J Mol Sci, 2019, 20(20): 5118. DOI: 10.3390/ijms20205118 |
| [33] | MAAROUF M, CHEN B, CHEN Y H, et al. Identification of lncRNA-155 encoded by MIR155HG as a novel regulator of innate immunity against influenza A virus infection[J]. Cell Microbiol, 2019, 21(8): e13036. |
| [34] | CHAI W J, LI J, SHANGGUAN Q L, et al. Lnc-ISG20 inhibits influenza a virus replication by enhancing ISG20 expression[J]. J Virol, 2018, 92(16): e00539–18. |
| [35] | MA Q M, LI L Y, TANG Y, et al. Analyses of long non-coding RNAs and mRNA profiling through RNA sequencing of MDBK cells at different stages of bovine viral diarrhea virus infection[J]. Res Vet Sci, 2017, 115: 508–516. DOI: 10.1016/j.rvsc.2017.09.020 |
| [36] | TANG X Y, LAN T, WU R T, et al. Analysis of long non-coding RNAs in neonatal piglets at different stages of porcine deltacoronavirus infection[J]. BMC Vet Res, 2019, 15(1): 111. DOI: 10.1186/s12917-019-1862-4 |
| [37] | YOU Z, ZHANG Q H, LIU C J, et al. Integrated analysis of lncRNA and mRNA repertoires in Marek's disease infected spleens identifies genes relevant to resistance[J]. BMC Genomics, 2019, 20(1): 245. DOI: 10.1186/s12864-019-5625-1 |
| [38] | CHEN J N, ZHANG C Y, ZHANG N, et al. Porcine endemic diarrhea virus infection regulates long noncoding RNA expression[J]. Virology, 2019, 527: 89–97. DOI: 10.1016/j.virol.2018.11.007 |
| [39] | ZHAO P S, LIU S D, ZHONG Z X, et al. Analysis of expression profiles of long noncoding RNAs and mRNAs in brains of mice infected by rabies virus by RNA sequencing[J]. Sci Rep, 2018, 8: 11858. DOI: 10.1038/s41598-018-30359-z |
| [40] | WANG P. The Opening of Pandora's Box:An emerging role of long noncoding RNA in viral infections[J]. Front Immunol, 2018, 9: 3138. |
| [41] | JIANG M H, ZHANG S K, YANG Z H, et al. Self-recognition of an inducible host lncRNA by RIG-I feedback restricts innate immune response[J]. Cell, 2018, 173(4): 906–919. e13. DOI: 10.1016/j.cell.2018.03.064 |
| [42] | WANG L, WEN M Y, CAO X T. Nuclear hnRNPA2B1 initiates and amplifies the innate immune response to DNA viruses[J]. Science, 2019, 365: eaav0758. DOI: 10.1126/science.aav0758 |
| [43] | ZHANG Y B, GUI J F. Molecular regulation of interferon antiviral response in fish[J]. Dev Comp Immunol, 2012, 38(2): 193–202. |
| [44] | OUYANG J, ZHU X M, CHEN Y H, et al. NRAV, a long noncoding RNA, modulates antiviral responses through suppression of interferon-stimulated gene transcription[J]. Cell Host Microbe, 2014, 16(5): 616–626. DOI: 10.1016/j.chom.2014.10.001 |
| [45] | HUANG X W, XU Y G, LIN Q Y, et al. Determination of antiviral action of long non-coding RNA loc107051710 during infectious bursal disease virus infection due to enhancement of interferon production[J]. Virulence, 2020, 11(1): 68–79. DOI: 10.1080/21505594.2019.1707957 |
| [46] | WANG P, XU J F, WANG Y J, et al. An interferon-independent lncRNA promotes viral replication by modulating cellular metabolism[J]. Science, 2017, 358(6366): 1051–1055. DOI: 10.1126/science.aao0409 |
| [47] | XIONG Y L, YUAN J, ZHANG C J, et al. The STAT3-regulated long non-coding RNA Lethe promote the HCV replication[J]. Biomed Pharmacother, 2015, 72: 165–171. DOI: 10.1016/j.biopha.2015.04.019 |
| [48] | ZHOU B, QI F, WU F Y, et al. Endogenous retrovirus-derived long noncoding RNA enhances innate immune responses via derepressing RELA expression[J]. mBio, 2019, 10(4): e00937–19. |
| [49] | MAO X H, SU Z Y, MOOKHTIAR A K. Long non-coding RNA:a versatile regulator of the nuclear factor-κB signalling circuit[J]. Immunology, 2017, 150(4): 379–388. DOI: 10.1111/imm.12698 |
| [50] | WU H W, LIU J F, LI W, et al. LncRNA-HOTAIR promotes TNF-α production in cardiomyocytes of LPS-induced sepsis mice by activating NF-κB pathway[J]. Biochem Biophys Res Commun, 2016, 471(1): 240–246. |
| [51] | JIN C Y, JIA L F, HUANG Y P, et al. Inhibition of lncRNA MIR31HG promotes osteogenic differentiation of human adipose-derived stem cells[J]. Stem Cells, 2016, 34(11): 2707–2720. DOI: 10.1002/stem.2439 |
| [52] | KE S, LI R C, MENG F K, et al. NKILA inhibits NF-κB signaling and suppresses tumor metastasis[J]. Aging, 2018, 10(1): 56–71. DOI: 10.18632/aging.101359 |
| [53] | CHEN L L, ZHOU Y, LI H J. LncRNA, miRNA and lncRNA-miRNA interaction in viral infection[J]. Virus Res, 2018, 257: 25–32. DOI: 10.1016/j.virusres.2018.08.018 |
| [54] | SALMENA L, POLISENO L, TAY Y, et al. A ceRNA hypothesis:the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353–358. DOI: 10.1016/j.cell.2011.07.014 |
| [55] | CAZALLA D, YARIO T, STEITZ J A, et al. Down-regulation of a host microRNA by a Herpesvirus saimiri noncoding RNA[J]. Science, 2010, 328(5985): 1563–1566. DOI: 10.1126/science.1187197 |
| [56] | CHEN W B, LIN C H, GONG L, et al. Comprehensive analysis of the mRNA-lncRNA Co-expression profile and ceRNA networks patterns in chronic hepatitis B[J]. Curr Genomics, 2019, 20(4): 231–245. DOI: 10.2174/1389202920666190820122126 |
| [57] | WANG Z Y, GUO Z D, LI J M, et al. Genome-wide search for competing endogenous RNAs responsible for the effects induced by ebola virus replication and transcription using a trVLP system[J]. Front Cell Infect Microbiol, 2017, 7: 479. DOI: 10.3389/fcimb.2017.00479 |
| [58] | DAI M M, FENG M, XIE T T, et al. Long non-coding RNA and MicroRNA profiling provides comprehensive insight into non-coding RNA involved host immune responses in ALV-J-infected chicken primary macrophage[J]. Dev Comp Immunol, 2019, 100: 103414. DOI: 10.1016/j.dci.2019.103414 |
| [59] | WANG L L, YOU Z, WANG M Y, et al. Genome-wide analysis of circular RNAs involved in Marek's disease tumourigenesis in chickens[J]. RNA Biol, 2020, 17(4): 517–527. DOI: 10.1080/15476286.2020.1713538 |
| [60] | ZHENG Z Q, LI Z X, ZHOU G Q, et al. Long noncoding RNA FAM225A promotes nasopharyngeal carcinoma tumorigenesis and metastasis by acting as ceRNA to sponge miR-590-3p/miR-1275 and upregulate ITGB3[J]. Cancer Res, 2019, 79(18): 4612–4626. DOI: 10.1158/0008-5472.CAN-19-0799 |



