葡萄糖是动物机体的主要供能物质,是维持细胞能量代谢和生命活动的重要原料。葡萄糖转运蛋白(glucose transporters, GLUT)介导的葡萄糖跨膜运输是葡萄糖代谢和血糖调节中的重要步骤,是细胞能量代谢不可或缺的膜蛋白[1]。
在哺乳动物体内,GLUTs家族有14个成员,由葡萄糖转运体SLC2A(solute linked carrier family 2, subfamily A)基因编码的约500个氨基酸残基组成[2]。基于其氨基酸序列的相似性可将GLUTs分为3类:Ⅰ类有GLUT-1、2、3、4和14;Ⅱ类有GLUT-5、7、9、11;Ⅲ类有GLUT-6、8、10、12、13或H+/肌醇转运体(H+/myoinositol transporter, HMIT)[3-5]。因不同的GLUT基因对糖类物质具有不同的转运动力学和调节特性,所以它们的生理功能和组织分布大不相同[6]。GLUT13又称为HMIT,属于Ⅲ类GLUT家族,大鼠HMIT主要储存在细胞质小泡中,并在细胞内钙水平升高或pH降低等刺激下被转运到膜上[7-8]。HMIT是一种非典型的SLC2A转运蛋白,它是该家族中唯一的H+偶联转运蛋白,并且是唯一有文献报道的肌醇转运蛋白[9-10]。目前,在大鼠大脑和PC12细胞中的研究表明,HMIT主要参与肌醇跨膜转运,与突触囊泡的形成、胞吐及细胞膜的更新等密切相关[11-12]。肌醇的代谢异常与糖尿病患者的胰岛素抵抗和糖尿病心血管并发症有关,有研究报道,肌醇代谢异常可能与葡萄糖稳态有关[13]。
随着对GLUT家族的深入研究,其各个成员的生理功能以及组织分布陆续被揭示出来,目前,国内外学者关于HMIT基因的研究主要集中在神经调节和信号通路调节方面[14],包括肌醇信号通路[8]、钙信号通路[15]、mTOR信号通路[16]、NRF2信号通路[17]等,研究对象仅限于鼠和兔子,HMIT基因在鸡上的研究鲜有报道,Merkin等[18]利用转录组测序技术在鸡肾内检测到HMIT的表达。本研究利用RT-PCR等技术对鸡HMIT基因的全长CDS区进行克隆,并对其编码蛋白的结构和功能进行分析,采用实时荧光定量PCR技术对鸡各组织表达谱及特定组织的表达调控模式进行研究,从而为鸡HMIT基因生物学功能的深入研究提供一定的理论依据。
1 材料与方法 1.1 样品采集试验选用同批孵化、相同条件下饲养的24日龄爱拔益加肉公鸡(AA肉鸡)由河南省开封市兴达禽业有限责任公司提供。随机挑选3只屠宰,采集大脑、肝、腹脂、腿肌、心、肾、空肠和回肠等8个组织用于HMIT基因克隆和组织表达分析。另随机挑选40只鸡分为试验组和对照组,试验组用胰岛素处理记为INS组,对照组用磷酸盐缓冲液(PBS)处理记为PBS组,禁食12 h后,对INS组进行腹部皮下注射5 IU·kg-1的胰岛素,PBS组注射同等剂量的PBS溶液,经外源注射后采用罗氏血糖仪分别测定在0、60、120、240 min(n=5)时的血糖浓度。试验期间所有鸡保持自由饮水,试验所用胰岛素用PBS进行稀释(胰岛素:PBS=1:9),所用胰岛素为诺和锐短效胰岛素。INS组和PBS组注射120、240 min后进行屠宰,采集大脑和肾组织,每个组织取部分样品于液氮中速冻后于-80 ℃冰箱保存。
1.2 主要试剂DL2000DNA Marker、2×Taq PCR MaserMix、PBS及其他常规试剂均购自天根生化科技(北京)有限公司,PrimeScriptTM RT Reagent Kit with gDNA Eraser(Perfect Real-time)反转录试剂盒购自TaKaRa公司,诺和锐短效胰岛素由诺和锐德(中国)制药有限公司生产,胰岛素的规格为3 mL= 300 IU(国际单位)。
1.3 RNA提取与cDNA合成采用Trizol法提取AA肉鸡各组织的总RNA,利用1.0%琼脂糖凝胶电泳检测RNA质量,分光光度计测定RNA浓度及OD260 nm/OD280 nm值。参照PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)反转录试剂盒说明书将RNA反转录为cDNA。反转录共包括两步反应:第一步是去除基因组DNA反应,即取1 μg总RNA配制混合体系去除gDNA;第二步是反转录反应,即在步骤一所得混合液中加入含有随机引物的混合体系。将反转录得到的cDNA于-20 ℃保存备用。
1.4 鸡HMIT基因全编码序列的扩增根据GenBank预测的鸡HMIT序列(登录号:XM_001232939.5),采用Primer Premier5.0软件设计引物(G1~G4)进行基因的全编码序列扩增(表 1),引物由生工生物工程(上海)股份有限公司合成。以反转录的各组织混合cDNA为模板进行PCR扩增。PCR反应体系20 μL:2×Taq PCR MasterMix 10 μL, 上、下游引物各0.5 μL, cDNA 2 μL, ddH2O 7 μL。反应条件:94 ℃预变性5 min;94 ℃变性30 s,68 ℃退火30 s,72 ℃延伸70 s,共35个循环;72 ℃延伸5 min。PCR产物进行2.0%琼脂糖凝胶电泳检测,对鉴定为单一条带的PCR产物送至生工生物工程(上海)股份有限公司进行直接PCR测序。
|
|
表 1 HMIT引物信息 Table 1 Primer information of HMIT |
测序结果采用DNAMAN软件进行序列拼接比对分析,运用NCBI上的ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)程序对鸡HMIT基因的开放阅读框进行分析,使用Protparam(https://web.expasy.org/protparam/)在线软件对鸡HMIT的理化性质进行预测;利用UCSC在线数据库的Blat查找鸡HMIT在基因组位置和外显子的分布情况(http://genome.ucsc.edu/cgi-bin/hgBlat?command=start);使用Genomicus(http://www.genomicus.biologie.ens.fr)在线软件对鸡HMIT基因进行共线性分析;使用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL (https://www.swissmodel.expasy.org/)分别预测HMIT蛋白质的二级结构和三级结构;使用TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构预测;利用PSORTII Prediction在线网站(https:1psort.hgc.jp/form2.html)进行蛋白质亚细胞定位预测;运用NCBI的Conserved Domain Search Service在线分析HMIT蛋白的保守结构域(https://www..ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)[19-20]。
1.6 鸡HMIT基因表达分析以β-actin为内参基因,采用荧光定量PCR检测鸡HMIT在不同组织(大脑、肝、腹脂、腿肌、心、肾、空肠和回肠)以及外源胰岛素处理后在大脑和肾中的表达量,每个样品3个重复。PCR反应体系为10 μL:SYBR Premix ExTaqTM(2×) 5 μL,G5上、下游引物(表 1)各0.25 μL,组织cDNA模板1 μL, ddH2O 3.5 μL。PCR反应程序:95 ℃ 30 s;95 ℃ 5s,60 ℃ 30 s,共39个循环。运用2-△△Ct法分析所得数据,利用SPSS 19. 0软件进行显著性检验分析,以P < 0.05为差异显著性判断标准。
1.7 鸡血糖的检测与分析鸡经过12 h禁食后,对INS组进行腹部皮下注射5 IU·kg-1的胰岛素,PBS组注射同等剂量的PBS溶液,INS组和PBS组均分别于注射后0、60、120、240 min采集翅下血,用罗氏血糖仪进行血糖的实时检测,所测血糖数据分析方法同“1.6”。
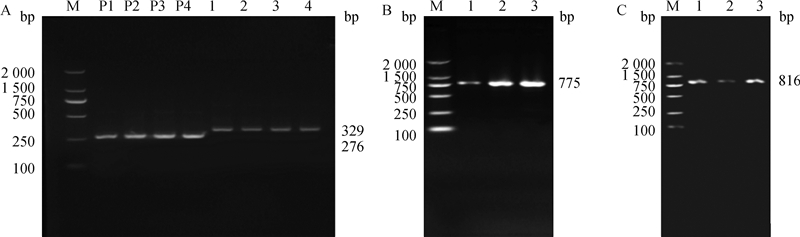
2 结果 2.1 鸡HMIT基因全编码序列的扩增GenBank上预测的鸡HMIT基因(XM_001232939.5)序列,编码区(CDS区)为229~2 169 bp,长度为1 941 bp。采用G1~G4引物(表 1)对HMIT基因CDS序列进行分段扩增,PCR扩增产物通过2.0%琼脂糖凝胶电泳检测,结果显示,扩增片段无杂带,条带单一明亮(图 1)。经多次的引物优化和尝试,采用G1和G2引物获得了位于预测的鸡HMIT基因CDS区的5′-端扩增产物。G1和G2引物具有相同的正向引物,而反向引物位于不同区间,G1预测扩增区间为53~904 bp,G2预测扩增区间为53~957 bp,G1和G2引物的扩增长度预测分别为852和905 bp,将条带单一明亮的PCR产物直接测序获得了G1和G2引物的扩增序列。结果表明,G1引物的扩增片段为276 bp(图 1A,P1~P4),与预期HMIT基因序列相比,缺少其86~661 bp区间;G2扩增片段为329 bp(图 1A,1~4),与预期序列相比,同样缺少86~661 bp。G3引物的预期扩增长度为775 bp,G4引物的预期扩增长度为816 bp,电泳结果显示,G3和G4引物均得到了预期的扩增片段长度(图 1B和图 1C),G3与G4引物的PCR产物直接测序表明得到了与XM_001232939.5相一致的序列。整合G1~G4引物扩增产物的测序结果,获得了1 694 bp的鸡HMIT基因序列。相关序列已经提交到NCBI数据库,序列登陆号为MN708211。
|
M. DNA相对分子质量标准; A.G1和G2引物的PCR扩增产物电泳图:P1~P4. G1的PCR扩增产物;1~4.G2引物的PCR扩增产物。B. G3引物的PCR扩增产物电泳图。C. G4引物的PCR扩增产物电泳图 M. DL2000 DNA marker; A. Electrophoretogram of PCR products with G1 and G2 primers: P1-P4.PCR amplification products of G1 primer; 1-4.PCR amplification products of G2 primer. B.Electrophoretogram of PCR products of G3 primer. C. Electrophoretogram of PCR products of G4 primer 图 1 鸡HMIT基因CDS序列的PCR扩增电泳图 Fig. 1 PCR amplification electrophoresis map for CDS sequence of chicken HMIT gene |
采用NCBI上的ORF Finder对本研究克隆得到的鸡HMIT序列分析发现,鸡HMIT含有一个长161 bp的5′UTR,一个长1 380 bp的ORF(open reading frame),一个长153 bp的3′UTR,预测编码一个由459个氨基酸组成的蛋白。利用UCSC在线数据库的Blat查找HMIT在基因组位置和外显子的分布情况,结果发现,鸡HMIT位于1号染色体上,CDS区跨越的基因组大小为102 545 bp,共包含9个外显子,长度在109~227 bp之间,且剪接供体和受体都符合GT-AG法则。将本研究克隆得到的鸡HMIT编码序列结构与已知的人HMIT (NM_052885.4)基因结构相比(表 2),克隆的鸡HMIT比人HMIT缺少第一外显子和第二外显子的部分序列。
|
|
表 2 鸡与人HMIT基因CDS区基因组结构比较 Table 2 Genomic structural comparison of HMIT CDS between chicken and human |
进行鸡HMIT基因CDS区及其预测的氨基酸序列同源性分析,结果发现,鸡HMIT基因的CDS区核苷酸序列与其它禽类一致性最高,与日本鹌鹑和火鸡预测序列的一致性高达98.0%和97.3%,其次是人(82.3%),与琥珀鱼的相似性最低(71.2%)。鸡HMIT基因编码氨基酸序列与日本鹌鹑和火鸡的一致性最高,为99.6%,与琥珀鱼的一致性最低,为75.2%(表 3,图 2)。说明HMIT基因序列在物种间高度保守。

|
鹅HMIT基因GenBank登录号:XM_013190750.1;斑胸草雀HMIT基因GenBank登录号:XM_002193899.3;人-L为人HMIT基因的长转录本(NM_052885.4);人-S为人HMIT基因的短转录本(XM_017018765.1);大鼠、小鼠、牛、绿头鸭和日本鹌鹑的HMIT基因参考表 3中GenBank号 Goose HMIT gene GenBank ID: XM_013190750.1; Zebra finch HMIT gene GenBank ID: XM_002193899.3; Human-L is the long transcript of human HMIT gene(NM_052885.4); Human-S is the short transcript of human HMIT gene(XM_017018765.1); The HMIT genes of rat, mice, cattle, anas platyrhynchos and coturnix japonica refer to the GenBank ID in Table 3 图 2 鸡与其他物种HMIT氨基酸序列比对 Fig. 2 The alignment of amino acid sequences of HMIT between AA broiler and other species |
|
|
表 3 鸡HMIT基因CDS区和编码氨基酸序列与其他物种间的一致性分析 Table 3 Identity analysis of HMIT CDS and encoded amino acids sequence between the chicken and other species |
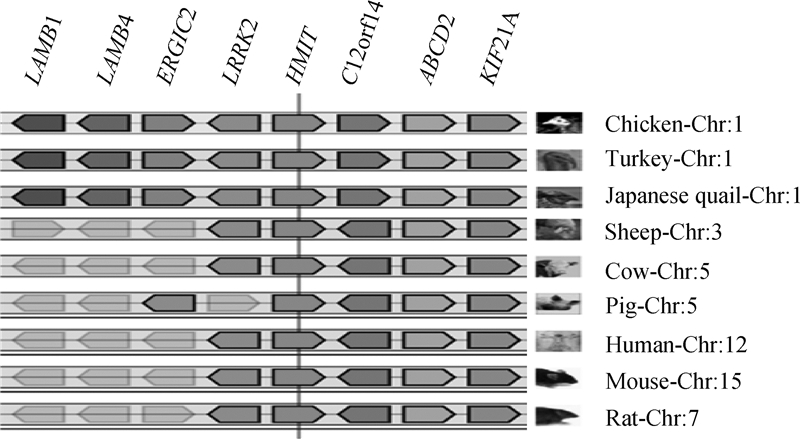
进一步分析显示(图 3),包含鸡HMIT-C12orf14-ABCD2-KIF21A基因的1号染色体区段与多种哺乳动物相应染色体区域(含有已被鉴定的HMIT基因)均具有保守的共线性,说明本研究获得序列是哺乳动物HMIT在鸡上的同源基因。
|
物种及染色体列在右侧,基因符号列在顶部。同一行中的不同方框呈现的是不同的基因。不同基因盒子指向的方向代表它们的转录方向 Species and chromosomes are listed on the right, gene symbols are listed at the top. Different boxes in the same line show different genes. Different gene boxes point to the direction in which they are transcripted 图 3 HMIT基因组区域的保守共线性 Fig. 3 Conservative collinearity of the genomic region of HMIT gene |
使用在线软件ProtParam对HMIT蛋白进行预测,结果显示,鸡HMIT蛋白编码459个氨基酸,其中异亮氨酸(Leu)含量最高。鸡HMIT蛋白分子式为C2 329H3 577N589O655S27,分子量为51.17 ku,半衰期为30 h,脂肪系数为92.38,理论等电点(pI)为6.04,不稳定系数为44.23,说明该蛋白质为酸性、不稳定蛋白质。运用ProtScale在线软件对鸡HMIT蛋白进行疏水性分析发现,在HMIT蛋白的328位点存在最大疏水指数(2.911),在111位点存在最小疏水指数(-2.700),亲水性平均值(GRAVY)为0.166,表明鸡HMIT属于亲水性蛋白。
2.4 鸡HMIT蛋白的结构预测分析2.4.1 二级和三级结构预测分析 采用SOPMA软件对AA肉鸡HMIT基因编码蛋白进行二级结构预测,结果显示,鸡HMIT基因编码蛋白的二级结构由无规则卷曲、α-螺旋、延伸链和β-转角构成,所占比例分别为39.87%、38.12%、16.56%和5.45%(图 4A)。使用SWISS-MODEL在线软件对鸡HMIT基因编码蛋白进行三级结构预测,结果发现,鸡HMIT蛋白主要由α-螺旋和无规则卷曲组成(图 4B),与二级结构预测结果一致。

|
A.鸡HMIT蛋白二级结构:h. α-螺旋;t. β-转角;c.无规则卷曲;e.延伸链。B.鸡HMIT蛋白三级结构 A. Secondary structure of chicken HMIT protein: h. Alpha helix; t. Beta turn; c. Random coil; e. Extended strand. B. Tertiary structure of chicken HMIT protein 图 4 鸡HMIT蛋白二级结构和三级结构预测 Fig. 4 Prediction of secondary and tertiary structure of chicken HMIT protein |
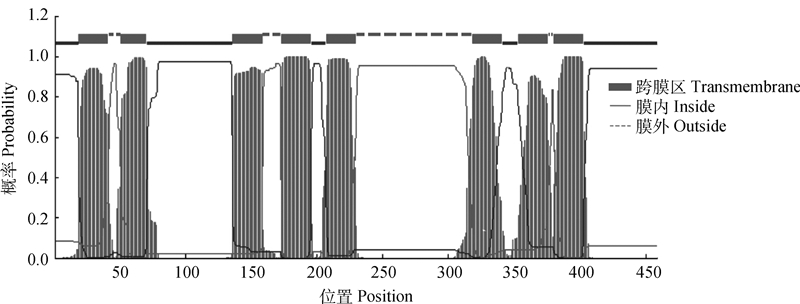
2.4.2 跨膜结构预测及蛋白亚细胞定位预测 利用在线跨膜区结构预测软件TMHMM Server v.2.0进行HMIT蛋白质序列跨膜区分析(图 5)。表明克隆的鸡HMIT蛋白含有8个跨膜结构域,分别在第19~41、51~70、136~158、173~195、207~229、318~340、353~375和380~402位氨基酸处;第1~18、71~135、196~206、341~352和403~459位氨基酸位于胞内区;第42~50、159~172、230~317和376~379位氨基酸位于胞外区。利用在线软件PSORT Ⅱ Prediction预测鸡HMIT蛋白亚细胞定位,结果表明,其定位于质膜、内质网、液泡、线粒体和高尔基体的比例分别为52.3%、21.7%、17.4%、4.3%和4.3%。
|
图 5 鸡HMIT蛋白跨膜区域分析 Fig. 5 The transmembrane region analysis of HMIT protein in chicken |
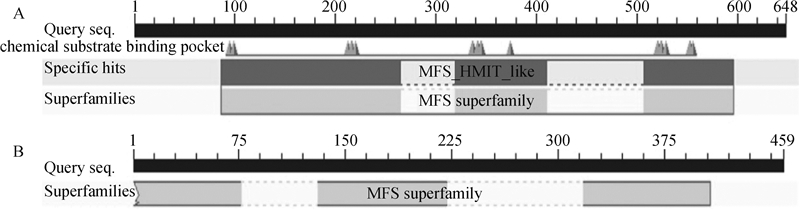
2.4.3 保守结构域预测分析 运用NCBI中的Conserved Domain Search Service(CD Search)在线分析人和鸡HMIT蛋白的保守结构域(图 6)。结果发现,人与鸡HMIT蛋白存在一个共同的保守功能结构域,为MFS结构域(图 6)。与人HMIT蛋白相比(图 6A),克隆的鸡HMIT缺少一个MFS_HMIT_like结合位点(图 6B)。此外,分析发现,鸡HMIT蛋白与第三类葡萄糖转运蛋白家族GLUT6、8、10和12具有相同的保守结构域。这也更进一步证实本研究所得序列为鸡HMIT基因。
|
A.人HMIT(NP_443117.3)蛋白结构域分析;B.鸡HMIT蛋白结构域分析 A. Domain analysis of human HMIT protein(NP_443117.3); B. Domain analysis of chicken HMIT protein 图 6 人和鸡HMIT蛋白结构域分析 Fig. 6 Domain analysis of HMIT protein in human and chicken |
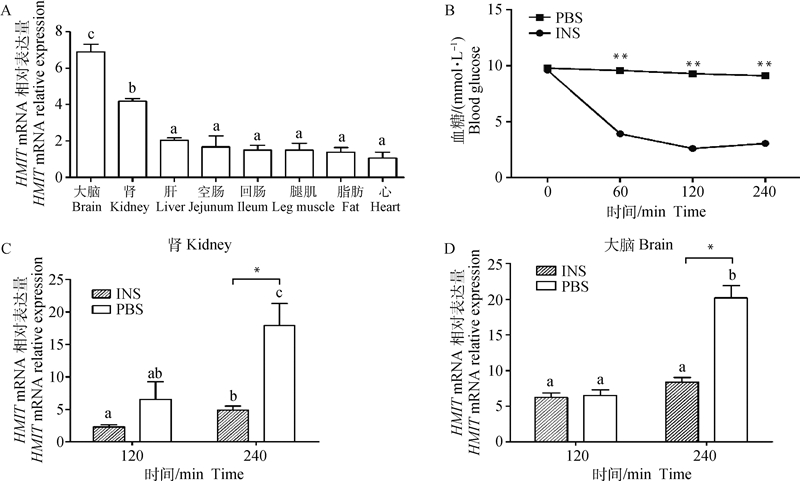
利用实时荧光定量PCR检测鸡HMIT基因在AA肉鸡不同组织中的mRNA相对表达量(图 7A),结果表明,HMIT基因在AA肉鸡大脑、肾、肝、空肠、腿肌、回肠、腹脂和心中均有表达,其中,在大脑和肾中表达量相对较高,且显著高于其他组织(P < 0.05),心和脂肪中表达相对较弱。
|
不同字母表示差异显著(P < 0.05);相同字母表示差异不显著(P>0.05)。同一时间点不同处理间:*表示差异显著(P < 0.05),**表示差异极显著(P < 0.01)。INS.胰岛素处理组;PBS. PBS对照组 Different letters indicate significant difference(P < 0.05), while the same letter indicate no significant difference(P>0.05). Between different treatment groups at the same time: * indicate the difference was significant(P < 0.05), ** indicate the difference was extremely significant (P < 0.01). INS. Insulin treatment group; PBS. PBS control group 图 7 鸡HMIT基因的表达 Fig. 7 Expression of chicken HMIT gene |
用罗氏血糖仪检测胰岛素和PBS处理后不同时间点鸡的血糖浓度,结果显示(图 7B),注射60、120和240 min后,INS组血糖浓度极显著低于PBS组(P < 0.01),且血糖浓度在120 min达到最低。组织表达结果显示,鸡HMIT在大脑和肾表达量最高,血糖检测结果显示,胰岛素处理后120和240 min血糖浓度较低。通过荧光定量PCR检测HMIT基因在肾和大脑中外源胰岛素处理120和240 min后的表达情况,结果显示,在肾的INS组中(图 7C),HMIT在240 min的表达量显著高于120 min(P < 0.05),在240 min时,肾中HMIT在INS组的表达量显著低于PBS组(P < 0.05)。在大脑的INS组中(图 7D),HMIT在240 min的表达量同样高于120 min,但未达到显著水平,在240 min时,大脑中HMIT在INS组的表达量显著低于PBS组(P < 0.05),表现出与肾中同样的表达规律。
3 讨论NCBI数据库的登录数据表明,HMIT基因在人上可能存在多个转录本,其目前已知的转录本(NM_052885.4)预测编码由648个氨基酸组成的蛋白,另外还发现一个短的转录本(XM_017018765.1)预测仅编码由459个氨基酸组成的蛋白。NCBI上预测的鸡HMIT序列(XM_001232939.5)类似于人类已知的长转录本,存在12个跨膜结构域[11, 21],包含一个MFS_HMIT_like结合位点和一个MFS超家族(major facilitator superfamily)结构域。本研究获得的鸡HMIT基因(NM_052885.4)相似于预测的人HMIT的短转录本,预测编码459个氨基酸,包含8个跨膜结构,与目前已知的人HMIT的蛋白结构相比正好缺失了一个MFS_HMIT_like结合位点,显示本研究获得了鸡HMIT的一个短转录本。本研究采用G1和G2引物的扩增产物直接进行测序均得到了短的转录本序列,也表明该短的转录本是鸡HMIT基因的优势转录产物,甚至可能是唯一的转录产物。考虑到保守结构域的潜在重要功能,MFS_HMIT_like结合位点在鸡HMIT蛋白的缺失可能导致其功能的改变,后期可进一步开展鸡HMIT基因的功能研究。此外,HMIT短转录本的表达是否是在禽类乃至哺乳动物具有保守性,鸡HMIT是否存在类似于人HMIT的长转录本,也值得进一步研究。
在哺乳动物中的研究表明,HMIT是一种跨膜蛋白。大鼠HMIT通常在神经细胞内和大脑中表达[22],Fagerberg等[23]利用转录组学和蛋白组学分析技术研究发现,HMIT在人类大脑、心、肾等27个组织均有表达,其中在大脑和肾组织中表达最高。本研究发现,HMIT基因在鸡大脑和肾中表达量最高,在心中表达较低,这与Barbosa-Morais等[24]报道HMIT基因在鸡大脑和肾中表达最高结果一致。HMIT主要参与肌醇跨膜转运,可激活肌醇摄入神经元和星形胶质细胞[25]。肌醇是一种天然的环状醇,主要在大脑和肾中合成。肾是调节渗透的重要器官和肌醇生成的重要场所[26-27]。因此,外源胰岛素注射后,选择大脑和肾用于后续研究分析。有研究表明,肌醇代谢与血糖调节有关,高血糖可诱导肌醇的消耗,通常糖尿病模型动物中发现细胞内肌醇的耗竭[28-30]。本研究发现,肉鸡外源胰岛素处理240 min后,肉鸡血糖水平显著低于对照组,而此时肉鸡大脑和肾中HMIT的水平也显著低于对照组,显示作为GLUT成员,胰岛素能够在一定程度上通过抑制肉鸡优势组织中HMIT的表达,进而影响肉鸡的血糖水平。
GLUTs家族属于MFS超家族成员[31-32]。鸡HMIT蛋白也被预测存在一个MFS保守结构域。MFS转运蛋白在许多生理过程中起着重要作用。比如,MFS蛋白可通过促进扩散跨膜转运各种离子和氨基酸[33-35]。研究表明,MFS转运蛋白的功能、表达或定位的变化与各种人类疾病相关[36]。例如,HMIT的异常表达与精神疾病、糖尿病、肿瘤、阿尔茨海默氏病[8, 37-39]等有一定的关系。HMIT通过转运肌醇可以改善动物和人类的葡萄糖耐受性,与胰岛素抵抗、2型糖尿病等存在一定关系[40]。研究表明,HMIT可作为口腔鳞状细胞等各种肿瘤干细胞的潜在标志物,同时,也可作为急性髓性白血病的潜在生物标志物[41]。目前,HMIT被鉴定为在脑中高度表达的新型γ分泌相关蛋白,因此,可作为治疗阿尔茨海默氏病的候选药物靶标。
4 结论本研究克隆了鸡HMIT基因的全编码序列(GenBank登录号:MN708211),研究表明,鸡HMIT的ORF全长1 380 bp,共有9个外显子,编码459个氨基酸。生物信息学分析表明,HMIT基因在物种间高度保守,鸡HMIT蛋白为酸性、不稳定、亲水性蛋白质,包含8个跨膜结构,存在1个MFS保守功能结构域。荧光定量PCR显示,鸡HMIT在肾、肝、心、腹脂等组织中均有表达,其中在大脑和肾组织中表达量最高,注射外源胰岛素可影响HMIT基因在大脑和肾中的表达,本研究为深入探索鸡HMIT基因的生理功能和调控机制奠定了基础。
| [1] | WANG J Q, YU J C, ZHANG Y Q, et al. Glucose transporter inhibitor-conjugated insulin mitigates hypoglycemia[J]. Proc Natl Acad Sci U S A, 2019, 116(22): 10744–10748. DOI: 10.1073/pnas.1901967116 |
| [2] | BYERS M S, HOWARD C, WANG X F. Avian and mammalian facilitative glucose transporters[J]. Microarrays, 2017, 6(2): 7. DOI: 10.3390/microarrays6020007 |
| [3] | MUECKLER M, THORENS B. The SLC2(GLUT) family of membrane transporters[J]. Mol Aspects Med, 2013, 34(2-3): 121–138. DOI: 10.1016/j.mam.2012.07.001 |
| [4] | DENG D, YAN N. GLUT, SGLT, and SWEET:structural and mechanistic investigations of the glucose transporters[J]. Protein Sci, 2016, 25(3): 546–558. DOI: 10.1002/pro.2858 |
| [5] | YAN N. A glimpse of membrane transport through structures-advances in the structural biology of the GLUT glucose transporters[J]. J Mol Biol, 2017, 429(17): 2710–2725. DOI: 10.1016/j.jmb.2017.07.009 |
| [6] | THORENS B, MUECKLER M. Glucose transporters in the 21st Century[J]. Am J Physiol Endocrinol Metab, 2010, 298(2): E141–145. DOI: 10.1152/ajpendo.00712.2009 |
| [7] | ULDRY M, STEINER P, ZURICH M G, et al. Regulated exocytosis of an H+/myo-inositol symporter at synapses and growth cones[J]. EMBO J, 2004, 23(3): 531–540. DOI: 10.1038/sj.emboj.7600072 |
| [8] | FARIAS V X, UCHOA P N, AQUINO C P, et al. Expression of myo-inositol cotransporters in the sciatic nerve and dorsal root ganglia in experimental diabetes[J]. Braz J Med Biol Res, 2019, 52(6): e8589. DOI: 10.1590/1414-431x20198589 |
| [9] | FERREIRA R S, PONS J L, LABESSE G. Insights into substrate and inhibitor selectivity among human GLUT transporters through comparative modeling and molecular docking[J]. ACS Omega, 2019, 4(3): 4748–4760. DOI: 10.1021/acsomega.8b03447 |
| [10] | EDAMATSU M, KONDO Y, ANDO M. Differential localizations of the myo-inositol transporters HMIT and SMIT1 in the cochlear stria vascularis[J]. Neurosci Lett, 2018, 674: 88–93. DOI: 10.1016/j.neulet.2018.03.028 |
| [11] | ULDRY M, IBBERSON M, HORISBERGER J D, et al. Identification of a mammalian H+-myo-inositol symporter expressed predominantly in the brain[J]. EMBO J, 2001, 20(16): 4467–4477. DOI: 10.1093/emboj/20.16.4467 |
| [12] | ULDRY M, THORENS B. The SLC2 family of facilitated hexose and polyol transporters[J]. Pflügers Arch, 2004, 447(5): 480–489. DOI: 10.1007/s00424-003-1085-0 |
| [13] | CROZE M L, SOULAGE C O. Potential role and therapeutic interests of myo-inositol in metabolic diseases[J]. Biochimie, 2013, 95(10): 1811–1827. DOI: 10.1016/j.biochi.2013.05.011 |
| [14] | DI DANIEL E, KEW J N, MAYCOX P R. Investigation of the H+-myo-inositol transporter (HMIT) as a neuronal regulator of phosphoinositide signalling[J]. Biochem Soc Trans, 2009, 37(5): 1139–1143. DOI: 10.1042/BST0371139 |
| [15] | LYU M, ZHOU J, CHEN H, et al. The genetic variants in calcium signaling related genes influence anti-tuberculosis drug induced liver injury:a prospective study[J]. Medicine (Baltimore), 2019, 98(44): e17821. DOI: 10.1097/MD.0000000000017821 |
| [16] | DONG J, WEN L, GUO X Y, et al. The increased expression of glucose transporters in human full-term placentas from assisted reproductive technology without changes of mTOR signaling[J]. Placenta, 2019, 86: 4–10. DOI: 10.1016/j.placenta.2019.08.087 |
| [17] | YANG W J, LIU H L, DUAN B S, et al. Three novel genetic variants in NRF2 signaling pathway genes are associated with pancreatic cancer risk[J]. Cancer Sci, 2019, 110(6): 2022–2032. |
| [18] | MERKIN J, RUSSELL C, CHEN P, et al. Evolutionary dynamics of gene and isoform regulation in Mammalian tissues[J]. Science, 2012, 338(6114): 1593–1599. DOI: 10.1126/science.1228186 |
| [19] |
刘宗立, 陈涛, 杨丹丹, 等. 猪Nrf2基因克隆、生物信息学分析及启动子区转录活性分析[J]. 畜牧兽医学报, 2019, 50(7): 1328–1339.
LIU Z L, CHEN T, YANG D D, et al. Molecular cloning, bioinformatics analysis and transcriptional activity of promoter of nuclear factor erythroid 2-related factor (Nrf2) gene in pig[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(7): 1328–1339. (in Chinese) |
| [20] |
郭晓红, 李萌, 高鹏飞, 等. 猪MYNN基因可变剪接体的克隆及表达特性研究[J]. 畜牧兽医学报, 2018, 49(3): 477–487.
GUO X H, LI M, GAO P F, et al. Molecular cloning on alternative splice variants of pig MYNN gene and their expression patterns[J]. Acta Veterinaria et Zootechnica Sinica, 2018, 49(3): 477–487. (in Chinese) |
| [21] | SCHNEIDER S. Inositol transport proteins[J]. FEBS Lett, 2015, 589(10): 1049–1058. DOI: 10.1016/j.febslet.2015.03.012 |
| [22] | AUGUSTIN R. The protein family of glucose transport facilitators:it's not only about glucose after all[J]. IUBMB Life, 2010, 62(5): 315–333. |
| [23] | FAGERBERG L, HALLSTRÖM B M, OKSVOLD P, et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics[J]. Mol Cell Proteomics, 2014, 13(2): 397–406. DOI: 10.1074/mcp.M113.035600 |
| [24] | BARBOSA-MORAIS N L, IRIMIA M, PAN Q, et al. The evolutionary landscape of alternative splicing in vertebrate species[J]. Science, 2012, 338(6114): 1587–1593. DOI: 10.1126/science.1230612 |
| [25] | INOUE M, HUR J Y, KIHARA T, et al. Human brain proteins showing neuron-specific interactions with γ-secretase[J]. FEBS J, 2015, 282(14): 2587–2599. DOI: 10.1111/febs.13303 |
| [26] | KAGE-NAKADAI E, UEHARA T, MITANI S. H+/myo-inositol transporter genes, hmit-1.1 and hmit-1.2, have roles in the osmoprotective response in Caenorhabditis elegans[J]. Biochem Biophys Res Commun, 2011, 410(3): 471–477. DOI: 10.1016/j.bbrc.2011.06.001 |
| [27] | MACFARLANE P M, DI FIORE J M. Myo-inositol effects on the developing respiratory neural control system[J]. Adv Exp Med Biol, 2018, 1071: 159–166. |
| [28] | SUN T H, HEIMARK D B, NGUYGEN T, et al. Both myo-inositol to chiro-inositol epimerase activities and chiro-inositol to myo-inositol ratios are decreased in tissues of GK type 2 diabetic rats compared to Wistar controls[J]. Biochem Biophys Res Commun, 2002, 293(3): 1092–1098. |
| [29] | CROZE M L, VELLA R E, PILLON N J, et al. Chronic treatment with myo-inositol reduces white adipose tissue accretion and improves insulin sensitivity in female mice[J]. J Nutr Biochem, 2013, 24(2): 457–466. DOI: 10.1016/j.jnutbio.2012.01.008 |
| [30] | VILLALBA H, SHAH K, ALBEKAIRI T H, et al. Potential role of myo-inositol to improve ischemic stroke outcome in diabetic mouse[J]. Brain Res, 2018, 1699: 166–176. DOI: 10.1016/j.brainres.2018.08.028 |
| [31] | IANCU C V, ZAMOON J, WOO S B, et al. Crystal structure of a glucose/H+ symporter and its mechanism of action[J]. Proc Natl Acad Sci U S A, 2013, 110(44): 17862–17867. DOI: 10.1073/pnas.1311485110 |
| [32] | BAZZONE A, ZABADNE A J, SALISOWSKI A, et al. A loose relationship:incomplete H+/sugar coupling in the MFS sugar transporter GlcP[J]. Biophys J, 2017, 113(12): 2736–2749. DOI: 10.1016/j.bpj.2017.09.038 |
| [33] | YAN N. Structural biology of the major facilitator superfamily transporters[J]. Annu Rev Biophys, 2015, 44(1): 257–283. DOI: 10.1146/annurev-biophys-060414-033901 |
| [34] | DENG D, XU C, SUN P C, et al. Crystal structure of the human glucose transporter GLUT1[J]. Nature, 2014, 510(7503): 121–125. DOI: 10.1038/nature13306 |
| [35] | YAN N. Structural advances for the major facilitator superfamily (MFS) transporters[J]. Trends Biochem Sci, 2013, 38(3): 151–159. DOI: 10.1016/j.tibs.2013.01.003 |
| [36] | WILLSON B J, DALZELL L, CHAPMAN L N M, et al. Enhanced functionalisation of major facilitator superfamily transporters via fusion of C-terminal protein domains is both extensive and varied in bacteria[J]. Microbiology, 2019, 165(4): 419–424. DOI: 10.1099/mic.0.000771 |
| [37] | TERANISHI Y, INOUE M, YAMAMOTO N G, et al. Proton myo-nositol cotransporter is a novel γ-ecretase associated protein that regulates Aβ production without affecting Notch cleavage[J]. FEBS J, 2015, 282(17): 3438–3451. DOI: 10.1111/febs.13353 |
| [38] | VAWTER M P, HAMZEH A R, MURADYAN E, et al. Association of Myoinositol transporters with schizophrenia and bipolar disorder:evidence from human and animal studies[J]. Mol Neuropsychiatry, 2019, 5(4): 200–211. DOI: 10.1159/000501125 |
| [39] | LEE D G, LEE J H, CHOI B K, et al. H+-myo-inositol transporter SLC2A13 as a potential marker for cancer stem cells in an oral squamous cell carcinoma[J]. Curr Cancer Drug Targets, 2011, 11(8): 966–975. DOI: 10.2174/156800911797264752 |
| [40] | GAO Y F, ZHANG M N, WANG T X, et al. Hypoglycemic effect of D-chiro-inositol in type 2 diabetes mellitus rats through the PI3K/Akt signaling pathway[J]. Mol Cell Endocrinol, 2016, 433: 26–34. DOI: 10.1016/j.mce.2016.05.013 |
| [41] | LAI B B, LAI Y L, ZHANG Y L, et al. The solute carrier family 2 genes are potential prognostic biomarkers in acute myeloid leukemia[J]. Technol Cancer Res Treat, 2020, 19. |



