2. 贵州大学动物疫病研究所, 贵阳 550025;
3. 贵州省动物疫病与兽医公共卫生重点实验室, 贵阳 550025;
4. 贵州省畜禽资源遗传管理站, 贵阳 550025;
5. 都匀市农业农村局, 都匀 558000;
6. 黔南民族职业技术学院, 都匀 558000
2. Institute of Animal Diseases, Guizhou University, Guiyang 550025, China;
3. Guizhou Key Laboratory of Animal Diseases and Veterinary Public Health, Guiyang 550025, China;
4. Guizhou Province Livestock and Poultry Resource Genetic Management Station, Guiyang 550025, China;
5. Agriculture and Rural Affairs Bureau of Duyun City, Duyun 558000, China;
6. Qiannan National Vocational and Technical College, Duyun 558000, China
鼠伤寒沙门菌是一种重要的人兽共患病原菌,常对畜禽健康、公共卫生以及食品安全造成威胁,是世界公共卫生安全重点监测的原核生物。鼠伤寒沙门菌小RNA GcvB是一种保守的Hfq蛋白依赖的非编码RNA,反式编码调节靶mRNA,与靶mRNA通过有限的并且不完美的碱基配对相互作用[1-2],反式编码调控小RNA与靶mRNA之间互补配对区域通常为相邻的6~8个碱基[3]。研究发现小RNA GcvB调控某些编码氨基酸与短多肽的转移蛋白,包括ABC转移蛋白家族中的Dpp和Opp蛋白等[4]。GcvB控制大的转录后调节子,通过其保守的富含G/U的结构域R1影响约1%的沙门菌基因,抑制一些氨基酸生物合成基因的表达[5]。到目前为止,仅验证了约24种鼠伤寒沙门菌GcvB的靶基因[6-7],仍有许多未知的靶基因有待发掘。
本研究通过TargetRNA2软件预测鼠伤寒沙门菌野生型菌株小RNA GcvB的直接作用靶标,并结合该菌株在敲除小RNA GcvB的情况下转录组数据产生的差异结果分析,采用荧光定量PCR检测的方法对相应基因的表达差异进行验证,筛选可能受小RNA GcvB调控的基因,为明确鼠伤寒沙门菌小RNA GcvB的调控机制提供基础资料。
1 材料与方法 1.1 主要实验材料野生型鼠伤寒沙门菌LT2 3409株和其gcvB基因敲除株10241株均由法国国家科学研究中心(CNRS)分子遗传学Bossi实验室惠赠。细菌DNA提取试剂盒购自天根生化科技有限公司;反转录试剂盒PrimeScript RT reagent Kit with gDNA Eraser、荧光定量PCR试剂盒SYBR Premix Ex TaqⅡ等,购自TaKaRa公司;其他试剂均为分析纯。
1.2 TargetRNA2对GcvB靶标的预测登陆网站(http://cs.wellesley.edu/~btjaden/TargetRNA2/),输入分析对象(Salmonella enterica subsp. enterica serovar Typhimurium str.LT2),GcvB R1区共识序列(GGTTGTGATGTTGTGTTGTTGTGTTTGCAATTGGTCTG)、R2区共识序列(ACTTCCTGTA)和R3共识序列(TACCCTGTCTGTCCATAGTGATTAAT)分3次输入,mRNA匹配区域为-70至+30 nt,sRNA窗口大小数值为13,最小杂交种子数为7,P值最大阈值为0.05,过滤尺寸值为400,点击搜索并导出结果,筛选评分较高的5个基因进行后续试验。
1.3 基于转录组KEGG与GO富集的分析基于前期鼠伤寒沙门菌gcvB基因敲除株转录组数据[8],将转录组分析结果注释到KEGG数据库进行pathway富集并通过与GO数据库对比,定位相关基因,分析其涉及通路和细胞成分(cellular component,CC)、分子功能(molecular function,MF)以及生物过程(biological process,BP)。
1.4 预测的靶基因荧光定量PCR验证登录GenBank (登录号: AE006468.2)设计预测的靶基因荧光定量PCR引物,引物由生工生物工程(上海)有限公司合成并进行特异性检测。以3409株作为对照组,GAPDH为内参基因,提取培养至对数早期(OD600 nm≈0.5)的3 409株及10 241株的总RNA样本,反转录为cDNA后进行荧光定量PCR反应,反应体系为SYBR Premix Ex TaqⅡ(Tli RNaseH Plus)5.0 μL、上下游引物各0.3 μL、cDNA 2.0 μL、ddH2O 2.4 μL;扩增条件为95 ℃ 30 s;95 ℃5 s,60 ℃30 s,39个循环;每个样本3个重复,采用2-ΔΔCt法计算相应基因的转录水平,检测并计算当gcvB基因敲除时narY、yeaK、trpB、STM2732和iciA基因在转录水平产生的差异表达倍数,结合转录组测序结果进行分析。
2 结果 2.1 小RNA GcvB的潜在靶标预测TargetRNA2预测结果显示,杂交能量值最高的4个基因narY、yeaK、trpB和STM2732以及阳性对照基因iciA被预测到。除STM2732基因功能未知外,其他4个基因分别编码硝酸还原酶、细胞质蛋白、色氨酸合成酶以及精氨酸转运系统的转录激活因子。5个基因靶位点的预测结果显示,邻近narY mRNA翻译起始位点上游的区域与GcvB R2区形成了7个连续碱基的互补,其余4个mRNA通过富含CA的区域与GcvB富含GU的R1区连续或不连续互补,其中以trpB较为特殊,该匹配区域包含了5′UTR和CDS区(图 1),结果表明,所选5个基因均能和GcvB产生至少7个连续的碱基互补,并且可能存在与此前研究不同的作用机制。
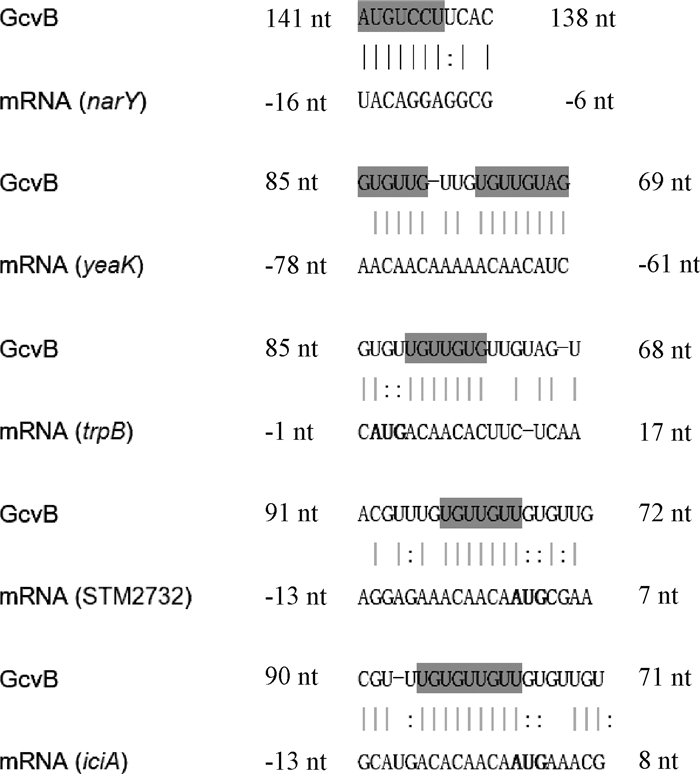
|
图 1 小RNA GcvB与靶mRNA的预测靶位点 Fig. 1 Predicted target sites of small RNA GcvB and target mRNA |
KEGG数据库注释结果显示narY、yeaK、trpB、iciA基因分别富集到硝酸还原酶、Ala-tRNA(Pro)脱酰酶、色氨酸合酶β链和LysR家族转录调节因子几个通路。在细胞成分方面,narY基因还与细菌的细胞质、硝酸盐、还原酶复合物和细胞膜相关,而其他基因未富集到相关功能。在分子功能方面,narY、yeaK和trpB基因分别涉及硝酸还原酶活性、脯氨酸氨酰-tRNA连接酶活性和色氨酸合成酶活性。在生物过程方面,narY、yeaK和trpB基因分别涉及无氧呼吸、翻译保真度的调节和色氨酸合成(表 1)。结果表明,除STM2732基因功能未知外,narY基因与鼠伤寒沙门菌的无氧呼吸过程相关,yeaK基因与蛋白质翻译时脯氨酸的识别相关,trpB基因与色基酸的合成有关,iciA基因与精氨酸的转运有关。转录组数据分析结果显示,5个基因narY、yeaK、trpB、STM2732和iciA分别在gcvB基因敲除后转录水平相对上调1.1、1.5、1.5、7.6和1.4倍,功能未知基因STM2732表达差异最大,其余基因差异表达倍数均小于2,结果表明,除STM2732基因外,其他基因包括阳性对照基因iciA均未达到差异表达阈值,其差异性需进一步验证。
|
|
表 1 KEGG和GO分析信息表 Table 1 KEGG and GO analysis information sheet |
荧光定量PCR引物的特异性经验证后符合要求。荧光定量PCR检测结果显示,包括阳性对照iciA在内的narY、yeaK、trpB和STM2732基因相比3409株均在转录水平表达上调超过2倍,经2-ΔΔCt法计算分析,narY、yeaK、trpB、STM2732和iciA基因分别上调3.4、9.8、12.1、18.37和5.4倍,5个基因所呈现的差异性高低趋势与转录组测序结果保持一致(图 2),以上结果表明,荧光定量PCR方法检测基因的差异表达在敏感性上整体高于转录组测序方法,narY、yeaK、trpB和STM2732是本研究筛选到的受GcvB直接调控的最优选靶基因。
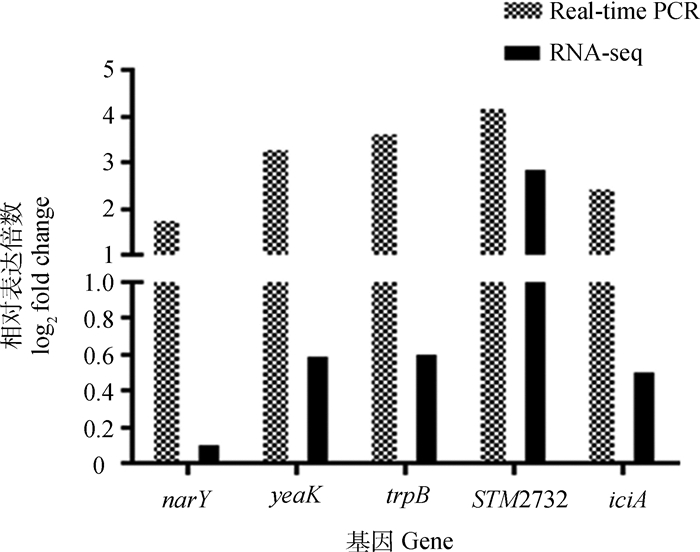
|
试验组与对照组的相对差异表达倍数值为n,代入log2n公式所得 The relative difference between the experimental group and the control group is n, which is obtained by substituting the log2n formula 图 2 预测基因的荧光定量PCR验证结果 Fig. 2 Results of real-time PCR for predicted genes |
所有已经过验证的鼠伤寒沙门菌小RNA GcvB的靶标中,70%的基因与细菌氨基酸的摄取和合成代谢相关,GcvB的功能也被假定为细菌节约能量[9]。TargetRNA2预测的5个基因在转录水平均表达上调,被假定为受GcvB的负调控。其中,narY与细菌的无氧呼吸有关[10-11],鼠伤寒沙门菌作为需氧及兼性厌氧型细菌,当其处于营养过剩的状态时,可能会抑制不必要的能量消耗,所以当细菌处于有氧呼吸时,无必要的无氧呼吸过程将可能被抑制。trpB基因GO富集到色氨酸合成过程[12],这与此前GcvB调控氨基酸的摄取和合成代谢两大功能不谋而合,通常GcvB作用于靶mRNA 5′UTR核糖体结合位点或位于其上游的区域,而预测结果显示GcvB与邻近trpB mRNA起始密码子的CDS区匹配,该机制较为少见。最近,Lalaouna等[13]通过MAPS试验结合生物计算验证了与大肠杆菌小RNA GcvB的靶基因asnB、ysgA、tcyJ、asnA、panD、nlpA和inaA,GcvB均与7个基因的mRNA CDS区不同区域产生碱基互补作用,并主导mRNA的降解或二级结构的释放。该研究支撑了trpB基因作为GcvB靶标的可能性。yeaK基因与脯氨酸氨酰-tRNA连接酶的激活有关[14],该酶对基因的翻译有着极其重要的作用,基因要实现无错误的蛋白质合成,在于氨酰-tRNA与相应氨基酸能否正确定位。STM2732基因的功能未知,但其与GcvB假定的结合位点与已验证的iciA基因具有极其相似的地方,iciA mRNA与STM2732 mRNA起始密码子上游-1至-7 nt的区域完全相同,富含CA的区域与GcvB富含GU的R1区完全互补,因此,其与GcvB的相互作用机制可能与靶基因iciA相同。
4 结论本研究预测了4个鼠伤寒沙门菌小RNA GcvB的靶基因narY、yeaK、trpB和STM2732与GcvB的假定连续碱基匹配数均大于7。4个基因除STM2732功能未知外,其他分别与鼠伤寒沙门菌无氧呼吸过程、硝酸还原酶的激活、脯氨酸氨酰tRNA连接酶的活化以及色氨酸合酶的生成有关。荧光定量PCR验证了4个基因的表达受GcvB的抑制,假定受直接负调控作用。
| [1] |
杨阳, 杨琦, 董然然, 等. 基于转录组测序筛选鼠伤寒沙门菌hfq基因缺失菌的差异表达基因[J]. 畜牧兽医学报, 2019, 50(12): 2578–2584.
YANG Y, YANG Q, DONG R R, et al. Screening of differentially expressed genes of Salmonella typhimurium hfq gene deletion bacteria based on transcriptome sequencing[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(12): 2578–2584. DOI: 10.11843/j.issn.0366-6964.2019.12.023 (in Chinese) |
| [2] | GRVLL M P, MASSÉ E. Mimicry, deception and competition: the life of competing endogenous RNAs[J]. WIREs RNA, 2019, 10(3): e1525. DOI: 10.1002/wrna.1525 |
| [3] | BOUVIER M, SHARMA C M, MIKA F, et al. Small RNA binding to 5' mRNA coding region inhibits translational initiation[J]. Mol Cell, 2008, 32(6): 827–837. DOI: 10.1016/j.molcel.2008.10.027 |
| [4] | PARKER A, GOTTESMAN S. Small RNA regulation of TolC, the outer membrane component of bacterial multidrug transporters[J]. J Bacteriol, 2016, 198(7): 1101–1113. DOI: 10.1128/JB.00971-15 |
| [5] | SHARMA C M, PAPENFORT K, PERNITZSCH S R, et al. Pervasive post-transcriptional control of genes involved in amino acid metabolism by the Hfq-dependent GcvB small RNA[J]. Mol Microbiol, 2011, 81(5): 1144–1165. DOI: 10.1111/j.1365-2958.2011.07751.x |
| [6] | BEISEL C L, STORZ G. The base-pairing RNA spot 42 participates in a multioutput feedforward loop to help enact catabolite repression in Escherichia coli[J]. Mol Cell, 2011, 41(3): 286–297. |
| [7] | PAPENFORT K, ESPINOSA E, CASADESÚS J, et al. Small RNA-based feedforward loop with AND-gate logic regulates extrachromosomal DNA transfer in Salmonella[J]. Prac Natl Acad Sci U S A, 2015, 112(34): E4772–E4781. DOI: 10.1073/pnas.1507825112 |
| [8] |
刘丽娟, 董然然, 王开功, 等. 敲除小RNA gcvB后沙门菌的转录组分析[J]. 畜牧兽医学报, 2019, 50(4): 840–850.
LIU L J, DONG R R, WANG K G, et al. mRNA-Seq whole-transcriptome analysis of sRNA gcvB deletion background in Salmonella[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(4): 840–850. (in Chinese) |
| [9] | MIYAKOSHI M, CHAO Y, VOGEL J. Cross talk between ABC transporter mRNAs via a target mRNA-derived sponge of the GcvB small RNA[J]. EMBO J, 2015, 34(11): 1478–1492. DOI: 10.15252/embj.201490546 |
| [10] | JOHN P. Aerobic and anaerobic bacterial respiration monitored by electrodes[J]. Microbiology, 1977, 98(1): 231–238. |
| [11] | AZHIKINA T L, IGNATOV D V, SALINA E G, et al. Role of small noncoding RNAs in bacterial metabolism[J]. Biochem (Moscow), 2015, 80(13): 1633–1646. DOI: 10.1134/S0006297915130015 |
| [12] | CRAWFORD I P, NICHOLS B P, YANOFSKY C. Nucleotide sequence of the trpB gene in Escherichia coli and Salmonella typhimurium[J]. J Mol Biol, 1980, 142(4): 489–502. |
| [13] | LALAOUNA D, EYRAUD A, DEVINCK A, et al. GcvB small RNA uses two distinct seed regions to regulate an extensive targetome[J]. Mol Microbiol, 2019, 111(2): 473–486. |
| [14] | KUMAR S, DAS M, HADAD C M, et al. Substrate and enzyme functional groups contribute to translational quality control by bacterial prolyl-tRNA synthetase[J]. J Phys Chem B, 2012, 116(23): 6991–6999. DOI: 10.1021/jp300845h |



