2. 国家现代农业产业技术体系四川省兽药创新团队, 成都 610041
2. Sichuan Veterinary Medicine and Drug Innovation Group of China Agricultural Research System, Chengdu 610041, China
Aichivirus B型包括两个成员,分别为牛库布病毒(bovine kobuvirus,BKoV)和羊库布病毒(sheep kobuvirus)[1-2]。目前,BKoV已在多个国家的牛粪便样本中检出[1, 3-13]。韩国、意大利和巴西的报道均显示牛腹泻样本的检出率高于非腹泻样本的检出率[10-11, 13]。国内北京、内蒙古、黑龙江和新疆的166份奶牛腹泻样本中BKoV的检出率为34.90%[7]。本实验室前期对辽宁、河南、山东和陕西96份腹泻粪便和77份非腹泻粪便样本进行BKoV检测,结果显示腹泻样本中阳性率显著高于非腹泻样本(35.42% vs 11.69%,P < 0.001),提示BKoV可能与牛腹泻有关[8]。现有的资料表明,BKoV在国内外奶牛中广泛流行,但目前未见关于牦牛源BKoV的流行病学资料。最近,在一头患有腹泻和神经系统疾病的小牛脑脊液中检出BKoV,表明该病毒可以引起全身感染[14]。至今,BKoV只在日本被成功分离,但还没有关于BKoV人工感染牛的试验报道[1],因此,BKoV对牛的致病性还有待进一步证实。
3D基因是库布病毒的保守区域,常用来作为分子检测的靶基因[15-16]。目前只有国外3篇关于BKoV RT-PCR方法的报道,其引物的靶基因位点均位于3D基因的7 357—7 987区域[1, 4, 10]。至今,未见有关检测BKoV的实时荧光定量RT-PCR的报道。本实验室之前用宏病毒组技术鉴定牦牛腹泻样本中病毒种类时,发现有BKoV核酸序列的存在[17],然而用这3篇已报道的RT-PCR方法却均未能检出,推测可能由于核酸序列变异或检测方法的灵敏度不高所致。因此,本研究的目的是建立检测BKoV的实时荧光定量RT-PCR方法,并对青藏高原地区牦牛腹泻样本中BKoV进行检测。
1 材料与方法 1.1 主要的试剂及仪器PrimeScriptTM、TrizolTM Reagent、克隆载体PMD19-T、TB Green Premix Ex Taq Ⅱ、反转录试剂盒等购于TaKaRa公司;DH5α感受态细胞、DNA Marker、质粒提取试剂盒购于宝生物工程(大连)股份有限公司;超微量核酸蛋白测定仪(广州仪涛科学仪器有限公司,中国);Doc2000凝胶成像系统(BioRad公司,美国);荧光定量PCR仪(BIOER,中国);高速冷冻离心机(Eppendorf公司,德国)。
1.2 临床样本和核酸用于本研究所建的方法和国外3篇已报道的RT-PCR方法比较的48份奶牛腹泻样本于2019年1月采集自河南和辽宁共5个牧场;24份牦牛腹泻样本于2017年8月采集自四川藏区和西藏共5个牧场。用于BKoV检测的131份牦牛腹泻样本于2018年5月—2019年5月分别采集自青海45份(4个牧场)、西藏34份(3个牧场)及四川藏区52份(8个牧场)。以上样本均为3月龄以内的犊牛腹泻粪便。所有样本于-80 ℃保存备用。
用以特异性检验的病毒核酸:BKoV毒株、牛冠状病毒(bovine coronavirus, BCoV)、牛轮状病毒(bovine rotavirus, BRV)、牛病毒性腹泻/黏膜病病毒(bovine viral diarrhea virus, BVDV)、纽布病毒(Nebovirus,NeV)、牛诺如病毒(bovine norovirus, BNoV)、牛肠道病毒(bovine enterovirus, BEV)Swun0510、牛源K99大肠杆菌分离株Swun4027、牛源都柏林沙门菌Swun3740、牛源产气荚膜梭菌Swun2931、牛源空肠弯曲杆菌Swun1644、牛源艾美尔球虫、牛源安氏隐孢子虫的核酸均由西南民族大学动物医学实验室保存。
空白对照均以RNase Free ddH2O代替模板。
1.3 核酸的提取和RNA的反转录用Trizol Reagent法提取总RNA,按照反转录试剂盒说明书的方法将RNA核酸反转录成cDNA;用酚-氯仿法提取样本的总DNA,以上获得的核酸均置于-20 ℃条件下保存备用。
1.4 引物的设计与合成依据GenBank中登录的BKoV 3D基因序列,利用Primer6.0设计一对特异性引物(表 1),目的片段长为157 bp,位于7 861—8 017 nt(GenBank No.MK080265),由上海生工生物工程有限公司合成。
|
|
表 1 4种方法的基本信息及临床样本的检出率 Table 1 Information of four methods and detection rate in clinical samples |
用BKoV的cDNA进行PCR反应及电泳检测并纯化回收目的片段。再将目的片段进行克隆转化,涂于含氨苄青霉素的LB平板上进行增菌。挑取菌落溶于LB溶液中,取2 μL进行PCR鉴定并送生工生物有限公司测序,以测序正确的质粒作为模板进行后续试验。用超微量核酸蛋白测定仪测定质粒的浓度,按照公式拷贝数(copies·μL-1)=(6.02×1023 copies·mol-1)×(浓度μg·mL-1)×10-9/(分子质量g·mol-1)计算质粒拷贝数。
1.6 反应体系及条件的优化采用四步法[18]以20 μL体系(TB Green Premix Ex TaqⅡ 10 μL,BKoV阳性标准品2.0 μL)对0.4~0.8 μL引物浓度和57~64 ℃退火温度进行矩形优化,以获得最高荧光强度及最低 Ct 值和具有特异性单峰的熔解曲线。基于TB Green建立检测BKoV的实时荧光定量RT-PCR方法。
1.7 标准曲线的建立将BKoV质粒以10倍递增稀释,用建立的实时荧光定量RT-PCR方法分别对108、107、106、105、104、103、102、10 copies·μL-1的质粒进行检测并确定线性范围。以循环的Ct值为纵坐标,起始模板浓度的对数为横坐标建立标准曲线。
1.8 特异性评价把“1.2”中列的常见的腹泻病原的核酸及空白对照用本研究建立的实时荧光定量RT-PCR方法进行检测,以鉴定该方法的特异性。
1.9 稳定性评价采用浓度为4.86×105~4.86×103 copies·μL-1的标准品为模板进行实时荧光定量RT-PCR反应。每份样本做3次重复试验用来检验组内重复效果。此外,在同一反应条件下做3次独立的实时荧光定量RT-PCR反应来检验该方法组间的重复性。
1.10 敏感性测定用灭菌超纯水对模板进行100~109系列稀释,利用优化后的实时荧光定量RT-PCR方法进行扩增,以此计算所能检出的最低模板拷贝数,同时设置空白对照。
1.11 4种检测方法对临床样本BKoV检出率的比较48份奶牛腹泻临床样本和24份牦牛腹泻临床样本分别采用本研究所建立的实时荧光定量RT-PCR方法和3篇有关RT-PCR报道的方法[1, 4, 10]进行检测,比较它们的检出率,全部阳性产物送公司测序。引物的基本信息见表 1。
1.12 牦牛腹泻临床样本中BKoV的检测131份牦牛腹泻样本用本研究建立的实时荧光定量RT-PCR方法进行检测,从检出的阳性样本中随机挑选出20%送公司测序。
2 结果 2.1 引物的特异性测序结果长度为157 bp, 符合预期大小。与GenBank中BKoV 3D基因序列的相似性为100%。
2.2 优化的反应体系及条件确定的20 μL反应体系:TB Green Premix Ex Taq Ⅱ 10 μL,上、下游引物各0.6 μL,cDNA 2.0 μL,最后ddH2O补足至20 μL。反应条件:95 ℃ 2 min; 95 ℃ 15 s,60 ℃ 20 s, 72 ℃ 15 s, 84 ℃ 5 s, 40个循环反应条件进行扩增,以35个循环为判定结果的标准。
2.3 标准曲线及熔解曲线克隆的质粒经超微量核酸蛋白测定仪在260 nm处测定其吸光度为154 μg·mL-1,计算其原始的拷贝数:4.86×1010 copies·μL-1。
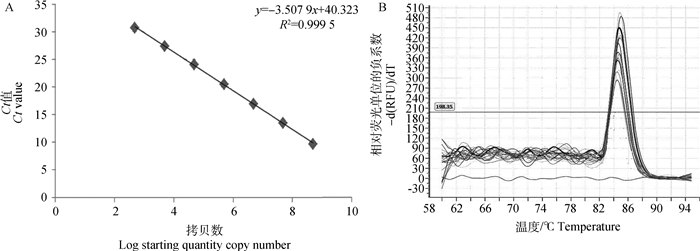
本研究所建方法在4.86×108~4.86×102 copies·μL-1标准品浓度范围内具有良好的线性关系和扩增效率(图 1A),标准曲线为 y=-3.507 9x+40.323,相关系数为0.999 5,扩增效率为92.75%。通过熔解曲线分析可知,PCR产物仅在84.5 ℃左右出现单一峰(图 1B)。
|
图 1 BKoV实时荧光定量RT-PCR方法的标准曲线(A)及熔解曲线(B) Fig. 1 Standard curve (A) and melting curve (B) of real-time RT-PCR for BKoV |
荧光扩增曲线显示仅BKoV具有荧光信号,其他“1.2”提到的无关致腹泻病原均为阴性,表明该方法的特异性好。
2.5 方法的稳定性所建方法的组内变异系数为0.88%~1.38%,组间变异系数为0.58%~0.98%(表 2),结果均小于2%,表明该方法具有良好的稳定性。
|
|
表 2 实时荧光定量RT-PCR稳定性试验 Table 2 Stable test of the real-time RT-PCR |
用所建立的实时荧光定量RT-PCR方法对100~109的稀释度样品进行检测,同时设置空白对照。结果显示稀释度102~109为特异性扩增曲线,该方法所能检出的最低拷贝数为4.86×102 copies·μL-1。
2.7 4种检测方法对临床样本BKoV检出率的比较对于48份奶牛腹泻样本,本研究所建的实时荧光定量RT-PCR方法的检出率明显高于国外3篇已报道的RT-PCR方法(表 1)。检出的阳性样本也包含了其他3种方法所检出的阳性样本,经测序证实均为BKoV的目的片段。
对于24份牦牛腹泻样本,本研究所建的实时荧光定量RT-PCR方法的检出率为37.50%,阳性PCR产物经测序证实均为BKoV核酸片段;其他3种方法均未检出(表 1)。
2.8 牦牛腹泻样本中BKoV的检出率本研究所建实时荧光定量RT-PCR方法对131份牦牛腹泻样本的检测结果显示,BKoV的阳性率为31.30%。青海、西藏和四川藏区的阳性率分别为44.44%、41.18%和13.46%,场阳性率分别为50.00%、100.00%和37.50%。随机挑选的20%阳性PCR产物经测序证实均为BKoV核酸片段,进一步证明该方法适用于牦牛BKoV的检测。
3 讨论国外3篇有关BKoV RT-PCR报道的检测方法[1, 4, 10],均为2011年以前所报道,但目前还没有关于BKoV的实时荧光定量RT-PCR检测方法的报道。本研究通过反应体系和条件的优化,成功建立了一种TB Green染料法实时荧光定量RT-PCR方法。此方法具有特异性和稳定性好、灵敏性高的特点,而且对奶牛和牦牛腹泻样本中BKoV均有很好的检出效果,明显优于目前3篇已报道的RT-PCR方法。进一步分析发现,与GenBank中以前登录的序列相比,2011年以后登录的BKoV 3D基因序列存在不同程度的核苷酸变异,导致这3种RT-PCR方法的引物扩增位点不完全匹配,如文献[4]报道的上、下游引物序列与2011以后GenBank登录的国内外毒株分别有7/23和4/21个碱基不同,这可能是影响上述RT-PCR方法检测效果的重要原因。
牦牛是青藏高原地区的主要养殖畜种[19],但关于BKoV在牦牛的流行情况仍不清楚。本试验结果显示,BKoV在青海、西藏和四川藏区的犊牦牛腹泻样本中检出率较高,提示了BKoV在青藏高原牦牛中广泛流行,为牦牛腹泻的防控提供了参考。值得注意的是,目前国外3篇已报道的有关BKoV的RT-PCR方法均不能检出牦牛源BKoV,GenBank数据库中也没有牦牛源BKoV的序列信息。因此,有必要加强牦牛源BKoV的分子生物学研究,了解牦牛源BKoV的遗传变异,为深入了解BKoV的遗传进化提供参考。
4 结论首次成功建立检测BKoV的实时荧光定量RT-PCR方法,其特异性、重复性好,灵敏度高,为BKoV的定量检测和流行病学调查提供了新的技术手段。首次证实BKoV在牦牛中的广泛流行,其与腹泻的关系值得进一步关注。
| [1] | YAMASHITA T, ITO M, KABASHIMA Y, et al. Isolation and characterization of a new species of kobuvirus associated with cattle[J]. J Gen Virol, 2003, 84(11): 3069–3077. DOI: 10.1099/vir.0.19266-0 |
| [2] | REUTER G, BOROS É, PANKOVICS P, et al. Kobuvirus in domestic sheep, Hungary[J]. Emerg Infect Dis, 2010, 16(5): 869–870. DOI: 10.3201/eid1605.091934 |
| [3] | KHAMRIN P, MANEEKARN N, PEERAKOME S, et al. Bovine kobuviruses from cattle with diarrhea[J]. Emerg Infect Dis, 2008, 14(6): 985–986. DOI: 10.3201/eid1406.070784 |
| [4] | REUTER G, EGYED L. Bovine kobuvirus in Europe[J]. Emerg Infect Dis, 2009, 15(5): 822–823. DOI: 10.3201/eid1505.081427 |
| [5] | BARRY A F, RIBEIRO J, ALFIERI A F, et al. First detection of kobuvirus in farm animals in Brazil and the Netherlands[J]. Infect Genet Evol, 2011, 11(7): 1811–1814. DOI: 10.1016/j.meegid.2011.06.020 |
| [6] | MOHAMED F F, MANSOUR S M G, ORABI A, et al. Detection and genetic characterization of bovine kobuvirus from calves in Egypt[J]. Arch Virol, 2018, 163(6): 1439–1447. DOI: 10.1007/s00705-018-3758-1 |
| [7] | CHANG J T, WANG Q, WANG F, et al. Prevalence and genetic diversity of bovine kobuvirus in China[J]. Arch Virol, 2014, 159(6): 1505–1510. DOI: 10.1007/s00705-013-1961-7 |
| [8] | LI H P, TANG C, YUE H. Molecular detection and genomic characteristics of bovine kobuvirus from dairy calves in China[J]. Infect Genet Evol, 2019, 74: 103939. DOI: 10.1016/j.meegid.2019.103939 |
| [9] | PARK S J, KIM H K, SONG D S, et al. Molecular detection and genetic characterization of kobuviruses in fecal samples collected from diarrheic cattle in Korea[J]. Infect Genet Evol, 2011, 11(5): 1178–1182. DOI: 10.1016/j.meegid.2011.02.019 |
| [10] | JEOUNG H Y, LIM J A, JEONG W, et al. Three clusters of bovine kobuvirus isolated in Korea, 2008-2010[J]. Virus Genes, 2011, 42(3): 402–406. |
| [11] | DI MARTINO B, DI PROFIO F, DI FELICE E, et al. Molecular detection of bovine kobuviruses in Italy[J]. Arch Virol, 2012, 157(12): 2393–2396. DOI: 10.1007/s00705-012-1439-z |
| [12] | RIBEIRO J, LORENZETTI E, ALFIERI A F, et al. Kobuvirus (Aichivirus B) infection in Brazilian cattle herds[J]. Vet Res Commun, 2014, 38(2): 177–182. DOI: 10.1007/s11259-014-9600-7 |
| [13] | CANDIDO M, BATINGA M C A, ALENCAR A L F, et al. Molecular characterization and genetic diversity of bovine Kobuvirus, Brazil[J]. Virus Genes, 2017, 53(1): 105–110. |
| [14] | MOREIRA A S D, RAABIS S M, GRAHAM M E, et al. Identification by next-generation sequencing of Aichivirus B in a calf with enterocolitis and neurologic signs:a cautionary tale[J]. J Vet Diagnostic Invest, 2017, 29(2): 208–211. DOI: 10.1177/1040638716685597 |
| [15] | REUTER G, BOROS Á, PANKOVICS P. Kobuviruses - a comprehensive review[J]. Rev Med Virol, 2011, 21(1): 32–41. DOI: 10.1002/rmv.677 |
| [16] | RIBEIRO J, DE ARRUDA LEME R, ALFIERI A F, et al. High frequency of Aichivirus C (porcine Kobuvirus) infection in piglets from different geographic regions of Brazil[J]. Trop Anim Health Prod, 2013, 45(8): 1757–1762. DOI: 10.1007/s11250-013-0428-x |
| [17] | CHEN X, ZHANG B, YUE H, et al. A novel astrovirus species in the gut of yaks with diarrhoea in the Qinghai-Tibetan Plateau, 2013[J]. J Gen Virol, 2015, 96(12): 3672–3680. DOI: 10.1099/jgv.0.000303 |
| [18] |
张驰宇, 张高红, 杨敏, 等. 四步法消除SYBR Green Ⅰ实时定量RT-PCR中引物二聚体的影响[J]. 中国生物化学与分子生物学报, 2004, 20(3): 387–392.
ZHANG C Y, ZHANG G H, YANG M, et al. Elimination of Primer-dimer Effect in SYBR Green Ⅰ real-time RT-PCR Using 4-step Program[J]. Chinese Journal of Biochemistry and Molecular Biology, 2004, 20(3): 387–392. DOI: 10.3969/j.issn.1007-7626.2004.03.018 (in Chinese) |
| [19] |
保善科, 张丽, 孔祥颖, 等. 不同部位高原牦牛肉品质评价[J]. 畜牧兽医学报, 2015, 46(3): 388–394.
BAO S K, ZHANG L, KONG X Y, et al. The quality evaluation of different muscles from plateau yak[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(3): 388–394. (in Chinese) |



