猪伪狂犬病(pseudorabies,PR)是由疱疹病毒科、α-疱疹病毒亚科中的猪伪狂犬病病毒(pseudorabies virus,PRV)引起猪的一种致死性、接触性、急性传染病[1]。PRV感染在全球养猪业普遍存在,是影响当今养猪业的主要病原之一[2]。我国于20世纪50年代首次报道PR的暴发,60至70年代PR给国内养猪业造成严重经济损失。后期由于基因缺失疫苗Bartha-K61等的成功引入及普遍推广使用,PR疫情曾得到有效防控[3-4]。然而,自2011年以来,我国黑龙江、吉林、山东等多个省份报道猪场发生PR暴发式疫情[5-6],多数已免疫了PRV疫苗的猪场仍然暴发PR,给养猪业造成了巨大经济损失[7-8]。
PRV属于猪疱疹病毒Ⅰ型[9],基因组为线状双链DNA,约143 kb,G+C碱基含量可达74%[10],gB、gE、TK基因常被作为了解PRV分子流行情况的主要参考基因[11]。gB基因作为PRV感染、复制的必须基因,其编码的囊膜蛋白是最保守的糖蛋白之一,能有效诱导机体产生两种中和抗体并参与病毒的细胞间传播[12],且若有补体存在,其诱导机体产生的中和能力更强[13]。gE基因作为区分PRV野毒株与疫苗株的主要毒力基因,其编码的糖蛋白主要介导病毒的细胞间扩散、融合[14-15]。TK基因作为PRV重要毒力基因,其缺失会导致病毒毒力减弱甚至消失,因此成为基因工程疫苗研发热点[16]。根据报道,近年常用的gE基因缺失疫苗Bartha-K61无法对猪群提供全面的保护[17-19]。基于本实验室前期研究成果,本研究对广西近年PRV流行株持续进行检测监测,并对流行分离株gB、gE、TK基因进行遗传变异分析,以了解近年广西PR的发生、流行态势以及PRV流行株遗传演化特点,为做好PR针对性防控及净化提供科学依据。
1 材料与方法 1.1 病料2013年4月至2018年1月,从广西各地已免疫PRV疫苗的疑似PR发病猪,采集脑、肺、脾等靶组织样品,-20 ℃保存备用。
1.2 主要试剂及仪器设备DMEM、胎牛血清(GIBCO);PBS(博士德);胰酶(Invitrogen);LA Taq聚合酶、内切酶、pMD18-T vector[宝生物工程(大连)]、体液病毒RNA/DNA小量制备试剂盒(Axyprep);凝胶回收试剂盒、质粒抽提试剂盒(Omega)、-80 ℃超低温冰箱(SANYO);PCR仪(BIO-RAD);凝胶紫外图像处理系统(上海天能)等。
gB、gE、TK基因扩增引物参照参考文献[16]。引物由上海杰李生物技术公司合成。引物序列见表 1。由于PRV的突变、进化等因素,导致PRV变异株、经典株及国外株在gB、gE基因核苷酸序列上存在不同,因此用同一引物扩增不同亚型毒株的相同基因会得到两个不同大小的目的片段。
|
|
表 1 引物序列 Table 1 The sequences of primers |
在NCBI数据库中下载国内外代表性PRV毒株序列作为参考序列,其名称、登录号、时间及地域信息如表 2所示。
|
|
表 2 参考的国内外基因序列 Table 2 The reference strains of domestic and overseas |
PRVE-F/PRVE-R gE特异性引物用于临床样品及分离病毒的PCR检测。将阳性样品组织剪碎研磨,加入PBS稀释成组织悬液,反复冻融3次,12 000 r·min-1离心5 min,上清经0.22 μm滤器过滤,滤液冻存于-80 ℃备用。待Vero细胞贴壁培养至80%密度后,取400 μL处理后的滤液接种Vero细胞,轻轻混匀后置于37 ℃ 5%CO2培养箱孵育1 h。孵育结束后弃去孵育液,加入含2%胎牛血清的DMEM培养液,同时设立阴性对照,观察并记录细胞生长及病变产生情况。病毒盲传3代,待细胞病变稳定且病变率达80%以上时收获病毒液,PCR检测确认为PRV阳性后,-80 ℃冻存备用。
1.5 阳性样本DNA提取及gB、gE、TK基因扩增克隆及测序依照相应试剂盒说明书提取阳性样本DNA。PCR体系及程序参照参考文献[16]。PCR扩增产物经电泳检测,按相应试剂盒说明书进行目的片段纯化回收并克隆至pMD18-T载体。挑选阳性重组质粒送至广州华大基因进行测序,测序结果用DNAStar完成序列的拼接,同时用Blast和DNAStar两种方法进行比对分析。
1.6 gB、gE、TK基因遗传进化分析测序获得38株分离毒株的gB、gE、TK基因核苷酸序列。对核苷酸序列进行推导,得出分离毒株及参考毒株(表 2)的gB、gE、TK基因编码氨基酸序列。利用DNAStar对分离毒株与参考毒株gB、gE、TK基因核苷酸序列以及氨基酸序列进行同源性及变异位点的比对分析;利用MEGA6.0构建系统进化树,进行遗传进化分析。
2 结果 2.1 样品检测2013年4月至2018年1月,共检测714份疑似PR病例病料样品,阳性175份,阳性率为24.5%(175/714)。由表 3可知,近年广西地区的PRV检测阳性率总体呈上升趋势,以2016年及2018年阳性率较高,达30%以上。为进一步了解PR发病特点,以不同季度为单位对发病情况进行分析,显示PR一年四季均可发病,季节性不显著。四个季度的样品阳性率分别如下:第一季度27.89%(41/147)、第二季度34.06%(47/138)、第三季度24.02%(49/204)、第四季度27.44%(87/317)。
|
|
表 3 疑似PR病料组织样品PRV检测结果 Table 3 Detection results of tissue samples from suspected clinical specimen |
将滤液接种Vero细胞后,细胞出现聚集、变圆、脱落及出现空泡等PRV典型细胞病变(CPE)。收集病毒液,利用PRV特异性引物对病毒液进行PCR检测,可获得预期目的条带(图 1),证实所分离病毒为PRV。共计获得38株PRV地方流行株。
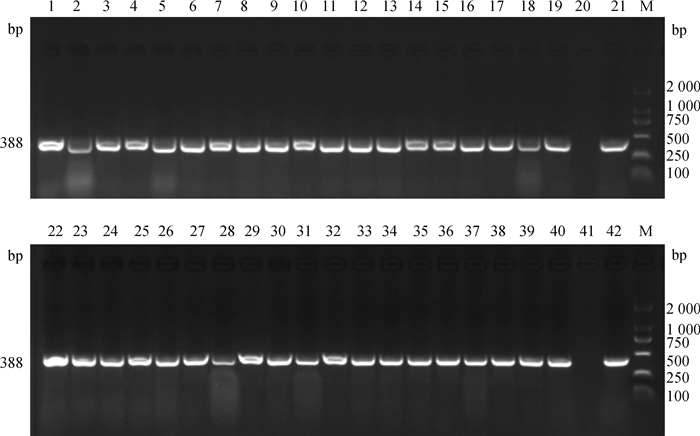
|
1~19、22~40.分离毒株;20、41.阴性对照;21、42.阳性对照;M. DL2000 DNA相对分子质量标准 1-19, 22-40. PRV isolates; 20, 41. Negative control; 21, 42. Positive control; M. DL2000 DNA marker 图 1 PRV分离株gE基因PCR鉴定结果 Fig. 1 PCR identification results of PRV gE genes |
参照表 1中的三对引物对38株PRV分离株gB、gE、TK基因进行PCR扩增,将目的片段连接至pMD18-T载体上,送至广州华大基因进行测序。38株PRV的3个基因PCR产物进行凝胶电泳试验检测发现,2016-GG-4株的TK基因扩增结果与其他分离株TK基因扩增结果电泳条带相比偏小。
2.3.1 gB、gE、TK基因的同源性分析将广西地方分离株与参考毒株的gB、gE、TK基因核苷酸和氨基酸使用MegAlign进行同源性的比对,结果如表 4所示。
|
|
表 4 广西PRV分离株与参考毒株gB、gE、TK基因的核苷酸和氨基酸相似性比对结果 Table 4 The results of gB, gE, TK nucleotide and amino acid sequence alignment of PRV isolates in Guangxi |
PRV可分为国内株G1及国外株G2(以NIA3、BECKER、kaplan、Bartha-K61株为代表)两大群。国内株可分为经典株G1-b(以SC、Ea为代表)和变异株G1-a(以TJ、JS-2012、HNX、HN1201、HLJ8、HeN1为代表)两个亚群。38株PRV广西分离株可分为3个亚群,其中2株属国外株亚群,5株属国内经典株亚群,其余31株分离毒株属国内变异毒株亚群(见图 2)。
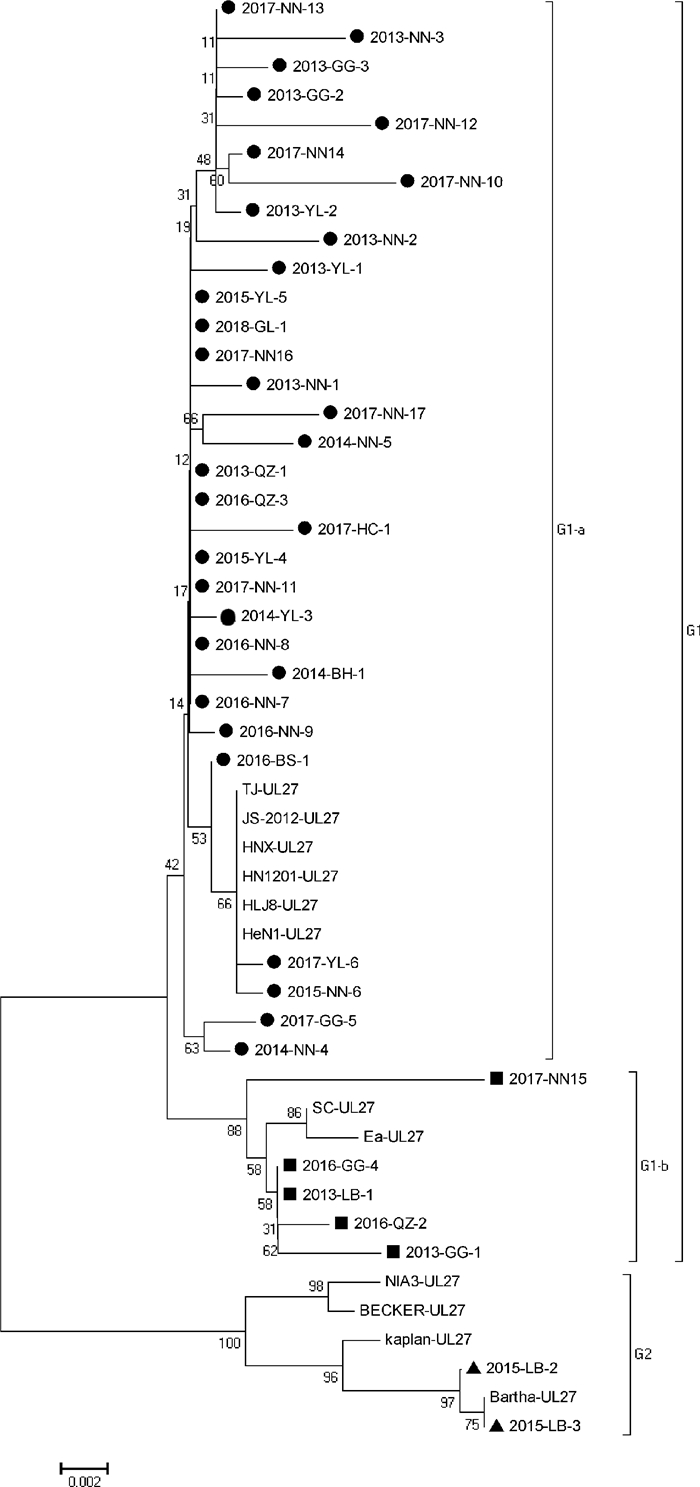
|
●.变异型的PRV毒株;■.经典型的PRV毒株;▲.国外PRV毒株 ●. Variant PRV strain; ■. Classical PRV strain; ▲. Foreign PRV strain 图 2 PRV广西分离毒株gB基因系统进化树 Fig. 2 Phylogenetic tree of gB gene of PRV |
根据遗传进化树的分析结果,比对不同亚型的gB基因编码的氨基酸序列,结果显示:G2型的毒株与国外毒株的氨基酸序列相同;G1型毒株与G2型毒株相比有以下相同的氨基酸变化规律,在以下位置出现如下变异:A53T、P55 T、T70 A、N81 D、D82 G、V83 F、A87 E、E93 D、F96 V、E102 P、G557 S、S678 G、T847 A、D854 E、G856 D,在第94位插入G;进一步分析发现,在G1型毒株中,G1-a型毒株出现R458K的独特变化,G1-b型毒株在以下位置出现如下突变:A85 T、Q567 H、A744T。
2.3.2.2 gE基因的遗传进化分析遗传进化分析显示,38株PRV广西分离株分属国内2个亚群(G1-a、G1-b),其中5株属国内经典株亚群(G1-b),其余分离毒株属国内变异毒株亚群(G1-a),见图 3。
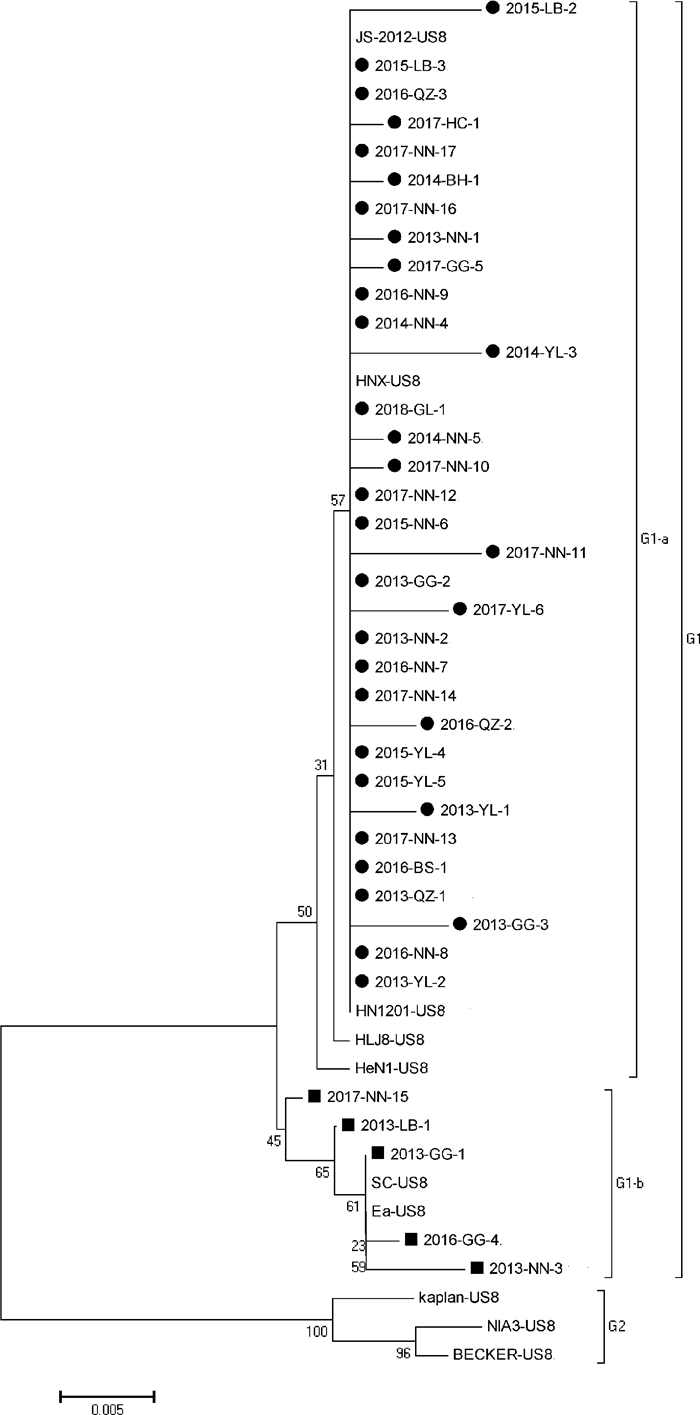
|
●.变异型的PRV毒株;■.经典型的PRV毒株 ●. Variant PRV strain; ■. Classical PRV strain 图 3 PRV广西分离株gE基因系统进化树 Fig. 3 Phylogenetic tree of gE gene of PRV |
根据遗传进化树的分析结果,比对不同亚型的gE基因编码氨基酸序列,结果发现G1型毒株同G2型毒株相比有相同氨基酸变化规律,在以下位置出现如下变异:D59 N、N63 D、V106 L、R149 M、T179 S、L215 A、A216 D、G471 R、R473 H、V502 A、A524 P、S571 N,在第48位插入D;同时G1-a型毒株出现独特的突变S518 P,且在496位插入D。
2.3.2.3 TK基因的遗传进化分析遗传进化分析显示TK基因相似性较高,38株PRV广西分离株中除2016-GG-4株因为存在大片段基因缺失导致与国内外参考毒株无法分在同一分支外,其他37株与参考毒株可分成国内株(G1)和国外株(G2)两个亚群,且处在G1亚群的国内经典株与变异株无明显分支。广西分离株中有5株与国内经典毒株Ea(G1)有较近的亲缘关系,4株与Bartha-K61、BECKER(G2)相似性较高(见图 4)。
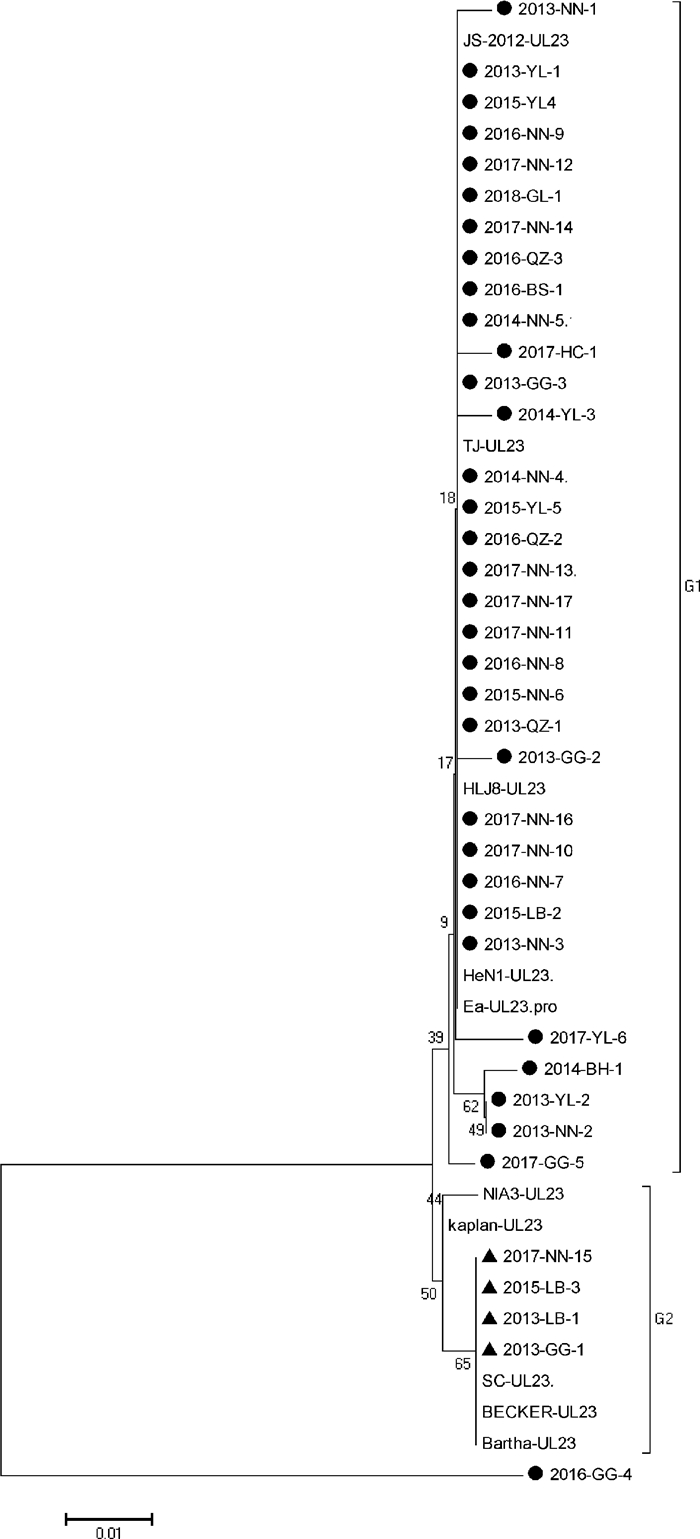
|
●.国内PRV毒株;▲.国外PRV毒株 ●. Domestic strains; ▲. Foreign PRV strains 图 4 PRV广西分离株TK基因系统进化树 Fig. 4 Phylogenetic tree of TK gene of PRV |
对TK基因编码氨基酸序列分析表明,TK基因较为保守,大部分广西分离株与国外毒株相比,仅出现了T215V、A284V突变。其中,测序发现2016-GG-4毒株TK基因缺失205个碱基,全长共758 bp,氨基酸的缺失情况如图 5所示,与HB98株TK基因缺失情况相同。

|
图 5 2016-GG-4株TK基因核苷酸缺失的位置及数目 Fig. 5 Location and number of TK deletion in 2016-GG-4 strain |
2011年以来,我国多地区已免疫PR疫苗的猪群发生PR疫情,导致仔猪和中大猪大量死亡,以及怀孕母猪流产。PRV疫苗免疫猪场PR疫情的出现给我国养猪业造成了重大的经济损失[20]。究其原因主要是PRV流行株发生了基因变异或重组,产生了变异毒株或重组毒株,新毒株抗原性发生了改变,致病力较经典PRV毒株更强[21-22],使得疫苗免疫保护作用下降,致使已经免疫了PR疫苗的猪群仍然发生PR疫情[23-24]。为较全面了解近年来广西PRV流行株基因变异与遗传演化特点,本研究对2013—2018年广西已免疫PRV疫苗猪场的PR疑似病例进行PRV检测监测,并对流行分离株gB、gE及TK基因进行遗传进化分析。结果表明,经PRV疫苗免疫的广西受检疑似PR发病猪场普遍存在PRV野毒感染,总体呈上升趋势,而且从不同年份获得的PRV流行株多数为变异毒株。该研究结果与国内相关报道基本一致[4, 5, 8, 16, 25]。这提示应加强对PRV变异株感染及防控的基础研究,为有效防控PRV变异毒株感染提供理论及技术支持。
相关研究表明,gB、gE和TK基因分别对于PRV的感染和复制、细胞融合以及病毒的侵染能力具有极为重要的作用[12, 16, 26]。研究这三个基因的遗传变异特点对于分析PRV流行株变异具有重要意义。为进一步较全面了解广西地区不同时空PRV流行株流行特点、基因变异与遗传演化特点,课题组对2013—2018年于广西各地区采集的疑似PR发病猪靶组织进行PRV检测以及病毒分离鉴定,并对分离流行株gB、gE和TK基因进行克隆测序及遗传变异分析。分离获得38株地方流行毒株,对gB、gE基因的同源性及遗传进化树分析结果表明,广西地区PRV主要流行毒株为变异株且不同亚型毒株之间存在相同的氨基酸变化特点,这些氨基酸位点的变化可以作为区分PRV不同亚型的毒株的分子特征,这与已有的研究结果相符[27-28]。其中,变异株所具有的独特氨基酸位点的变化,其对病毒毒力的改变以及病毒的免疫逃避是否产生影响需要进一步研究验证。
对广西PRV流行株的gB、gE基因系统分析研究发现,PRV分离株中2015-LB-2及2015-LB-3毒株的gB基因与疫苗株gB基因相似性较高,而gE基因与变异株gE基因相似性较高,推测其属于国内变异株与疫苗株发生重组产生的变异重组毒株,表明在广西地区PRV变异毒株可能是在疫苗免疫压力的作用下产生的病毒重组毒株,对此需进一步深入研究加以证实。
TK基因保守性较高,该基因遗传进化树无明显分支,这与陈超[29]的研究结果相同。分离株中有5株分离株与疫苗株有较高亲缘性。同时发现,2016-GG-4的TK基因在第634—838位缺失205 bp核苷酸,与疫苗株HB98在TK基因缺失的区域相同,且经测序可知无LacZ基因插入导致的gG基因沉默情况存在[20],分析推测2016-GG-4株不是HB98疫苗株,而是国内经典毒株与HB98疫苗株的重组毒株,此现象在先前的PRV流行病学调查中均未有过报道,该改变是否会影响常规疫苗对该重组毒株攻击的保护仍未明确。对2016-GG-4株进一步研究显示[10],该毒株与其他PRV毒株相比,在Vero细胞上增殖特性保持一致,但对小鼠致病性显著降低,极有潜力研发成新的疫苗候选株以应对新型PRV流行株感染。
自2011年PRV变异株出现以来,国内针对变异株的基因缺失疫苗进行了大量的研究,这些基因缺失苗虽然仍能对PRV变异株的感染提供较好的保护,但是无法做到完全的保护[30]。由于临床中存在PRV疫苗株与野毒株的重组情况,新变异毒株的出现可能导致现有基因缺失疫苗保护效果不佳、甚至免疫失败的情况出现。因此,针对当前PRV流行毒株感染的新态势,需要研发针对性免疫防控及净化新策略,杜绝及减少PRV流行变异毒株对养猪业的危害。
4 结论近年广西受检猪场较普遍存在PRV感染,而且PRV流行株以变异毒株为主,可能存在两种不同重组变异形式产生的重组毒株,猪场需重视及实施针对性PR免疫防控计划。广西PRV流行株的TK基因相对保守,gB、gE基因与2011年后国内其他流行的变异株同源性较高,同属一个分支,与国外株和国内经典株相比发生了较为显著的变异。本研究从分子水平解释了广西地区2013—2018年PRV的流行特征,为进一步采取针对性综合防控措施提供了可靠的科学依据。
| [1] |
殷震.
动物病毒学[M]. 2版. 北京: 科学出版社, 1997.
YIN Z. Animal virology[M]. 2nd ed. Beijing: Science Press, 1997. (in Chinese) |
| [2] |
刘涛, 王瑞, 曲哲会, 等. 2014-2016年豫南地区规模化猪场猪主要病毒性传染病流行病学调查分析[J]. 黑龙江畜牧兽医, 2018(20): 106–108.
LIU T, WANG R, QU Z H, et al. Epidemiological survey and analysis of major viral infectious diseases in large-scale pig farms in southern Henan from 2014 to 2016[J]. Heilongjiang Animal Science and Veterinary Medicine, 2018(20): 106–108. (in Chinese) |
| [3] |
赵雪丽, 班付国, 马震原, 等. 猪伪狂犬病免疫抗体及感染抗体消长规律研究[J]. 现代牧业, 2019, 3(1): 14–16.
ZHAO X L, BAN F G, MA Z Y, et al. Study on the change law of anti-pseudorabies virus immune antibody and infected antibody[J]. Modern Animal Husbandry, 2019, 3(1): 14–16. (in Chinese) |
| [4] |
张青占.变异猪伪狂犬病毒的分离鉴定及生物学特性分析[D].北京: 中国农业科学院, 2013.
ZHANG Q Z. Isolation and identification of a mutated swine pseudorabies virus and its biological characteristics analysis[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. (in Chinese) http://cdmd.cnki.com.cn/article/cdmd-82101-1013357459.htm |
| [5] |
彭金美, 安同庆, 赵鸿远, 等. 猪伪狂犬病病毒新流行株的分离鉴定及抗原差异性分析[J]. 中国预防兽医学报, 2013, 35(1): 1–4.
PENG J M, AN T Q, ZHAO H Y, et al. Identification and antigenic variation of new epidemiology of pseudorabies virus from swine[J]. Chinese Journal of Preventive Veterinary Medicine, 2013, 35(1): 1–4. (in Chinese) |
| [6] |
王国栋. 江山地区猪伪狂犬的流行现状与防控措施[J]. 畜禽业, 2019, 30(3): 47.
WANG G D. The epidemic status and control measures of pig pseudorabies in Jiangshan area[J]. Livestock and Poultry Industry, 2019, 30(3): 47. (in Chinese) |
| [7] |
李海琴, 康昭风, 谭美芳, 等. 江西省猪伪狂犬病原流行病学调查及其gB、gE及TK基因遗传变异分析[J]. 江西农业大学学报, 2019, 41(2): 332–339.
LI H Q, KANG Z F, TAN M F, et al. Epidemiological survey of pseudorabies in Jiangxi province and analysis of genetic variation of gB, gE and TK genes[J]. Acta Agriculturae Universitatis Jiangxiensis, 2019, 41(2): 332–339. (in Chinese) |
| [8] | LUO Y Z, LI N, CONG X, et al. Pathogenicity and genomic characterization of a pseudorabies virus variant isolated from Bartha-K61-vaccinated swine population in China[J]. Vet Microbiol, 2014, 174(1-2): 107–115. |
| [9] | DEAN H J, MILLER J M, ACKERMANN M R, et al. Replication and pathogenicity after intranasal and intracranial inoculation of swine with a recombinant pseudorabies virus containing a deletion at the UL/IR junction[J]. Virology, 1996, 223(1): 19–28. |
| [10] |
秦树英.猪伪狂犬病毒GXLB-2015和GXGG-2016毒株的全基因组序列分析和生物学特性研究[D].南宁: 广西大学, 2017.
QIN S Y. Complete genome amalysis and biological characteristics of PRV GXLB-2015 and GXGG-2016 strain[D]. Nanning: Guangxi University, 2017. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10593-1017225751.htm |
| [11] |
庞旋飞, 练斯南, 伍建敏, 等. 伪狂犬病病毒FS-2015株gE和gB基因序列分析[J]. 中国畜牧兽医, 2018, 45(12): 3524–3534.
PANG X F, LIAN S N, WU J M, et al. Sequence analysis of gE and gB genes of pseudorabies virus FS-2015 strain[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(12): 3524–3534. (in Chinese) |
| [12] | LUKÀCS N, THIEL H J, METTENLEITER T C, et al. Demonstration of three major species of pseudorabies virus glycoproteins and identification of a disulfide-linked glycoprotein complex[J]. J Virol, 1985, 53(1): 166–173. |
| [13] | LI X D, YANG F L, HU X L, et al. Two classes of protective antibodies against Pseudorabies virus variant glycoprotein B:implications for vaccine design[J]. PLoS Pathog, 2017, 13(12): e1006777. |
| [14] |
任卫科, 池晶晶, 李秀丽, 等. 伪狂犬病毒主要毒力基因的研究进展[J]. 畜牧兽医科学, 2017(8): 12–13.
REN W K, CHI J J, LI X L, et al. Research progress on the main virulence genes of Pseudorabies virus[J]. Graziery Veterinary Sciences, 2017(8): 12–13. (in Chinese) |
| [15] |
李宁, 吴志明, 闫若潜, 等. 2012-2014年河南省猪伪狂犬病病毒gE基因和TK基因的遗传变异分析[J]. 中国兽医学报, 2016, 36(10): 1653–1657.
LI N, WU Z M, YAN R Q, et al. Genetic variation analysis of gE and TK genes of pseudorabies virus in Henan province from 2012 to 2014[J]. Chinese Journal of Veterinary Science, 2016, 36(10): 1653–1657. (in Chinese) |
| [16] |
薛辉.猪伪狂犬病毒变异株的分离鉴定及主要基因序列分析和生物学特性研究[D].南宁: 广西大学, 2015.
XUE H. Isolation and identification, major gene sequence analysis of pseudorabies virus variant strain and its biological characteristics study[D]. Nanning: Guangxi University, 2015. (in Chinese) |
| [17] |
周金柱. Bartha-k61株疫苗对猪伪狂犬病病毒变异株的免疫保护及变异株生物学特性的初步研究[D].扬州: 扬州大学, 2018.
ZHOU J Z. Protection efficacy of Bartha-k61 vaccine strain in growing pigs against emerging variant pseudorabies virus challenge and the preliminary biological characteristics of the variant pseudorabies viruses[D]. Yangzhou: Yangzhou University, 2018. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-11117-1018020324.htm |
| [18] | AN T Q, PENG J M, TIAN Z J, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J]. Emerg Infect Dis, 2013, 19(11): 1749–1755. |
| [19] |
白挨泉. 新型猪伪狂犬病流行情况及净化措施[J]. 兽医导刊, 2016(3): 22–24.
BAI A Q. Epidemic situation and purification measures of new swine pseudorabies[J]. Veterinary Orientation, 2016(3): 22–24. (in Chinese) |
| [20] |
陈焕春, 周复春, 方六荣, 等. 伪狂犬病病毒鄂A株TK-/gG-/LacZ+突变株的构建[J]. 病毒学报, 2001, 17(1): 69–74.
CHEN H C, ZHOU F C, FANG L R, et al. Construction of TK-/gG-/LacZ+ mutant of pseudorabies virus strain Ea[J]. Chinese Journal of Virology, 2001, 17(1): 69–74. (in Chinese) |
| [21] |
余秋颖, 常洪涛, 陈文定, 等. 2012-2013年新流行猪伪狂犬病病毒的分离鉴定及其gE、TK、gD基因序列分析[J]. 中国兽医学报, 2014, 34(10): 1573–1578, 1583.
YU Q Y, CHANG H T, CHEN W D, et al. Isolation and identification of pseudorabies virus prevailing from 2012 to 2013 and sequence analysis of gE, TK, gD gene[J]. Chinese Journal of Veterinary Science, 2014, 34(10): 1573–1578, 1583. (in Chinese) |
| [22] |
周春陵, 谭鑫, 许拓, 等. PRV变异株的分离及抗原差异性分析[J]. 中国兽医学报, 2017, 37(3): 404–409.
ZHOU C L, TAN X, XU T, et al. Identification and antigenic variation of variant PRV[J]. Chinese Journal of Veterinary Science, 2017, 37(3): 404–409. (in Chinese) |
| [23] |
叶超.猪伪狂犬病病毒HeN1株全基因组测序与病毒遗传变异分析[D].北京: 中国农业科学院, 2015.
YE C. Genomic sequencing of pseudorabies virus HeN1 strain and genetic diversity analysis of the virus[D]. Beijing: Chinese Academy of Agricultural Sciences, 2015. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2787515 |
| [24] |
王一鹏, 王亚文, 徐瑞涛, 等. 一株分离自免疫猪场的伪狂犬病病毒的鉴定与变异分析[J]. 畜牧兽医学报, 2019, 50(10): 2070–2078.
WANG Y P, WANG Y W, XU R T, et al. Indentification and variation analysis of pseudorabies virus from vaccinated pig farm[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(10): 2070–2078. (in Chinese) |
| [25] |
王宇, 李段, 张乐宜, 等. 猪伪狂犬病毒的分离鉴定及其gE、TK全基因序列遗传变异分析[J]. 广东农业科学, 2016, 43(7): 133–144.
WANG Y, LI D, ZHANG L Y, et al. Isolation and identification of porcine pseudorabies virus and genetic variation analysis of gE, TK gene[J]. Guangdong Agricultural Sciences, 2016, 43(7): 133–144. (in Chinese) |
| [26] |
孙佳楠, 王楠楠, 王瑞, 等. 猪伪狂犬病病毒LN1301株分离鉴定及其gE和TK基因的变异分析[J]. 中国兽医学报, 2016, 36(8): 1324–1329.
SUN J N, WANG N N, WANG R, et al. Isolation and identification of pig pseudorabies virus strain LN1301 and its gE and TK gene mutation analysis[J]. Chinese Journal of Veterinary Science, 2016, 36(8): 1324–1329. (in Chinese) |
| [27] |
赵鸿远.猪伪狂犬病病毒变异株的分离鉴定及其生物学特性的研究[D].北京: 中国农业科学院, 2014.
ZHAO H Y. Identification of pseudorabies virus variants in swine and the research of their biological characteristics[D]. Beijing: Chinese Academy of Agricultural Sciences, 2014. (in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-82101-1014326499.htm |
| [28] |
高泽文, 范桂芳, 赵婷婷, 等. 猪伪狂犬病病毒流行株遗传进化及序列比对分析[J]. 中国兽医学报, 2018, 38(1): 1–10.
GAO Z W, FAN G F, ZHAO T T, et al. Phylogenetic and sequence alignment analysis of epidemic swine Pseudorabies virus[J]. Chinese Journal of Veterinary Science, 2018, 38(1): 1–10. (in Chinese) |
| [29] |
陈超.山东省免疫猪场猪伪狂犬病毒分离鉴定及gE、gC、gD和TK基因的序列分析[D].泰安: 山东农业大学, 2015.
CHEN C. Sequence analysis of gE, gC, gD and TK gene among pseudorabies virus strains[D]. Tai'an: Shandong Agricultural University, 2015. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2786254 |
| [30] |
华利忠, 刘剑锋, 冯志新, 等. 猪伪狂犬病病毒新流行变异毒株的研究进展[J]. 江苏农业学报, 2017, 33(2): 476–480.
HUA L Z, LIU J F, FENG Z X, et al. Progress in novel pseudorabies virus variants prevalent in China[J]. Jiangsu Journal of Agricultural Sciences, 2017, 33(2): 476–480. (in Chinese) |



