猪流感是由猪流感病毒(swine influenza viruses,SIVs)引起的一种急性、热性、高度接触性的呼吸道传染病[1-2],通常在猪场内呈现大规模的暴发或者地方性的流行,临床上多以突发性、群发性、咳嗽、呼吸困难为主要特征[3]。目前在世界范围内流行的SIVs主要包括H1N1、H1N2和H3N2三种不同亚型[4]。不同年龄、性别和品种的猪均能够感染SIVs,猪群感染率可高达100%,但死亡率却非常低[5]。猪的呼吸道上皮细胞表面存在着人流感病毒受体(唾液酸α-2, 6半乳糖苷,SAα-2, 6-Gal)和禽流感病毒受体(唾液酸α-2, 3半乳糖苷,SAα-2, 3-Gal)[6],因而猪被视作是“禽-猪-人”流感病毒跨种传播过程中重要的中间宿主[7],同时也是多种异源流感病毒基因重配的“混合容器”[8],更是产生人类流感大流行毒株的“孵育器”[9]。虽然与其他猪的烈性或致死性传染病相比,SIVs对养猪业的危害并不算特别严重,但是它作为一种能够引发人畜共患病的病毒,具有较为深远的公共卫生学意义[10]。
2009年甲型H1N1大流感(2009 Pandemic H1N1,pdm/09 H1N1)是21世纪以来第一次人类流感大流行,在经历过两轮大暴发之后,逐渐成为了季节性流感病毒毒株之一[11]。pdm/09 H1N1毒株的出现进一步证实了SIV可以感染人,并引发全球大流行[12]。它在人体内的持续存在极大地改变了流感病毒流行病学和生态学的面貌[13]。尤其是近几年来,pdm/09 H1N1已经潜入到本地的猪群中,其内部基因与当地流行的SIVs发生了重配,重配后的病毒经过在猪体内的适应与进化,很有可能再次地演变成为新的流行性毒株[14]。在我们的前期研究中,He等[15]发现携带有欧亚类禽H1N1谱系(Eurasian Avian-like,EA)表面基因(HA和NA),pdm/09 H1N1谱系内部基因(PB2、PB1、PA、NP)和古典型H1N1猪流感谱系(Classical Swine H1N1,CS H1N1)内部基因NS的新型三重重配类人H3N2已经在广西的猪群中流行传播。pdm/09 H1N1的内部基因盒(internal gene cassette, IGC)已经能够通过进化适应一系列不同的表面基因,进行重组成为具有不同抗原性的病毒,这增加了人类被感染的风险[16]。通过对猪场的持续监测,2019年我们从同一个猪场再次分离获得了携带有pdm/09 H1N1内部基因盒的H3N2亚型SIVs。为进一步了解这类病毒经历6年的遗传进化规律和分子特征,我们对其进行了深入分析,为今后更好地防控SIV提供理论依据。
1 材料与方法 1.1 材料 1.1.1 细胞和病料样品犬肾细胞(Madin-Darby canine kidney cells,MDCK)由广西壮族自治区疾病预防控制中心惠赠。在前期研究中,本实验室从南宁地区某个规模化猪场先后发现并分离获得毒株A/swine/Guangxi/JGB4/2013(H3N2)和A/swine/Guangxi/JG1/2014(H3N2)[15]。2018—2019年间,本实验室再次采集了该猪场的鼻拭子、气管和肺组织等猪呼吸道症状临床样品,共计205份。
1.1.2 主要试剂dNTP、M-MLV反转录酶、DNA Marker和RNAiso Plus购自宝生物工程(大连)有限公司;Green Taq Mix酶、RNasin抑制剂购自诺唯赞生物科技有限公司;DPBS粉末购自博士德生物工程有限公司;硫酸链霉素和青霉素钠购自山东鲁抗医药股份有限公司(用于样品处理);DMEM培养液、0.05%Trypsin-EDTA购自CORNING公司;FBS血清和Pen-Strep Solution青链双抗购自Biological Industry公司(用于细胞培养)。
1.1.3 引物设计与合成利用DNAStar和Primer Premier软件,根据M基因设计一对鉴定引物M30F和M264R[17]。参照文献设计HA、NA、NP、M和NS基因扩增引物[18],其余基因分段扩增引物序列未列出[19]。引物由上海杰李生物技术有限公司合成。
1.2 方法 1.2.1 病毒RNA的提取与RT-PCR取300 μL的组织研磨液、棉拭子处理液或者细胞悬液,加入700 μL RNAiso Plus,按照说明书提取病毒总RNA。用流感病毒反转录通用引物Uni12[18],并按照M-MLV反转录酶使用说明书进行反转录合成cDNA,具体体系如下:5×Buffer 2.5 μL,2.5 mmol·L-1 dNTP mix 1 μL,Uni12 0.5 μL,M-MLV反转录酶0.25 μL,RNasin抑制剂0.25 μL,RNA模板8 μL。置PCR仪42 ℃反转录1 h。所得的cDNA进行后续PCR扩增,具体体系如下:Green Taq Mix酶12.5 μL,ddH2O 8.5 μL,M30F和M264R各0.5 μL,模板cDNA为3 μL,反应程序:95 ℃ 5 min,95 ℃ 30 s,50 ℃ 30 s,72 ℃ 30 s,72 ℃ 10 min,30个循环。针对不同基因片段调整它们的退火和延伸条件(HA、NA、M、NS、NP为59 ℃ 2 min,PA为50 ℃ 2 min 30 s,PB1和PB2为54 ℃ 3 min)。
1.2.2 病毒分离鉴定与全基因组扩增测序将鉴定为阳性样品进行过滤处理后,接种单层MDCK细胞,48 h后观察细胞病变,盲传至少3代,取细胞悬液进行RT-PCR鉴定。以反转录所得的cDNA为模板,分段扩增8个基因节段[19],并将PCR产物经胶回收纯化后送至上海生物工程股份有限公司进行测序。
1.2.3 基因序列分析及其遗传演化分析借助DNAStar中的Seqman组件进行序列拼接,并在NCBI-BLAST(http://www.ncbi.nlm.nih.gov/)上进行检索确认。利用MEGA7.0软件,采用Clustal W的方法进行序列比对,选用“邻接法”(Neighbor-Joining)构建系统发育进化树,Bootstrap设置为1 000。
1.2.4 HA蛋白同源建模分析利用SWISS-MODEL(https://swissmodel.expasy.org)对JGB4/2013、JG1/2014、JG13/2019、JG20/2019四株H3N2亚型SIVs进行HA蛋白同源建模。根据模拟后的蛋白构型变化,通过Pymlol软件分析并评估氨基酸突变对蛋白结构与功能可能产生的影响。
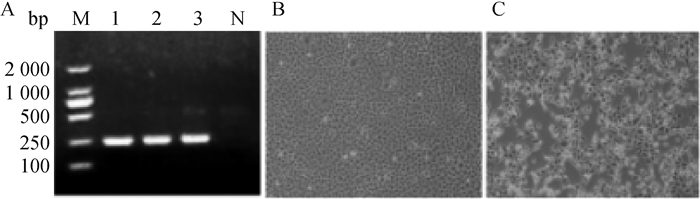
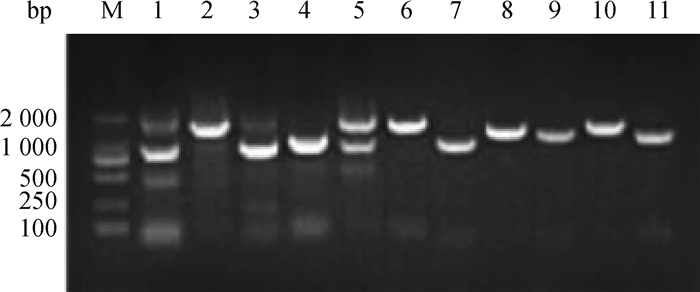
2 结果 2.1 病毒的分离鉴定和全基因组扩增本研究将RT-PCR检测为阳性的2份肺组织样品接种于单层MDCK细胞上,连续盲传3代后,结果显示,接种阳性样品的细胞出现变圆、皱缩、拉丝状等明显细胞病变(cytopathic effect,CPE),而阴性孔无变化(如图 1)。分别取上清液进行RT-PCR鉴定,结果显示扩增获得约250 bp的片段,与预期相符(如图 1)。利用特异性引物扩增病毒的全基因组,所扩增的片段大小均与理论值相符(如图 2),将这两株分离株分别命名为A/swine/Guangxi/JG13/2019(简称JG13/2019)和A/swine/Guangxi/JG20/2019(简称JG20/2019)。
|
A. RT-PCR检测(M. DL2000 DNA相对分子质量标准;1~2. JG13/2019、JG20/2019扩增产物;3.阳性对照;N.阴性对照);B.阴性对照;C.分离株造成的细胞病变 A. RT-PCR detection(M. DL2000 DNA marker; 1-2. RT-PCR products of JG13/2019 and JG20/2019;3. Positive control; N. Negative control); B. Negative control; C. The CPE induced by isolated virus 图 1 分离株的RT-PCR检测电泳图和MDCK细胞病变图 Fig. 1 Detection of SIVs by RT-PCR and the results of CPE in MDCK |
|
M. DL2000 DNA相对分子质量标准;1~11.分别为HA、NA、NP、NS、M、PA1、PA2、PB11、PB12、PB21、PB22片段的RT-PCR产物 M. DL2000 DNA marker; 1-11. RT-PCR products of HA gene, NA gene, NP gene, NS gene, M gene, PA1, PA2, PB11, PB12, PB21, PB22 segments, respectively 图 2 SIVs全基因组扩增电泳图 Fig. 2 Amplification of SIVs complete genome by RT-PCR |
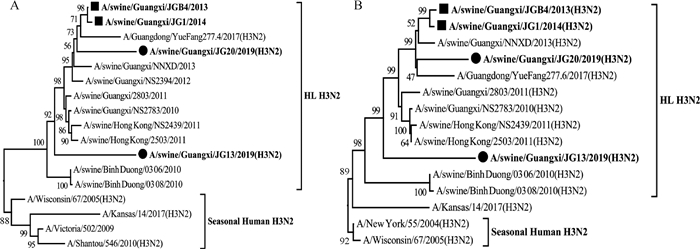
针对分离获得的2株SIVs进行RT-PCR扩增和全基因组测序(GenBank收录号为MN865146~MN865161),并构建HA和NA基因的核苷酸遗传进化树,如图 3A和B所示。序列比较结果发现,所获得毒株JG13/2019和JG20/2019均为H3N2亚型,并且与早期同一个猪场的毒株(JGB4/2013和JG1/2014)的基因型相似。表面基因HA和NA均来源于类人H3N2流感谱系(Human-Like H3N2,HL H3N2),内部基因PB2、PB1、PA、NP和M均来源于pdm/09 H1N1谱系,NS来源于CS H1N1 SIV谱系。
|
●表示本研究所分离获得的毒株;■表示前期研究分离获得的毒株 The viruses isolated in our study are shown in black circles, and those in previous study are shown in black square 图 3 分离株SIVs的HA (A)和NA (B)基因核苷酸序列系统进化树 Fig. 3 Phylogenetic trees of HA gene (A) and NA gene (B) of the isolated H3N2 SIVs |
通过对HA和NA基因的序列比较,结果发现JG13/2019与早期毒株(JGB4/2013和JG1/2014)HA基因的核苷酸相似性分别为95.3%和95.5%,而JG20/2019的核苷酸相似性均为97.4%。JG13/2019毒株的NA基因与早期毒株的核苷酸相似性相对较低,平均为93.9%,JG20/2019的核苷酸相似性为97.0%。分子特征分析发现,所获得4个毒株均为低致病性株,其HA蛋白裂解区域均为单一碱性氨基酸裂解位点。JG13/2019和JG20/2019不仅保持了早期分离株能够结合人流感病毒SAα-2, 6-Gal受体的典型位点(190D、226I和228S),而且还出现了V223I和P227S新位点上的氨基酸突变。NA蛋白的茎部区域均没有出现缺失,也都未在已经报道过的关键位点上出现E119V/I、H274Y和R292K等氨基酸的突变(如表 1所示)。
|
|
表 1 2013、2014和2019年H3N2亚型SIVs分离株之间关键氨基酸位点差异比较 Table 1 Comparison of differences at key amino acid sites among H3N2 SIV isolates in 2013, 2014 and 2019 |
内部基因(NP、M、PA、PB1和PB2)相似性比较发现,JG13/2019和JG20/2019与早期毒株的核苷酸相似性为96.2%~98.1%。NS基因的核苷酸相似性为97.1%~97.6%。序列分析发现,JG13/2019在PA蛋白上出现R356K的突变,在PB2蛋白上出现I588T的突变。而JG20/2019与早期毒株相比,却未发现明显的氨基酸突变。
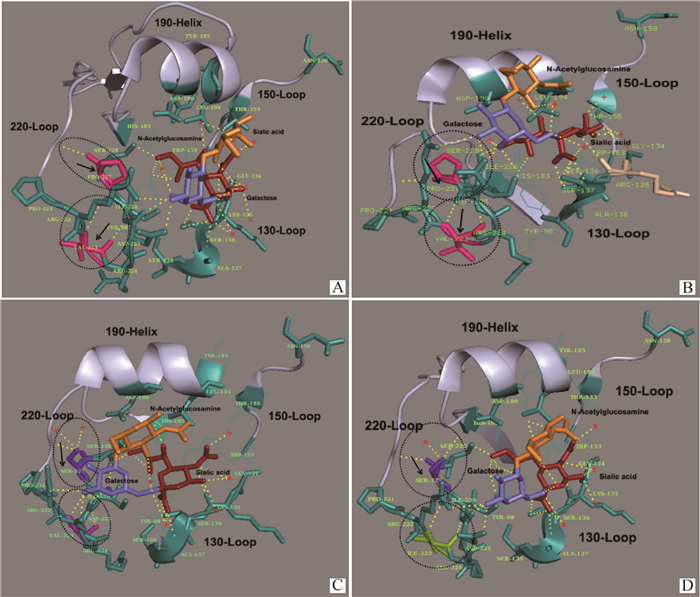
2.4 3D蛋白模型分析HA蛋白受体结合位点的氨基酸突变情况通过比较JG13/2019和JG20/2019与早期毒株JGB4/2013和JG1/2014 HA蛋白受体结合区域蛋白构象之间的差异,结果发现226位点上的异亮氨酸(I)和228位点上的丝氨酸(S),均通过氢键与N-乙酰葡萄糖胺(N-acetylglucosamine)相连接。由于227位点处于这两个关键位点之间,且与两者紧密相连,因此227位氨基酸的改变很可能会间接影响到病毒与人流感病毒受体SAα-2, 6-Gal的结合能力;疏水基团脯氨酸(P)有利于蛋白质高级结构的形成,但是因为脯氨酸具有较强的刚性,较容易出现在β-转角处,因而P227→S的突变可能会改变HA蛋白的空间构型(图 4A和B)。此外,223位点上的氨基酸通过氢键与225位点上的天冬氨酸(D)相连接,而225位上的氨基酸也同样通过氢键的方式与人源受体类似物(human receptor analog,HRA)上的半乳糖(galactose)相结合,所以223位点氨基酸的改变同样能够间接影响到病毒的受体结合能力;由于缬氨酸(V)和异亮氨酸(I)均为非极性R基因氨基酸,属于疏水基团,参与维持蛋白质的结构与功能,本研究中V223→I的突变并没有导致223位点和225位点之间氢键的断裂,由此推断这种突变对于HA蛋白的构象,特别是受体结合域的影响可能不显著(图 4C和D)。
|
A. JGB4/2013; B. JG1/2014; C. JG13/2019; D. JG20/2019;黑色箭头指示的为本研究中HA蛋白受体结合位点上的突变;黄色点状虚线代表氢键 A. JGB4/2013; B. JG 1/2014; C. JG13/2019; D. JG20/2019;Black arrow points out the mutation of receptor binding site in HA protein; Yellow dot line stands for hydrogen bonds 图 4 分离株SIVs HA蛋白受体结合区域的氨基酸突变模型 Fig. 4 Structural model for the amino acid mutations of the isolated SIVs in HA protein's receptor binding domain |
2009年甲型H1N1流感大流行过后,一种携带了pdm/09 H1N1内部基因片段的新型H3N2重配“变异毒株”在美国的猪群中被鉴定出来,该病毒于2011年7月首次出现在美国的人群中,随后在美国多地造成了零星感染甚至局部暴发[20]。该病毒基因组包括了来自禽、人、猪流感病毒的基因片段以及pdm/09 H1N1毒株的M基因。研究证实,正是这种M基因的获得使得病毒比其他SIV更容易感染人[21-22]。在此之后,越来越多不同重配方式的H3N2甲流重组变异株在世界范围内的猪群中被鉴定出来,它们均携带有1~6个pdm/09 H1N1的内部基因片段。美国疾病防控中心(Centers for Disease Control and Prevention,CDC)将这类重组病毒称作“H3N2v(H3N2 Variants)”。H3N2v通常在猪体内循环,于特定的环境下感染人,并有过导致人患严重肺炎而死亡的报道[23]。H3N2v不仅具备人季节性H3N2流感的表面基因HA和NA,同时还具备pdm/09 H1N1流感的内部基因,因而更可能在人群中快速、高效和持续地传播。季节性流感疫苗不能预防H3N2v,且年龄≤9岁的儿童对H3N2v病毒几乎没有免疫力[20]。
目前,中国各地陆续鉴定出携带多个pdm/09 H1N1基因片段的新型甲流重组SIVs的报道。Liang等[24]在2009—2012年期间总共分离得到了387株SIVs,并从中鉴定出17种携带有pdm/09 H1N1基因片段的不同重配病毒,其中H3N2v病毒占4种[25]。He等[15]在2013年至2015年期间从广西的猪群中分离出共计15株SIVs,并进一步鉴定出5种含有pdm/09 H1N1基因片段的不同重组基因型,其中H3N2v病毒占2种。
通过对同一个猪场的持续监测,时隔6年,本研究连续分离获得两株H3N2亚型SIVs(JG13/2019和JG20/2019),它们与早期分离的JGB4/2013和JGB1/2014毒株的基因型完全一致。进一步证实pdm/09 H1N1内部基因盒已经在这些毒株中稳定存在,而且这些毒株已经逐渐发展成为地方优势毒株,在本地猪群中广泛流行。核苷酸同源性分析表明,JG13/2019和JG20/2019分别与JGB4/2013和JG1/2014相比较,均出现了一定程度的变异,其中JG13/2019的变异程度较大,平均相似性仅为95.4%。分子特征分析发现,JG13/2019和JG20/2019均保留了重要位点上的氨基酸功能特性,主要包括HA蛋白上(190D、226I和228S)能够结合人流感病毒SAα-2, 6-Gal受体的特异性[26-28],NA蛋白上(E119V/I、H274Y、R292K)对神经氨酸酶抑制剂类药物的敏感性[29-30],M2蛋白上(31N)对金刚烷胺类药物的耐药性[31],PB1蛋白上(622G)[32]、PB2蛋白上(271A、590S、591R)增强病毒致病力和跨种传播能力的特性[33]。有趣的是,与早期毒株(JGB4/2013和JG1/2014)相比,JG13/2019在HA蛋白上出现了P227S的改变,在PA蛋白上出现了R356K的改变,在PB2蛋白上出现了I588T等一系列新的氨基酸突变,而JG20/2019则是在HA蛋白上223I和227S同过去的毒株有所不同。虽然目前极少有关HA蛋白上223位和227位上氨基酸功能的报道,但是它们与HA蛋白上受体结合位点(receptor binding site,RBS)构成相关联,是否影响其受体结合特性需要进一步验证。流感病毒的RNA聚合酶复合体蛋白是由PB2蛋白、PB1蛋白以及PA蛋白三个亚单位组成,它们均参与病毒基因组的复制和转录,同病毒的感染性和致病力密切相关。研究表明,H9N2亚型禽流感病毒PA蛋白上出现的K356R氨基酸变化能够提升病毒本身的复制能力以及对哺乳动物的致病性[34]。pdm/09 H1N1病毒PB2蛋白上出现的T558I氨基酸突变可以通过提高病毒复制水平和加强对β干扰素表达的抑制作用来增强病毒自身的毒力[35],而JG13/2019在这些位点上的突变是否会影响其复制能力和致病性,也需要体内外试验加以验证。综上所述,我们推测JG13/2019对哺乳动物的致病力相较于JGB4/2013和JG1/2014可能会有所下降,而且JG13/2019和JG20/2019的受体结合特性相较于过去的毒株可能会有所改变。
2019年,Lu等[36]最新报道了一起有关“新型三源重组H3N2亚型SIV感染人”的事件。该毒株(A/Guangdong/YueFang277/2017/H3N2,简称GD-YF)是从一个10岁女童的鼻咽拭子中鉴定出来的,其内部基因仍旧携带了pdm/09 H1N1的内部基因盒(PB2、PB1、PA和NP基因),表面基因HA和NA来源于HL-H3N2谱系。同源性分析进一步显示了,猪源H3N2亚型人流感病毒株GD-YF的HA和NA基因同2012—2019年期间流行在我国广东、广西、香港等南方地区的H3N2亚型SIV的亲缘关系比较接近,这表明了H3N2亚型SIV在这些地方的猪群中建立了稳定的遗传谱系,对人类的健康构成了潜在的威胁。
4 结论2018年至2019年,通过对广西地区猪场的监测,本研究发现猪场一直持续流行一种以类人谱系为表面基因(HA和NA)和pdm/09 H1N1谱系为内部基因(PB2、PB1、PA、M、NP)的H3N2亚型SIV,而且这种携带pdm/09 H1N1内部基因盒的H3N2亚型流行毒株已经获得了选择优势,并在猪群中建立起了稳定的遗传谱系,仍然保持感染人受体的结合特性。因此,继续加强对这种三重重配SIV的监测,及时了解其遗传进化规律和分子特征,将为今后防控人流感大暴发提供重要的科学依据。
| [1] | SANDROCK C. Influenza[M]//MAINOUS Ⅲ A G, POMEROY C. Management of Antimicrobials in Infectious Diseases. Totowa: Humana Press, 2010: 109-126. |
| [2] | JAFRI S S I, ILYAS M, IDREES M. Swine flu: a threat to human health[J]. Biotechnol Mol Biol Rev, 2010, 5(3): 46–50. |
| [3] | WEN F, YU H, YANG F R, et al. Efficacy of a high-growth reassortant H1N1 influenza virus vaccine against the classical swine H1N1 subtype influenza virus in mice and pigs[J]. Arch Virol, 2014, 159(11): 2957–2967. |
| [4] | BROWN I H. The epidemiology and evolution of influenza viruses in pigs[J]. Vet Microbiol, 2000, 74(1-2): 29–46. |
| [5] |
陈樱, 於庆雄, 银凤桂, 等. 2009~2013年广西H1N1和H3N2亚型猪流感病毒血清学调查[J]. 南方农业学报, 2015, 46(1): 155–159.
CHEN Y, YU Q X, YIN F G, et al. Serological investigation of H1N1 and H3N2 subtype swine influenza virus in Guangxi from 2009 to 2013[J]. Journal of Southern Agriculture, 2015, 46(1): 155–159. (in Chinese) |
| [6] | SUZUKI Y. Receptor sialylsugar chains as determinants of host range of influenza viruses[J]. Nihon Rinsho, 2000, 58(11): 2206–2210. |
| [7] | BROWN I H. The pig as an intermediate host for influenza A viruses between birds and humans[J]. Int Congr Ser, 2001, 1219: 173–178. |
| [8] | MA W J, KAHN R E, RICHT J A. The pig as a mixing vessel for influenza viruses: human and veterinary implications[J]. J Mol Genet Med, 2009, 3(1): 158–166. |
| [9] |
顾春英, 张宏伟, 曹广文. 2009年新型甲型H1N1流感病毒进化过程中猪作为宿主及"混合器"的作用[J]. 第二军医大学学报, 2009, 29(6): 605–609.
GU C Y, ZHANG H W, CAO G W. Role of swine as host and a mixing vessel in evolution of 2009 novel influenza virus A/H1N1[J]. Academic Journal of Second Military Medical University, 2009, 29(6): 605–609. (in Chinese) |
| [10] | HAQUE N, BARI M S, BILKIS L, et al. Swine flu: a new emerging disease[J]. Mymensingh Med J, 2010, 19(1): 144–149. |
| [11] | SCALERA N M, MOSSAD S B. The first pandemic of the 21st century: review of the 2009 pandemic variant influenza A (H1N1) virus[J]. Postgrad Med, 2009, 121(5): 43–47. |
| [12] | GARTEN R J, DAVIS C T, RUSSELL C A, et al. Antigenic and genetic characteristics of swine-origin 2009 A(H1N1) influenza viruses circulating in humans[J]. Science, 2009, 325(5937): 197–201. |
| [13] | KONG W L, WANG F B, DONG B, et al. Novel reassortant influenza viruses between pandemic (H1N1) 2009 and other influenza viruses pose a risk to public health[J]. Microb Pathog, 2015, 89: 62–72. |
| [14] | VIJAYKRISHNA D, POON L L M, ZHU H C, et al. Reassortment of pandemic H1N1/2009 influenza a virus in swine[J]. Science, 2010, 328(5985): 1529. |
| [15] | HE P, WANG G J, MO Y N, et al. Novel triple-reassortant influenza viruses in pigs, Guangxi, China[J]. Emerg Microbes Infect, 2018, 7(1): 85. |
| [16] | FAN X H, ZHU H C, ZHOU B P, et al. Emergence and dissemination of a swine H3N2 reassortant influenza virus with 2009 pandemic H1N1 genes in pigs in China[J]. J Virol, 2012, 86(4): 2375–2378. |
| [17] | FOUCHIER R A M, BESTEBROER T M, HERFST S, et al. Detection of influenza A viruses from different species by PCR amplification of conserved sequences in the matrix gene[J]. J Clin Microbiol, 2000, 38(11): 4096–4101. |
| [18] | HOFFMANN E, STECH J, GUAN Y, et al. Universal primer set for the full-length amplification of all influenza A viruses[J]. Arch Virol, 2001, 146(12): 2275–2289. |
| [19] |
莫彦宁.广西H3N2亚型和H1N1亚型宠物犬流感病毒的分子流行病学调查及其遗传进化分析[D].南宁: 广西大学, 2014.
MO Y N. Molecular epidemiology and phylogenetic analysis of H3N2 and H1N1 influenza among pet dogs in Guangxi[D]. Nanning: Guangxi University, 2014. (in Chinese) http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D523872 |
| [20] | Centers for Disease Control and Prevention. Specific Variant Flu Viruses [EB/OL]. https://www.cdc.gov/flu/swineflu/variant/index.html?CDC_AA_refVal=https%3A%2F%2Fwww.cdc.gov%2Fflu%2Fswineflu%2Fh3n2v-case-count.htm. [2020-01-30] |
| [21] | LINDSTROM S, GARTEN R, BALISH A, et al. Human infections with novel reassortant influenza A(H3N2)v viruses, United States, 2011[J]. Emerg Infect Dis, 2012, 18(5): 834–837. |
| [22] | BOWMAN A A, NELSON S W, PAGE S L, et al. Swine-to-human transmission of influenza A(H3N2) virus at agricultural fairs, Ohio, USA, 2012[J]. Emerg Infect Dis, 2014, 20(9): 1472–1480. |
| [23] | MA J J, SHEN H G, LIU Q F, et al. Pathogenicity and transmissibility of novel reassortant H3N2 influenza viruses with 2009 pandemic H1N1 genes in pigs[J]. J Virol, 2015, 89(5): 2831–2841. |
| [24] | LIANG H Y, LAM T T Y, FAN X H, et al. Expansion of genotypic diversity and establishment of 2009 H1N1 pandemic-origin internal genes in pigs in China[J]. J Virol, 2014, 88(18): 10864–10874. |
| [25] |
高灵茜, 崔梦一, 樊晓晖. 猪流感病毒基因池中的新成员:新甲型H1N1流感病毒基因片段的存在及潜在威胁[J]. 中国人兽共患病学报, 2016, 32(9): 832–837.
GAO L Q, CUI M Y, FAN X H. The new member of the swine influenza virus gene pool: the existence of the 2009 pandemic H1N1 virus gene fragments and potential threats[J]. Chinese Journal of Zoonoses, 2016, 32(9): 832–837. (in Chinese) |
| [26] | TENG Q Y, XU D W, SHEN W X, et al. A single mutation at position 190 in hemagglutinin enhances binding affinity for human type sialic acid receptor and replication of H9N2 avian influenza virus in mice[J]. J Virol, 2016, 90(21): 9806–9825. |
| [27] | WAN H Q, PEREZ D R. Amino acid 226 in the hemagglutinin of H9N2 influenza viruses determines cell tropism and replication in human airway epithelial cells[J]. J Virol, 2007, 81(10): 5181–5191. |
| [28] | RAMOS I, BERNAL-RUBIO D, DURHAM N, et al. Effects of receptor binding specificity of avian influenza virus on the human innate immune response[J]. J Virol, 2011, 85(9): 4421–4431. |
| [29] | OKOMO-ADHIAMBO M, DEMMLER-HARRISON G J, DEYDE V M, et al. Detection of E119V and E119I mutations in influenza A (H3N2) viruses isolated from an immunocompromised patient: challenges in diagnosis of oseltamivir resistance[J]. Antimicrob Agents Chemother, 2010, 54(5): 1834–1841. |
| [30] | STEAIN M C, DWYER D E, HURT A C, et al. Detection of influenza A H1N1 and H3N2 mutations conferring resistance to oseltamivir using rolling circle amplification[J]. Antiviral Res, 2009, 84(3): 242–248. |
| [31] | SAITO R, LI D J, SHIMOMURA C, et al. An off-seasonal amantadine-resistant H3N2 influenza outbreak in Japan[J]. Tohoku J Exp Med, 2006, 210(1): 21–27. |
| [32] | FENG X X, WANG Z, SHI J Z, et al. Glycine at position 622 in PB1 contributes to the virulence of H5N1 avian influenza virus in mice[J]. J Virol, 2016, 90(4): 1872–1879. |
| [33] | LIU Q F, QIAO C L, MARJUKI H, et al. Combination of PB2 271A and SR polymorphism at positions 590/591 is critical for viral replication and virulence of swine influenza virus in cultured cells and in vivo[J]. J Virol, 2012, 86(2): 1233–1237. |
| [34] | XU G L, ZHANG X X, GAO W H, et al. Prevailing PA mutation K356R in avian influenza H9N2 virus increases mammalian replication and pathogenicity[J]. J Virol, 2016, 90(18): 8105–8114. |
| [35] | ZHAO Z Z, YI C Y, ZHAO L Z, et al. PB2-588I enhances 2009 H1N1 pandemic influenza virus virulence by increasing viral replication and exacerbating PB2 inhibition of beta interferon expression[J]. J Virol, 2014, 88(4): 2260–2267. |
| [36] | LU J, YI L N, JING Y, et al. A human infection with a novel reassortant H3N2 swine virus in China[J]. J Infect, 2019, 79(2): 174–187. |



