2. 中国农业科学院北京畜牧兽医研究所, 农业农村部动物遗传育种与繁殖重点实验室, 北京 100193
2. Key Laboratory of Animal Genetics, Breeding and Reproduction of Ministry of Agriculture and Rural Affairs, Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193, China
绵羊是最主要的经济动物之一,在生产中羊角因易造成家畜或人员伤害而制约着产业的发展,通过人工去角违背动物福利且增加工作量,故不可取。绵羊拥有多个无角品种,如澳洲美利奴羊、湖羊[1],说明有无角受到遗传基因控制,通过分子育种筛选无角绵羊是目前绵羊产业的一个重要发展方向。因此,筛选绵羊角功能基因和分子标记对生产和研究都具有重大价值。
胰岛素样因子3(insulin-like 3,INSL3)属于胰岛素样激素超家族。在人和小鼠中,INSL3与睾丸沉降[2-3]、生殖细胞存活[4]、骨代谢调控相关[5-6],还能加速伤口愈合[7]。男性血液中INSL3浓度随着睾丸发育而升高,在青春期达到最高,随着睾丸衰老,浓度不断降低,因此INSL3成为了一种新型的性腺功能和分化的标记物[8-10]。该基因主要在性腺组织中表达,但存在典型的性别特异性,在雄性胎儿羊水中可以检测到INSL3,雌性胎儿则检测不到[11-12]。而且在牛上的研究发现,INSL3主要在卵巢和睾丸中表达,并发现它是雄性胎儿一种主要的性别特异性循环激素[13-14]。在绵羊中,INSL3基因相关功能研究较少,仅Anand-Ivell等[15]通过特异的酶联免疫试验检测了绵羊血液中INSL3的表达,发现未怀孕母羊INSL3的表达量是怀孕母羊的4倍。然而,INSL3基因的受体基因松弛素亚族肽受体2(RXFP2)在绵羊一直被广泛关注,RXFP2基因不仅影响角的有无[16],也影响角的长度和周长[17],最近通过重测序发现,RXFP2基因上的一个错义突变与绵羊角大小密切相关[18],因此,作为RXFP2配体的INSL3可能也与绵羊角有关。
角为头部皮肤的衍生物,是一个完整独立的器官。绵羊角属于洞角,由角质外鞘和骨质角心构成。Dove[19]通过初生小牛的角牙组织移殖试验,发现骨心的形成是角发育的关键点。在人和小鼠中,INSL3- RXFP2信号直接影响骨代谢,绵羊角的骨心是由骨质层、间质层和髓质层的细胞构成,INSL3是否参与了绵羊角的骨心构建以及在哪一层起着最重要作用仍然未知。有研究发现,INSL3-RXFP2信号参与骨代谢是通过作用于MAPK通路实现的,影响到下游ALP、COL1A1、COL6A1、SPARC、BSP-1和TGF-β 等基因的表达[20]。随后的研究发现,INSL3 -RXFP2信号能使得维生素D实现25羟基化,维生素D与人类佝偻病有关,调控体内钙浓度,促使骨的矿物化[21],使用25-羟维生素D3可以显著治疗病人腰椎的骨质疏松症[22-23]。而最近一些研究表明,INSL3在骨生成中起着重要作用[24-25]。
以上研究表明,INSL3可能是影响绵羊角骨形成的功能基因,但仍需验证。组织表达谱和SNP关联分析是探究基因功能的方式,可以初步判断基因在绵羊上的主要功能。本课题组在关联和功能分析上做了一些研究[26-28],因此,本研究拟使用荧光定量PCR对绵羊INSL3基因组织表达谱进行探究,并通过10个绵羊群体重测序数据SNP位点分析,初步解析该基因在绵羊上的功能。
1 材料与方法 1.1 试验动物和组织样品采集选取发育良好、健康、角型正常、1岁的小尾寒羊成年母羊和公羊各3只,所有羊购于山东郓城小尾寒羊种羊场,公母羊饲养于相同环境和条件。绵羊经屠宰后立即采样,分别采集软角、角骨膜、心、肾、卵巢、子宫、大脑皮层、骨骼肌等组织,置于冻存管内,放入液氮中保存。
1.2 RNA提取和反转录使用Trizol (Thermo Scientific,Wilmington,USA)法提取组织总RNA,DNase Ⅰ(天根,北京)处理,应用NANODROP 2000 (Thermo Scientific,Wilmington,USA)检测RNA浓度和1%琼脂糖凝胶电泳进行质量检测,RNA置于-80 ℃。
根据PrimeScript TM RT reagent kit反转录试剂盒(RR047A,TaKaRa,大连)操作程序进行反转录。
1.3 引物设计INSL3基因引物设计参照NCBI(http://www.ncbi.nlm.nih.gov/)中NM_001024738.1序列,利用Primer express 3.0软件设计,由宝生物工程(大连)有限公司合成。引物序列和大小见表 1。
|
|
表 1 Real-time PCR引物 Table 1 Primers used for real-time PCR |
试剂购于天根生化科技(北京)有限公司,PCR扩增反应体系为25 μL:10×PCR Buffer(+MgCl2) 2.5 μL,dNTPs 2 μL,PCR Forward Primer 0.5 μL,PCR Reverse Primer 0.5 μL,Taq DNA polymerase 0.125 μL,cDNA 1 μL,ddH2O 18.375μL。反应条件:95 ℃预变性8 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,循环33次;72 ℃延伸5 min;4 ℃保存。PCR产物送至宝生物工程(大连)有限公司测序。
1.5 INSL3基因实时荧光定量PCR检测以制备的各组织样品cDNA为模板。利用表 1中的引物对β-actin和INSL3基因在不同组织中的表达量分别进行分析。实时荧光定量PCR使用SYBR Premix TaqTM Ⅱ荧光定量试剂盒(TaKaRa,大连)和罗氏480荧光定量仪,反应体系为20 μL:SYBR Premix Ex TaqTM Ⅱ(2×)10 μL,PCR Forward Primer 0.8 μL,PCR Reverse Primer 0.8 μL,cDNA 2 μL,ddH2O 6.4 μL。程序:95 ℃ 5 min; 95 ℃ 15 s,60 ℃ 60 s,40个循环;熔解曲线分析。每个样品设置3个技术重复,并设置阴性对照(ddH2O)。
1.6 INSL3在不同物种中的表达谱分析进入EBI数据库(https://www.ebi.ac.uk/gxa/home),输入INSL3进行搜索,结果展示了INSL3在不同物种不同组织中RNA-seq的表达数据,选择较为完善的群体,分别下载人[29]、猕猴[30]、小鼠[31]、大鼠[32]、牛[30]、绵羊[33]的TPM表达值数据,通过R软件构建热图。
1.7 INSL3区域主成分分析PCA前期通过重测序,获得了10个品种99只绵羊个体的全基因组SNPs位点[18, 34],使用linux cat命令将INSL3基因及其上下游各2 000 bp所有SNPs位点调出,10个品种分别为6个蒙古系绵羊(小尾寒羊STH、乌珠穆沁羊WZ、滩羊T、湖羊H、策勒黑羊CB和巴音布鲁克羊BY)、3个藏系绵羊品种(河谷型藏羊VT、草原型藏羊PT、欧拉羊OL)和1个欧洲绵羊(澳洲美利奴羊AM)。其中湖羊和澳洲美利奴羊完全无角,小尾寒羊、乌珠穆沁羊、滩羊、策勒黑羊和巴音布鲁克羊部分有角,河谷型藏羊、草原型藏羊和欧拉羊有角。计算各品种所有SNP位点等位基因频率,将其中一个等位基因频率导入R 3.61软件中,使用R软件prcomp程序计算PCA,并作图。
1.8 INSL3基因潜在功能位点查找利用上述10个群体数据,计算各群体差异值
通过UCSC数据库获取INSL3上下游2 000 bp序列,将上游2 000 bp序列放入AnimalTFDB 3.0[35]进行转录因子结合位点预测,对获得的因子进行筛选,可信分值(score)>17,P-value<0.01,Q-value<0.01。将下游序列放入FindTerm(http://www.softberry.com/berry.phtml?topic=indterm&group=programs&subgroup=gfindb)寻找终止子。
1.10 数据处理与统计分析相对定量结果采用2-ΔΔCt方法处理,差异显著性分析使用SPSS 17.0软件T检验。
2 结果 2.1 RT-PCR检测INSL3在绵羊各组织的表达以各组织总RNA的反转录产物cDNA为模板,采用普通PCR法对所设计的2对引物(INSL3、β-actin)进行扩增,经1%的琼脂糖凝胶电泳检测,结果见图 1。目的基因的PCR扩增产物条带清晰明亮,无杂带,说明产物具有很高的特异性,同时结果表明INSL3具有广泛性表达,在所检测多组织中均有表达。

|
M.DNA相对分子质量标准;1~8.心、肌肉、卵巢/睾丸、子宫、肾、大脑皮层、角骨膜、软角组织 M.DL2000 marker; 1-8. Heart, muscle, ovary/testis, uterus, kidney, cerebrum, horn periosteum, soft horn 图 1 INSL3和β-actin基因RT-PCR产物琼脂糖电泳 Fig. 1 The agarose electrophoresis of RT-PCR products of INSL3 and β-actin genes |
荧光定量结果(图 2)表明,INSL3基因在各组织均表达,这与RT-PCR结果一致;在卵巢和睾丸中表达量最高,在软角组织和心中也有较高表达,其它具有少量表达。公羊睾丸表达量极显著高于母羊卵巢组织(P<0.01),公羊软角组织表达显著高于母羊(P<0.05)。
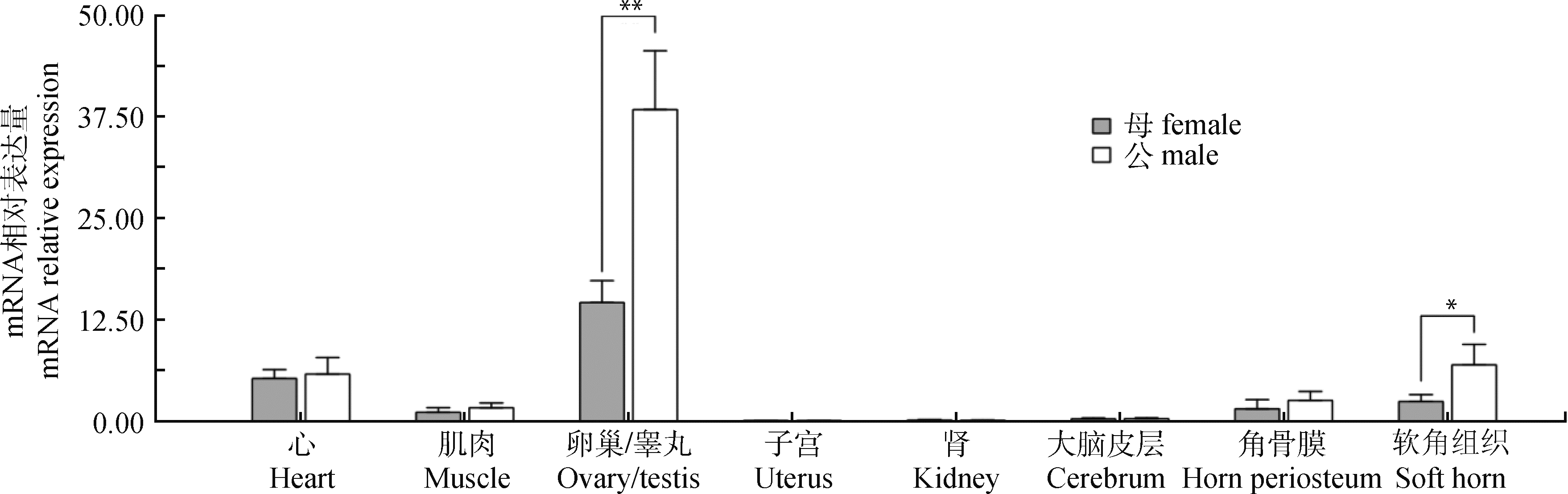
|
**.差异极显著(P<0.01);*.差异显著(P<0.05) **. Means extremely significant difference (P < 0.01); *. Means significant difference (P < 0.05) 图 2 INSL3基因在公母羊不同组织中的表达 Fig. 2 The mRNA expression of INSL3 in tissues of male and female sheep |
由结果(图 3)可以看出,绵羊INSL3在不同组织之间特异性表达非常明显,在睾丸和卵巢中特异性高表达,说明该基因与绵羊的繁殖力相关。同时,INSL3具有高度的表达保守性,在不同物种间表达规律基本一致,都是在睾丸和卵巢高表达。
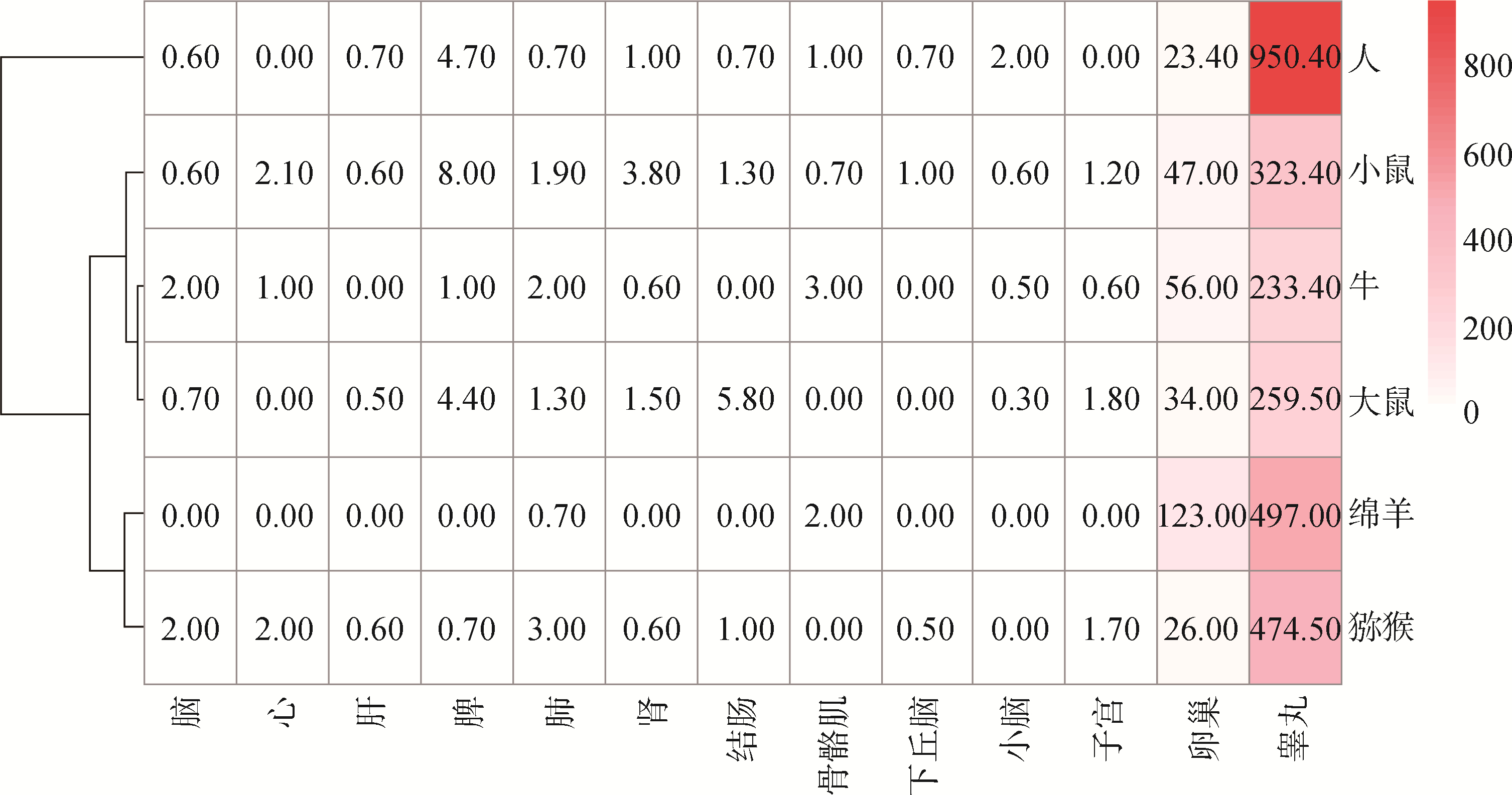
|
图中的数字来自于RNA-seq的TPM值 The number in the figure is TPM values from RNA-seq 图 3 INSL3基因在不同物种不同组织中的RNA-seq表达差异 Fig. 3 The difference of RNA-seq expression of INSL3 gene in different tissues of different species |
根据PCA结果(图 4),无角的澳洲美利奴羊(AM)和湖羊(H)靠得最近,小尾寒羊(STH)母羊无角或小角,因此也靠近湖羊。而藏羊中尤其是草原型藏羊PT离的最远,可能是因为该群体大部分有角。该结果表明,此区域突变位点虽不能完全将有角或无角分开,但在一定程度上能分开角区别非常明显的群体,说明INSL3基因区域SNP位点在有角或无角性状中可能起到一定作用。
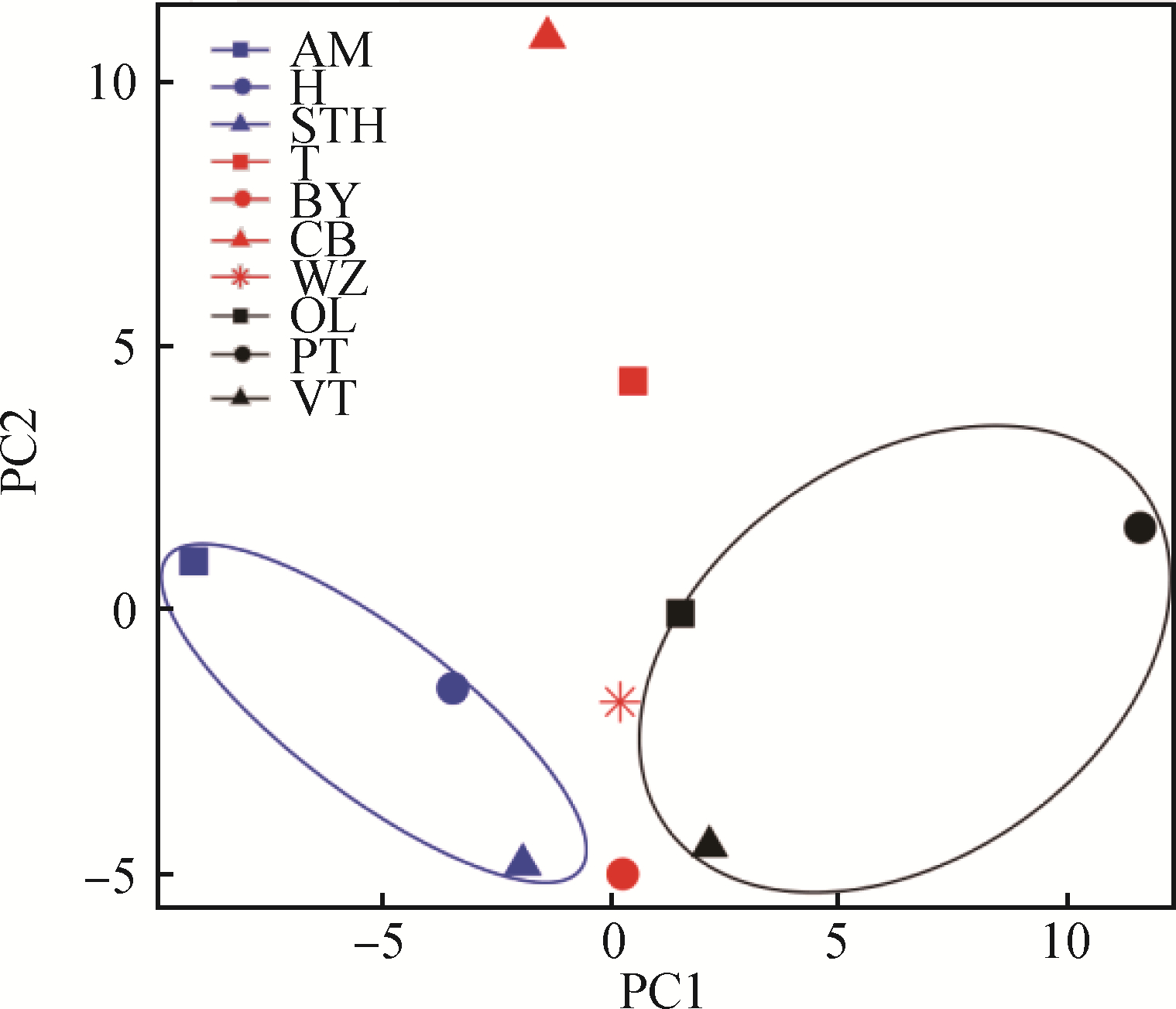
|
STH.小尾寒羊;WZ.乌珠穆沁羊;T.滩羊;H.湖羊;CB.策勒黑羊;BY.巴音布鲁克羊;VT.河谷型藏羊;PT.草原型藏羊;OL.欧拉羊;AM.澳洲美利奴羊 STH. Small-tailed Han; WZ. Wuzhumuqin; T. Tan; H. Hu; CB. Cele Black; BY. Bayinbuluke; VT. Valley Tibetan; PT. Prairie Tibetan; OL. Oula; AM. Australian Merino 图 4 INSL3基因区域群体主成分分析 Fig. 4 Principal component analysis (PCA) of INSL3 gene region |
通过筛选,初步确定了6个潜在功能位点(表 2)。SNP1、SNP2、SNP3位于启动子上游区域,具有较高的Fst值,说明这些位点在有角和无角群体中分化显著,SNP4是INSL3上的错义突变,该位点在有角和无角群体中分化不明显,但也有可能在某个群体中起作用。SNP5是同义突变,该位点具有较高遗传多样性,但在有角和无角群体中分化不明显,可能与其他性状相关。SNP6位于基因下游,该位点同样在有角和无角群体中分化明显。
|
|
表 2 INSL3基因潜在功能位点 Table 2 Potential functional loci of INSL3 gene |
通过AnimalTFDB
3.0预测,并通过严格的筛选,发现SNP1(5 054 755)处于KDM1A转录因子结合位点上,而SNP3(5 054 996)位于ESR1结合位点。终止子预测结果显示SNP6(5 059 168)处于终止子区域(图 5、图 6)。
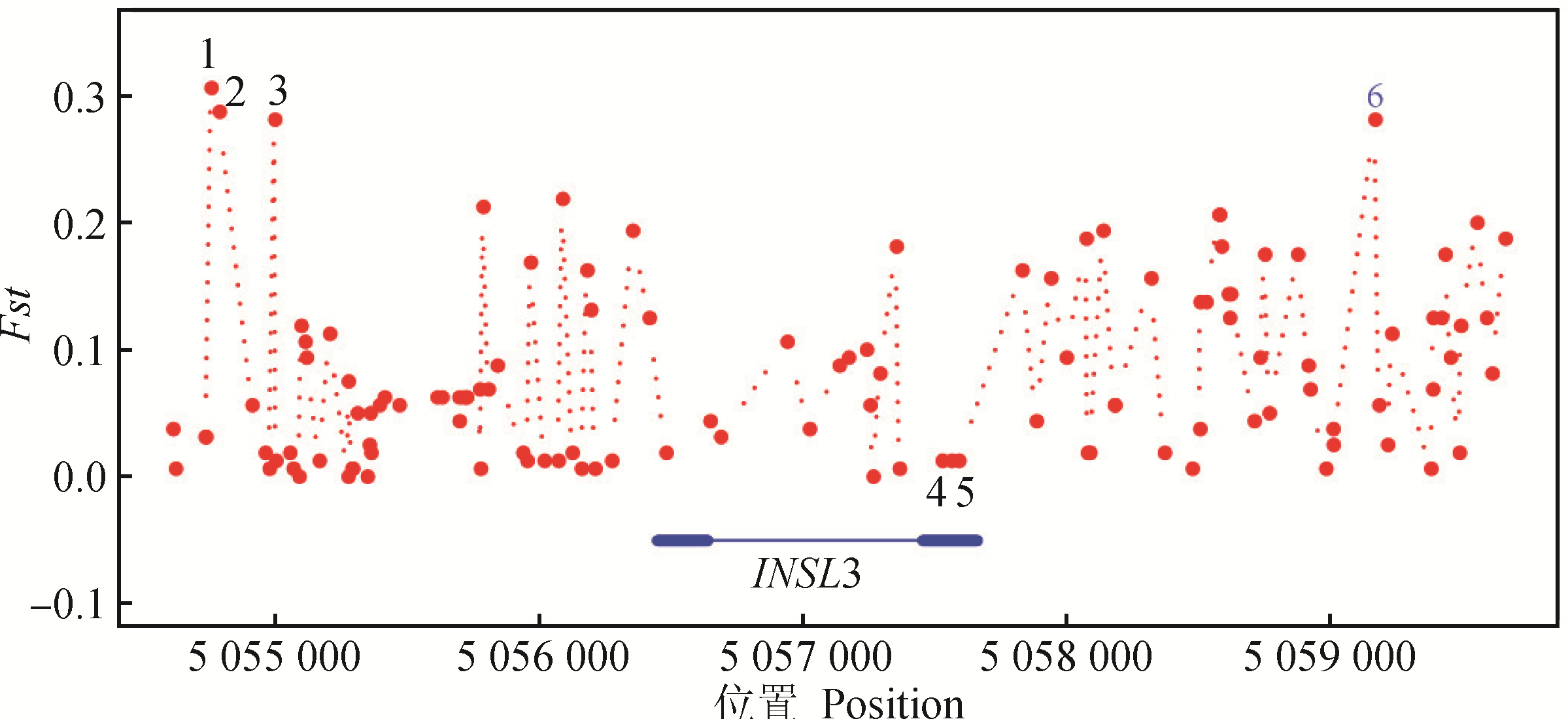
|
图 5 INSL3基因区域SNPs差异分布(Fst) Fig. 5 Difference distribution (Fst) of SNPs in INSL3 gene region |

|
图 6 INSL3关键位点所处转录调控位点 Fig. 6 Transcriptional regulatory sites of the key SNPs in INSL3 |
INSL3属于类胰岛素超家族成员。在小鼠上的研究发现,INSL3敲除后会出现隐睾症、卵泡闭锁加快和黄体提前凋亡的现象[36],说明该基因对生殖功能非常重要。本研究发现,INSL3基因在卵巢和睾丸高表达,说明该基因确实与生殖功能密切相关。而角作为绵羊一种特殊的生殖特征,也跟生殖密切相关,在野生环境下,公羊通过显耀角或者通过角斗来获得交配权利[37],这初步说明了INSL3可能与角有关。角在公羊和母羊中存在显著差异,公羊往往带有巨大的角,而母羊的角相对小很多。本研究结果发现,INSL3在公羊睾丸表达量极显著高于母羊卵巢组织(P<0.01),这表明公羊巨大的角有可能是其高表达INSL3导致,而本研究公羊软角组织表达显著高于母羊(P<0.05)也证明了这一点。因此可以推测,在自然选择过程中,获得INSL3高水平表达的公羊个体因为角更大,而得到交配机会,从而保留下来完成进化。本研究结果发现,在有角和无角群体中分化显著的SNP位点都位于上下游调控区,这进一步说明该基因是通过表达来发挥功能的。
根据重测序数据PCA分析发现,在一定程度上能分开有角或无角别非常明显的群体,以往的研究发现,RXFP2与角的有无有关,但发现该基因仅能解释部分现象[38],说明有角和无角并不是单位点或单基因控制的性状,而本研究发现INSL3可能也与角的有无相关。
本研究发现,SNP1(5 054 755)位点处于赖氨酸特异性去甲基酶1A(lysine (K)-specific demethylase 1A,KDM1A)转录因子结合位点上,KDM1A是第一个被发现的组蛋白去甲基化酶[39],这种酶在组织特异性分化以及卵母细胞生长中具有重要作用[40],这与本研究结果INSL3在卵巢中高表达相符,说明SNP1极可能起调控表达作用。SNP3(5 054 996)位于雌激素受体α(ESR1)结合位点,ESR1是由雌激素激活的核受体,通过接受激素的信号来调控核内基因的转录[41],雌激素是众所周知的性激素,而INSL3基因又与生殖有关,SNP3位点突变很容易改变与ESR1基因结合,进一步影响基因的表达,所以该位点也很有可能是一个功能位点。一些研究表明,3′UTR区域的SNP会影响基因的表达[42-43],而SNP6(5 059 168)处于3′UTR终止子区域,因为在有角和无角群体中存在差异分布,所以该位点也有可能是一个功能位点。
4 结论本研究发现,INSL3在绵羊卵巢和睾丸表达量最高,在软角和心脏组织中也有较高表达。INSL3可能与公母绵羊角大小的差异有关,同时也可能与角的有无有关。本研究发现了多个潜在功能SNPs位点,其中3个最有可能与角性状存在关联,分别是处于KDM1A转录因子结合位点上的SNP1、位于ESR1结合位点的SNP3、处于终止子区域的SNP6。此结果为今后研究INSL3功能机制奠定基础。
| [1] |
国家畜禽遗传资源委员会.
中国畜禽遗传资源志-羊志[M]. 北京: 中国农业出版社, 2011: 1-16.
National Commission of Livestock and Poultry Genetic Resources. Animal genetic resources in China:sheep and goats[M]. Beijing: China Agriculture Press, 2011: 1-16. (in Chinese) |
| [2] | NEF S, PARADA L F. Cryptorchidism in mice mutant for Insl3[J]. Nat Genet, 1999, 22(3): 295–299. DOI: 10.1038/10364 |
| [3] | FENG S, FERLIN A, TRUONG A, et al. INSL3/RXFP2 signaling in testicular descent:mice and men[J]. Ann N Y Acad Sci, 2009, 1160(1): 197–204. DOI: 10.1111/j.1749-6632.2009.03841.x |
| [4] | IVELL R, ANAND-IVELL R. Insulin-like peptide 3 (INSL3) is a major regulator of female reproductive physiology[J]. Hum Reprod Update, 2018, 24(6): 639–651. DOI: 10.1093/humupd/dmy029 |
| [5] | FERLIN A, DE TONI L, AGOULNIK A I, et al. Protective role of testicular hormone INSL3 from atrophy and weakness in skeletal muscle[J]. Front Endocrinol, 2018, 9: 562. DOI: 10.3389/fendo.2018.00562 |
| [6] | IVELL R, BATHGATE R A D. Reproductive biology of the relaxin-like factor (RLF/INSL3)[J]. Biol Reprod, 2002, 67(3): 699–705. DOI: 10.1095/biolreprod.102.005199 |
| [7] | HAMPEL U, KLONISCH T, SEL S, et al. Insulin-like factor 3 promotes wound healing at the ocular surface[J]. Endocrinology, 2013, 154(6): 2034–2045. DOI: 10.1210/en.2012-2201 |
| [8] | IVELL R, ANAND-IVELL R. Biology of Insulin-like Factor 3 in human reproduction[J]. Hum Reprod Update, 2009, 15(4): 463–476. DOI: 10.1093/humupd/dmp011 |
| [9] | FERLIN A, GAROLLA A, RIGON F, et al. Changes in serum insulin-like factor 3 during normal male puberty[J]. J Clin Endocrinol Metab, 2006, 91(9): 3426–3431. DOI: 10.1210/jc.2006-0821 |
| [10] |
赵丽丽, 聂敏, 伍学焱. 胰岛素样肽3的功能及其研究进展[J]. 生殖医学杂志, 2016, 25(4): 374–377.
ZHAO L L, NIE M, WU X Y. Function insulin-like peptide 3 and its research progress[J]. Journal of Reproductive Medicine, 2016, 25(4): 374–377. DOI: 10.3969/j.issn.1004-3845.2016.04.017 (in Chinese) |
| [11] | BAY K, COHEN A S, J∅RGENSEN F S, et al. Insulin-like factor 3 levels in second-trimester amniotic fluid[J]. J Clin Endocrinol Metab, 2008, 93(10): 4048–4051. DOI: 10.1210/jc.2008-0358 |
| [12] | HARRISON S M, BUSH N C, WANG Y, et al. Insulin-Like Peptide 3 (INSL3) serum concentration during human male fetal life[J]. Front Endocrinol, 2019, 10: 596. DOI: 10.3389/fendo.2019.00596 |
| [13] | BATHGATE R, BALVERS M, HUNT N, et al. Relaxin-like factor gene is highly expressed in the bovine ovary of the cycle and pregnancy:sequence and messenger ribonucleic acid analysis[J]. Biol Reprod, 1996, 55(6): 1452–1457. DOI: 10.1095/biolreprod55.6.1452 |
| [14] | BATHGATE R, MONIAC N, BARTLICK B, et al. Expression and regulation of relaxin-like factor gene transcripts in the bovine ovary:differentiation-dependent expression in theca cell cultures[J]. Biol Reprod, 1999, 61(4): 1090–1098. DOI: 10.1095/biolreprod61.4.1090 |
| [15] | ANAND-IVELL R, HIENDLEDER S, VIÑOLES C, et al. INSL3 in the ruminant:a powerful indicator of gender-and genetic-specific feto-maternal dialogue[J]. PLoS One, 2011, 6(5): e19821. DOI: 10.1371/journal.pone.0019821 |
| [16] | ANAND-IVELL R, HIENDLEDER S, VIÑOLES C, et al. INSL3 in the ruminant: a powerful indicator of gender- and genetic-specific feto-maternal dialogue[J]. PLoS One, 2011, 6(5): e19821. DOI: 10.1371/journal.pone.0019821 |
| [17] | DOMINIK S, HENSHALL J M, HAYES B J. A single nucleotide polymorphism on chromosome 10 is highly predictive for the polled phenotype in Australian Merino sheep[J]. Anim Genet, 2012, 43(4): 468–470. |
| [18] | JOHNSTON S E, MCEWAN J C, PICKERING N K, et al. Genome-wide association mapping identifies the genetic basis of discrete and quantitative variation in sexual weaponry in a wild sheep population[J]. Mol Ecol, 2011, 20(12): 2555–2566. DOI: 10.1111/j.1365-294X.2011.05076.x |
| [19] | PAN Z Y, LI S D, LIU Q Y, et al. Whole-genome sequences of 89 Chinese sheep suggest role of RXFP2 in the development of unique horn phenotype as response to semi-feralization[J]. Gigascience, 2018, 7(4): giy019. |
| [20] | DOVE W F. The physiology of horn growth:a study of the morphogenesis, the interaction of tissues, and the evolutionary processes of a mendelian recessive character by means of transplantation of tissues[J]. J Exp Zool, 1935, 69(3): 347–405. DOI: 10.1002/jez.1400690302 |
| [21] | FERLIN A, PERILLI L, GIANESELLO L, et al. Profiling insulin like factor 3 (INSL3) signaling in human osteoblasts[J]. PLoS One, 2011, 6(12): e29733. DOI: 10.1371/journal.pone.0029733 |
| [22] | FERLIN A, SELICE R, CARRARO U, et al. Testicular function and bone metabolism—beyond testosterone[J]. Nat Rev Endocrinol, 2013, 9(9): 548–554. DOI: 10.1038/nrendo.2013.135 |
| [23] | FERLIN A, SELICE R, DI MAMBRO A, et al. Role of vitamin D levels and vitamin D supplementation on bone mineral density in Klinefelter syndrome[J]. Osteoporos Int, 2015, 26(8): 2193–2202. DOI: 10.1007/s00198-015-3136-8 |
| [24] | FERLIN A, DE TONI L, SANDRI M, et al. Relaxin and insulin-like peptide 3 in the musculoskeletal system:from bench to bedside[J]. Brit J Pharmacol, 2017, 174(10): 1015–1024. DOI: 10.1111/bph.13490 |
| [25] | DE TONI L, AGOULNIK A I, SANDRI M, et al. INSL3 in the muscolo-skeletal system[J]. Mol Cell Endocrinol, 2019, 487: 12–17. DOI: 10.1016/j.mce.2018.12.021 |
| [26] | DI NISIO A, DE TONI L, ROCCA M S, et al. Negative association between sclerostin and INSL3 in isolated human osteocytes and in klinefelter syndrome:new hints for testis-bone crosstalk[J]. J Clin Endocrinol Metab, 2018, 103(5): 2033–2041. DOI: 10.1210/jc.2017-02762 |
| [27] |
潘章源, 贺小云, 刘秋月, 等. 绵羊GDF9基因mRNA、DNA和调控区序列克隆及其在11个品种中遗传多态性检测[J]. 畜牧兽医学报, 2016, 47(8): 1555–1564.
PAN Z Y, HE X Y, LIU Q Y, et al. Cloning and genetic polymorphism analysis of mRNA, DNA and regulatory region of ovine GDF9 gene in 11 breeds[J]. Acta Veterinaria et Zootechnica Sinica, 2016, 47(8): 1555–1564. (in Chinese) |
| [28] |
卢璐璐, 孙晓笛, 储明星, 等. 绵羊AA-NAT基因mRNA表达与常年发情相关性研究[J]. 畜牧兽医学报, 2015, 46(4): 542–548.
LU L L, SUN X D, CHU M X, et al. Association between AA-NAT mRNA expression and year-round estrus in sheep[J]. Acta Veterinaria et Zootechnica Sinica, 2015, 46(4): 542–548. (in Chinese) |
| [29] |
潘章源, 贺小云, 刘秋月, 等. 全基因组测序(WGS)在畜禽群体进化和功能基因挖掘中的应用[J]. 农业生物技术学报, 2016, 24(12): 1945–1954.
PAN Z Y, HE X Y, LIU Q Y, et al. Application of whole genome sequencing (WGS) in population evolution and gene discovery of domestic animals[J]. Journal of Agricultural Biotechnology, 2016, 24(12): 1945–1954. (in Chinese) |
| [30] | KAWAJI H, LIZIO M, ITOH M, et al. Comparison of CAGE and RNA-seq transcriptome profiling using clonally amplified and single-molecule next-generation sequencing[J]. Genome Res, 2014, 24(4): 708–717. DOI: 10.1101/gr.156232.113 |
| [31] | MERKIN J, RUSSELL C, CHEN P, et al. Evolutionary dynamics of gene and isoform regulation in mammalian tissues[J]. Science, 2012, 338(6114): 1593–1599. DOI: 10.1126/science.1228186 |
| [32] | HUNTLEY M A, LOU M, GOLDSTEIN L D, et al. Complex regulation of ADAR-mediated RNA-editing across tissues[J]. BMC Genomics, 2016, 17(1): 61. DOI: 10.1186/s12864-015-2291-9 |
| [33] | YU Y, FUSCOE J C, ZHAO C, et al. A rat RNA-Seq transcriptomic BodyMap across 11 organs and 4 developmental stages[J]. Nat Commun, 2014, 5: 3230. |
| [34] | JIANG Y, XIE M, CHEN W B, et al. The sheep genome illuminates biology of the rumen and lipid metabolism[J]. Science, 2014, 344(6188): 1168–1173. DOI: 10.1126/science.1252806 |
| [35] | PAN Z Y, LI S D, LIU Q Y, et al. Rapid evolution of a retro-transposable hotspot of ovine genome underlies the alteration of BMP2 expression and development of fat tails[J]. BMC Genomics, 2019, 20(1): 261. DOI: 10.1186/s12864-019-5620-6 |
| [36] | HU H, MIAO Y R, JIA L H, et al. AnimalTFDB 3.0:a comprehensive resource for annotation and prediction of animal transcription factors[J]. Nucleic Acids Res, 2019, 47(D1): D33–D38. DOI: 10.1093/nar/gky822 |
| [37] | SPANEL-BOROWSKI K, SCHÄFER I, ZIMMERMANN S, et al. Increase in final stages of follicular atresia and premature decay of corpora lutea in Insl3-deficient mice[J]. Mol Reprod Dev, 2001, 58(3): 281–286. DOI: 10.1002/1098-2795(200103)58:3<281::AID-MRD6>3.0.CO;2-0 |
| [38] | JOHNSTON S E, GRATTEN J, BERENOS C, et al. Life history trade-offs at a single locus maintain sexually selected genetic variation[J]. Nature, 2013, 502(7469): 93–95. DOI: 10.1038/nature12489 |
| [39] | LVHKEN G, KREBS S, ROTHAMMER S, et al.The 1.78-kb insertion in the 3'-untranslated region of RXFP2 does not segregate with horn status in sheep breeds with variable horn status[J].Genet Sel Evol, 2016, 48(1): 78. |
| [40] | SHI Y J, LAN F, MATSON C, et al. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1[J]. Cell, 2004, 119(7): 941–953. DOI: 10.1016/j.cell.2004.12.012 |
| [41] | PEDERSEN M T, HELIN K. Histone demethylases in development and disease[J]. Trends Cell Biol, 2010, 20(11): 662–671. DOI: 10.1016/j.tcb.2010.08.011 |
| [42] | DAHLMAN-WRIGHT K, CAVAILLES V, FUQUA S A, et al. International union of pharmacology.LXIV.Estrogen receptors[J]. Pharmacol Rev, 2006, 58(4): 773–781. DOI: 10.1124/pr.58.4.8 |
| [43] | DI PAOLA R, FRITTITTA L, MISCIO G, et al. A variation in 3' UTR of hPTP1B increases specific gene expression and associates with insulin resistance[J]. Am J Hum Genet, 2002, 70(3): 806–812. DOI: 10.1086/339270 |
| [44] | NOSSENT A Y, HANSEN J L, DOGGEN C, et al. SNPs in microRNA binding sites in 3'-UTRs of RAAS genes influence arterial blood pressure and risk of myocardial infarction[J]. Am J Hypertens, 2011, 24(9): 999–1006. DOI: 10.1038/ajh.2011.92 |



