伪狂犬病病毒(pseudorabies virus, PRV)可以感染引起母猪繁殖障碍、仔猪神经症状和呼吸系统疾病,严重危害我国养猪业的健康发展[1-3]。PRV是一种双链DNA病毒,基因组全长约143 kb,可编码100多种蛋白质[4-5]。在PRV的主要结构蛋白中,膜蛋白gB和gC主要激发细胞和体液免疫反应[6-7],gE蛋白是PRV对猪致病的主要毒力因子之一[8-9]。这三个蛋白的编码基因被广泛用于监测PRV的遗传进化[5, 10-15]。
PRV在我国的首次暴发见于20世纪50年代。1979年我国从匈牙利引入PRV弱毒疫苗Bartha-K61用于猪伪狂犬病的预防[16]。该疫苗的广泛使用使得我国猪伪狂犬病在1990—2011年间得到了较好的控制[17]。然而,2011年底,我国部分地区出现了疑似猪伪狂犬病的大规模暴发,很多免疫过Bartha-K61疫苗的猪场也未能幸免,给我国养猪业造成了巨大的经济损失。后续研究表明,PRV变异毒株的流行是此次猪伪狂犬病再度暴发的主要原因,并且许多研究显示Bartha-K61疫苗无法针对PRV变异毒株提供充足有效的保护[9, 12, 18-19]。这意味着当前在我国流行的PRV的病原学特征已经发生了某些重要的变化。因此,本研究针对2018年我国部分地区规模化猪场开展了PRV的PCR检测和病毒的分离鉴定,并对所分离的病毒开展遗传变异分析,旨在了解我国当前PRV的流行特征及其遗传变异。
1 材料与方法 1.1 试验材料样品来源于2018年1—12月我国辽宁、黑龙江、吉林、广东、广西、海南、天津、北京、山西、河北、内蒙古、湖北、湖南、河南、云南、贵州、重庆、四川、安徽、山东、江苏、浙江、福建、上海、江西、陕西、甘肃、新疆等28个省、市、自治区规模化猪场(存栏母猪≥100头)各年龄段疑似猪伪狂犬病发病猪肺、死胎、脑、脾、扁桃体、淋巴结等组织样品1 328份。样品的处理方法如下:用无菌眼科剪取5 g左右样品,置于无菌2 mL离心管中,剪碎,加入1 mL无菌PBS液(含终浓度为100 IU·mL-1的青链霉素),加入1颗灭菌钢珠进行匀浆处理,匀浆液置于-80 ℃保存备用。
1.2 病毒检测和分离利用康为世纪动物组织基因组提取试剂盒(Cat. No. CW0546)提取组织样品的基因组DNA作为模板,利用PCR检测PRV-gE基因,检测引物如表 1所示。PCR反应体系:12.5 μL 2× Taq Master Mix(Dye Plus),8.5 μL ddH2O及上游引物、下游引物、DMSO和DNA模板各1 μL;反应条件:95.0 ℃预变性5 min;95.0 ℃变性30 s,55.0 ℃退火30 s,72.0 ℃延伸1 min,35个循环;72.0 ℃延伸10 min。PCR产物经0.8%琼脂糖凝胶电泳进行检测。PRV-gE呈阳性的样品组织匀浆液经0.22 μm滤器过滤后,滤液接种长满单层的PK-15细胞,置于37 ℃、5% CO2的条件下吸附1~2 h后,弃去维持液(含3%胎牛血清的DMEM培养基),加入5 mL维持液,于37 ℃、5% CO2条件下培养4~5 d,观察是否有病变产生。如果无病变,-80 ℃冻融2~3次,再继续盲传3代,若再无病变,则废弃。选取第一代出现明显病变的细胞培养物进行空斑纯化,空斑纯化并稳定传代的PRV第一代病毒液用于提取DNA,利用表 1中的引物对病毒的gB、gC、gE基因进行PCR扩增,PCR反应体系和反应条件如上所示,退火温度见表 1。PCR产物送至武汉天一辉远股份有限公司进行测序。
|
|
表 1 本研究所用的引物及其序列 Table 1 Primers for gene amplification |
针对本研究中所分离毒株的gB、gC和gE序列进行生物信息学分析,同时选取NCBI中已经公布全基因组序列或者gB、gC和gE序列的毒株作为参考毒株(如表 2所示)。利用CLUSTAL W软件[20]进行多序列比对分析,同时利用MEGA X软件[21]进行遗传进化分析。
|
|
表 2 本研究所引用PRV参考毒株信息表 Table 2 PRV reference strains used in this study |
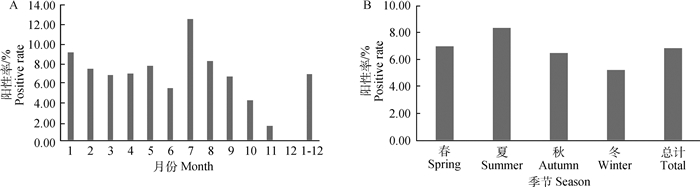
经PCR检测PRV-gE基因发现,1 328份临床样品中有92份为PRV-gE基因阳性,阳性检出率为6.93%。就不同月份而言,所有月份PRV-gE的阳性检出率均低于15%,其中7月份PRV-gE的阳性检出率最高,为12.63%;12月份PRV-gE的阳性检出率最低,为零检出(图 1A)。就不同季节而言,不同季度PRV-gE阳性检出率位于5.28%~8.41%,其中夏季最高,为8.41%,而冬季最低,为5.28%(图 1B)。就不同地区而言,东北(辽宁/黑龙江/吉林)、华南(广东/广西/海南)、华北(天津/北京/山西/河北/内蒙古)、华中(湖北/湖南/河南)、西南(云南/贵州/重庆/四川)、华东(安徽/山东/江苏/浙江/福建/上海/江西)和西北(陕西/甘肃/新疆)阳性检出率分别为12.20%(5/41)、11.58%(11/95)、10.48%(11/105)、8.66%(46/531)、7.69%(4/52)、3.42%(14/409)和1.05%(1/95),所有地区的阳性检出率均低于20%,东北地区阳性检出率(12.20%)最高,西北地区阳性检出率(1.05%)最低(表 3)。
|
A.不同月份PRV的阳性检出率;B.不同季节PRV的阳性检测率 A. The positive rate of PRV in different months; B. The positive rate of PRV in different seasons 图 1 2018年不同月份和季节我国PRV的阳性检出率 Fig. 1 Positive detection rate of PRV in different months and seasons of 2018 |
|
|
表 3 2018年我国不同地区PRV的阳性检出率 Table 3 Positive rate of PRV detection in different regions of China in 2018 |
从92份阳性样品中,成功分离到13株PRV,对应的样品分别来源于湖北、河南、广西、广东等地,13株PRV的TCID50·0.1 mL-1值为106.12~107.23。PRV分离株的信息如表 4所示。
|
|
表 4 13株PRV分离株具体信息表 Table 4 The information of 13 PRV isolates |
针对gB基因进行相似性分析发现,13株PRV分离株与国外毒株(Becker和Bartha等)的核苷酸和氨基酸相似性分别为94.1%~97.5%和92.2%~97.3%;13株PRV分离株与我国2011年以前PRV流行毒株的核苷酸和氨基酸相似性分别为96.4%~ 99.9%和95.5%~100%;而13株PRV分离株与我国2011年以后PRV流行毒株相似性分别为96.4%~100%和95.9%~100%;13株PRV分离株核苷酸和氨基酸相似性分别为96.4%~100%和95.5%~100%(表 5)。
|
|
表 5 核苷酸和氨基酸的相似性分析 Table 5 Homology analysis of nucleotide and amino acid sequences |
基于gB基因进行遗传进化分析显示,PRV分为两个基因簇,其中Bartha、Becker等国外分离毒株位于一个基因簇,而中国流行毒株包括HeN1、Ea和P-PRV等位于另一个基因簇(图 2A)。值得注意的是,本研究中的一株分离株HBZX-China-2018与Bartha、Becker等国外分离毒株位于一个基因簇(图 2A)。

|
A. gB;B. gC;C. gE 图 2 不同PRV毒株间的遗传进化分析 Fig. 2 Phylogenetic analysis among different PRV isolates |
gB蛋白的氨基酸序列比对结果显示,与国外毒株(Becker、Bartha和Kaplan等)相比,13株分离株的gB蛋白存在5个位点的氨基酸突变:87位点A→E、93位点E→D、96—97位点FT→VS、102位点E→D;大多数分离株有9个氨基酸位点突变(除HuBZX-China-2018分离株外):分别为53位点A→T、70位点T→A、55位点P→T、72—73位点VP→GT、81—83位点NDV→DGF、78位点L→A。另外,个别分离株还有特殊的氨基酸位点替换。例如:HBAL-China-2018分离株在40位点A→T、43位点L→V,HBWQL-China-2018和LNCY-China-2018在56—57位点发生连续两个位点突变CG→VR,HBZX-China-2018在67位点有特殊的突变A→V。WHBD-China-2018分离株在第65—77位点均存在SA氨基酸的连续缺失(图 3)。与国外毒株(Bartha)相比,所有分离株在第75—77位点均连续缺失三个氨基酸(SPG), 第94位点插入氨基酸G。

|
图 3 PRV gB基因氨基酸序列结果 Fig. 3 Alignment of partial amino acid sequences of PRV gB |
针对gC基因进行相似性分析发现,13株PRV分离株与国外毒株(Becker和Bartha等)核苷酸和氨基酸相似性分别为94.2%~97.7%和89.4%~94.4%;与我国2011年以前的PRV流行毒株的核苷酸和氨基酸相似性分别为94.9%~97.7%和93.2%~99.3%;与我国2011年以后的PRV流行毒株的相似性分别为94.8%~100%和93.2%~100%;13株PRV分离株的核苷酸和氨基酸相似性分别为98.4%~100%和93.5%~100%(表 5)。
基于gC基因进行遗传进化分析显示,本研究所分离的13株PRV分离株均同处于一个独立的进化分支,与Bartha等国外经典毒株处于不同的分支。其中4株分离株位于一个相对独立的分支,与Ea株等早期经典毒株亲缘关系较近(图 2B)。
通过对13株PRV分离株gC蛋白的氨基酸序列进行比对分析发现,与国外毒株(Becker、Bartha和Kaplan等)相比,分离株的gC蛋白存在31个氨基酸的替换(图 4);所有分离毒株均在第63位点存在7个氨基酸(AAASTPA)的连续插入。与Ea等国内经典毒株相比,这13株PRV分离株均在第34、99、194位氨基酸存在替换,分别为T→N、E→K、G→E(图 4)。其中2株分离株HBQJR-China-2018和WHBD-China-2018在107位点还有一个氨基酸替换R→H。此外,个别分离毒株在个别位点存在氨基酸的替换:GD-1-China-2018在75位点R→P,在210和227位点A→T;HBWQL-China-2018在176位点N→T; HBZX-1-China-2018在227位点A→T(图 4)。

|
图 4 PRV gC基因氨基酸序列比对结果分析 Fig. 4 The results of the complete amino acid sequence of the PRV gC gene |
针对gE基因进行相似性分析发现,13株PRV分离株与国外毒株(Becker和Bartha等)的核苷酸和氨基酸相似性分别为97.6%~98.1%和92.1%~96.3%;分离株与我国2011年以前PRV流行毒株的核苷酸和氨基酸相似性分别为98.3%~99.7%和97.3%~99.3%;分离株与我国2011年以后的PRV流行毒株的核苷酸和氨基酸相似性分别为98.7%~100%和97.3%~100%;与此同时,13株分离株核苷酸和氨基酸相似性分别为98.0%~100%和96.3%~100%(表 5)。
基于gE基因进行遗传进化分析显示,绝大多数2011年以后的PRV中国分离株位于同一进化分支,与2011年以前的PRV中国分离株及国外分离株的亲缘关系较远(图 2C)。
通过对13株PRV分离株gE蛋白的氨基酸序列进行分析发现,与国外经典毒株(Becker和Kaplan等)相比,所有的分离毒株的gE蛋白存在17个氨基酸位点的突变:59位点D→N、106位点V→L、122位点A→S、125位点M→T、149位点R→M、163位点Q→R、179位点T→S、181位点R→L、215—216位点LA→AD、472位点G→R、474位点R→H、503—504位点DA→VI、509位点S→A、522位点V→A、526位点A→P;此外,所有分离毒株均在第48位点插入1个天冬氨酸(D);绝大多数分离株在497位点插入1个天冬氨酸(D)(除HBQJR-China-2018和GXGG-China-2018分离株外)。此外,个别分离株还存在个别氨基酸突变:GXGG-China-2018分离株在100和171位点发生替换R→H、P→R;HBZX-China-2018分离株在335—337位点发生3个氨基酸的连续替换FDA→LRR;JXGA-China-2018分离株在159位点发生突变V→G。与此同时,与Ea等国内经典毒株相比,这13株PRV野毒株在第54、404及520位点均发生突变(图 5)。

|
图 5 PRV gE基因氨基酸序列比对结果分析 Fig. 5 The results of the complete amino acid sequence of the PRV gE gene |
本研究对2018年来源于我国28个省、市、自治区的1 328份疑似猪伪狂犬病发病猪的组织样品开展PRV的检测和毒株的分离鉴定,结果显示PRV的平均阳性检出率为6.93%。这一数据略低于我国2012—2017年的阳性检出率(约为8.27%)[22],提示过去几年我国针对PRV的防控取得了一定的成效。就不同季节而言,夏季PRV的阳性检出率最高,而冬季最低(图 1B),提示PRV的流行表现出一定的季节性。就不同地区而言,东北地区的PRV阳性检出率最高,而西北地区的阳性检出率最低(表 3)。不同地区PRV的检测结果的差异可能一方面与不同地区样品来源的总量有关,另一方面也可能与我国不同地区的生猪养殖规模以及猪场基础条件和生物安全体系建设、生产管理等因素有关。
本研究从92份PRV-gE阳性样品中成功分离13株PRV。对13株分离株gB、gC和gE基因进行遗传变异分析发现,13株PRV分离株与Bartha等国外毒株之间的遗传关系较远(图 2)。有学者根据gC基因将PRV分为两个基因型,即基因Ⅰ型和基因Ⅱ型,并且发现当前在中国流行的PRV主要为基因Ⅱ型,而非中国毒株(如Bartha等)主要为基因Ⅰ型[23]。在本研究中,基于gC进行遗传进化分析发现所有PRV中国分离株与国外毒株分别位于两个不同的遗传进化分支,其中中国分离株为基因Ⅱ型,而非中国毒株为基因Ⅰ型(图 2B);基于gB和gE进行遗传进化分析也得到了相似的结果(图 2A和C)。这或许可以从遗传学的角度解释Bartha K61疫苗无法对当前我国流行的PRV提供充分保护的原因[21]。值得一提的是在本研究中发现有一株PRV分离株HBZX-China-2018基于gC基因进行遗传进化分析属于基因Ⅱ型,与Bartha等国外毒株的亲缘关系较远,而基于gB基因进行遗传进化分析却发现其与Bartha等国外毒株的亲缘关系较近,而与中国流行毒株的亲缘关系较远(图 2A和B)。经调查发现,分离出HBZX-China-2018的样品所来源的猪场仅免疫过Bartha-K61疫苗,提示该毒株的产生可能是由于流行毒株和疫苗株发生重组。
gB是PRV病毒粒子的主要囊膜蛋白和重要免疫原蛋白,在细胞融合和病毒感染过程中发挥着重要的作用[24]。有研究证实gB的913个氨基酸是病毒感染不可缺少的[6, 25]。此外,gB蛋白作为PRV的主要免疫原性蛋白,能刺激机体产生补体依赖性及非补体依赖性中和抗体[26]。在本研究中,我们发现13株PRV分离株相对于国外毒株而言,其gB蛋白均存在与已有文献报道[27]一致的氨基酸插入、缺失和替换。这些氨基酸变异主要位于gB蛋白的中和表位(59—279氨基酸)区域,它们的存在有可能使得gB蛋白中和表位发生改变,这也可能是传统疫苗(Bartha-K61)免疫失败的一个重要原因[27]。除了gB外,在13株的gC蛋白也存在与文献报道[28]一致的氨基酸插入和替换;这些特征性的变异位点均位于gC的功能区内。已知gC蛋白是PRV的中和抗原,能影响病毒毒力的变化及介导病毒和细胞间的吸附过程[29]。当前流行的PRV毒株的gC蛋白中存在的这些氨基酸变化可能是新分离毒株毒力增强的原因之一,然而相关报道还不多见,需要更多的研究作进一步的验证。gE也是PRV主要的毒力决定因素[13]。有报道显示gE蛋白中只需几个氨基酸位点的改变就能显著影响病毒的毒力[30]。与国外经典毒株(Becker和Kaplan等)相比,本研究中分离的13株PRV在gE蛋白中均有17个氨基酸位点的突变;其中在第48位点插入1个天冬氨酸(D),这些位点突变均位于gE蛋白的氨基酸表位上(52—238)。此外,与Ea等国内经典毒株相比,这13株PRV分毒株在第54、404及520位点均存在差异。这些变异可能也与PRV的毒力变化有关。
4 结论针对2018年我国绝大多数省、市疑似PRV感染的病猪组织样品进行检测。结果表明,PRV仍然在我国大多数省份流行,且各个地区PRV阳性检出率存在一定差异。病毒的分离鉴定表明当前我国流行的PRV主要为基因Ⅱ型。此外,当前流行的PRV毒株相对于国外毒株及我国早期流行的PRV而言,在gB、gC和gE蛋白中存在一些特征性的氨基酸变异,这些变异可能是造成已有疫苗免疫不足及毒株毒力改变的重要原因之一。因此,在我国当前的PRV防控中需要筛选与我国流行毒株相匹配的PRV毒株进行疫苗的研发,从而保证我国PRV的疫苗防控质量。
| [1] | METTENLEITER T C. Aujeszky's disease (pseudorabies) virus:the virus and molecular pathogenesis——state of the art, June 1999[J]. Vet Res, 2000, 31(1): 99–115. |
| [2] | NAUWYNCK H, GLORIEUX S, FAVOREEL H, et al. Cell biological and molecular characteristics of pseudorabies virus infections in cell cultures and in pigs with emphasis on the respiratory tract[J]. Vet Res, 2007, 38(2): 229–241. DOI: 10.1051/vetres:200661 |
| [3] | SUN Y, LUO Y Z, WANG C H, et al. Control of swine pseudorabies in China:opportunities and limitations[J]. Vet Microbiol, 2016, 183: 119–124. DOI: 10.1016/j.vetmic.2015.12.008 |
| [4] | KLUPP B G, HENGARTNER C J, METTENLEITER T C, et al. Complete, annotated sequence of the pseudorabies virus genome[J]. J Virol, 2004, 78(1): 424–440. DOI: 10.1128/JVI.78.1.424-440.2004 |
| [5] | MVLLER T, HAHN E C, TOTTEWITZ F, et al. Pseudorabies virus in wild swine:a global perspective[J]. Arch Virol, 2011, 156(10): 1691–1705. DOI: 10.1007/s00705-011-1080-2 |
| [6] | OBER B T, TEUFEL B, WIESMVLLER K H, et al. The porcine humoral immune response against pseudorabies virus specifically targets attachment sites on glycoprotein gC[J]. J Virol, 2000, 74(4): 1752–1760. DOI: 10.1128/JVI.74.4.1752-1760.2000 |
| [7] | OBER B T, SUMMERFIELD A, MATTLINGER C, et al. Vaccine-induced, pseudorabies virus-specific, extrathymic CD4+ CD8+ memory T-helper cells in swine[J]. J Virol, 1998, 72(6): 4866–4873. DOI: 10.1128/JVI.72.6.4866-4873.1998 |
| [8] | KIMMAN T G, DE WIND N, OEI-LIE N, et al. Contribution of single genes within the unique short region of Aujeszky's disease virus (suid herpesvirus type 1) to virulence, pathogenesis and immunogenicity[J]. J Gen Virol, 1992, 73(2): 243–251. DOI: 10.1099/0022-1317-73-2-243 |
| [9] | WANG C H, YUAN J, QIN H Y, et al. A novel gE-deleted pseudorabies virus (PRV) provides rapid and complete protection from lethal challenge with the PRV variant emerging in Bartha-K61-vaccinated swine population in China[J]. Vaccine, 2014, 32(27): 3379–3385. DOI: 10.1016/j.vaccine.2014.04.035 |
| [10] |
何启盖, 方六荣, 吴斌, 等. 猪伪狂犬病基因缺失疫苗的制备、安全性、免疫原性、保存期测定及区域试验[J]. 畜牧兽医学报, 2005, 36(10): 1055–1063.
HE Q G, FANG L R, WU B, et al. The preparation of gene-deleted vaccine against swine pseudorabies, measurement of its safety, immunogenicity, shelf life and the evaluation of vaccine by field trials[J]. Acta Veterinaria et Zootechnica Sinica, 2005, 36(10): 1055–1063. DOI: 10.3321/j.issn:0366-6964.2005.10.014 (in Chinese) |
| [11] | SOZZI E, MORENO A, LELLI D, et al. Genomic characterization of pseudorabies virus strains isolated in Italy[J]. Transbound Emerg Dis, 2014, 61(4): 334–340. DOI: 10.1111/tbed.12038 |
| [12] | YU X L, ZHOU Z, HU D M, et al. Pathogenic pseudorabies virus, China, 2012[J]. Emerg Infect Dis, 2014, 20(1): 102–104. DOI: 10.3201/eid2001.130531 |
| [13] | WANG Y B, QIAO S L, LI X W, et al. Molecular epidemiology of outbreak-associated pseudorabies virus (PRV) strains in central China[J]. Virus Genes, 2015, 50(3): 401–409. DOI: 10.1007/s11262-015-1190-0 |
| [14] | XIA L M, SUN Q Y, WANG J J, et al. Epidemiology of pseudorabies in intensive pig farms in Shanghai, China:herd-level prevalence and risk factors[J]. Prev Vet Med, 2018, 159: 51–56. DOI: 10.1016/j.prevetmed.2018.08.013 |
| [15] | LIU C, LIU Y H, TIAN Y, et al. Genetic characterization and mutation analysis of Qihe547 Aujeszky's disease virus in China[J]. BMC Vet Res, 2018, 14: 218. DOI: 10.1186/s12917-018-1492-2 |
| [16] | AN T Q, PENG J M, TIAN Z J, et al. Pseudorabies virus variant in Bartha-K61-vaccinated pigs, China, 2012[J]. Emerg Infect Dis, 2013, 19(11): 1749–1755. DOI: 10.3201/eid1911.130177 |
| [17] |
童光志, 陈焕春. 伪狂犬病流行现状及我国应采取的防制措施[J]. 中国兽医学报, 1999, 19(1): 1–2.
TONG G Z, CHEN H C. Epidemic present status and prophylactico-therapeutic measures of pseudorabies taken in China[J]. Chinese Journal of Veterinary Science, 1999, 19(1): 1–2. DOI: 10.3969/j.issn.1005-4545.1999.01.001 (in Chinese) |
| [18] | LUO Y Z, LI N, CONG X, et al. Pathogenicity and genomic characterization of a pseudorabies virus variant isolated from Bartha-K61-vaccinated swine population in China[J]. Vet Microbiol, 2014, 174(1-2): 107–115. DOI: 10.1016/j.vetmic.2014.09.003 |
| [19] |
王一鹏, 王亚文, 徐瑞涛, 等. 一株分离自免疫猪场的伪狂犬病病毒的鉴定与变异分析[J]. 畜牧兽医学报, 2019, 50(10): 2070–2078.
WANG Y P, WANG Y W, XU R T, et al. Identification and variation analysis of pseudorabies virus from vaccinated pig farm[J]. Acta Veterinaria et Zootechnica Sinica, 2019, 50(10): 2070–2078. DOI: 10.11843/j.issn.0366-6964.2019.10.013 (in Chinese) |
| [20] | THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 1994, 22(22): 4673–4680. DOI: 10.1093/nar/22.22.4673 |
| [21] | KUMAR S, STECHER G, LI M, et al. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol, 2018, 35(6): 1547–1549. DOI: 10.1093/molbev/msy096 |
| [22] | SUN Y, LIANG W, LIU Q Y, et al. Epidemiological and genetic characteristics of swine pseudorabies virus in mainland China between 2012 and 2017[J]. PeerJ, 2018, 6: e5785. DOI: 10.7717/peerj.5785 |
| [23] | YE C, ZHANG Q Z, TIAN Z J, et al. Genomic characterization of emergent pseudorabies virus in China reveals marked sequence divergence:evidence for the existence of two major genotypes[J]. Virology, 2015, 483: 32–43. DOI: 10.1016/j.virol.2015.04.013 |
| [24] | METTENLEITER T C. Pathogenesis of neurotropic herpesviruses:role of viral glycoproteins in neuroinvasion and transneuronal spread[J]. Virus Res, 2003, 92(2): 197–206. DOI: 10.1016/S0168-1702(02)00352-0 |
| [25] | BEN-PORATH E, WANDS J R, MARCINIAK R A, et al. Structural analysis of hepatitis B surface antigen by monoclonal antibodies[J]. J Clin Invest, 1985, 76(4): 1338–1347. DOI: 10.1172/JCI112108 |
| [26] | OKAZAKI K. Proteolytic cleavage of glycoprotein B is dispensable for in vitro replication, but required for syncytium formation of pseudorabies virus[J]. J Gen Virol, 2007, 88(7): 1859–1865. DOI: 10.1099/vir.0.82610-0 |
| [27] | CURANOVIC D, ENQUIST L W. Virion-incorporated glycoprotein B mediates transneuronal spread of pseudorabies virus[J]. J Virol, 2009, 83(16): 7796–7804. DOI: 10.1128/JVI.00745-09 |
| [28] | XIAO S B, CHEN H C, FANG L R, et al. Comparison of immune responses and protective efficacy of suicidal DNA vaccine and conventional DNA vaccine encoding glycoprotein C of pseudorabies virus in mice[J]. Vaccine, 2004, 22(3-4): 345–351. DOI: 10.1016/j.vaccine.2003.08.010 |
| [29] | KARGER A, SCHMIDT J, METTENLEITER T C. Infectivity of a pseudorabies virus mutant lacking attachment glycoproteins C and D[J]. J Virol, 1998, 72(9): 7341–7348. DOI: 10.1128/JVI.72.9.7341-7348.1998 |
| [30] | METTENLEITER T C, KLUPP B G, WEILAND F, et al. Characterization of a quadruple glycoprotein-deleted pseudorabies virus mutant for use as a biologically safe live virus vaccine[J]. J Gen Virol, 1994, 75(7): 1723–1733. DOI: 10.1099/0022-1317-75-7-1723 |



