哺乳动物生长发育过程中,骨骼肌参与了机体储能、产热、防护及运动等重要生理过程[1],其作为家畜动物体中占比较大的组织,直接决定家畜的产肉性能,关系到最终的生产效益。家畜肌纤维数量和直径与其产肉性能密切相关,但家畜肌纤维数量在胚胎期即达到发育极值,出生后只能依靠肌纤维横截面积的增加而提高产肉量。若适当提高肌纤维数量极值,则可在一定程度上有助于实现高产肉量性能家畜的培育[2]。因此,肌纤维发育机制的研究成为家畜育种的热点[3]。早在1972年,Ashmore等[4]对不同胚龄绵羊(D50、D60、D70、D100、D125和D140)骨骼肌组织进行了组织学检测,结果发现,肌纤维数量的增加始于D50~D60,在D100~D125之间肌纤维数量趋于稳定,D140后基本停止增长。Fahey等[5]通过对不同时间节点(D45、D55、D70、D85、D100、D115和D130)绵羊胚胎肌肉组织切片检测和转录组测序,并对IGF-I、IGF-II、GH受体(GHR)和肌肉生长抑制素(myostatin)进行q-PCR定量[6],分析基因表达和组织切片结果得出成肌细胞增殖主要发生在D85之前,而后是肌纤维膨大和增粗期。Xu等[7]在研究两个品种绵羊的胚胎肌肉发育时,采用D70、D85、D100、D120、D135胚胎背最长肌,构建肌肉增长高相关的基因共表达网络,鉴定出LMNB1、TADA3和FBP2等与胎儿体重相关的基因,并指出在绵羊妊娠发育晚期有多个基因参与肌纤维及肌间脂肪发育的调控。
目前为止,调控绵羊肌纤维数量和类型转换的分子机制仍不清楚。由于骨骼肌发育的每个阶段都有众多基因、转录物和蛋白共同参与,单一因素的分子研究并不能完全阐述机体内分子互作机理。因此,研究鉴定发育动态性表达差异转录因子的变化是解析绵羊肌纤维发育过程的重要基础[8]。本试验采用高通量RNA-seq技术对不同阶段胚胎背最长肌进行全转录组测序,构建骨骼肌全转录组图谱,表达谱大数据集的分析可以促进复杂遗传性状调节机制的解析[9]。分析基因表达变化是了解基因间复杂的互作模式和网络结构的有效途径,加权基因共表达网络分析(weighted gene co-expression network analysis, WGCNA)被用于分析高度相关基因的模块/簇,将模块链接到数量性状以分析分子作用机制以及网络关系的生物信息学方法[10-11]。WGCNA网络构建方法能够整合个体基因,发挥系统生物学的功能[12]。共表达网络中高度连接基因的表达水平较其他基因稳定,对调节动物体某阶段的核心生物功能至关重要。因此,可以使用WGCNA方法研究基因共表达调控网络,探讨并发掘肌纤维发育过程中的关键基因及转录因子。该方法始于2005年,目前已成功应用在人[13-14]、小鼠[15]、牛[16]、鸡[17]、狗[18]等相关研究中,且WGCNA网络有利于预测lncRNA的潜在功能[19-20]。
本试验以中国美利奴羊为研究对象,发掘在妊娠中后期影响肌纤维发育的重要候选基因,分析肌纤维数量与质量生长变化的窗口期,为深入解析哺乳动物肌纤维增生和肥大的分子机制奠定坚实的基础。
1 材料与方法 1.1 试验样品采集与测序本试验选择年龄相近、体况良好、体重在55~60 kg的成年中国美利奴羊(购自新疆巴州种畜场),经埋栓同期发情处理后进行人工授精配种,通过超数排卵获得胚胎并移植到体况相近的受体母羊。妊娠母羊均按照国家农业行业肉羊饲养标准(NY/T 816-2004)进行饲喂。胚胎背最长肌组织样品分别在妊娠第85(D85N)、105(D105N)和135天(D135N)时通过外科手术取出,每组采样个体为3只,设置D85N vs D105N、D105N vs D135N和D85N vs D135N 3个比较组。通过Illumina HiSeq System高通量测序技术对样本进行测序处理,统计reads count数量以估计lncRNA和mRNA的表达水平。然后与绵羊(Ovis aries)、山羊(Capra aegagrus hircus)、牛(Bos taurus)、人(Homo sapiens)、小鼠(Mus musculus)等物种基因组、lncRNA数据库进行比对和映射,整合并注释了绵羊胚胎期骨骼肌的全转录组图谱,并上传至NCBI公共功能基因组数据库(GSE127187)。
1.2 统计原理及肌肉组织差异表达lncRNA和mRNA的筛选为使不同基因和不同样本间基因的表达水平具有可比性,将每个基因的表达水平标准化为FPKM(fragments per kilobase of exon model per million mapped reads)。在消除差异干扰的基础上,通过StringTie[21]计算mRNA和lncRNA的表达水平。将3个发育时期lncRNA和mRNA的表达谱分别按胚龄分组对比,以|log2FC| ≥ 1且P≤0.05为标准,选择差异表达的mRNA(DEMs)和lncRNA(DELs)。差异表达RNA的筛选是基于ANOVA的Fisher精确检验、卡方2 × 2检验、卡方n × n检验及Student t检验等方法。在每次检验中,显著性阈值设定为0.01和0.05。
1.3 肌肉组织miRNA的靶向预测在哺乳动物中,miRNA的作用机制是通过成熟miRNA的种子区域(seed)与AGO蛋白形成复合物后结合在mRNA的3′端非翻译区(untranslated regions, UTR),导致mRNA的翻译受到抑制。miRNA靶基因预测是基于miRNA与mRNA的作用原理,使用TargetScan[22-23]、miRanda[24-25]软件对miRNA分别进行靶基因预测。TargetScan采用默认参数,miRanda采用Max_Energy小于-10。TargetScan算法中去除context score percentile小于50的靶基因,miRanda算法中去除最大自由能(max energy)大于-10的靶基因。为确保精度,最后取TargetScan和miRanda预测结果的交集作为miRNA的靶基因。
1.4 WGCNA加权共表达网络的构建加权共表达网络的分析基于R语言WGCNA包,首先从表达矩阵中提取差异表达值,计算两两间的Pearson相关系数,构建DEMs和DELs表达相关系数矩阵。为放大相关性差距,对基因相关关系进行无尺度化处理并建立无尺度网络关系。网络由节点(node)和边(edge)组成,节点代表DEMs和DELs,边代表两者之间的关系。节点的度(degree)又叫节点的连通性(connectivity),它代表了该节点所连接的边的数量。本研究采用pick-SoftThreshold函数计算无尺度拓扑拟合指数并取合理β值作为构建网络的软阈值,将相关系数矩阵转换为邻接矩阵。然后通过拓扑重叠方法评估两基因间直接或间接关系,构建拓扑重叠矩阵(topological overlap matrix,TOM),并根据基因间的TOM值进行层级聚类,将表达模式高度相似的基因进行分组,使用混合动态树剪切法将分组剪切区分产生表达模块。使用average-linkage层次聚类对各个模块进行聚类,合并相似度高的模块,确定最终的共表达模块。然后用DAVID软件进行富集分析,设置P≤0.05作为显著性阈值,筛选出肌肉发育相关的共表达模块,并利用R语言的ggplot2软件包[26]对富集结果进行可视化。
1.5 lncRNA-miRNA-mRNA共表达网络的构建根据lncRNA和miRNA的非编码特性,lncRNA上的miRNA结合位点的预测基于它们的全长序列。同时为减少假阳性,每个lncRNA保留至少两个miRNA结合位点。在WGCNA加权共表达分析基础上,结合mRNA和lncRNA的靶向预测结果,选择lncRNA-mRNA对中均被靶向于某常见miRNA的共表达网络。使用Perl脚本整合ceRNA关系,将包括筛选出的所有相互作用对的信息导入Cytoscapes软件[27](http://www.cytoscape.org)以构建可视化互作网络。
2 结果 2.1 绵羊胎儿肌肉组织转录本差异表达测序结果如表 1所示,每个测序文库获得质控有效数据约占原始数据的97%,Q-score>20和Q-score>30的reads都超过97%。在有效读段中,约85% reads比对到绵羊参考基因组上,其中超过82%是唯一比对。因此证明样本测序质量可靠,符合下游数据分析要求。本试验是基于全转录组测序结果进行的lncRNA功能分析,首先在全转录组图谱中,通过edgeR包筛选出差异表达的lncRNAs和mRNAs,去除在3个时间节点重复出现的差异表达RNA使其保留唯一,最后共得到78个显著差异表达已知lncRNAs以及33个novel lncRNAs,2 592个差异表达mRNAs(表 2),用于WGCNA加权共表达分析和lncRNA-miRNA-mRNA共表达网络构建。
|
|
表 1 不同样本测序数据质控结果 Table 1 Quality control results of sequencing data of different samples |
|
|
表 2 差异表达lncRNAs和mRNAs数目的统计 Table 2 Statistics of number of differentially expressed lncRNAs and mRNAs |
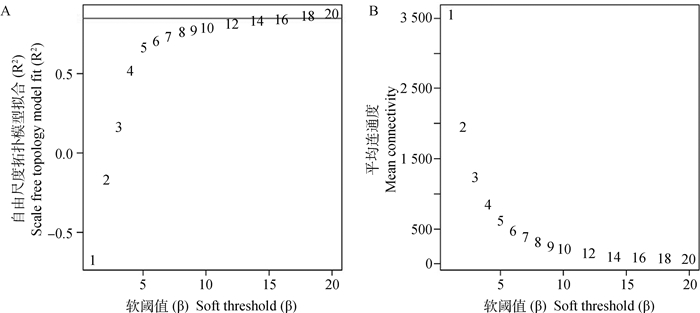
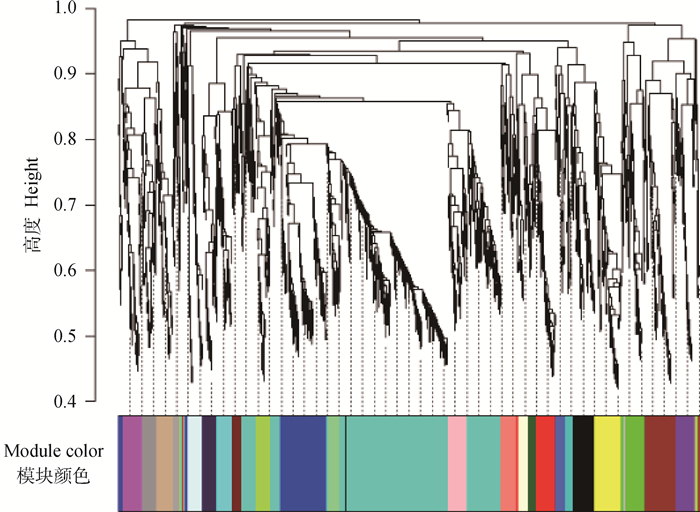
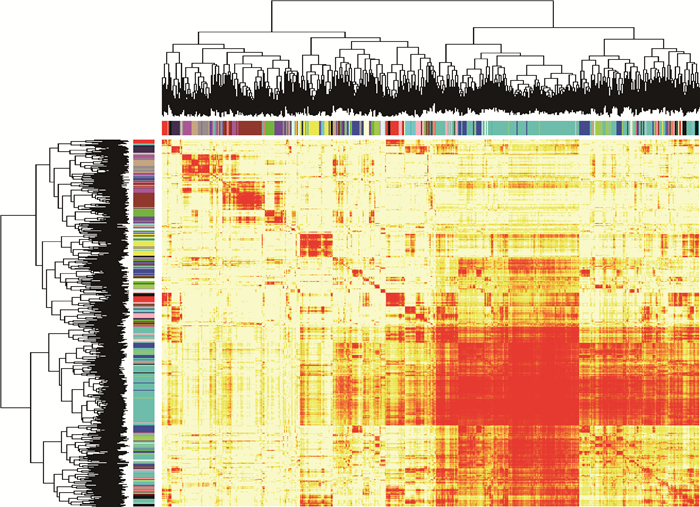
为使网络接近无尺度分布,需要选择适合参数β值的R2。理论上R2越接近1,模型越符合无尺度分布,本研究以R2>0.85为例,β=14作为构建共表达模块的软阈值较为合理(图 1)。然后根据所获得基因间TOM值进行层级聚类,构建基因聚类树(图 2),若某些基因在同一生理/病理过程或不同组织中具有类似的表达变化,那么就认定这些基因在功能上是相关的,可以将他们的集合定义为一个模块。分析中通过混合动态树剪切法对聚类树分支进行剪切区分,共产生25个不同的模块,其中灰色模块代表无法归入任何一个模块的基因,即在共表达分析时未被接受的基因。为探究各模块之间的相关性,根据基因间表达量对模块进行拓扑重叠热图绘制,图中行和列都表示单个基因,深黄色和红色表示高度的拓扑重叠(图 3)。其中蓝绿色模块共表达聚类结果显著,表明该模块内基因表达相关性高。相关性较高的目标模块内部分基因KEGG富集结果及相关信息见表 3。
|
A图纵轴代表对应的网络中log(k)与log(p(k))相关系数的平方;B图的纵轴代表相应的基因模块中基因邻接函数的均值 The vertical axis in A figure represents the square of the correlation coefficient between log(k) and log(p(k)) in the network; The vertical axis in B figure represents the mean of the gene adjacency function in the gene module 图 1 lncRNA-mRNA加权共表达软阈值的筛选 Fig. 1 Screening of lncRNA-mRNA weighted co-expression soft threshold |
|
横轴表示聚类模块 Horizontal axis are clustering modules 图 2 基因聚类树与模块构建 Fig. 2 Gene clustering tree and module construction |
|
图中行和列都表示单个基因,深黄色和红色表示高度的拓扑重叠 Both rows and columns in the figure represent the single gene, dark yellow and red indicate a high topological overlap 图 3 拓扑重叠热图 Fig. 3 Topological overlapping heat map |
|
|
表 3 目标模块KEGG富集结果 Table 3 KEGG enrichment results of the target modules |
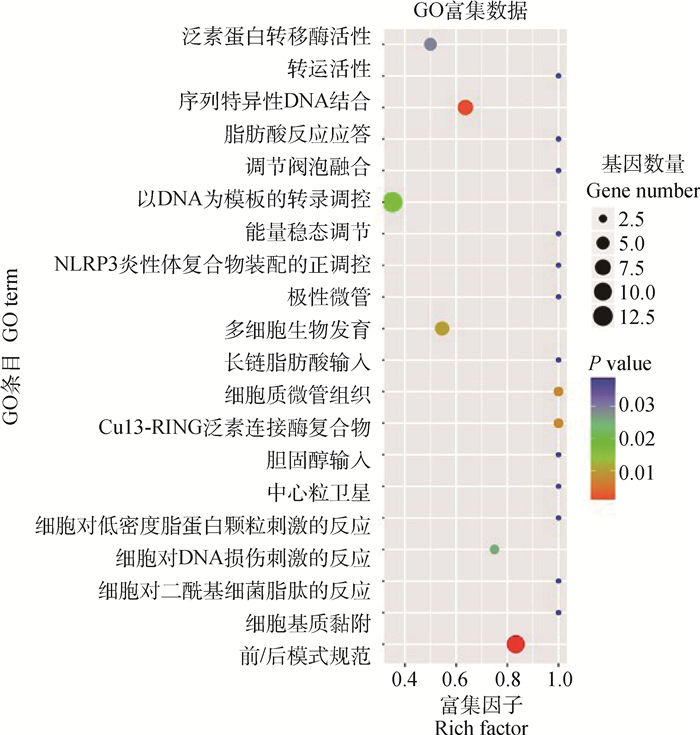
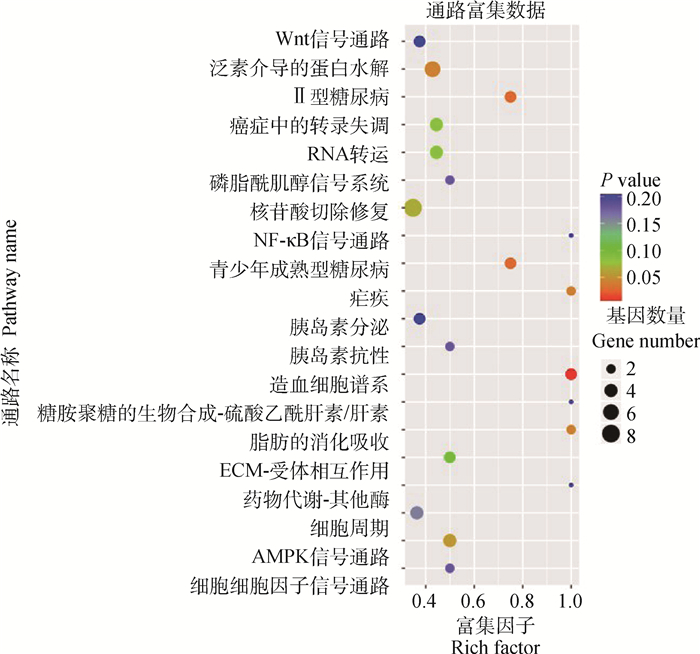
此外,通过DAVID 6.8在线分析软件(https://david.ncifcrf.gov/summary.jsp)对各个模块中的基因进行功能富集分析,以P≤0.05作为显著性阈值,GO分析结果表明,这些基因主要参与细胞周期、细胞凋亡的正调节、内胚层细胞分化、有丝分裂、蛋白质磷酸化调节、囊泡融合调节、钾离子跨膜转运蛋白活性的负调控、钠离子跨膜转运的负调控、蛋白质泛素化、细胞-基质粘附、脂质代谢等生物过程(图 4)。KEGG分析中,模块基因主要富集在AMPK信号通路、造血细胞谱系、粘着力、mTOR、Akt、ECM-受体相互作用、泛素介导的蛋白水解、脂肪消化吸收等脂代谢途径和免疫系统相关通路(图 5)。
|
气泡大小反映了富集到某个GO条目的差异表达基因的数量/背景中所有基因的数量;颜色反映了富集在某过程的显著性 Bubble size reflect the number of differentially expressed genes enriched to a certain GO term/the number of all genes in the background; Color reflect the significance of differentially expressed genes enriching in a certain process 图 4 模块中核心基因的GO富集分析 Fig. 4 GO analysis of core genes in modules |
|
气泡大小表示富集到某个通路中的核心基因的数量/背景中所有基因的数量;颜色反映了富集在某通路的显著性 Bubble size reflect the number of core genes enriched to a certain pathway/the number of all genes in the background; Color reflect the significance of differentially expressed lncRNAs enriching in a certain pathway 图 5 模块中核心基因的KEGG通路分析 Fig. 5 KEGG pathway analysis of core genes in modules |
根据lncRNA与miRNA之间的靶向作用及与lncRNA与mRNA之间的连通度,各转录物连通度信息见表 4,筛选出4个lncRNAs、4个miRNAs和152个mRNAs构建共表达网络,共形成370个互作关系。结果显示,大多数mRNA与核心lncRNA、miRNA具有复杂的网络连接,这表明它们在胎儿发育过程中对肌肉发育和能量代谢的调节具有中心作用。
|
|
表 4 miRNA和lncRNA连通度 Table 4 Connectivity degree of miRNAs and lncRNAs |
在表达模块中,与成纤维细胞增殖和分化相关的基因包含WAPL、CDKN1A、ITGB3,SGPL1、SMAD3、PIP5K1A;其他与肌肉发育相关的基因有ACTN3、IGF2、MYH1、TMOD4、TNNI2、TPM1;筛选到参与肌肉发育过程的肌球蛋白复合物,包括MYH1、MYL6B、MYH2、PPP3CA、TPM1、ATP2A1、MYL1等等。此外,发现网络中与肌肉发育相关基因互作的多数基因的功能与免疫系统、血液系统、神经系统、干细胞维持、破骨细胞分化、脂代谢、能量代谢相关,如基因PDK4、TRRAP、FGFR1等。通过靶基因预测,找到了MSTRG.3903、MSTRG.10154、MSTRG.1629、MSTRG.10496、MSTRG.9559、MSTRG.10178、MSTRG.10521、MSTRG.3911、MSTRG.4586、MSTRG.7232等10个与肌肉发育、肌肉疾病、细胞增殖相关的lncRNAs。复杂多样的基因表达模式显示出调控肌纤维发育和相关生理过程的相关基因间潜在的复杂调控网络,根据连通度推测PIP5K1A、PDK4、TNNI2基因可能在胚胎发育后期发挥重要作用。
2.5 实时荧光定量检测对全转录组测序结果的验证为确保测序结果的准确性和有效性,随机挑选lncRNA和miRNA各4个进行qRT-PCR验证。引物通过NCBI官网Blast进行设计,actin与U6分别为内参基因(表 5)。如图 6实时荧光定量检测结果表明,8条RNA的检测差异表达水平与深度测序结果趋势基本一致。后续对功能聚类后筛选出的关键RNA进行分析,以D85N vs D105N,D105N vs D135N和D85N vs D135N进行组间显著性检验。设定P≤0.05为差异显著,P≤0.01为差异极显著。结果显示,MSTRG.8724.1、chi-miR-133a-5p、hsa-miR-410-5p、bta-miR-365-3p和oar-miR-150在各组间差异显著。MSTRG.5112.1在D105N vs D135N差异显著,而MSTRG.10496.1和MSTRG.10702.1在D85N vs D135N和D105N vs D135N两组中都差异极显著,符合数据鉴定和功能分析的结果。
|
|
表 5 荧光定量引物信息 Table 5 The information of primers for qRT-PCR detection |

|
图中为RNA-seq和qRT-PCR的结果;折线图表示测序的FPKM值,柱状图表示相对定量结果。*. P≤0.05,**. P≤0.01, 此处表示的是qRT-PCR结果的显著性。**/**.左边的结果是D85N vs D135N组间差异,右边的结果是D105N vs D135N组间的差异 The figure shows the results of RNA-seq and qRT-PCR; the polyline graph shows the FPKM value of the sequencing and the histogram shows the relative quantitative results. *. P≤0.05, **. P≤0.01, which indicate the significance of qRT-PCR result. **/**.The result on the left is the difference between the D85N vs D135N groups, and the result on the right is the difference between the D105N vs D135N groups 图 6 全转录组测序结果的验证 Fig. 6 Verification of whole transcriptome sequencing results |
前期通过对3个节点(D85N、D105N和D135N)组织进行切片检测初步分析,发现D85N与D135N时期染色切片具有明显差异[5]。在美利奴绵羊D85N骨骼肌中可见中空管状的初级肌纤维典型结构,周围出现大量次级肌纤维,而D105N和D135N切片中只有次级肌纤维结构,无初级肌纤维结构特征。肌纤维数量方面,D105N与D135N时切片视野内基本相同,明显多于D85N时期。本研究在转录水平上对中国美利奴羊胎儿背最长肌不同发育阶段的分子表达差异进行分析和解释。转录组结果中,D135N时期内差异表达RNA数量最多,但与肌原纤维增殖、迁移相关基因较少。D85N处于胚胎发育的中后期,组织内仍具有与成肌细胞增殖和分化相关的基因,表明胚胎在发育中后期仍存在肌纤维增殖行为。通过对模块内基因的GO和KEGG分析,发现肌原纤维的生长和类型转换中伴随着其他多个复杂的生物学过程,其中相关度较高的是脂肪的分解和代谢。此外,肌纤维快慢肌的组成对动物机体的运动和生存具有重要意义[28],红白肌类型的构成也与家畜肌肉品质和风味密切相关[29]。
本研究中,分析网络模块发现胚胎发育的3个阶段均存在着与脂质合成、代谢和能量代谢相关的基因,其与肌肉相关因子密切相关。试验中富集到的与动物脂肪调控过程相关的代谢相关信号通路有脂肪酸代谢、丙酸代谢、PPAR信号通路、脂肪酸生物合成等,参与这些通路的差异基因包括RXRG、PDK4、LDHA、ACSL1、FABP3。PDK4是一种在D135N中显著上调的基因,是一种关键的调节酶,参与调节生理能源转变(从葡萄糖转换为脂肪酸)以响应生理变化[30]。丙酮酸脱氢酶复合物在肌肉中间代谢中占据中心和战略位置,主要受磷酸化/去磷酸化调节[31],有研究报道,PDK4与肌内脂肪和肌肉含水量显著相关[32]。FABP3(H-FABP)基因参与细胞内长链脂肪酸转运[33],可以将脂肪酸从细胞膜上转运到脂肪酸氧化和甘油三酯及磷酸的合成位置,调节肌间脂质代谢,通常脂质含量高的组织FABP3表达水平高。在畜禽肌内脂肪含量和肌间脂肪沉积研究中,将FABP3基因视作改善肉质的候选基因[34]。
本研究功能富集显示,与肌纤维发育相关的差异表达关键基因有CDKN1A、TNNI2、PIP5K1A等。增殖细胞核抗原(PCNA)是δ和εDNA聚合酶的辅助因子[35],CDKN1A/p21可与PCNA结合,促进肌原细胞的持续增殖和存活[36]。Cayrol等[37]证明, CDKN1A/p21的激活可通过抑制PCNA功能缩短细胞周期进程,导致细胞停滞于G1和G2期而影响肌细胞凋亡或分化[38]。Shen等[39]指出,CDKN1A/p21参与肌肉卫星细胞的维持,当肌肉组织损伤再生或发生肌疾病恢复时,CDKN1A/p21参与肌细胞的增殖调控。CDKN1A的表达在D85N至D105N期间较稳定,D105N到D135N期间表达量下降。因此,根据CDKN1A的表达情况可以看出肌原纤维在胚胎发育的中期仍有增殖小高峰,而后期持续低水平增生。肌钙蛋白I(TnI)是肌纤维细丝中肌钙蛋白复合物的抑制亚基,在钙调节收缩和松弛中起重要作用。脊椎动物TnI分为TNNI1、TNNI2和TNNI3三个亚型[40]。在3个阶段转录组表达谱中,TNNI2的表达强度大于TNNI1,调节快慢肌发育转换(从慢肌切换到快肌)的TnI异构体开关在肌纤维发育期间稳定表达[41],这可能与胚胎期快慢肌的发育特征相关。TNNI2在D85N与D105N期间表达基本不变,而在D135N时期表达量是之前的3倍以上,这表明快肌在D105N至D135N期间快速生长,TNNI2等调控基因在该阶段发挥积极影响。
通过网络构建和共表达分析,发现肌纤维的增生和肥大是一个复杂的多基因调控过程,在肌纤维发育的同时涉及到多个生理发育的生物学过程。本试验筛选出的模块核心基因主要与肌肉发育相关,模块中部分基因还参与脂肪合成、能量代谢、免疫防御、神经系统和血液系统相关的信号通路,推测这可能与胚胎在发育后期的生理需求有关,如骨骼肌的收缩、快慢肌的调控受神经支配等。此外,还有少部分基因参与肌肉内脂肪沉积、糖酵解/糖异生代谢、线粒体氧化代谢,具体调控方式还需要进一步深入研究。
4 结论中国美利奴羊胎儿3个时间节点背最长肌中lncRNA、mRNA的表达谱表明,胚胎D135N阶段转录物种类和基因表达水平不同于其他两个阶段(D85N和D105N)。D135N阶段差异表达非编码RNA和基因主要参与胚胎肌肉的发育和脂质代谢过程,还调控能量运输和糖代谢信号通路,如mTOR、Akt信号通路等。通过WGCNA方法成功构建了lncRNA-miRNA-mRNA共表达网络,找到CDKN1A、TNNI2、PIP5K1A、FABP3、PDK4等与妊娠后期胎儿骨骼肌发育相关的潜在候选基因,为深入研究非编码RNA在中国美利奴羊胚胎发育过程中对骨骼肌的发育调控机制奠定了基础。
| [1] | HORAK M, NOVAK J, BIENERTOVA-VASKU J. Muscle-specific microRNAs in skeletal muscle development[J]. Dev Biol, 2016, 410(1): 1–13. DOI: 10.1016/j.ydbio.2015.12.013 |
| [2] | LUO W, ABDALLA B A, NIE Q H, et al. The genetic regulation of skeletal muscle development:insights from chicken studies[J]. Front Agric Sci Eng, 2017, 4(3): 295–304. |
| [3] | MOK G F, LOZANO-VELASCO E, MVNSTERBERG A. microRNAs in skeletal muscle development[J]. Semin Cell Dev Biol, 2017, 72: 67–76. DOI: 10.1016/j.semcdb.2017.10.032 |
| [4] | ASHMORE C R, ROBINSON D W, RATTRAY P, et al. Biphasic development of muscle fibers in the fetal lamb[J]. Exp Neurol, 1972, 37(2): 241–255. |
| [5] | FAHEY A J, BRAMELD J M, PARR T, et al. Ontogeny of factors associated with proliferation and differentiation of muscle in the ovine fetus[J]. J Anim Sci, 2005, 83(10): 2330–2338. DOI: 10.2527/2005.83102330x |
| [6] | ZHANG D L, DU Q, DJEMLI A, et al. Cord blood insulin, IGF-I, IGF-II, leptin, adiponectin and ghrelin, and their associations with insulin sensitivity, β-cell function and adiposity in infancy[J]. Diabet Med, 2018, 35(10): 1412–1419. DOI: 10.1111/dme.13671 |
| [7] | XU L Y, ZHAO F P, REN H X, et al. Co-expression analysis of fetal weight-related genes in ovine skeletal muscle during mid and late fetal development stages[J]. Int Biol Sci, 2014, 10(9): 1039–1050. DOI: 10.7150/ijbs.9737 |
| [8] |
石田培, 张莉. 全转录组学在畜牧业中的应用[J]. 遗传, 2019, 41(3): 193–205.
SHI T P, ZHANG L. Application of whole transcriptomics in animal husbandry[J]. Hereditas (Beijing), 2019, 41(3): 193–205. DOI: 10.3760/cma.j.issn.1673-4386.2019.03.002 (in Chinese) |
| [9] | KADARMIDEEN H N, VON ROHR P, JANSS L L G. From genetical genomics to systems genetics:potential applications in quantitative genomics and animal breeding[J]. Mamm Genome, 2006, 17(6): 548–564. DOI: 10.1007/s00335-005-0169-x |
| [10] | WOMACK J E. Advances in livestock genomics:opening the barn door[J]. Genome Res, 2005, 15(12): 1699–1705. DOI: 10.1101/gr.3809105 |
| [11] | ANDERSSON L, GEORGES M. Domestic-animal genomics:deciphering the genetics of complex traits[J]. Nat Rev Genet, 2004, 5(3): 202–212. DOI: 10.1038/nrg1294 |
| [12] | TIAN H L, GUAN D H, LI J M. Identifying osteosarcoma metastasis associated genes by weighted gene co-expression network analysis (WGCNA)[J]. Medicine (Baltimore), 2018, 97(24): e10781. DOI: 10.1097/MD.0000000000010781 |
| [13] | STUART J M, SEGAL E, KOLLER D, et al. A gene-coexpression network for global discovery of conserved genetic modules[J]. Science, 2003, 302(5643): 249–255. DOI: 10.1126/science.1087447 |
| [14] | JORDAN I K, MARINÑO-RAMIÍREZ L, WOLF Y I, et al. Conservation and coevolution in the scale-free human gene coexpression network[J]. Mol Biol Evol, 2004, 21(11): 2058–2070. DOI: 10.1093/molbev/msh222 |
| [15] | FULLER T F, GHAZALPOUR A, ATEN J E, et al. Weighted gene coexpression network analysis strategies applied to mouse weight[J]. Mamm Genome, 2007, 18(6-7): 463–472. DOI: 10.1007/s00335-007-9043-3 |
| [16] | HUDSON N J, REVERTER A, WANG Y H, et al. Inferring the transcriptional landscape of bovine skeletal muscle by integrating co-expression networks[J]. PLoS One, 2009, 4(10): e7249. DOI: 10.1371/journal.pone.0007249 |
| [17] | STANLEY D, WATSON-HAIGH N S, COWLED C J E, et al. Genetic architecture of gene expression in the chicken[J]. BMC Genomics, 2013, 14(1): 13. DOI: 10.1186/1471-2164-14-13 |
| [18] | KOGELMAN L J A, BYRNE K, VUOCOLO T, et al. Genetic architecture of gene expression in ovine skeletal muscle[J]. BMC Genomics, 2011, 12(1): 607. DOI: 10.1186/1471-2164-12-607 |
| [19] | GAO P F, GUO X H, DU M, et al. LncRNA profiling of skeletal muscles in Large White pigs and Mashen pigs during development[J]. J Anim Sci, 2017, 95(10): 4239–4250. DOI: 10.2527/jas2016.1297 |
| [20] |
周瑞, 王以鑫, 龙科任, 等. LncRNA调控骨骼肌发育的分子机制及其在家养动物中的研究进展[J]. 遗传, 2018, 40(4): 292–304.
ZHOU R, WANG Y X, LONG K R, et al. Regulatory mechanism for lncRNAs in skeletal muscle development and progress on its research in domestic animals[J]. Hereditas (Beijing), 2018, 40(4): 292–304. (in Chinese) |
| [21] | PERTEA M, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads[J]. Nat Biotechnol, 2015, 33(3): 290–295. |
| [22] | HIRAI H, VERMA M, WATANABE S, et al. MyoD regulates apoptosis of myoblasts through microRNA-mediated down-regulation of Pax3[J]. J Cell Biol, 2015, 191(2): 347–365. |
| [23] | CRIST C G, MONTARRAS D, PALLAFACCHINA G, et al. Muscle stem cell behavior is modified by microRNA-27 regulation of Pax3 expression[J]. Proc Natl Acad Sci U S A, 2009, 106(32): 13383–13387. DOI: 10.1073/pnas.0900210106 |
| [24] | CESANA M, CACCHIARELLI D, LEGNINI I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2): 358–369. |
| [25] | LI H, YANG J M, WEI X F, et al. CircFUT10 reduces proliferation and facilitates differentiation of myoblasts by sponging miR-133a[J]. J Cell Physiol, 2018, 233(6): 4643–4651. DOI: 10.1002/jcp.26230 |
| [26] | VILLANUEVA R A M, CHEN Z J. ggplot2:elegant graphics for data analysis (2nd ed.)[J]. Meas:Interdiscipl Res Perspect, 2019, 17(3): 160–167. DOI: 10.1080/15366367.2019.1565254 |
| [27] | MUETZE T, LYNN D J. Using the Contextual Hub Analysis Tool (CHAT) in Cytoscape to identify contextually relevant network hubs[J]. Curr Protoc Bioinformatics, 2017, 59(1): 8.24.1–8.24.13. |
| [28] | CHOI Y M, RYU Y C, KIM B C. Influence of myosin heavy- and light chain isoforms on early postmortem glycolytic rate and pork quality[J]. Meat Sci, 2007, 76(2): 281–288. DOI: 10.1016/j.meatsci.2006.11.009 |
| [29] | GIL M, OLIVER M À, GISPERT M, et al. The relationship between pig genetics, myosin heavy chain I, biochemical traits and quality of M.Longissimus thoracis[J]. Meat Sci, 2003, 65(3): 1063–1070. DOI: 10.1016/S0309-1740(02)00324-8 |
| [30] | REN H X, LI L, SU H W, et al. Histological and Transcriptome-wide level characteristics of fetal Myofiber hyperplasia during the second half of gestation in texel and Ujumqin Sheep[J]. BMC Genomics, 2011, 12(1): 411. DOI: 10.1186/1471-2164-12-411 |
| [31] | ABBOT E L, MCCORMACK J G, REYNET C, et al. Diverging regulation of pyruvate dehydrogenase kinase isoform gene expression in cultured human muscle cells[J]. FEBS J, 2005, 272(12): 3004–3014. DOI: 10.1111/j.1742-4658.2005.04713.x |
| [32] | LAN J, LEI M G, ZHANG Y B, et al. Characterization of the porcine differentially expressed PDK4 gene and association with meat quality[J]. Mol Biol Rep, 2009, 36(7): 2003–2010. DOI: 10.1007/s11033-008-9411-4 |
| [33] | CHMURZYŃSKA A. The multigene family of Fatty Acid-Binding Proteins (FABPs):function, structure and polymorphism[J]. J Appl Genet, 2006, 47(1): 39–48. |
| [34] | LI X P, KIM S W, CHOI J S, et al. Investigation of porcine FABP3 and LEPR gene polymorphisms and mRNA expression for variation in intramuscular fat content[J]. Mol Biol Rep, 2010, 37(8): 3931–3939. DOI: 10.1007/s11033-010-0050-1 |
| [35] | OKE M, ZAHER M S, HAMDAN S M. PCNA Structure and interactions with partner proteins[M]. New York: Springer, 2018. |
| [36] | CALURA E, CAGNIN S, RAFFAELLO A, et al. Meta-analysis of expression signatures of muscle atrophy:gene interaction networks in early and late stages[J]. BMC Genomics, 2008, 9(1): 630. DOI: 10.1186/1471-2164-9-630 |
| [37] | CAYROL C, KNIBIEHLER M, DUCOMMUN B. p21 binding to PCNA causes G1 and G2 cell cycle arrest in p53-deficient cells[J]. Oncogene, 1998, 16(3): 311–320. DOI: 10.1038/sj.onc.1201543 |
| [38] | ZAREMBA-CZOGALLA M, HRYNIEWICZ-JANKO-WSKA A, TABOLA R, et al. A novel regulatory function of CDKN1A/p21 in TNFα-induced matrix metalloproteinase 9-dependent migration and invasion of triple-negative breast cancer cells[J]. Cell Signal, 2018, 47: 27–36. DOI: 10.1016/j.cellsig.2018.03.010 |
| [39] | SHEN J, MA J J, LEE C, et al. How muscles recover from paresis and atrophy after intramuscular injection of botulinum toxin A:study in juvenile rats[J]. J Orthop Res, 2006, 24(5): 1128–1135. DOI: 10.1002/jor.20131 |
| [40] | SHENG J J, JIN J P. TNNI1, TNNI2 and TNNI3:evolution, regulation, and protein structure-function relationships[J]. Gene, 2016, 576(1): 385–394. DOI: 10.1016/j.gene.2015.10.052 |
| [41] | STEVENS L, BASTIDE B, KISCHEL P, et al. Time-dependent changes in expression of troponin subunit isoforms in unloaded rat soleus muscle[J]. Am J Physiol Cell Physiol, 2002, 282(5): C1025–C1030. DOI: 10.1152/ajpcell.00252.2001 |



